一步一步学heatmap.2函数
荧光定量pcr 2-ct值统计方法

荧光定量pcr 2-ct值统计方法荧光定量PCR(qPCR)是一种高灵敏度、高特异性的PCR技术,已经广泛应用于生物学、医学、环境科学等领域的分子遗传学研究中。
在qPCR实验中,常用的数据分析方法是2-CT值法(Delta-CT method)和标准曲线法(Standard curve method)。
本文将介绍2-CT 值法的统计方法及其应用。
1. 原理2-CT值法是一种基于CT值测定样品中目标基因表达水平的方法。
CT值指的是PCR循环数的阈值点,即当荧光信号强度到达一定程度时,PCR反应被认为已进入指数增长期。
CT值越低,表示目标基因的表达量越高。
实验中,通常以内参基因(如β-actin、GAPDH 等)作为对照,计算目标基因和内参基因的CT值差(Delta CT值,ΔCT),代表样品中目标基因的表达水平。
如下所示:ΔCT = CT(目标基因) - CT(内参基因)2. 统计方法使用2-CT值法,需要进行数据标准化、统计和图形展示。
2.1 数据标准化数据标准化是指将ΔCT值转换为相对表达量(Relative Quantification)或折叠变化(Fold Change),用于比较不同样品之间目标基因表达水平的差异。
相对表达量是一种无单位的值,其计算公式为:Relative Quantification = 2^-ΔCT其中,2为基数,-ΔCT表示将ΔCT值取相反数后再进行指数计算。
折叠变化是指样品中目标基因表达量的倍数差异。
其计算公式为:Fold Chang e = 2^ΔΔCT其中,ΔΔCT表示将两个样品的ΔCT值差(如对照组和实验组)做差。
(1)平均值和标准误差(Mean and Standard Error):计算每组样品的相对表达量或折叠变化的平均值和标准误差,用于比较不同样品组之间的差异。
(2)t检验(t-test):比较不同样品之间的平均值是否显著不同。
(4)基因表达热图(Heatmap):根据每个样品的相对表达量或折叠变化进行聚类分析和热图展示。
关于热力型地图(HeatMap)的制作及简单应用

关于热力型地图(HeatMap)的制作及简单应用一直以来都对某些杂志及网页上的热力型地图(HeatMap,以下简称:HM)羡慕不已(如上图的美国太空总署(NASA)公布的全球PM2.5数值图),亦或有时在接受上级CDC培训时,讲师也经常在讲到某种传染病地区分布时,放出一些精美的HM,很是让人眼热。
当时就在想能不能自己制作一个以后放在ppt里或是疫情简报里让想要表达的数剧更加直观。
可惜由于找不到基于区县级地图的傻瓜模版,更找不到相关的DIY教程,以上设想很长一段时间都没能实现。
直到经过一个比较曲折的学习过程,终于用excel完成了一个煞形,以后再讲到疾病的三间分布,也可以拿出一个像模像样的HeatMap了,希望对大家有所帮助。
注:由于excel的地图模块已剥离单独售卖,在网上搜索数据地图的制作工具,基本没有相关的东西。
有一个国产数据地图控件,居然叫价800元,看来是想钱想疯了。
毛主席说:自力更生,丰衣足食,自己动手花点时间做个小工具,按数据大小分档层析填色,完成数据地图制作,实在是一劳永逸。
其实HeatMap的制图思路简单归纳起来有两点:1、绘制目标地区的地图轮廓。
2、地图模型的构建。
个人觉得地图的绘制是最废时间也是最考验一个人学习耐心的步骤,由于一般在网上都找不到免费的矢量地图(基于区、县一级的矢量图更是别想),这时一般只有自行绘制。
鄙人在用鼠标绘制本地区地图时数次差点放弃……由于我区行政区划复杂,又有长江穿过,饶是我耐心再好,中途也几次差点放弃。
要不是想到一次完成可以长期使用,也许早就跑去玩了。
关于用word或excel绘制地图轮廓,网上教程很多,大家百度一下便知,此次不进行赘述。
至于第二步地图模型的构建,首先,用拼音首字字母为图形命名。
如选中德感的自选图形,在Excel左上角的名称框中输入dg,Enter 即可,后面会用这个名称来引用这个图形。
按此方法逐一完成所有区域图形的命名。
(PS:如果某些地区被大江大河划为两部分,则必须在B例的地名中占用两行,数值行用同一数据对应,命名则可加1,2,3...区别,如白沙被长江分为两北两部分,刚可用bs、bs1表示)接下来,设置数据分档阀值和图例一般HeatMap用5档进行分档填色,在在I列设置填色样式,J列填入文字标签,在L列设置分档阀值,填入J列标签的下限,M列是为引用准备的名称。
第二讲 MATLAB 绘图

绘制y=1-exp(0.3*t).*cos(0.7*t)
t=6*pi*(0:100)/100; y=1-exp(-0.3*t).*cos(0.7*t); tt=t(find(abs(y-1)>0.05)); ts=max(tt); plot(t,y,'r-'); grid on; axis([0,6*pi,0.6,max(y)]); title('y=1-exp(-\alpha*t)*cos(\omega*t)'); hold on; plot(ts,0.95,'bo'); hold off; set(gca,'xtick',[2*pi,4*pi,6*pi],'ytick',[0.95,1,1.05,max(y)]); grid on;
绘制曲线
x t cos(3t ) , t 2 y t sin t
t = -pi:pi/100:pi; x = t.*cos(3*t); y = t.*sin(t).^2; plot(x,y)
2015-5-14 10
图形标识
图形标识包括:
图名(title) 坐标轴名(xlabel、ylabel) 图形文本注释(text) 图例(legend)
2015-5-14 22
双纵坐标:plotyy指令
plotyy指令调用格式:
plotyy(x1, y1, x2, y2)
x1-y1曲线y轴在左, x2-y2曲线y轴在右。
例3.7:
x = 0:0.01:20; y1 = 200*exp(-0.05*x).*sin(x); y2 = 0.8*exp(-0.5*x).*sin(10*x); plotyy(x,y1,x,y2);
一步一步学heatmap.2函数

一步一步学heatmap.2函数数据如下:library(gplots)data(mtcars)x <- as.matrix(mtcars)rc <- rainbow(nrow(x), start=0, end=.3)cc <- rainbow(ncol(x), start=0, end=.3)X就是一个矩阵,里面是我们需要画热图的数据。
Rc是一个调色板,有32个颜色,渐进的Cc也是一个调色板,有11个颜色,也是渐进的首先画一个默认的图:heatmap.2(x)然后可以把聚类数可以去掉:就是控制这个dendrogram参数heatmap.2(x, dendrogram="none")然后我们控制一下聚类树heatmap.2(x, dendrogram="row") 只显示行向量的聚类情况heatmap.2(x, dendrogram="col")只显示列向量的聚类情况下面还是在调控聚类树,但是我没看懂跟上面的参数有啥子区别!heatmap.2(x, keysize=2) ## default - dendrogram plotted and reordering done.heatmap.2(x, Rowv=FALSE, dendrogram="both") ## generate warning!heatmap.2(x, Rowv=NULL, dendrogram="both") ## generate warning!heatmap.2(x, Colv=FALSE, dendrogram="both") ## generate warning!接下来我们可以调控行列向量的label的字体大小方向首先我们调控列向量,也就是x轴的labelheatmap.2(x, srtCol=NULL)heatmap.2(x, srtCol=0, adjCol = c(0.5,1) )heatmap.2(x, srtCol=45, adjCol = c(1,1) )heatmap.2(x, srtCol=135, adjCol = c(1,0) )heatmap.2(x, srtCol=180, adjCol = c(0.5,0) )heatmap.2(x, srtCol=225, adjCol = c(0,0) ) ## not very useful heatmap.2(x, srtCol=270, adjCol = c(0,0.5) )heatmap.2(x, srtCol=315, adjCol = c(0,1) )heatmap.2(x, srtCol=360, adjCol = c(0.5,1) )然后我们调控一下行向量,也就是y轴的labelheatmap.2(x, srtRow=45, adjRow=c(0, 1) )heatmap.2(x, srtRow=45, adjRow=c(0, 1), srtCol=45, adjCol=c(1,1) )heatmap.2(x, srtRow=45, adjRow=c(0, 1), srtCol=270, adjCol=c(0,0.5) )设置offsetRow/offsetCol 可以把label跟热图隔开!## Show effect of offsetRow/offsetCol (only works when srtRow/srtCol is ## not also present)heatmap.2(x, offsetRow=0, offsetCol=0)heatmap.2(x, offsetRow=1, offsetCol=1)heatmap.2(x, offsetRow=2, offsetCol=2)heatmap.2(x, offsetRow=-1, offsetCol=-1)heatmap.2(x, srtRow=0, srtCol=90, offsetRow=0, offsetCol=0)heatmap.2(x, srtRow=0, srtCol=90, offsetRow=1, offsetCol=1)heatmap.2(x, srtRow=0, srtCol=90, offsetRow=2, offsetCol=2)heatmap.2(x, srtRow=0, srtCol=90, offsetRow=-1, offsetCol=-1)## Show effect of z-score scaling within columns, blue-red color scale##hv <- heatmap.2(x, col=bluered, scale="column", tracecol="#303030")hv是一个热图对象!!!> names(hv)可以看到hv对象里面有很多子对象"rowInd" "colInd" "call" "colMeans" "colSDs" "carpet" "rowDendrogram" "colDendrogram" "breaks" "col" "vline" "colorTable"## Show the mapping of z-score values to color binshv$colorTable## Extract the range associated with white我们得到了热图的颜色的数值映射矩阵,接下来就可以进行一系列的操作~!!!hv$colorTable[hv$colorTable[,"color"]=="#FFFFFF",]首先得到了白色所对应的数值区间!然后还可以通过一下命令,直接求出属于白色区间的那些数值。
hive函数大全

收藏limma转录组定量中文操作指南

对于一个给定的实验,通常有几种等价的方法可以创建一个合适的设计矩阵。比如说,~0+group+lane去除了第一个因子group的截距,但第二个因子lane的截距被保留。此外也可以使用~group+lane,来自group和la 情况下能更直截了当地设定模型中的对比。 用于细胞群之间成对比较的对比可以在limma中用makeContrasts函数设定。 contr.matrix <-> BasalvsLP = Basal-LP, BasalvsML = Basal - ML, LPvsML = LP - ML, levels = colnames(design)) limma的线性模型方法的核心优势之一便是其可以适应不同的实验复杂程度。无论这篇教程中关于细胞类型和批次的简单实验设计,还是更复杂的因子设计和含有交互作用项的模型,都能够得到相对简单的处理。 对于RNA-seq原始计数或其log-CPM值,方差与均值并不独立,而使用负二项分布来模拟计数的方法假设均值与方差间具有二次的关系。在limma中,假设log-CPM值符合正态分布,并使用由voom函数计算得到的精确 下图左侧展示了这个数据集log-CPM值的均值-方差关系。通常而言,voom图中均值随着方差增加而递减。生物学差异高的实验通常会有更平坦的趋势,其方差值在高表达处稳定。生物学差异低的实验更倾向于急剧下 此外,如果对于低表达基因的过滤不够充分,在图上表达低的一端可以观察到方差水平的下降。如果观察到了这种情况,应当回到最初的过滤步骤并提高用于该数据集的表达阈值。
数据通信原理实验指导书

实验一编码与译码一、实验学时:2学时二、实验类型:验证型三、实验仪器:安装Matlab软件的PC机一台四、实验目的:用MATLAB仿真技术实现信源编译码、过失操纵编译码,并计算误码率。
在那个实验中咱们将观看到二进制信息是如何进行编码的。
咱们将要紧了解:1.目前用于数字通信的基带码型2.过失操纵编译码五、实验内容:1.经常使用基带码型(1)利用MATLAB 函数wave_gen 来产生代表二进制序列的波形,函数wave_gen 的格式是:wave_gen(二进制码元,‘码型’,Rb)此处Rb 是二进制码元速度,单位为比特/秒(bps)。
产生如下的二进制序列:>> b = [1 0 1 0 1 1];利用Rb=1000bps 的单极性不归零码产生代表b的波形且显示波形x,填写图1-1:>> x = wave_gen(b,‘unipolar_nrz’,1000);>> waveplot(x)(2)用如下码型重复步骤(1)(提示:能够键入“help wave_gen”来获取帮忙),并做出相应的记录:a 双极性不归零码b 单极性归零码c 双极性归零码d 曼彻斯特码(manchester)x 10-3x 10-3图1-1 单极性不归零码图1-2双极性不归零码x 10-3x 10-32.过失操纵编译码(1) 利用MATLAB 函数encode 来对二进制序列进行过失操纵编码, 函数encode 的格式是:A .code = encode(msg,n,k,'linear/fmt',genmat)B .code = encode(msg,n,k,'cyclic/fmt',genpoly)C .code = encode(msg,n,k,'hamming/fmt',prim_poly)其中A .用于产生线性分组码,B .用于产生循环码,C .用于产生hamming 码,msg 为待编码二进制序列,n 为码字长度,k 为分组msg 长度,genmat 为生成矩阵,维数为k*n ,genpoly 为生成多项式,缺省情形下为cyclpoly(n,k)。
MATLAB中文手册

Matlab中文手册目录 (1)第1章MATLAB 6.5环境 (11)1.1 MATLAB简介 (11)1.1.1 MATLAB工具箱 (11)1.1.2 MATLAB功能和特点 (12)1.2 MATLAB 6.5环境设置 (13)1.2.1 菜单栏 (13)1.2.2 工具栏 (18)1.2.3 通用操作界面窗口 (18)1.3 MATLAB 6.5帮助 (32)1.4 MATLAB 6.5其他管理 (35)1.4.1 MATLAB用户文件格式 (35)1.4.2设置搜索路径 (36)1.4.3文件管理命令 (37)1.4.4 退出MATLAB (39)1.5 一个实例 (39)第2章MATLAB数值计算 (43)2.1 变量和数据 (43)2.1.1数据类型 (43)2.1.2数据 (43)2.1.3变量 (46)2.2 矩阵和数组 (47)2.2.1矩阵输入 (47)2.2.2矩阵元素和操作 (54)2.2.3字符串 (65)2.2.4矩阵和数组运算 (72)2.2.5多维数组 (92)2.3稀疏矩阵 (98)2.3.1稀疏矩阵的建立 (98)2.3.2稀疏矩阵的存储空间 (103)2.3.3稀疏矩阵的运算 (105)2.4多项式 (105)2.4.1多项式的求值、求根和部分分式展开 (105)2.4.2多项式的乘除法和微积分 (109)2.4.3多项式拟合和插值 (112)2.5元胞数组和结构数组 (115)2.5.1元胞数组 (116)2.5.2结构数组 (121)2.6数据分析 (127)2.6.1数据统计和相关分析 (127)2.6.2差分和积分 (129)2.6.3卷积和快速傅里叶变换 (134)2.6.4向量函数 (137)第3章MATLAB符号计算 (138)3.1 符号表达式的建立 (138)3.1.1 创建符号常量 (138)3.1.2 创建符号变量和表达式 (141)3.1.3 符号矩阵 (143)3.2符号表达式的代数运算 (145)3.2.1符号表达式的代数运算 (146)3.2.2 符号数值任意精度控制和运算 (149)3.2.3 符号对象与数值对象的转换 (152)3.3符号表达式的操作和转换 (154)3.3.1符号表达式中自由变量的确定 (154)3.3.2符号表达式的化简 (156)3.3.3符号表达式的替换 (161)3.3.4求反函数和复合函数 (164)3.3.5 符号表达式的转换 (166)3.4 符号极限、微积分和级数求和 (169)3.4.1符号极限 (169)3.4.2符号微分 (171)3.4.3符号积分 (174)3.4.4符号级数 (176)3.5 符号积分变换 (178)3.5.1傅里叶(Fourier)变换及其反变换 (178)3.5.2拉普拉斯(Laplace)变换及其反变换 (180)3.5.3 Z变换及其反变换 (181)3.6符号方程的求解 (183)3.6.1代数方程 (183)3.6.2符号常微分方程 (185)3.7符号函数的可视化 (186)3.7.1符号函数的绘图命令 (186)3.7.2图形化的符号函数计算器 (189)3.8 Maple函数的使用 (189)3.8.1访问Maple函数 (190)3.8.2 获得Maple的帮助 (191)第4章MATLAB计算的可视化和GUI设计 (192)4.1二维曲线的绘制 (192)4.1.1基本绘图命令plot (192)4.1.2绘制曲线的一般步骤 (198)4.1.3多个图形绘制的方法 (199)4.1.4曲线的线型、颜色和数据点形 (202)4.1.5设置坐标轴和文字标注 (204)4.1.6交互式图形命令 (210)4.2 MATLAB的三维图形绘制 (211)4.2.1绘制三维线图命令plot3 (211)4.2.2绘制三维网线图和曲面图 (212)4.2.3立体图形与图轴的控制 (217)4.2.4色彩的控制 (220)4.3 MATLAB的特殊图形绘制 (225)4.3.1条形图 (225)4.3.2面积图和实心图 (227)4.3.3直方图 (228)4.3.4饼图 (230)4.3.5离散数据图 (231)4.3.6对数坐标和极坐标图 (232)4.3.7等高线图 (234)4.3.8复向量图 (235)4.4图形窗口的功能 (236)4.5对话框 (238)4.6句柄图形 (242)4.6.1句柄图形体系 (242)4.6.2图形对象的操作 (242)4.6.3图形对象属性的获取和设置 (248)4.7图形用户界面(GUI)设计 (251)4.7.1可视化的界面环境 (252)4.7.2菜单 (253)4.7.5回调函数 (258)4.7.6 GUI应用举例 (258)4.8动画 (261)4.8.1以电影方式产生动画 (261)4.8.2以对象方式产生动画 (262)第5章MATLAB程序设计 (264)5.1脚本文件和函数文件 (264)5.1.1 M文本编辑器 (265)5.1.2 M文件的基本格式 (265)5.1.3 M脚本文件 (266)5.1.4 M函数文件 (268)5.2程序流程控制 (269)5.2.1 for ... end循环结构.. (269)5.2.2 while ... end循环结构. (271)5.2.3 If...else...end条件转移结构 (272)5.2.4 switch...case开关结构.. (273)5.2.5 try... catch... end试探结构 .. (275)5.2.6流程控制语句 (276)5.3函数调用和参数传递 (280)5.3.1子函数和私有函数 (280)5.3.2局部变量和全局变量 (281)5.3.4程序举例 (287)5.4 M文件性能的优化和加速 (289)5.4.1 P码文件 (289)5.4.2 M文件性能优化 (290)5.4.3 JIT和加速器 (292)5.5内联函数 (295)5.6利用函数句柄执行函数 (298)5.6.1函数句柄的创建 (298)5.6.2用feval命令执行函数 (299)5.7利用泛函命令进行数值分析 (301)5.7.1求极小值 (302)5.7.2求过零点 (304)5.7.3数值积分 (305)5.7.4微分方程的数值解 (306)第6章线性控制系统分析与设计 (309)6.1线性系统的描述 (309)6.1.1状态空间描述法 (309)6.1.2传递函数描述法 (311)6.1.3零极点描述法 (312)6.1.4离散系统的数学描述 (313)6.2线性系统模型之间的转换 (317)6.2.1连续系统模型之间的转换 (317)6.2.2连续系统与离散系统之间的转换 (325)6.2.3模型对象的属性 (330)6.3结构框图的模型表示 (334)6.4线性系统的时域分析 (348)6.4.1零输入响应分析 (348)6.4.2脉冲响应分析 (350)6.4.3阶跃响应分析 (351)6.4.4任意输入的响应 (353)6.4.5系统的结构参数 (356)6.5线性系统的频域分析 (358)6.5.1频域特性 (358)6.5.2连续系统频域特性 (359)6.5.3幅值裕度和相角裕度 (366)6.5.4离散系统频域分析 (367)6.6线性系统的根轨迹分析 (367)6.6.1绘制根轨迹 (367)6.6.2根轨迹的其它工具 (370)6.7线性系统的状态空间设计 (373)6.7.1极点配置法 (373)6.7.2最优二次型设计 (374)第7章Simulink仿真环境 (376)7.1演示一个Simulink的简单程序 (376)7.2 Simulink的文件操作和模型窗口 (379)7.2.1 Simulink的文件操作 (379)7.2.2 Simulink的模型窗口 (380)7.3 模型的创建 (382)7.3.1模块的操作 (382)7.3.2信号线的操作 (385)7.3.3给模型添加文本注释 (387)7.4 Simulink的基本模块 (387)7.4.1基本模块 (387)7.4.2常用模块的参数和属性设置 (390)7.5复杂系统的仿真与分析 (395)7.5.1仿真的设置 (395)7.5.2连续系统仿真 (397)7.5.3离散系统仿真 (400)7.5.4仿真结构参数化 (402)7.6子系统与封装 (403)7.6.1建立子系统 (403)7.6.2条件执行子系统 (405)7.6.3子系统的封装 (407)7.7用MATLAB命令创建和运行Simulink模型 (413)7.7.1用MATLAB命令创建Simulink模型 (413)7.7.2用MATLAB命令运行Simulink模块 (417)7.8以Simulink为基础的模块工具箱简介 (418)第8章MATLAB高级应用 (419)8.1 MATLAB应用接口 (419)8.1.1 MEX文件 (419)8.1.2 使用MATLAB编译器生成MEX和EXE文件 (424)8.2 低级文件的输入输出 (426)8.2.1打开和关闭文件 (426)8.2.2读写格式化文件 (428)8.2.3读写二进制数据 (433)8.2.4文件定位 (435)8.3 图形文件的转储 (437)8.4 Notebook (438)8.4.1 Notebook的安装 (438)8.4.2 Notebook的启动 (438)8.4.3 Notebook的使用 (441)8.4.4 Notebook中MATLAB的使用 (445)第1章MATLAB 6.5环境1.1MATLAB简介●MATLAB(Matrix Laborator)是MathWorks公司开发科学与工程计算软件;●广泛应用于自动控制、数学运算、信号分析、计算机技术、图像信号处理、财务分析、航天工业、汽车工业、生物医学工程、语音处理和雷达工程等行业;●国内外高校和研究部门科学研究的重要工具;●MATLIB 已成为数学计算工具方面事实上的标准,MATLIB 6.5是最新版本。
sufer操作手册

§2 基本图形的绘制..................................................................................................................6
2.1 建立XYZ数据文件..............................................................................................................6 2.2 数据文件的网格化(Grid) ..............................................................................................6 2.3 绘制和设置等高线..............................................................................................................7
§3 图形绘制的技巧................................................................................................................17
ABAQUS常见问题汇总

ABAQUS 常见问题汇总 - 2.0 版
目录 点击小节标题,可以跳到相应的内容(有些 WORD 版本可能需要按住 ctrl 键)
0. ABAQUS 入门资料.......................................................................................................................... 4
6.1 ABAQUS 安装方法 ................................................................................................................. 12 6.2 ABAQUS 显示异常(无法显示栅格、显卡冲突、更改界面颜色).......................................... 21 6.3 Document 无法搜索................................................................................................................. 21 6.4 磁盘空间不足 ........................................................................................................................... 22 6.5 Linux 系统................................................................................................................................ 22 6.6 死机后恢复模型 ....................................................................................................................... 23
Icepak培训教程

目录什么是Icepak? (2)程序结构 (2)软件功能 (3)练习1 翅片散热器 (6)练习2 辐射的块和板 (41)练习3 瞬态分析 (56)练习4 笔记本电脑 (75)练习5 修改的笔记本电脑 (104)练习6 由IGES导入的发热板模型 (114)练习7 非连续网格 (138)练习8 Zoom-in建模 (149)什么是Icepak?Icepak是强大的 CAE 仿真软件工具,它能够对电子产品的传热,流动进行模拟,从而提高产品的质量,大量缩短产品的上市时间。
Icepak能够计算部件级,板级和系统级的问题。
它能够帮助工程师完成用试验不可能实现的情况,能够监控到无法测量的位置的数据。
Icepak采用的是FLUENT计算流体动力学 (CFD) 求解引擎。
该求解器能够完成灵活的网格划分,能够利用非结构化网格求解复杂几何问题。
多点离散求解算法能够加速求解时间。
Icepak提供了其它商用热分析软件不具备的特点,这些特点包括:图软件架构非矩形设备的精确模拟接触热阻模拟各向异性导热率非线性风扇曲线集中参数散热器外部热交换器辐射角系数的自动计算程序结构Icepak软件包包含如下内容:Icepak, 建模,网格和后处理工具FLUENT, 求解器Icepak本身拥有强大的建模功能。
你也可以从其它 CAD 和 CAE 软件包输入模型. Icepak然后为你的模型做网格, 网格通过后就是进行CFD求解。
计算结果可以在Icepak中显示, 如图所示.软件功能所有的功能均在Icepak界面下完成。
总述鼠标控制的用户界面o鼠标就能控制模型的位置,移动及改变大小o误差检查灵活的量纲定义几何输入IGES, STEP, IDF, 和 DXF格式库功能在线帮助和文档o完全的超文本在线帮助 (包括理论和练习册) 支持平台o UNIX 工作站o Windows NT 2000/XP 的PC机建模基于对象的建模o cabinets 机柜o networks 网络模型o heat exchangers 热交换器o wires 线o openings 开孔o grilles 过滤网o sources 热源o printed circuit boards (PCBs) PCB板o enclosures 腔体o plates 板o walls 壁o blocks 块o fans (with hubs) 风扇o blowers 离心风机o resistances 阻尼o heat sinks 散热器o packages 封装macros 宏o JEDEC test chambers JEDEC试验室o printed circuit board (PCB)o ducts 管道o compact models for heat sinks 简化的散热器2D object shapes 2D模型o rectangular 矩形o circular 圆形o inclined 斜板o polygon 多边形板complex 3D object shapes 3D模型o prisms 四面体o cylinders 圆柱o ellipsoids 椭圆柱o elliptical and concentric cylinders 椭圆柱o prisms of polygonal and varying cross-section 多面体o ducts of arbitrary cross-section 任意形状的管道网格自动非结构化网格生成o六面体,四面体,五面体及混合网格网格控制o粗网格生成o细网格生成o网格检查o非连续网格材料综合的材料物性数据库各向异性材料属性随温度变化的材料物理模型层流/湍流模型稳态/瞬态分析强迫对流/自然对流/混合对流传导流固耦合辐射体积阻力混合长度方程(0-方程), 双方程(标准 - 方程), RNG - , 增强双方程 (标准 - 带有增强壁面处理), 或Spalart-Allmaras 湍流模型接触阻尼体积阻力模型非线性风扇曲线集中参数的fans, resistances, and grilles边界条件壁和表面边界条件:热流密度, 温度, 传热系数, 辐射,和对称边界条件开孔和过滤网风扇热交换器时间相关和温度相关的热源随时间变化的环境温度求解引擎对于求解器FLUENT,是采用的有限体积算法。
零基础-绝对掌握-热图(Heatmap)-绘制细节,只要请我喝一瓶华农酸奶
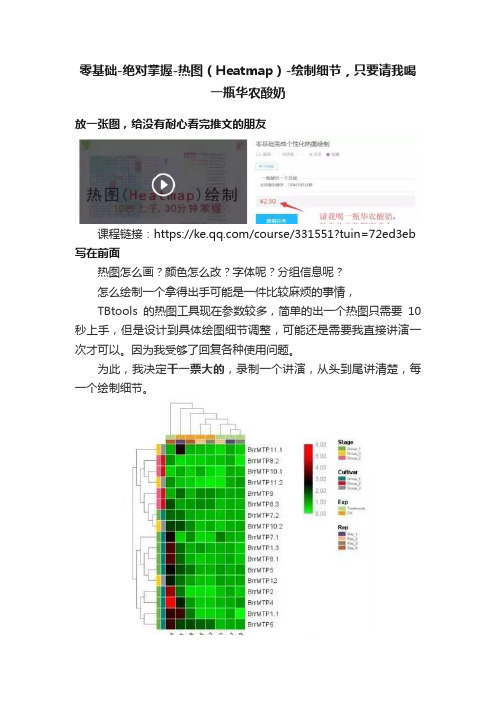
零基础-绝对掌握-热图(Heatmap)-绘制细节,只要请我喝
一瓶华农酸奶
放一张图,给没有耐心看完推文的朋友
课程链接:https:///course/331551?tuin=72ed3eb 写在前面
热图怎么画?颜色怎么改?字体呢?分组信息呢?
怎么绘制一个拿得出手可能是一件比较麻烦的事情,
TBtools的热图工具现在参数较多,简单的出一个热图只需要10秒上手,但是设计到具体绘图细节调整,可能还是需要我直接讲演一次才可以。
因为我受够了回复各种使用问题。
为此,我决定干一票大的,录制一个讲演,从头到尾讲清楚,每一个绘制细节。
这一次,我们干一票大的
你请我喝一瓶华农酸奶,我告诉你一个画热图不再求人的解法。
示例图片
写在后面
写这一段的主要原因是,原创声明需要至少300字。
既然如此,索性我就大概讲讲为什么TBtools突然间有这个热图功能?
TBtools的每一个工具,或者是课题组课题需要(其实整套基因家族分析相关功能,都是因为课题组有人要用到),而剩下的部分功能,则是水到渠成。
热图属于后者。
我正好为了实现RNA二级结构可视化,所以优化了newick类似的文本的解析器,同时JIGplot的拼图功能刚实现。
两者结合起来,便是热图。
Anyway,所有我知道的,我都放在课程里面了。
如果你想要,那就去看吧。
课程中闲扯的比较多,大概时长是 1 hours。
也就是,你请我喝一瓶酸奶,你还要忍受我讲一个小时的话,同时看着我操作。
愿不愿意在你,我只想说,以后凡是热图绘制问题。
先看这个课,再来问我。
哈哈。
matlab中heatmap用法

Matlab中heatmap用法概述在Matlab中,heatmap函数用于创建热图,它是一种可视化工具,用于展示数据的分布情况。
热图通过使用不同颜色的方块来表示数据的大小或者密度,从而使数据的模式和趋势更加清晰可见。
基本用法使用heatmap函数创建热图的基本语法如下:heatmap(data)其中,data是一个二维矩阵,表示要展示的数据。
矩阵的每个元素对应一个方块,方块的颜色根据元素的值来确定。
自定义颜色映射默认情况下,heatmap函数使用默认的颜色映射来表示数据的大小。
如果希望自定义颜色映射,可以使用colormap参数。
例如,以下代码将热图的颜色映射设置为红色到蓝色的渐变:heatmap(data, 'Colormap', 'redblue')调整方块大小可以使用CellSize参数来调整热图中方块的大小。
例如,以下代码将方块的大小设置为20像素:heatmap(data, 'CellSize', 20)添加标题和标签可以使用Title参数来为热图添加标题,使用XLabel和YLabel参数来添加横轴和纵轴的标签。
例如,以下代码为热图添加标题和标签:heatmap(data, 'Title', 'My Heatmap', 'XLabel', 'X Axis', 'YLabel', 'Y Axis')调整热图尺寸可以使用FigureSize参数来调整热图的尺寸。
例如,以下代码将热图的宽度设置为800像素,高度设置为600像素:heatmap(data, 'FigureSize', [800, 600])添加颜色条可以使用Colorbar参数来为热图添加颜色条,颜色条用于表示方块颜色对应的数值。
例如,以下代码为热图添加颜色条:heatmap(data, 'Colorbar', true)高级用法除了基本用法之外,heatmap函数还支持许多其他参数和选项,用于进一步自定义热图的样式和属性。
R语言之heatmap.2篇

R语言之heatmap.2篇R语言gplots包heatmap.2函数是用来画热图的, 主要子图包括以下几个: 颜色键值图, 列系统树图, 行系统树图, 单元格图像, ColSideColors图, RowSideColors图以及额外图像. heatmap.2的参数很多, 声明如下:heatmap.2 (x,# dendrogram controlRowv = TRUE,Colv=if(symm)"Rowv"else TRUE,distfun = dist,hclustfun = hclust,dendrogram = c("both","row","column","none"),symm = FALSE,# data scalingscale = c("none","row", "column"),na.rm=TRUE,# image plotrevC = identical(Colv, "Rowv"),add.expr,# mapping data to colorsbreaks,symbreaks=min(x < 0, na.rm=TRUE) || scale!="none",# colorscol="heat.colors",# block seprationcolsep,rowsep,sepcolor="white",sepwidth=c(0.05,0.05),# cell labelingcellnote,notecex=1.0,notecol="cyan",na.color=par("bg"),# level tracetrace=c("column","row","both","none"), tracecol="cyan",hline=median(breaks),vline=median(breaks),linecol=tracecol,# Row/Column Labelingmargins = c(5, 5), ColSideColors,RowSideColors,cexRow = 0.2 + 1/log10(nr),cexCol = 0.2 + 1/log10(nc),labRow = NULL,labCol = NULL,srtRow = NULL,srtCol = NULL,adjRow = c(0,NA),adjCol = c(NA,0),offsetRow = 0.5,offsetCol = 0.5,# color key + density infokey = TRUE,keysize = 1.5,=c("histogram","density","none"), denscol=tracecol,symkey = min(x < 0, na.rm=TRUE) || symbreaks, densadj = 0.25,# plot labelsmain = NULL,xlab = NULL,ylab = NULL,# plot layoutlmat = NULL,lhei = NULL,lwid = NULL,# extrasextrafun=NULL,...)x: 是矩阵, 是热图的每一个单元的取值;系统树图的控制Rowv: 用于指定行是否需要重新排序, 如果是TURE, 则还用每一行的均值进行重排序, 如果是由整数组成的向量, 则按向量的次序重排序, 如果是dendrogram对象, 相当用"as-is", 没有重排序;Colv: 与Rowv类似, 另外若x是方阵, 则默认Colv = "Rowv", 表示分组跟Rowv相同;distfun: 用于指定如何计算各行和各列之间的距离, 默认是dist函数, 而dist函数默认使用欧几里得距离;hclustfun: 用于指定横坐标或纵坐标不是系统树图时分组聚类的方法;dendrogram: 可为"none", "row", "column"或者"both", 用于如何做系统树图;symm: 指定x矩阵是否对称处理, 只有在x是方阵时可以为TRUE.数据规范化scale: 指定x是否按行或按列规范化, 默认值为"none";na.rm: NA值是否需要移除(只是将单元格移除).作图revC: 纵轴是否需要反向作图; Colv为"Rowv"时默认为TRUE, 其他情况默认为FALSE;add.expr: 一个表达式, 在image函数调用后计算, 可以为图像添加成分. (源代码只有以下两行包括add.expr参数, heatmap函数也有这参数, 尚不清楚有何用处, 望广大网友指教)if (!missing(add.expr))eval(substitute(add.expr))映射数值到颜色breaks: 数值向量指定相邻两个数值之间的值为同一个颜色, 或者数值指定分成多少breaks以确定颜色值(min(x)到max(x)等值分割).symbreaks: breaks是否为对称, 默认如果有负数, 则为TRUE, 否则为FALSE.颜色col: 单元格颜色, 默认使用heat.colors函数.单元格分割colsep: 纵轴分割线的坐标;rowsep: 横轴分割线的坐标;sepcolor: 分割线的颜色;sepwidth: 分割线的宽度, 是二元向量, 默认取值为c(0.05, 0.05).单元格标注cellnote: 矩阵或者字符型变量, 标注在单元格内;notecex: 单元格内标注的大小;notecol: 单元格内标注的颜色;na.color: 当单元格为NA时, 单元格的颜色, 默认为背景颜色.痕迹量级trace: 指定是否用实线穿过单元格, 可取值为"row", "column", "both", "none", 偏移单元格中线的程度与以下值成正比:颜色键值如果是对称的话, 则是单元格对应数值相对0的偏移值, 如果不对称, 则是单元格对应数值相对于最大值和最小值的和的0.5倍的偏移值;tracecol: 上述直线的颜色;hline: 数值向量指定虚线从单元格水平穿过, 在trace为"row"或"both"时有效, 默认值为median(breaks);vline: 数值向量指定虚线从单元格垂直穿过, 在trace为"col"或"both"时有效, 默认值为median(breaks);linecol: 上述虚线的颜色.行列标注margins: 长度为2的数值向量, 是par(mar = c(margins[1], 0, 0, margins[2]))的参数, 也会影响其他绘图区域比如ColSideColors和RowSideColors;ColSideColors: 长度ncol(x)的指示颜色的字符型向量;RowSideColors: 长度nrow(x)的指示颜色的字符型向量;cexRow: 指定行标注的大小;cexCol: 指定列标注的大小;labRow: 指定行的标注;labCol: 指定列的标注;strRow: 指定行标注的斜角;strCol: 指定列标注的斜角;adjRow: 指定行标注对齐方式, 默认值为c(0, NA);adjCol: 指定列标注对齐方式, 默认值为c(NA, 0);offsetRow: 指定行标注远离作图区域多少空格;offsetCol: 指定列标注远离作图区域多少空格;颜色键值和密度分布信息key: 颜色键值图是否应该标出;keysize: 颜色键值图大小, 默认为1.5;: 可设置为"histogram", "density", "none", 分别是直方图, 密度图, 不画;denscol: 字符型给出上述曲线的颜色, 默认值为tracecol;symkey: 颜色键值是否要对称, 默认情况下, 如果x中有负值, symkey取TRUE;densadj: density函数的adjust参数, 可控制density图像的y轴取值, 默认值为0.25.图像标注main: 图像的主标题;xlab: 图像的x轴标注;ylab: 图像的y轴标注.图像布局lmat: 控制四个主要元素(颜色键值图编号为4, 列系统树图编号为3, 行系统树图编号为2, 单元格图像为编号1)的相对位置, heatmap.2的作图顺序与这个编号次序一致,其默认值为rbind(c(4, 3), c(2, 1));lhei: 行的相对高度, 默认值为(keysize, 4), 如果存在ColSideColors, 则为(keysize, 0.2, 4);lwid: 列的相对宽度, 默认值为(keysize, 4), 如果存在RowSideColors, 则为(keysize, 0.2, 4);需要注意的是, 当ColSideColors和RowSideColors其中一个存在时, 上述四个图像元素的编号为原始编号加1, 编号1为这个增加的元素; 如果都存在, 上述四个主图像的编号依次加2, RowSideColors的编号为1, ColSideColors的编号为2. 这些情形下, lmat的默认值会相应变化.额外部分extrafun: 一个函数, 在所有工作完成之后调用.需要注意的是, 如果这个函数是作图, 那么这个函数里面的第一个图像编号为上述四个主图像和两个副图像的最大编号加1, 如果还有图像,编号依次加1; 如果刚开始没给这些图像预留位置(lmat), 则会在新的一页画图.library(gplots)data(mtcars)x <- as.matrix(mtcars)lmat <- rbind( c(5,3,4), c(2,1,4) )lhei <- c(1.5, 4)lwid <- c(1.5, 4, 0.75)myplot <- function() {oldpar <- par("mar")par(mar=c(5.1, 4.1, 0.5, 0.5))plot(mpg ~ hp, data=x)}heatmap.2(x, lmat=lmat, lhei=lhei, lwid=lwid, key=FALSE, ex trafun=myplot)著作权归作者所有•分类:R。
plotheatmap参数

plotheatmap参数
`plotly.express`绘制热图时,常用的参数有:
- `px.imshow()`: 该函数用于显示热图(以及全彩图像)。
它接受类似数组的对象,如列表和`numpy`或`xarray`数组的列表,以及`pandas.DataFrame`对象。
- `text_auto`: 将其设置为`True`,将在条形图上显示值,并将其设置为`d3-format`格式化字符串将控制输出格式。
- `aspect`: 默认情况下,`px.imshow()`生成带有方形图块的热图,但将`aspect`参数设置为`"auto"`将使用非方形图块用热图填充绘图区域。
- `x, y, labels`: 可以使用这些参数自定义热图的显示,并使用`.update_xaxes()`将`x`轴刻度标签移动到顶部。
你可以根据实际需求调整这些参数,以获得满意的热图效果。
mlvisual使用方法

mlvisual使用方法摘要:1.MLVisual简介2.MLVisual安装与配置3.MLVisual主要功能与操作方法4.MLVisual实战案例与应用场景5.总结与建议正文:MLVisual是一款强大的机器学习可视化工具,可以帮助数据科学家和工程师更直观地理解和优化模型。
本文将介绍MLVisual的使用方法,包括安装与配置、主要功能与操作方法,以及实战案例与应用场景。
最后给出总结与建议,以帮助读者更好地利用这款工具。
1.MLVisual简介MLVisual是一款基于Python的开源可视化库,专为机器学习模型设计。
它支持多种流行的深度学习框架,如TensorFlow、PyTorch等。
通过MLVisual,用户可以轻松地可视化神经网络结构、训练过程、模型性能等,从而更好地分析与优化模型。
2.MLVisual安装与配置要使用MLVisual,首先需要安装它。
您可以使用以下命令安装:```pip install mlvisual```安装完成后,导入MLVisual库并配置相关参数,如下所示:```pythonimport mlvisual as mvmv.set(__model__, "your_model_path")mv.set(__title__, "Your Model Title")mv.set(__description__, "Your Model Description")```3.MLVisual主要功能与操作方法MLVisual提供了丰富的功能,可以帮助您全面展示机器学习模型。
以下是一些主要功能及其操作方法:- 展示神经网络结构:使用`mv.model_structure()`函数可视化模型结构。
- 绘制训练过程曲线:使用`mv.train_curve()`函数展示训练过程中的损失值和验证损失值。
- 分析模型性能:使用`mv.heatmap()`函数绘制权重矩阵,以便找出关键特征。
WGCNA新手入门笔记2(含代码和数据)

WGCNA新手入门笔记2(含代码和数据)上次我们介绍了WGCNA的入门(WGCNA新手入门笔记(含代码和数据)),大家在安装WGCNA包的时候,可能会遇到GO.db这个包安装不了的问题。
主要问题应该是出在电脑的防火墙,安装时请关闭防火墙。
如果还有问题,请先单独安装AnnotationDbi这个包,biocLite("AnnotationDbi")再安装GO.db,并尝试从本地文件安装该包。
如果还有问题,请使用管理员身份运行R语言,尝试上述步骤。
另外如果大家问题解决了请在留言处留个言,告知大家是在哪一步解决了问题,谢谢!因为本人没有进行单因素实验,不知道到底是哪个因素改变了实验结果。
今天给大家过一遍代码。
网盘中有代码和数据。
链接:/s/1bpvu9Dt密码:w7g4##导入数据##library(WGCNA)options(stringsAsFactors =FALSE)enableWGCNAThreads()enableWGCNAThreads()是指允许R语言程序最大线程运行,像我这电脑是4核CPU的,那么就能用上3核:当然如果当前电脑没别的事,也可以满负荷运作samples=read.csv( 'Sam_info.txt',sep = 't',s =1)expro=read.csv( 'ExpData.txt',sep = 't',s =1)dim(expro)这部分代码是为了让R语言读取外部数据。
当然了在读取数据之前首先改变一下工作目录,这一点在周二的文章中提过了。
R语言读取外部数据的方式常用的有read.table和read.csv,这里用的是read.csv,想要查看某一函数的具体参数,可以用?函数名查看,比如:大家可以注意到read.table和read.csv中header参数的默认值是不同的,header=true表示第一行是标题,第二行才是数据,header=false则表示第一行就是数据,没有标题。
ComplexHeatmap绘制热图详细步骤

ComplexHeatmap绘制热图详细步骤pheatmap是⼀个⾮常受欢迎的绘制热图的R包。
ComplexHeatmap包即是受之启发⽽来。
你可以发现Heatmap()函数中很多参数都与pheatmap()相同。
在pheatmap的时代(请允许我这么说),pheatmap意思是pretty heatmap,但是随着时间推进,技术发展,各种新的数据出现,pretty is no more pretty,我们需要更加复杂和更有效率的热图可视化⽅法对庞⼤的数据进⾏快速并且有效的解读,因此我开发并且⼀直维护和改进着ComplexHeatmap 包。
为了使庞⼤并且“陈旧”的(对不起,我不应该这么说)pheatmap⽤户群能够迅速并且⽆痛的迁移⾄ComplexHeatmap,从2.5.2版本开始,我在ComplexHeatmap包中加⼊了⼀个pheatm ap()函数,它涵盖了pheatmap::pheatmap()所有的功能,也就是说,它提供了和pheatmap:: pheatmap()⼀模⼀样的参数,并且⽣成的热图的样式也⼏乎相同。
同时,ComplexHeatmap: :pheatmap()函数也能使⽤ComplexHeatmap独有的功能,⽐如对⾏和列进⾏切分,加⼊⾃定义的annotation,多个热图和annotation的连接,或者创建⼀个互动的热图(interactive heatmap, 通过ht_shiny()函数)ComplexHeatmap::pheatmap()包含了pheatmap::pheatmap()中所有的参数,这意味着,当你从pheatmap迁移⾄ComplexHeatmap时,你⽆需添加任何额外的步骤,你只需要载⼊ComplexHeatmap⽽不是pheatmap包,然后重新运⾏你原始的pheatmap代码。
剩下的你只是去见证奇迹的发⽣。
注意如下五个pheatmap::pheatmap()的参数在ComplexHeatmap::pheatmap()中被忽视:kmeans_k:在pheatmap::pheatmap()中,如果这个参数被设定,输⼊矩阵会进⾏k均值聚类,然后每个cluster使⽤其均值向量表⽰。
教你画一个掰弯的热图(Heatmap),展示更多的基因表达量
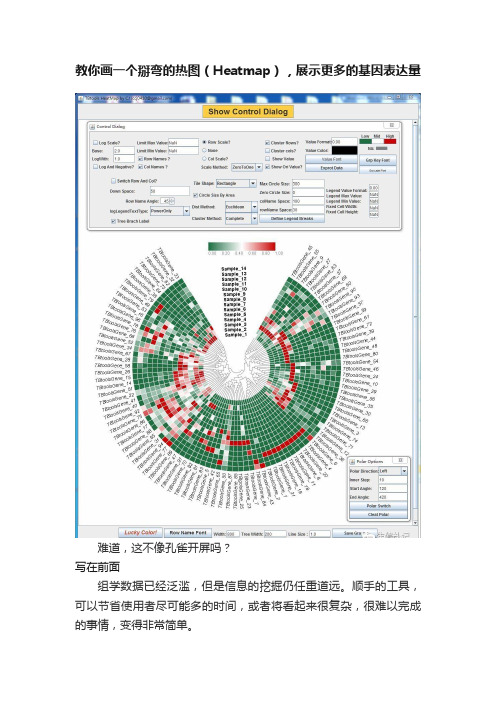
教你画一个掰弯的热图(Heatmap),展示更多的基因表达量难道,这不像孔雀开屏吗?写在前面组学数据已经泛滥,但是信息的挖掘仍任重道远。
顺手的工具,可以节省使用者尽可能多的时间,或者将看起来很复杂,很难以完成的事情,变得非常简单。
图形的掰弯,这个是我很久以前已经实现的功能,当然这个功能的灵感(而不是实现逻辑)来至《The Grammer of Graphics》(这本书也是R包ggplot2的实现基础)。
图形的掰弯,正如本公众号早前的推文介绍,事实是笛卡尔坐标系像极坐标系转换的过程。
坐标的转换是一个事情,而图形的变换是另一个事情,比如矩形要办成中空的扇形等。
热图,我已经不止一次谈及,如果早前有关注生信札记公众号的朋友,那么应该知道,TBtools有两个热图功能,Amazing Heatmap和Fancy Heatmap Browser,各有所长。
一般用过的人,大部分时候都会觉得真的是Amazing。
后者目前似乎用的人不多,不过我们课题组有一些相关成果会应用上。
或许有时候,过于非常规的出图逻辑(往往也是更优秀的发表级图片)并不能为多数人所接受。
所以没有人用,也是意料之内。
优秀的人,总是会突破已有局限的。
我写Amazing Heatmap在先,而后续才实现了掰弯逻辑。
掰弯逻辑的实现,本身是为了Circos图工具的实现。
但是我并没有想到,会有TBtools用户在我之前挖掘出了掰弯的热图的使用逻辑。
掰弯的热图,目前已可在数个已发表工作中见到。
往往,使用这一功能的目的,可能是尽可能展示更多的基因。
这个需求其实很容易产生。
有时候,我们展示十几个基因,那么常规的矩形热图效果就很不错;但是,当基因数目上升到数十上百,那么可能图版就不好放下。
此时,圆环状的优势就会体现出来。
工具的使用首先,应该升级TBtools到最新版本,然后,还是打开Amazing Heatmap这个工具然后输入数据,我直接用一个好基友的数据(屏蔽了样品和基因名字),于是得到下图,恩,基因数太多,一般可以缩小文字,当然,更多的时候,是调整图片的大小,同时也调一下配色,得到下图很明显,此时图片太长。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
一步一步学heatmap.2函数数据如下:library(gplots)data(mtcars)x <- as.matrix(mtcars)rc <- rainbow(nrow(x), start=0, end=.3)cc <- rainbow(ncol(x), start=0, end=.3)X就是一个矩阵,里面是我们需要画热图的数据。
Rc是一个调色板,有32个颜色,渐进的Cc也是一个调色板,有11个颜色,也是渐进的首先画一个默认的图:heatmap.2(x)然后可以把聚类数可以去掉:就是控制这个dendrogram参数heatmap.2(x, dendrogram="none")然后我们控制一下聚类树heatmap.2(x, dendrogram="row") 只显示行向量的聚类情况heatmap.2(x, dendrogram="col")只显示列向量的聚类情况下面还是在调控聚类树,但是我没看懂跟上面的参数有啥子区别!heatmap.2(x, keysize=2) ## default - dendrogram plotted and reordering done.heatmap.2(x, Rowv=FALSE, dendrogram="both") ## generate warning!heatmap.2(x, Rowv=NULL, dendrogram="both") ## generate warning!heatmap.2(x, Colv=FALSE, dendrogram="both") ## generate warning!接下来我们可以调控行列向量的label的字体大小方向首先我们调控列向量,也就是x轴的labelheatmap.2(x, srtCol=NULL)heatmap.2(x, srtCol=0, adjCol = c(0.5,1) )heatmap.2(x, srtCol=45, adjCol = c(1,1) )heatmap.2(x, srtCol=135, adjCol = c(1,0) )heatmap.2(x, srtCol=180, adjCol = c(0.5,0) )heatmap.2(x, srtCol=225, adjCol = c(0,0) ) ## not very useful heatmap.2(x, srtCol=270, adjCol = c(0,0.5) )heatmap.2(x, srtCol=315, adjCol = c(0,1) )heatmap.2(x, srtCol=360, adjCol = c(0.5,1) )然后我们调控一下行向量,也就是y轴的labelheatmap.2(x, srtRow=45, adjRow=c(0, 1) )heatmap.2(x, srtRow=45, adjRow=c(0, 1), srtCol=45, adjCol=c(1,1) )heatmap.2(x, srtRow=45, adjRow=c(0, 1), srtCol=270, adjCol=c(0,0.5) )设置offsetRow/offsetCol 可以把label跟热图隔开!## Show effect of offsetRow/offsetCol (only works when srtRow/srtCol is ## not also present)heatmap.2(x, offsetRow=0, offsetCol=0)heatmap.2(x, offsetRow=1, offsetCol=1)heatmap.2(x, offsetRow=2, offsetCol=2)heatmap.2(x, offsetRow=-1, offsetCol=-1)heatmap.2(x, srtRow=0, srtCol=90, offsetRow=0, offsetCol=0)heatmap.2(x, srtRow=0, srtCol=90, offsetRow=1, offsetCol=1)heatmap.2(x, srtRow=0, srtCol=90, offsetRow=2, offsetCol=2)heatmap.2(x, srtRow=0, srtCol=90, offsetRow=-1, offsetCol=-1)## Show effect of z-score scaling within columns, blue-red color scale##hv <- heatmap.2(x, col=bluered, scale="column", tracecol="#303030")hv是一个热图对象!!!> names(hv)可以看到hv对象里面有很多子对象"rowInd" "colInd" "call" "colMeans" "colSDs" "carpet" "rowDendrogram" "colDendrogram" "breaks" "col" "vline" "colorTable"## Show the mapping of z-score values to color binshv$colorTable## Extract the range associated with white我们得到了热图的颜色的数值映射矩阵,接下来就可以进行一系列的操作~!!!hv$colorTable[hv$colorTable[,"color"]=="#FFFFFF",]首先得到了白色所对应的数值区间!然后还可以通过一下命令,直接求出属于白色区间的那些数值。
whiteBin <- unlist(hv$colorTable[hv$colorTable[,"color"]=="#FFFFFF",1:2]) rbind(whiteBin[1] * hv$colSDs + hv$colMeans,whiteBin[2] * hv$colSDs + hv$colMeans )调整scale参数选择按照列还是行来进行数据的标准化heatmap.2(x, col=bluered, scale="column", tracecol="#303030")heatmap.2(x, col=bluered, scale="row", tracecol="#303030")如果选择了标准化,那么还可以手工调整标准化的参数:rowMeans, rowSDsmean and standard deviation of each row: only present if scale="row" colMeans, colSDsmean and standard deviation of each column: only present if scale="column"通过hclustfun参数来调整聚类方法参考:/foreverycc/archive/2012/10/19/2730391.htmlCluster_Method<-c( "ward", "single", "complete", "average", "mcquitty", "median", "centroid")#R语言里面自带的hclust函数共有7种聚类方法for (i in 1:length(Cluster_Method)){#make a function to extract the cluster methodmyclust<-function(x){hclust(x,method=Cluster_Method[i])}#make heatmap by jpegjpeg(filename=paste(Cluster_Method[i],'.jpg'),width=1024,height=728)heatmap.2(as.matrix(Data_Top1k_Var),trace='none',hclustfun=myclust,labRow=NA,ColSideColors=c('black',grey(0.4),'lightgrey')[as.factor(CellLine_Anno$Type)],xlab='CellLines',ylab='Probes',main=Cluster_Method[i],col=greenred(64))dev.off()}这样就可以一下子把七种cluster的方法依次用到heatmap上面来。
而且通过对cluster树的比较,我们可以从中挑选出最好、最稳定到cluster方法,为后续分析打好基础!对下面这个数据聚类:require(graphics)hc <- hclust(dist(USArrests), "ave")plot(hc)首先对一个数据框用dist函数处理得到一个dist对象!Dist对象比较特殊,专门为hclust函数来画聚类树的!。
