Sequenom_SNP实验过程说明
SNP实验标准操作规程

一、原理SNP (Single Nucleotide Polymorphism)即单核苷酸多态性,是由于单个核苷酸改变而导致的核酸序列多态性(Polymorphism)。
据估计,在人类基因组中,大约每千个碱基中有一个SNP,无论是比较于限制性片段长度多态性(RFLP)分析还是微卫星标记(STR),都要广泛得多。
SNP是我们考察遗传变异的最小单位,据估计,人类的所有群体中大约存在一千万个SNP位点。
一般认为,相邻的SNPs倾向于一起遗传给后代。
于是,我们把位于染色体上某一区域的一组相关联的SNP等位位点称作单体型(haplotype)。
大多数染色体区域只有少数几个常见的单体型(每个具有至少5%的频率),它们代表了一个群体中人与人之间的大部分多态性。
一个染色体区域可以有很多SNP位点,但是我们一旦掌握了这个区域的单体型,就可以只使用少数几个标签SNPs(tagSNP)来进行基因分型,获取大部分的遗传多态模式。
二、样品准备1. DNA抽提① 1、取新鲜肌肉组织约100mg,PBS漂洗干净,置于离心管中,加入液氮,迅速磨碎。
②加200μl 缓冲液GA,震荡至彻底悬浮。
加入20μl 蛋白酶K(20mg/ml)溶液,混匀③加220μl 缓冲液GB,充分混匀,37℃消化过夜,溶液变清亮。
加220μl 无水乙醇,充分混匀,此时可能会出现絮状沉淀。
④将上述一步所得溶液和絮状沉淀都加入一个吸附柱CB 中,(吸附柱放入废液收集管中)12000rpm 离心30 秒,弃掉废液。
⑤加入500μl 去蛋白液GD(使用前请先检查是否已加入无水乙醇),12000rpm 离心30 秒,弃掉废液。
⑥加入700μl 漂洗液GW(使用前请先检查是否已加入无水乙醇),12000rpm离心30 秒,弃掉废液。
加入500μl 漂洗液GW, 12000rpm 离心30 秒,弃掉废液。
将吸附柱CB 放回废液收集管中,12000rpm 离心2 分钟,尽量除去漂洗液。
SNP的原理以及应用原理

SNP的原理以及应用原理SNP(单核苷酸多态性)的定义SNP (Single Nucleotide Polymorphism),即单核苷酸多态性,是指基因组中存在的单个核苷酸的位置变异。
这种变异可能是由于单个碱基的替换、插入或删除引起的。
SNP是遗传变异中最常见的形式,也是人类基因组中最常见的遗传变异类型之一。
SNP的原理1.比对参考基因组:首先,SNP的测序团队会将被测个体的DNA样本与一个参考基因组进行比对。
参考基因组是一个代表人类基因组的模型序列。
2.寻找变异位点:接下来,比对结果会被分析软件使用,并寻找与参考基因组不同的位点,即潜在的SNP位点。
3.重测序:对于潜在的SNP位点,需要进行一个额外的步骤来确认该变异是否真的存在。
这个步骤被称为重测序,即对该位点进行多次测序,以保证准确性和可靠性。
4.鉴别基因型:在确认SNP位点后,就需要确定该位点的基因型。
基因型指的是一个SNP位点上两个等位基因的组合方式。
在人类中,一个等位基因可以来自父亲,另一个等位基因可以来自母亲。
5.数据分析:最后,SNP数据需要经过严格的分析以确定每个个体具体的基因型。
这种数据分析需要运用一系列统计学、计算机科学和生物学的方法。
SNP的应用原理SNP作为一种常见的遗传变异类型,具有广泛的应用。
以下是SNP在医学和生物研究中的应用原理的一些例子:1. 疾病相关性研究SNP在疾病的发病机制研究中发挥了重要作用。
通过比较在患病和正常人群中SNP的频率和分布情况,可以找到与某种疾病相关的SNP位点。
这种位点的发现有助于揭示疾病的遗传风险因素,并且为疾病的早期预测、诊断和治疗提供了基础。
2. 药物反应个体化SNP也可以帮助确定特定个体对药物的反应。
通过分析某些药物代谢酶基因上的SNP位点,可以预测一个人对某种药物的敏感性和药代动力学。
这使得医生能够根据个体的基因型来优化药物治疗,从而提高疗效和减少不良反应。
3. 种群遗传学研究SNP可以用于研究不同种群之间的遗传差异。
SNP检测原理和应用

变性梯度凝胶电泳(DGGE)
原理:是利用长度相同的双链DNA片段解链温度不同 的原理,通过梯度变性胶将DNA片段分开的电泳技术。 电泳开始时,DNA在胶中的迁移速率仅与分子大小 有关,而一旦DNA泳动到某一点时,即到达该DNA变 性浓度位置时,使得DNA双链开始分开,从而大大降 低了迁移速率。当迁移阻力与电场力平衡时,DNA片 段在凝胶中基本停止迁移。由于不同的DNA片段的碱 基组成有差异,使得其变性条件产生差异,从而在凝胶 上形成不同的条带。
特点:使用高效液相色谱检测SNPs具有检测效率高,便于自 动 化 的 优 点 , 对 未 知 SNPs 的 准 确 率 可 达 95% 以 上 。 但 DHPLC检测对所用试剂和环境要求较高,容易产生误差, 不能检测出纯合突变。
阅微基因提供的SNP检测方法
基于荧光定量PCR平台的TaqMan探针法; 基于ABI遗传分析仪平台的SnapShot法; 基于Sequenom质谱仪平台的MassArray法
它包括单碱基的转换,颠换、 插入及缺失等形式。
SNP在基因组内的形式:
一是遍布于基因组的大量单碱基变异; 二是分布在基因编码区(coding region) ,称其 为cSNP,属功能性突变。
SNP在单个基因或整个基因组的分布是不均匀的: (1)非转录序列要多于转录序列; (2)在转录区非同义突变的频率,比其他方式突变的频率
SnapShot法
该技术由美国应用生物公司(ABI)开发,基于荧光标记 单碱基延伸原理的分型技术,也称小测序,主要针对中等 通量的SNP分型项目。
在一个含有测序酶,四种荧光标记的ddNTP,紧挨多 态位点5’端的不同长度延伸引物和PCR产物模板的反应体 系中,引物延伸一个碱基即终止,经ABI测序仪电泳后, 根据峰的颜色可知掺入的碱基种类,从而确定该样本的基 因型,根据峰移动的胶位置确定该延伸产物对应的SNP位 点。
SNP检测详细步骤

SNP检测(中文)Part I:样本基因组DNA的提取1.取50 μl血样于离心管中,加PBS缓冲液至1.5mL,轻轻地摇匀。
冷冻离心机6500 rpm离心10 min,去掉上清液,保留沉淀物。
重复洗2次。
2.向保留沉淀物的离心管中加入DNA提取液500 μl,15 μl的蛋白酶K,混匀放入55℃水浴锅中消化过夜。
3.将消化过夜的反应液冷却至室温,加入等体积冰冷的饱和酚溶液,盖紧离心管盖,缓慢地来回颠倒10 min(在冰上进行),形成均匀的乳浊液。
4.冷冻离心机12000 rpm离心10min。
5.小心地吸取上层水相至新管,用等体积饱和酚再抽提一次。
6.用等体积的氯仿再抽提一次。
7.离心后再取上清液于另一离心管中,加入1∕10体积3mol/L的NaAc使终浓度达到0.3mol/L,并加2倍体积冷无水乙醇,上下倒置混匀,置-20℃冰箱沉淀30-60min。
8.冷冻离心机12000 rpm离心10 min,弃上清液。
9.加入500 μl 70%冷乙醇小心洗涤沉淀。
冷冻离心机6500 rpm离心5 min,弃上清,用干净的吸水纸或用吸头将管壁残留的乙醇去除,干燥10~15 min,不要等沉淀完全干燥,否则难以溶解。
10.沉淀于100 μl超纯水中。
11.将提取的基因组DNA进行琼脂糖凝胶电泳及浓度的测定。
Part II:SNP分型检测1.引物的设计与合成(1)查阅文献,参考文献中的引物,直接合成;(2)根据SNP的位置找到其序列,设计引物并合成2.PCR扩增片段(1)PCR扩增体系:Components Volume (μl)DNA template1PrimeSTAR0.5dNTPs (2.5 mM)1Primer-F (10 μM)1Primer-R (10 μM)15*PS buffer(Mg2+)10ddH2O1(2)PCR扩增程序:(3)将PCR产物进行琼脂糖凝胶电泳检测。
(4)A. 测序法:对目的条带进行切胶回收纯化测序,根据测序结果统计分析各个样本下该SNP的基因型。
SNP开发验证的研究方法和技术路线

SNP开发/验证的研究方法和技术路线1分子标记:分子标记,我想这部分是我们分子标记组最核心的任务。
现在,我们没有任何可用的标记检测我们的定位材料。
即使想要验证已经定位的QTLs,我们也需要相对应的区间内的分子标记,尤其是SNP标记。
1.1全基因组SNP—Asymetrix芯片:一套完整的全基因组的SNP芯片,相对于Douglas体系,其操作简单,高通量。
可以直接对定位群体进行初定位的扫描或是对育种材料的背景进行分析。
在国家玉米改良中心,有一套3k的lllumina芯片,就是用来对玉米材料进行高通量检测,基因型检测结果通常可以用来QTLs初定位,育种材料的群体划分与纯度鉴定以及低密度的关联分析等。
在此,我建议我们应该开发一套番茄基因型检测的芯片。
目前,只是查找到lllumina芯片有一套全基因SNP信息,包含7,720条探针。
而Affymetrix公司目前并没有相应的产品。
但是通过跟Affymetrix公司了解,可以利用lllumina芯片已有的结果进行开发。
番茄目前测序结果显示其全基因组大小为~760Mb,而玉米为~2,500Mb,但是他们包括的基因数目-30,000个,整体情况相近。
另外,番茄作为自交植物, 其LD 的衰减值应该更大,有效的历史重组会更少,遗传多样性低。
因此,综合考虑,我建议我们可以开发~3k芯片,应该可以满足大多数研究材料、育种材料的基因型检测需求。
虽然目前下一代测序技术蓬勃发展,但是对于用于基因型检测来讲,其数据分析与成本相对于芯片都要更复杂和更高。
总之,我们番茄处于刚刚发展阶段,我认为就基因型检测方面,芯片有其很高的应用价值。
即使像玉米,这样测序技术发展很多年的材料,芯片技术也在应用。
1.2 全基因组SNP—Douglas:当用Affymetrix芯片检测鉴定完番茄基因型并完成基因型分析之后,1)对于优良的QTLs或是基因,我们可以直接选择覆盖整个区间的分子标记运行Douglas系统进行分子标记辅助育种,2)对于需要进一步验证的QTLs,我们也可以利用Douglas系统只检测材料覆盖定位区间的基因型,而不需要再一次利用Affymetrix芯片或是其他方法进行全基因检测(图1.1)。
SEQUENOM-iPLEX标准操作流程
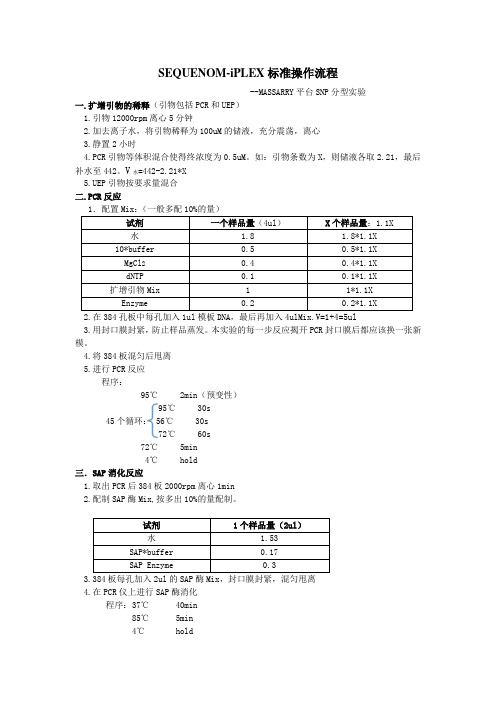
SEQUENOM-iPLEX标准操作流程--MASSARRY平台SNP分型实验一.扩增引物的稀释(引物包括PCR和UEP)1.引物12000rpm离心5分钟2.加去离子水,将引物稀释为100uM的储液,充分震荡,离心3.静置2小时4.PCR引物等体积混合使得终浓度为0.5uM。
如:引物条数为X,则储液各取2.21,最后补水至442。
V水=442-2.21*X5.UEP引物按要求量混合二.PCR反应1.配置Mix:(一般多配10%的量)试剂一个样品量(4ul)X个样品量:1.1X水 1.8 1.8*1.1X 10*buffer 0.5 0.5*1.1XMgCl20.4 0.4*1.1XdNTP 0.1 0.1*1.1X 扩增引物Mix 1 1*1.1XEnzyme 0.2 0.2*1.1X2.在384孔板中每孔加入1ul模板DNA,最后再加入4ulMix.V=1+4=5ul3.用封口膜封紧,防止样品蒸发。
本实验的每一步反应揭开PCR封口膜后都应该换一张新模。
4.将384板混匀后甩离5.进行PCR反应程序:95℃ 2min(预变性)95℃ 30s45个循环: 56℃ 30s72℃ 60s72℃ 5min4℃ hold三.SAP消化反应1.取出PCR后384板2000rpm离心1min2.配制SAP酶Mix,按多出10%的量配制。
试剂1个样品量(2ul)水 1.53SAP*buffer 0.17SAP Enzyme 0.33.384板每孔加入2ul的SAP酶Mix,封口膜封紧,混匀甩离4.在PCR仪上进行SAP酶消化程序:37℃ 40min85℃ 5min4℃ hold四.UEP延伸1. 取出SAP反应板2000rpm离心1min2. 配制延伸Mix,按多出10%的量配制。
试剂1个样本量(2ul)水0.619Gold*buffer 0.2Termination mix 0.2UEP引物 Mix 0.94Enzyme 0.0413.384板每孔加入2ul的延伸Mix,封口膜封紧,混匀甩离4.在PCR仪上进行下列反应程序:94℃ 30s94℃ 5s52℃ 5s 40个外部循环80℃ 5 5个内部循环72℃ 3min4℃ hold五.树脂纯化1.将384板2000rpm离心2min,每孔加入16ul水。
干货分享SNP验证及使用_专题(上)

⼲货分享SNP验证及使⽤_专题(上)前⾔利⽤基因组测序(全基因组重测序、简化基因组测序)或基因芯⽚技术可获得⼤量的SNP标记,那么接下来⼤家都会考虑⼀个问题:我们该如何去验证和使⽤它们呢?针对不同实验⽬的,⼩编给出以下建议。
情境⼀由公司完成SNP分型并执⾏相关分析要求(如群体遗传分析)之后,如果我们后续的研究不再需要SNP标记进⾏辅助,仅单纯想验证⼀下SNP分型结果的准确性。
那么⼩编推荐使⽤Sanger 测序法、Taqman探针法、Sequenom、SNaPshot等技术,可⼀次对⼗⼏个或⼏⼗个SNP位点进⾏验证,并能对分型结果的可靠性作出较准确的评估。
情境⼆如果我们后续的研究中仍需要使⽤SNP标记进⾏辅助,如品种特异分⼦标记开发、图位克隆、分⼦标记辅助育种(MAS)等。
那么⼩编建议在验证SNP分型准确性的同时,可将其转化成以PCR为基础的分⼦标记,以⽅便各位在⾃⼰实验室开展后续⼯作。
此类型的⽅法主要有CAPS、dCAPS、AS-PCR三种,讲到这,本专题的3位主⾓终于登场。
鉴于前两位的技术原理类似,故将它们⼀起作为本专题的第⼀发与⼤家见⾯。
(满满的⼲货奉上 ~)CAPS标记1.全称CleavedAmplified Polymorphism Sequences(CAPS)——酶切扩增多态性序列2.原理简介若SNP刚好处于某⼀限制性内切酶的识别位点上,则可通过PCR技术与RFLP技术的结合,将SNP转化为CAPS标记。
基本步骤是在SNP位点的两侧设计引物,PCR特异性扩增,使⽤相应识别位点的限制性内切酶对PCR产物进⾏酶切后电泳检其多态性。
CAPS为共显性分⼦标记,可判断纯杂合。
(讲到这⾥⼤家可能还有较多疑惑,接着往下看)3.实例解析以拟南芥研究中经常会使⽤的两种⽣态型材料Col与Ler为例,经⾼通量测序发现两⽣态型材料在基因组某处存在纯合差异的SNP位点(灰⾊背景位置),观察发现在Col材料中该SNP位点刚好位于EcoRI的酶切识别位点上(红⾊字体,5’-G▼AATTC-3’),⽽Ler材料中该位点为G碱基,不存在EcoRI识别位点。
sequenom-iPLEX实验操作

iPLEX实验操作一、引物设计用mysequenom网上工具设计(步骤略)二、扩增引物的稀释和UEP引物质量检测1.合成回来的扩增引物加去离子水,稀释为100uM的储液。
之后等体积混合使得终浓度为0.5uM。
如做14个Plex的反应,共28条扩增引物。
每条引物取1ul混合共28ul,再加入172ul的去离子水,总体积为200ul,即为引物Mix(0.5μM)。
2.合成回来的UEP引物按照“UEP引物稀释表”中的黄色高亮部分输入:ID号,Assay的命名,UEP引物的Mass,合成的管中的总OD量,总共有n 个Plex信息。
根据表格计算出每个管中需要加的去离子水量,进行引物重溶。
之后每个管中取等体积引物充分混合即为UEP Mix。
3.将前述UEP Mix 2ul加入40ul去离子水中混合。
加入384孔板中,可用于直接点芯片,打质谱。
这一步检测UEP的质量是必须的,如果质量不好必须重新设计。
三、DNA样品准备基因组DNA样品应电泳检测完整性,并测浓度。
用去离子水或Tris-Cl稀释为浓度10-20ng/ul。
由于样品量大,必须做好样品标记。
四、PCR反应1.在384孔板上一定要做好标记:横6,纵4的24个孔为一组。
以一组为将来点样时候的一针。
2.按多出来10-15%的量来配制Mix,如需检测40个样,则配50-55个样的量反应体系:均按ul计算模板DNA(10ng/ul) 1引物Mix(0.5μM)0.1µM 110*Buffer(含Mg2+)2mM MgCl2 0.5MgCl2(25mM) 2mM 0.4dNTP(25mM) 500µM 0.1Hotstar(5U/μl) 1 Unit 0.1*水 1.9总量5ul*如果做27个Plex以上的PCR,应用0.2 ul3.在384孔板中每孔加入4ul Mix,最后在加入1ul模板DNA。
4.用ABI的PCR封口膜封紧,防止样品蒸发。
全基因组snp分型步骤

全基因组snp分型步骤1.引言1.1 概述全基因组SNP分型是一种用于分析人类基因组中的单核苷酸多态性(Single Nucleotide Polymorphism,SNP)的方法。
SNP是指基因组中单个核苷酸的变异,这种变异可能与遗传疾病、药物反应等多种生物学特征相关。
全基因组SNP分型通过对整个基因组中的SNP进行分析,可以帮助我们了解人类基因组的个体差异,从而更好地理解遗传病理学、个体化医疗以及演化等方面的问题。
全基因组SNP分型的研究步骤包括样本准备、DNA提取和测序、数据处理和质量控制以及SNP分型算法。
首先,我们需要准备研究所需的样本,并对样本进行处理以获取所需的DNA。
接着,通过测序技术对DNA 进行测序,得到原始的测序数据。
在数据处理和质量控制阶段,我们需要对原始数据进行处理和过滤,以确保数据的准确性和可靠性。
最后,我们使用各种SNP分型算法对处理后的数据进行分析和解读,以获取SNP位点的基因型信息。
全基因组SNP分型具有广泛的应用前景。
在科学研究领域,它可以帮助我们研究遗传病理学、复杂疾病的致病机制以及人类演化历史等重要问题。
在临床医学中,全基因组SNP分型可以帮助医生进行个体化医疗决策,根据患者的基因信息选择最适合的治疗方案,提高治疗效果。
此外,全基因组SNP分型还可以应用于人口遗传学研究、药物研发与评价等方面,为我们提供更多关于人类基因组的信息。
本文将详细介绍全基因组SNP分型的步骤,希望能够为读者提供一个清晰的了解和入门指南,并展示全基因组SNP分型在生命科学领域的重要性和应用前景。
1.2 文章结构文章结构部分的内容可以包括以下内容:本文将按照以下顺序介绍全基因组SNP分型的步骤。
首先,我们将在引言部分进行概述,介绍全基因组SNP分型的定义、背景知识和研究目的。
接下来,在正文部分,我们将详细介绍全基因组SNP分型的步骤。
其中,包括样本准备、DNA提取和测序、数据处理和质量控制以及SNP 分型算法的介绍。
SNP实验标准操作规程

一、原理SNP (Single Nucleotide Polymorphism)即单核苷酸多态性,是由于单个核苷酸改变而导致的核酸序列多态性(Polymorphism)。
据估计,在人类基因组中,大约每千个碱基中有一个SNP,无论是比较于限制性片段长度多态性(RFLP)分析还是微卫星标记(STR),都要广泛得多。
SNP是我们考察遗传变异的最小单位,据估计,人类的所有群体中大约存在一千万个SNP位点。
一般认为,相邻的SNP s倾向于一起遗传给后代。
于是,我们把位于染色体上某一区域的一组相关联的SNP等位位点称作单体型(haplotype)。
大多数染色体区域只有少数几个常见的单体型(每个具有至少5%的频率),它们代表了一个群体中人与人之间的大部分多态性。
一个染色体区域可以有很多SNP位点,但是我们一旦掌握了这个区域的单体型,就可以只使用少数几个标签SNPs(tagSNP)来进行基因分型,获取大部分的遗传多态模式。
二、样品准备1. DNA抽提①1、取新鲜肌肉组织约100mg,PBS漂洗干净,置于1.5ml离心管中,加入液氮,迅速磨碎。
②加200μl 缓冲液GA,震荡至彻底悬浮。
加入20μl 蛋白酶K(20mg/ml)溶液,混匀③加220μl 缓冲液GB,充分混匀,37℃消化过夜,溶液变清亮。
加220μl 无水乙醇,充分混匀,此时可能会出现絮状沉淀。
④将上述一步所得溶液和絮状沉淀都加入一个吸附柱CB 中,(吸附柱放入废液收集管中)12000rpm 离心30 秒,弃掉废液。
⑤加入500μl 去蛋白液GD(使用前请先检查是否已加入无水乙醇),12000rp m 离心30 秒,弃掉废液。
⑥加入700μl 漂洗液GW(使用前请先检查是否已加入无水乙醇),12000rpm 离心30 秒,弃掉废液。
加入500μl 漂洗液GW, 12000rpm 离心30 秒,弃掉废液。
将吸附柱CB 放回废液收集管中,12000rpm 离心2 分钟,尽量除去漂洗液。
StepOneV2.X SNP实验简易操作流程

applied biosystemsStepOne/StepOnePlus 荧光定量PCR 仪SNP 实验简易操作流程SDS 2.21StepOne/StepOnePlus 定量PCR 仪SNP 实验简易操作流程1. 双击桌面图标 ,或从Start > All Programs > Applied Biosystems > StepOne Software> StepOne Software V2.2开启软件。
进入主界面后选择Advanced Setup 。
2. 进入Setup 下的Experiment Properties 界面:2.1 输入实验名称(Experiment Name )2.2 确认仪器型号2.32.3 在实验类型中,选择Genotyping22.4 选择试剂种类2.5 选择运行模式2.6 在定量仪器上使用默认设置进行预读板及扩增的过程3. 进入Setup 下的Plate Setup 界面,编辑SNP 位点信息及样本信息:3.1 在 界面中设置需要检测的SNP 位点。
选择3下的Edit SNP assay ,编辑assay 的名称及碱基种类,设置Report (报告基团)和 quencher (淬灭基团)。
若要添加其他SNP 位点,点击 增加。
3.2 在 界面中编辑样本信息。
3.3 在右侧 界面中编辑样品板。
利用鼠标单选或者拖拽以选择反应孔,然后选择界面左侧的SNP assay 和Sample ,并在SNP assay 的Task 一栏中指定该样本的种类(Unknown 未知样本,Negative Control 阴性对照或Positive Control 阳性对照)。
4. 进入Run Method界面,设定反应条件。
45. 点击 ,将文件存储成Experiment Document Single files (*.eds) 格式,然后按下 开始反应。
56. 实验结束后,先点击界面右上方的Analyze 进行分析,然后进入Allelic Discrimination Plot 界面观察分型结果。
基因组survey的数据snp位点开发

基因组survey的数据snp位点开发
基因组survey的数据,包括SNP位点,是通过高通量测序技术来开发的。
这些技术可以同时检测整个基因组中的大量位点,包括SNP。
开发SNP位点数据需要对样本进行DNA测序,然后使用计算方法进行数据分析,以确定哪些位点是SNP。
这些数据可用于研究遗传学、疾病关联和基因组进化等领域。
具体来说,基因组survey的数据开发过程大致如下:
1.样本收集: 首先需要收集大量的DNA样本,这些样本可以来自
人类、动物或植物等生物。
2.DNA测序: 接下来对样本中的DNA进行测序。
目前,最常用的
测序技术是高通量测序技术,如NGS(next-generation
sequencing)。
这些技术可以同时检测整个基因组中的大量位
点。
3.数据分析: 测序得到的数据需要进行分析。
首先,使用软件将原
始数据进行质量控制和过滤,然后使用计算方法进行数据分析,确定哪些位点是SNP。
4.数据整理: 最后将分析得到的SNP数据整理成数据库或文件,供
研究人员使用。
这些数据可用于研究遗传学、疾病关联和基因组进化等领域。
需要注意的是,这是一个简化的过程描述,在实际操作中,还需要考虑很多其他因素,如数据格式、数据管理、数据可视化等。
蜜蜂序列组装分析及SNP位点检测

蜜蜂序列组装分析及SNP位点检测蜜蜂是我们非常熟悉的昆虫之一,也是非常重要的生态系统组成部分。
在蜜蜂的研究中,基因组学技术也越来越受到关注和应用。
本文将介绍蜜蜂基因组组装和SNP位点分析的相关内容。
一、蜜蜂基因组组装基因组组装是将测序数据转换为完整的基因组序列的过程。
蜜蜂基因组组装的过程和其他生物物种的基因组组装类似,但由于其基因组大小较小,组装过程相对较容易。
蜜蜂基因组组装的第一步是建立一个高质量的基因组序列库。
这包括用不同的方法制备高质量的DNA样品、建立测序文库并进行高通量测序等。
蜜蜂的基因组测序是高度复杂的过程,需要通过多个测序平台(如Illumina HiSeq、PacBio等)进行组合。
在获得测序数据后,需要对数据进行预处理,如去除低质量序列、去除冗余序列、纠正测序错误等。
然后,将这些清洗后的序列通过不同的软件进行组装,并利用其他评估工具对组装质量进行评估。
最终的基因组序列可以通过验证和加工来达到最终的精度。
二、SNP位点检测SNP(single nucleotide polymorphism)是指基因组中的单个碱基差异。
SNP是生物基因组中最常见的组成成分之一,也是进化研究和基因组组装等生物信息学研究中广泛应用的工具之一。
在蜜蜂研究中,SNP位点分析可以帮助我们了解种群群体、家系和探测基因功能等。
SNP位点检测的步骤包括:(1)基因组序列和基因序列的比对;(2)确立SNP位点;(3)SNP位点筛选和统计;(4)SNP位点功能分析。
首先,需要将测序数据比对到参考基因组序列上,然后使用SNP检测软件如SAMtools、GATK等,通过生物统计学方法筛选SNP位点。
接下来,使用过滤器将SNP位点进行分组和筛选,去除无效SNP位点,比如低质量位点。
最终,SNP位点的功能分析可以通过注释工具进行。
这包括检测SNP位点是否对蛋白质编码区域有影响、是否为突变位点等。
三、应用和展望蜜蜂基因组组装和SNP位点检测技术对于我们了解蜜蜂适应性进化、抗逆性、基因结构和基因功能都有着重要的意义。
Sequenom_SNP实验过程说明

Sequenom SNP实验过程说明文件目录一、实验基本流程及原理 (1)二、实验过程 (2)1、引物设计 (2)2、DNA提取 (3)3、PCR扩增 (3)4、PCR产物碱性磷酸酶处理 (3)5、单碱基延伸 (4)6、树脂纯化 (5)7、芯片点样 (5)8、质谱检测 (5)三、附录实验所需的试剂、耗材和仪器设备 (5)一、实验基本流程及原理Sequenom MassARRAY®SNP检测过程结合多重PCR技术、MassARRAY iPLEX单碱基延伸技术,和基质辅助激光解吸附电离飞行时间质谱分析质谱技术(matrix-assisted laser desorption/ionization–time of flight,MALDI-TOF)进行分型检测。
将包含SNP位点区域的DNA模板通过PCR技术扩增,再使用特异的延伸引物与PCR产物进行单碱基延伸反应。
由于多态性位点碱基不同,延伸产物不同的末端碱基将导致延伸后的产物分子量的差异,因此由SNP多态性引起的碱基差异通过分子量的差异而体现,通过基质辅助激光解吸附电离飞行时间质谱分析质谱技术,检测延伸产物分子量的大小,应用专用的分析软件,通过判断分子量的差异而进行SNP分型检测。
SNP检测实验基本步骤如下图所示:图1 Sequenom MassARRAY® SNP检测实验基本步骤二、实验过程1、引物设计使用Sequenom公司Genotyping Tools 及MassARRAY Assay Design软件设计待测SNP位点的PCR扩增引物及单碱基延伸引物,并交由生物公司合成。
2、DNA提取使用Wizard Genomic DNA purification Kit (Promega)或NucleoSpin Tissue (MN)等试剂盒或类似产品,提取组织、细胞或血样中的DNA。
用分光光度计定量,琼脂糖凝胶电泳质检,基因组DNA电泳条带通常不小于20kb。
sequenom-iPLEX实验操作

iPLEX实验操作一、引物设计用mysequenom网上工具设计(步骤略)二、扩增引物的稀释和UEP引物质量检测1.合成回来的扩增引物加去离子水,稀释为100uM的储液。
之后等体积混合使得终浓度为0.5uM。
如做14个Plex的反应,共28条扩增引物。
每条引物取1ul混合共28ul,再加入172ul的去离子水,总体积为200ul,即为引物Mix(0.5μM)。
2.合成回来的UEP引物按照“UEP引物稀释表”中的黄色高亮部分输入:ID号,Assay的命名,UEP引物的Mass,合成的管中的总OD量,总共有n 个Plex信息。
根据表格计算出每个管中需要加的去离子水量,进行引物重溶。
之后每个管中取等体积引物充分混合即为UEP Mix。
3.将前述UEP Mix 2ul加入40ul去离子水中混合。
加入384孔板中,可用于直接点芯片,打质谱。
这一步检测UEP的质量是必须的,如果质量不好必须重新设计。
三、DNA样品准备基因组DNA样品应电泳检测完整性,并测浓度。
用去离子水或Tris-Cl稀释为浓度10-20ng/ul。
由于样品量大,必须做好样品标记。
四、PCR反应1.在384孔板上一定要做好标记:横6,纵4的24个孔为一组。
以一组为将来点样时候的一针。
2.按多出来10-15%的量来配制Mix,如需检测40个样,则配50-55个样的量反应体系:均按ul计算模板DNA(10ng/ul) 1引物Mix(0.5μM)0.1µM 110*Buffer(含Mg2+)2mM MgCl2 0.5MgCl2(25mM) 2mM 0.4dNTP(25mM) 500µM 0.1Hotstar(5U/μl) 1 Unit 0.1*水 1.9总量5ul*如果做27个Plex以上的PCR,应用0.2 ul3.在384孔板中每孔加入4ul Mix,最后在加入1ul模板DNA。
4.用ABI的PCR封口膜封紧,防止样品蒸发。
Senquenom_SNP引物设计
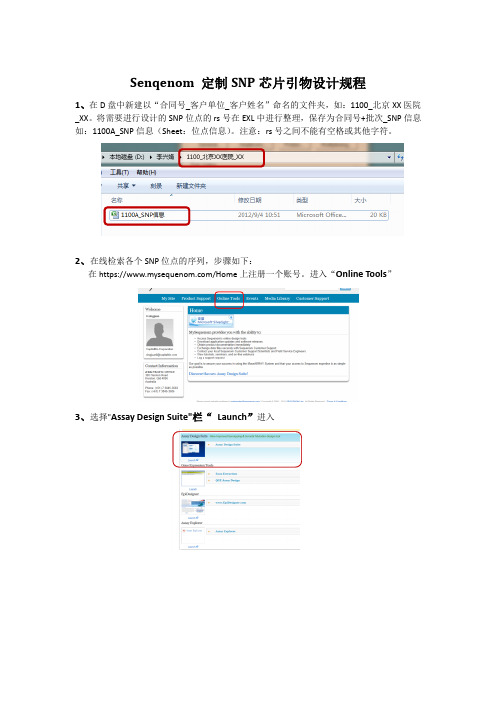
Senqenom 定制SNP芯片引物设计规程1、在D盘中新建以“合同号_客户单位_客户姓名”命名的文件夹,如:1100_北京XX医院_XX。
将需要进行设计的SNP位点的rs号在EXL中进行整理,保存为合同号+批次_SNP信息如:1100A_SNP信息(Sheet:位点信息)。
注意:rs号之间不能有空格或其他字符。
2、在线检索各个SNP位点的序列,步骤如下:在https:///Home上注册一个账号。
进入“Online Tools”3、选择"Assay Design Suite"栏“Launch”进入4、出现“Genotyping Design ”设计界面,如下图Design Name:合同号+批次或客户姓名拼音Vesion:设计版本(1,2,3...)rs or FASTA:在此输入SNP位点的rs号。
Orgnism:物种Database:数据库版本Chemistry :iPLEXMultiplex Level: 36 (每个WELL反应重数)5、点击“Edite Text Input”,将整理好的rs号粘贴至对话框中,点击“Save”,如图:6、根据检测需求单中样品的信息,更改“物种,数据库版本,反应重数”7、以上参数修改完成后,在第“4 Design Assays”项中右击选择“Run up to the previous step”进行前3项的在线运行。
运行结束后如下图:8、点击“Export All”出现如下窗口,选择“Export All Results”,保存在步骤1中建立的该项目文件夹中。
命名格式为:合同号+批次_设计版本(V1/V2/V3)。
9、右击该压缩文件,解压缩后如图,含有三个文件夹:Pretend,Proxsnp,Sequence Retrieval。
10、"Pretend"文件下有2个“1100A_V1”文本,一个为本次在线检索的信息,另一个“1100A_V1”文本为本次检索的“SNP_ID Sequence”信息,如图。
基因组snp遗传多样性分析流程

基因组snp遗传多样性分析流程基因组SNP遗传多样性分析流程1. 样本准备和DNA提取- 收集研究对象的样本,如植物、动物或人类样本- 从样本中提取高质量、高纯度的DNA2. 基因组测序- 利用高通量测序技术(如Illumina测序或纳米孔测序)对DNA样本进行全基因组测序- 获得大量原始测序数据3. 数据质控和过滤- 对原始测序数据进行质量评估和过滤- 去除低质量reads和接头序列等- 得到高质量的clean reads4. 比对参考基因组- 将clean reads比对到参考基因组序列上- 使用生物信息学工具(如BWA或Bowtie2)进行比对5. 变异检测- 基于比对结果,使用变异检测软件(如GATK或Samtools)检测SNP 和InDel等变异位点- 生成变异位点文件(VCF格式)6. 变异过滤- 根据变异质量值、缺失率、深度等参数对变异位点进行过滤- 去除低质量或可疑的变异位点7. 群体结构分析- 利用过滤后的SNP数据,分析种群或群体的遗传结构- 使用软件如STRUCTURE、ADMIXTURE或PCA等进行群体分层和聚类分析8. 遗传多样性分析- 计算各群体或种群的遗传多样性指数,如等位基因多样性、杂合度等- 评估不同群体间的遗传分化程度9. 选择压力分析- 基于SNP数据,检测是否存在遗传hitchhiking或选择性扫除的信号- 识别可能受到正向或负向选择作用的基因或基因组区域10. 关联分析- 对表型数据(如性状或疾病状态)与SNP数据进行关联分析- 鉴定与目标性状或疾病相关的基因或SNP位点11. 结果可视化和解释- 使用统计图表和绘图工具对分析结果进行可视化展示- 综合解释遗传多样性、群体结构、选择压力和关联分析结果12. 报告撰写- 总结分析过程和主要发现- 撰写科学论文或报告,描述研究目的、方法、结果和讨论该流程适用于利用SNP数据分析物种或群体的遗传多样性、群体结构、选择压力和基因型-表型关联等,是基因组学研究的重要环节。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
Sequenom SNP实验过程说明文件
目录
一、实验基本流程及原理 (1)
二、实验过程 (2)
1、引物设计 (2)
2、DNA提取 (3)
3、PCR扩增 (3)
4、PCR产物碱性磷酸酶处理 (3)
5、单碱基延伸 (4)
6、树脂纯化 (5)
7、芯片点样 (5)
8、质谱检测 (5)
三、附录实验所需的试剂、耗材和仪器设备 (5)
一、实验基本流程及原理
Sequenom MassARRAY®SNP检测过程结合多重PCR技术、MassARRAY iPLEX单碱基延伸技术,和基质辅助激光解吸附电离飞行时间质谱分析质谱技术(matrix-assisted laser desorption/ionization–time of flight,MALDI-TOF)进行分型检测。
将包含SNP位点区域的DNA模板通过PCR技术扩增,再使用特异的延伸引物与PCR产物进行单碱基延伸反应。
由于多态性位点碱基不同,延伸产物不同的末端碱基将导致延伸后的产物分子量的差异,因此由SNP多态性引起的碱基差异通过分子量的差异而体现,通过基质辅助激光解吸附电离飞行时间质谱分析质谱技术,检测延伸产物分子量的大小,应用专用的分析软件,通过判断分子量的差异而进行SNP分型检测。
SNP检测实验基本步骤如下图所示:
图1 Sequenom MassARRAY® SNP检测实验基本步骤
二、实验过程
1、引物设计
使用Sequenom公司Genotyping Tools 及MassARRAY Assay Design软件设计待测SNP位点的PCR扩增引物及单碱基延伸引物,并交由生物公司合成。
2、DNA提取
使用Wizard Genomic DNA purification Kit (Promega)或NucleoSpin Tissue (MN)等试剂盒或类似产品,提取组织、细胞或血样中的DNA。
用分光光度计定量,琼脂糖凝胶电泳质检,基因组DNA电泳条带通常不小于20kb。
质检合格的DNA将浓度调整到50ng/µl,转移至384孔板,-20℃储存备用。
℃
4、 PCR产物碱性磷酸酶处理
(1)在PCR反应结束后,将PCR产物用SAP(shrimp alkaline phosphatase,虾碱性磷酸酶)处理,以去除体系中游离的dNTPs。
(2)配制碱性磷酸酶处理反应液,SAP Mix。
(3)使用24通道加样器,调节加样体积为2µL,将SAP Mix加入384孔PCR反应板。
(4)将384孔板放置在兼容384孔板的PCR仪上,设定PCR反应条件:
I. 94º C for 30 seconds
II. 94º C for 5 seconds
III. 52º C for 5 seconds
IV. 80º C for 5 seconds
V. GOTO III, 4 more times
VI. GOTO II, 39 more times
VII. 72º C for 3 minutes
VII. 4º C forever
启动PCR仪进行单碱基延伸反应。
6、树脂纯化
(1)将Clean Resin树脂平铺到6mg的树脂板中;
(2)加16μl水到延伸产物的对应孔内;
(3)将干燥后的树脂倒入延伸产物板中,封膜,低速垂直旋转30分钟,使树脂与反应物充分接触;
(4)离心使树脂沉入孔底部。
7、芯片点样
启动MassARRAY Nanodispenser RS1000点样仪,将树脂纯化后的延伸产物移至384孔SpectroCHIP (Sequenom)芯片上。
8、质谱检测
将点样后的SpectroCHIP芯片使用MALDI-TOF(matrix-assisted laser desorption / ionization–time of fligh,基质辅助激光解吸附电离飞行时间质谱)分析,检测结果使用TYPER 4.0软件(sequenom)分型并输出结果。
三、附录实验所需的试剂、耗材和仪器设备
关键试剂
Code Reagents Manufacturer Cat No.
1 Complete iPLEX® Gold
Genotyping Reagent Set 384
Sequenom
10148-2
关键仪器
Code Instruments Manufacturer
1 S1000TM Thermal Cycler BIORAD
2 Pipette single channel Eppendorf
3 Electronic pipette 2
4 channel Rainin
4 MassARRAY Nanodispenser SEQUENOM
5 MassARRAY Compact System SEQUENOM。
