最新pymol作图的一个实例
pymol做图简单教程
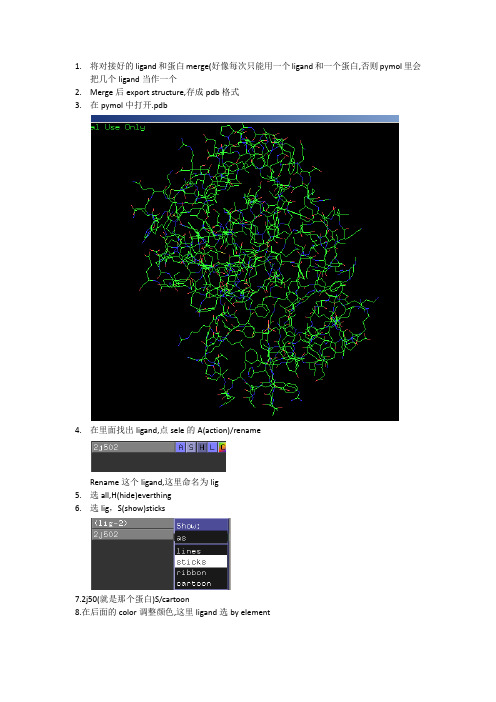
1.将对接好的ligand和蛋白merge(好像每次只能用一个ligand和一个蛋白,否则pymol里会把几个ligand当作一个2.Merge后export structure,存成pdb格式3.在pymol中打开.pdb4.在里面找出ligand,点sele的A(action)/renameRename这个ligand,这里命名为lig5.选all,H(hide)everthing6.选lig,S(show)sticks7.2j50(就是那个蛋白)S/cartoon8.在后面的color调整颜色,这里ligand选by element2j50选white9.下面显示氢键,点击2j50的Action,选择find/polar contacks/to other atoms in object,氢键就出来了10.然后再找氢键作用的残基,可以在上面直接点选,但最好找出氨基酸代码来选取(因为这里面点不准)Rename它们,以便以后好找,这里rename为res,show它们为line,color为by element11.再选display,将background改成white12.然后分别点选这几个res(因为颜色不同,所以很容易找出来),右键label/residuesbel出来后,还可以调整它们的位置,点一下左图viewing,就变成了右图的editing,这时就可以按住ctrl键拖动label的位置了12.大体已经完成了,再进行一些修饰Setting/transparency/cartoon/50% 蛋白的透明度Setting/label/size 18调节字体的大小Setting/edit allcartoon的颜色改回白色(在filter里输cartoon可以快点找出来,改完后按回车) //或者输入命令Pymol>set cartoon_color white在改氢键的线型,在dash gap length width进行调整把虚线改好看点Stick也可以改细点13.电子密度1)首先,登陆http://eds.bmc.uu.se/eds/搜索你的蛋白2)download maps3)选ccp4格式,generate map4)下载后解压,重命名为*p45)在pymol里打开,在打开的.map,action/mesh,color/blue6)选中lig,在PyMOL输入isomesh map,2j50.map,2.0,sele,carve=1.67)这样就从原来的map里iso出lig周围的蛋白的电子密度,把2j50.map_mesh关了就可以看见了然后再调整它的粗细电子密度周围的线好像有些奇怪,可能设置里哪个地方还没调好吧.另外,ray 1900,980就能生成1900*980分辨率的图片,然后file/save image as输出.png。
摩尔条纹 python

摩尔条纹 python摩尔条纹(Moiré Pattern)是一种常见的视觉现象,它产生的原因是由于两个或多个具有规则或周期性结构的图案叠加在一起,形成了新的图案。
Python作为一种强大的编程语言,在图像处理和模式生成方面有着广泛的应用。
本文将探讨如何使用Python生成摩尔条纹,并介绍一些相关的技术和应用。
一、摩尔条纹的生成原理摩尔条纹的生成原理可以通过空间频率重叠的原理来解释。
当两个图案具有相似的空间频率时,它们在叠加时会产生干涉,从而形成摩尔条纹。
比如,当两个正方形网格相互叠加时,就会产生明暗相间的条纹。
在Python中,可以通过使用Matplotlib库和NumPy库来生成摩尔条纹。
Matplotlib库提供了丰富的绘图功能,而NumPy库用于进行数组运算和数值计算。
二、生成简单的摩尔条纹下面我们将使用Python生成一个简单的摩尔条纹示例:```pythonimport numpy as npimport matplotlib.pyplot as plt# 定义两个正方形网格的空间频率freq1 = 10freq2 = 12# 生成x、y坐标网格x, y = np.meshgrid(np.linspace(-1, 1, 500), np.linspace(-1, 1, 500))# 计算两个正方形网格的干涉图案pattern = np.sin(2*np.pi*freq1*x) + np.sin(2*np.pi*freq2*y)# 绘制摩尔条纹图像plt.imshow(pattern, cmap='gray')plt.axis('off')plt.show()```以上代码首先定义了两个正方形网格的空间频率,然后生成了x、y 坐标的网格。
接着,通过计算两个正方形网格的干涉图案,生成了摩尔条纹的图像。
最后,使用Matplotlib库中的imshow函数绘制了摩尔条纹的图像。
pymol教程

pymol教程
Pymol是一种用于分子可视化的软件工具,它可以让科学家和
研究人员更好地理解和展示分子结构和相互作用。
本教程将引导您使用Pymol进行一些基本的分子可视化操作。
1. 安装Pymol
在开始之前,您需要在计算机上安装Pymol软件。
您可以从
官方网站上下载适用于您的操作系统的最新版本。
安装完成后,启动Pymol。
2. 加载分子数据
在Pymol中,您可以加载各种不同格式的分子数据文件,包
括PDB、MOL2、XYZ等。
您可以从菜单栏中选择“File”-
>“Open”来加载一个分子文件。
3. 调整分子的显示样式
一旦分子文件加载完成,您可以使用Pymol的各种工具来调
整分子的显示样式。
例如,您可以选择隐藏或显示原子、调整原子的颜色和大小等。
4. 旋转和平移分子
Pymol允许您通过鼠标来旋转和平移分子。
按住鼠标左键并
拖动可以旋转分子的视角,按住鼠标右键并拖动可以平移分子的位置。
5. 添加和调整分子的标签
Pymol还支持添加和调整分子上的标签。
您可以选择一个原
子或残基,并在菜单栏中选择“Label”->“Atom”或“Residue”,然后在弹出的对话框中输入您想要显示的标签。
6. 创建和编辑分子图形
Pymol还提供了一些工具来创建和编辑分子图形。
您可以使用线条、球棒、面片等工具来可视化分子的结构和相互作用。
以上是使用Pymol进行基本分子可视化的简要教程。
希望这对您有所帮助!。
pymol使用教程

pymol使用教程PyMOL是一种图形化的分子可视化工具,它可以用于展示和分析蛋白质、DNA、RNA等分子结构。
下面是一份PyMOL使用教程,帮助你了解如何使用PyMOL进行分子可视化。
1.安装PyMOL2.打开和导入结构在PyMOL中,你可以通过点击菜单中的“File”->“Open”来打开一个已有的结构文件。
PyMOL支持多种结构文件格式,包括PDB、MOL2、SDF 等。
你也可以通过在命令行中输入“load 文件路径”来导入结构文件。
3.调整视图PyMOL提供了多种视图设置选项,可以通过鼠标右键点击图像来进行操作。
你可以旋转、平移和缩放结构,以便更好地观察分子。
此外,你还可以通过鼠标滚轮或点击菜单中的“View”->“Zoom”来放大或缩小结构。
4.显示分子PyMOL允许你显示分子的不同组成部分,例如原子、氢键、键、半径等。
你可以通过点击菜单中的“Display”进行选择。
你还可以通过命令行输入“show 选项”来显示特定的分子部分,例如“show sticks”用于显示分子键。
5.颜色设置PyMOL允许你为分子的不同组成部分设置不同的颜色。
你可以通过点击菜单中的“Color”进行设置。
例如,你可以将蛋白质的α-螺旋设置为红色。
“color 红色, 选择项”命令可以实现相同的效果。
6.制作表面PyMOL还支持制作分子表面,以更好地展示分子的形状和结构。
你可以点击菜单中的“Action”->“Surface”来创建分子表面。
你还可以通过命令行输入“show surface”来实现相同的效果。
7.保存和导出在PyMOL中,你可以点击菜单中的“File”->“Save Image”将当前视图保存为图片文件。
此外,你还可以使用命令行输入“pn g 文件路径”将图片保存为指定的路径。
PyMOL还支持导出结构文件,你可以点击菜单中的“File”->“Export”来导出结构文件。
pymol作图的一个实例演示教学

p y m o l作图的一个实例Pymol作图的实例这是一个只是用鼠标操作的初步教程Pdb文件3ODU.pdb打开文件pymol右侧All指所有的对象,2ODU指刚才打开的文件,(sele)是选择的对象按钮A:代表对这个对象的各种action,S:显示这个对象的某种样式,H:隐藏某种样式,L:显示某种label,C:显示的颜色下面是操作过程:点击all中的H,选择everything,隐藏所有点击3ODU中的S,选择cartoon,以cartoon形式显示蛋白质点击3ODU中的C,选择by ss,以二级结构分配颜色,选择点击右下角的S,窗口上面出现蛋白质氨基酸序列,找到1164位ITD,是配体点击选择ITD ,此时sele中就包含ITD这个残基,点击(sele)行的A,选择rename selection,窗口中出现,更改sele为IDT,点击(IDT)行的S选择sticks,点击C,选择by element,选择,,调整窗口使此分子清楚显示。
寻找IDT与蛋白质相互作用的氢键:IDT行点击A 选择find,选择polar contacts,再根据需要选择,这里选择to other atoms in object ,分子显示窗口中出现几个黄色的虚线,IDT行下面出现了新的一行,这就是氢键的对象,点击这一行的C,选择red red,把氢键显示为红色。
接着再显示跟IDT形成氢键的残基点击3ODU行的S,选择lines,显示出所有残基的侧链,使用鼠标转动蛋白质寻找与IDT以红色虚线相连的残基,分别点击选择这些残基。
注意此时selecting要是residures。
选择的时候要细心。
取消选择可以再次点击已选择的残基。
使用上述的方法把选择的残基(sele)改名为s1。
点击S1行的S选择sticks,C选择by elements,点击L选择residures显示出残基名称.在这个例子中发现其中有一个N含有3个氢键有两个可以找到与其连接的氨基酸残基,另一个找不到,这是因为这个氢键可能是与水分子形成的,水分子在pdb文件中只用一个O表示,sticks显示方式没有显示出来水分子,点击all行S选择nonbonded,此时就看到一个水与N形成氢键,点击分子空白处,然后点击选择这个水分子,更改它的名字为w。
非常好的pymol教程

非常好的pymol教程Pymol是一款强大的分子可视化工具,它能够帮助生物学家、化学家和结构生物学家分析和可视化蛋白质、小分子和复杂化合物的结构。
本教程将向你展示Pymol的基本功能和操作步骤,以便你能够更好地理解和使用这个工具。
1. 安装Pymol:2.打开和导入分子结构:打开Pymol软件后,你将看到一个主界面。
你可以通过点击菜单栏上的“File”选项或使用快捷键Ctrl+O来打开分子结构文件。
Pymol支持多种主流的分子结构文件格式,如PDB、CIF和XYZ等。
选择你想要导入的分子结构文件,然后点击“Open”按钮。
3.调整视图和颜色:一旦成功导入分子结构,你可以通过使用鼠标右键和滚动调整视图。
拖动鼠标右键可以旋转分子,滚动滚轮可以缩放分子。
此外,你还可以在菜单栏的“Display”选项下调整分子的颜色、图形和材质。
4.选择和操作分子的部分结构:要选择和操作分子的部分结构,你可以使用菜单栏上的“Select”选项或使用快捷键Ctrl+Shift+A。
在打开的“Selection”对话框中,你可以输入选择命令来选择具体的分子结构,比如选择指定的氨基酸残基或原子。
选择完成后,你可以对所选结构进行操作,如旋转、平移、放大等。
5.显示分子间的非共价相互作用:Pymol能够计算并显示分子间的非共价相互作用,比如氢键、离子键和范德华力等。
你可以在菜单栏的“Display”选项下选择“Nonbonded”来显示非共价相互作用。
这将帮助你更好地理解和分析分子的结构和性质。
7.绘制分子的电子密度图:Pymol允许你绘制分子的电子密度图,以更好地理解其结构和性质。
你可以在菜单栏的“Display”选项下选择“Map”来打开“Maps”功能。
然后选择你想要的地图类型和参数,点击“Apply”按钮来生成电子密度图。
你可以使用鼠标右键和滚动调整绘制的电子密度图。
通过以上步骤,你已经了解了Pymol的基本功能和操作。
绘制洛伦兹曲线实例

绘制洛伦兹曲线实例洛伦兹曲线是描述物理学中的一种曲线,用于描述流体或气体的速度分布。
这里我们将使用Python的matplotlib库来绘制洛伦兹曲线的实例。
首先,我们需要导入matplotlib库和numpy库,用于绘制曲线和进行数学计算。
请确保你已经安装了这些库。
```pythonimport matplotlib.pyplot as pltimport numpy as np```接下来,我们定义洛伦兹方程的微分方程组,即速度变化的微分方程。
洛伦兹方程包括三个变量:x、y和z,以及三个参数:σ、ρ和β。
这些参数用于控制曲线的形状。
```pythondef lorenz_equations(state, t, sigma, rho, beta):x, y, z = statedxdt = sigma * (y - x)dydt = x * (rho - z) - ydzdt = x * y - beta * zreturn [dxdt, dydt, dzdt]```然后,我们定义绘制洛伦兹曲线的函数。
该函数使用scipy库的odeint函数来求解微分方程,并用matplotlib库绘制曲线。
```pythondef plot_lorenz_curve(sigma, rho, beta):t = np.arange(0, 30, 0.01)initial_state = [1, 1, 1]states = odeint(lorenz_equations, initial_state, t, args=(sigma, rho, beta))x = states[:, 0]y = states[:, 1]z = states[:, 2]fig = plt.figure()ax = fig.gca(projection='3d')ax.plot(x, y, z)ax.set_xlabel('X')ax.set_ylabel('Y')ax.set_zlabel('Z')plt.show()```最后,我们调用plot_lorenz_curve函数,并传入sigma、rho和beta的值来绘制洛伦兹曲线。
pymol做图简单教程

pymol做图简单教程1.将对接好的ligand和蛋白merge(好像每次只能用一个ligand和一个蛋白,否则pymol里会把几个ligand当作一个2.M erge后export structure,存成pdb格式3.在pymol中打开.pdb4.在里面找出ligand,点sele的A(action)/renameRename这个ligand,这里命名为lig5.选all,H(hide)everthing6.选lig,S(show)sticks7.2j50(就是那个蛋白)S/cartoon8.在后面的color调整颜色,这里ligand选by element2j50选white9.下面显示氢键,点击2j50的Action,选择find/polar contacks/to other atoms in object,氢键就出来了bel出来后,还可以调整它们的位置,点一下左图viewing,就变成了右图的editing,这时就可以按住ctrl键拖动label的位置了12.大体已经完成了,再进行一些修饰Setting/transparency/cartoon/50% 蛋白的透明度Setting/label/size 18调节字体的大小Setting/edit allcartoon的颜色改回白色(在filter里输cartoon可以快点找出来,改完后按回车)//或者输入命令Pymol>set cartoon_color white在改氢键的线型,在dash gap length width进行调整把虚线改好看点Stick也可以改细点13.电子密度1)首先,登陆http://eds.bmc.uu.se/eds/搜索你的蛋白2)d ownload maps3)选ccp4格式,generate map4)下载后解压,重命名为*p45)在pymol里打开,在打开的.map,action/mesh,color/blue6)选中lig,在PyMOL输入isomesh map,2j50.map,2.0,sele,carve=1.67)这样就从原来的map里iso出lig周围的蛋白的电子密度,把2j50.map_mesh关了就可以看见了然后再调整它的粗细电子密度周围的线好像有些奇怪,可能设置里哪个地方还没调好吧.另外,ray 1900,980就能生成1900*980分辨率的图片,然后file/save image as输出.png。
pymol作图的一个实例

Pymol作图的实例这是一个只是用鼠标操作的初步教程Pdb文件3ODU.pdb打开文件pymol右侧All指所有的对象,2ODU指刚才打开的文件,(sele)是选择的对象按钮A:代表对这个对象的各种action,S:显示这个对象的某种样式,H:隐藏某种样式,L:显示某种label,C:显示的颜色下面是操作过程:点击all中的H,选择everything,隐藏所有点击3ODU中的S,选择cartoon,以cartoon形式显示蛋白质点击3ODU中的C,选择by ss,以二级结构分配颜色,选择点击右下角的S,窗口上面出现蛋白质氨基酸序列,找到1164位ITD,是配体点击选择ITD ,此时sele中就包含ITD这个残基,点击(sele)行的A,选择rename selection,窗口中出现,更改sele为IDT,点击(IDT)行的S选择sticks,点击C,选择by element,选择,,调整窗口使此分子清楚显示。
寻找IDT与蛋白质相互作用的氢键:IDT行点击A 选择find,选择polar contacts,再根据需要选择,这里选择to other atoms inobject ,分子显示窗口中出现几个黄色的虚线,IDT行下面出现了新的一行,这就是氢键的对象,点击这一行的C,选择red red,把氢键显示为红色。
接着再显示跟IDT形成氢键的残基点击3ODU行的S,选择lines,显示出所有残基的侧链,使用鼠标转动蛋白质寻找与IDT 以红色虚线相连的残基,分别点击选择这些残基。
注意此时selecting要是residures。
选择的时候要细心。
取消选择可以再次点击已选择的残基。
使用上述的方法把选择的残基(sele)改名为s1。
点击S1行的S选择sticks,C选择by elements,点击L选择residures显示出残基名称.在这个例子中发现其中有一个N含有3个氢键有两个可以找到与其连接的氨基酸残基,另一个找不到,这是因为这个氢键可能是与水分子形成的,水分子在pdb文件中只用一个O表示,sticks显示方式没有显示出来水分子,点击all行S选择nonbonded,此时就看到一个水与N形成氢键,点击分子空白处,然后点击选择这个水分子,更改它的名字为w。
PyMOL概述及实例简析

http://eds.bmc.uu.se/eds/
13
三.应用实例-黄素氧还蛋白
黄素氧还蛋白
– 细菌中的常见蛋白,通过结合 FMN 参与电子传递。
异咯嗪
核糖醇 磷酸
黄素单核苷酸(FMN)
14
Sancho J. (2006) Cell Mol Life Sci
黄素氧还蛋白家族分类
长链蛋白 短链蛋白
更改透明度
PyMOL>set transparency=0.25
26
->action->preset->ligand sites->cartoon
27
实例3.晶体对称性
PyMOL>symexp sym, 1AHN,1AHN,5 #创建对称相关的对象
PyMOL>hide(not(1AHN expand 5)) #隐藏距离1AHN大于5Å
16
大肠杆菌中的黄素氧还蛋白
90‗s 50‘s
FldA FldB MioC YqcA
长链蛋白 短链蛋白
FldA: flavodoxin A FldB: flavodoxin B MioC: locates next to the chromosomal replication initiation origin (oriC) YqcA:??
载入结构 上色
PyMOL> color purple, ss h PyMOL> color yellow, ss s PyMOL> color green, ss ―‖
静电力学
->Action ->generate ->vaccum electrostatics
[转载]PyMOL用法(教程四)
![[转载]PyMOL用法(教程四)](https://img.taocdn.com/s3/m/0369c47049d7c1c708a1284ac850ad02de800772.png)
[转载]PyMOL⽤法(教程四)原⽂地址:PyMOL⽤法(教程四)作者:娴⽽静学关于label在显⽰⼀个蛋⽩结构的某个细节的时候,常常会需要给某些重要的氨基酸打上标签,这就需要⽤到label命令。
label的命令格式如下:Pymol> label [selection, [expression]]selection当然就是你要加标签的对象,expression就是标签的内容,可选的有:name, resn, resi, chain等等。
你也可以组合使⽤它们。
expression也可以是你⾃定义的⼀段内容,这时候只要把内容⽤引号包含起来就⾏:Pymol> label selection, "user-defined expression"在下⾯这个例⼦中,我想把glucosyltransferase中UDP-Glucose的binding pocket标注出来:⾸先说明⼀下,该pdb⽂件中A链是蛋⽩质,B链是UDP-Glucose。
Pymol> load glucosyltransferase.pdb, tmpPymol> extract upg, chain bPymol> extract pro, chain aPymol> delete tmpPymol> select near, pro within 4.5 of upgPymol> hide allPymol> show sticks, upgPymol> show lines, nearPymol> label near, ("%s/%s") % (resn, resi)#("%s/%s"): 设定显⽰格式。
上⾯的图看起来有点乱,因为默认Pymol在每个原⼦上都打上了标签。
要想看起来顺眼点,需要⼀点加⼯。
在这之前,让我们先看⼀下关于label的其他设置:投影模式投影模式,可选值0(⽆投影),1(object有投影到label上,但是label本⾝⽆投影),2(object有投影到label上,label也有投影),3(object不投影到label上,label本⾝有投影):Pymol> set label_shadow_mode, 3⽂字颜⾊⽂字颜⾊:Pymol> set label_color, color-name, selection标签⽂字的轮廓的颜⾊,这样就让在例如⽩⾊背景上加⽩⾊标签成为了可能:标签⽂字的轮廓的颜⾊Pymol> set label_outline_color, [color-name, [selection]]字体,pymol内置了12种字体,编号从5-16。
PyMOL最新版本教程:手把手教你作出大神级蛋白结构图(终结篇)

PyMOL最新版本教程:手把手教你作出大神级蛋白结构图(终结篇)展开全文大家好,经过前面两节课的学习,我们初步了解了PyMOL的基本鼠标操作以及如何用PyMOL来观察受体配体相互作用的方法。
但是,PyMOL的操作可不止这一点点,今天,小编就教大家一些更加酷炫的使用方法。
TEXT和Linux一样,想要更好地使用PyMOL,就不能浪费了PyMOL所支持的命令行输入模式,我们可以向external GUI窗口中键入命令行,命令行输入可以代替所有的鼠标操作,如果练习运用的好的话,操作会更加精准,速度也会更快。
下图就是external GUI的界面。
☛☛☛视频连接:https:///x/page/n0537l6gm00.htmlPyMOL命令的语法首先我们需要知道的是虽然PyMOL命令是区分大小写的,但是目前PyMOL命令都还是只用小写,所以以下我们看到的所有命令都是用小写字母的。
那么命令行的格式是怎样的呢?PyMOL命令一般都是由keyword(关键词) 和argument(变量)组成,具体如下:PyMOL> keyword argument但并不是所以命令都需要变量,比如退出命令,我们就可以直接输入PyMOL>quit,然而需要变量的命令依旧占据了大多数,可以分为单变量命令和多变量命令,单变量命令指的是对于某些命令,我们只可以输入一个变量,比如放大命令zoom,但是同时你会发现不加任何变量这个命令也可以被执行,这是因为PyMOL对单变量命令有一个默认的变量,对于放大命令zoom来说,all就是它的默认变量,即PyMOL>zoom等同于PyMOL>zoom all多变量命令可以带多个变量,比如说假如我想将对象的的颜色变为红色,我们可以使用PyMOL>color red这个命令,但是操作后我们会发现这样的命令没有办法指定某一部分变色,于是我们可以选中我们需要的部分,采用以下的命令PyMOL>color green,sele这里的sele就是除了颜色以外的第二个变量。
pymol用法(教程二)

[转载]PyMOL用法(教程二)基础Pymol命令这里主要介绍一下Pymol的一些基本命令操作。
就像Linux 一样,要想更好的操作Pymol,掌握一些常用的命令是必不可少的。
Pymol是区分大小写的,不过目前为止Pymol还是只用小写,所以记住,所有的命令都是使用小写字母的。
当你开始用Pymol来完成一个项目时,你也许想会让Pymol自动保存你所有输入过的命令,以方便日后你再次读取并修改。
这个可以通过创建一个log文件来达到,该文件的后缀名应为.pml,记住,Pymol像Linux一样支持Tab键命令补全:Pymol> log_open log-file-name.pml如果你想终止记录,只需要键入:Pymol> log_close好了,现在载入pdb文件(继续前用的pdb文件):Pymol> load 2vlo.pdb现在Pymol就创建了一个叫2vlo的对象,你可以在内部GUI窗口里面看见这个项目的名字。
但是你也可以自己定义该项目的名字(如test):Pymol> load 2vlo.pdb, test下面说说如何来操作你新建的对象。
首先:Pymol> show representationPymol> hide representation其中representation可以为:cartoon, ribbon,dots, spheres, surface和mesh。
使用这2个命令可以让Pymol以不同的方式显示蛋白质结构。
例如当我们键入:Pymol> hide linesPymol> show ribbon我们将得到如下结果:也许你已经注意到结构中有2个一模一样的蛋白质分子,只是方向不同而已,那么如何让Pymol只显示当中的一个分子呢?首先输入如下命令:Pymol> label all, chains这个命令的作用是让Pymol给蛋白质结构中的“链”编号,你会发现,第一个分子由“链”A-E组成,第二个则由F-J组成。
使用python的pyplot绘制函数实例
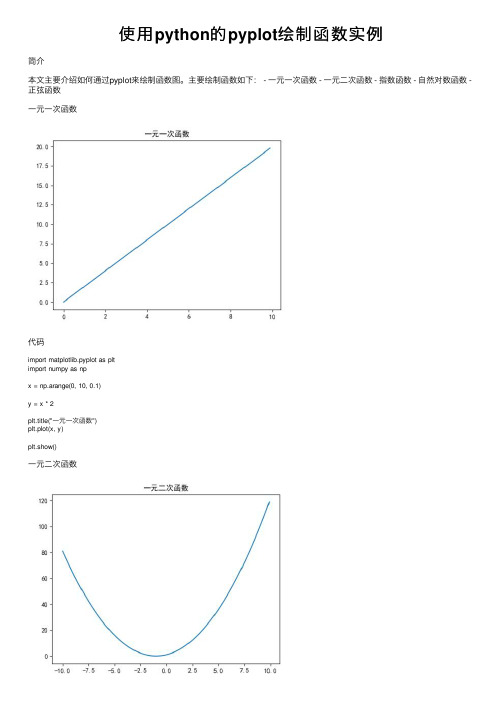
使⽤python的pyplot绘制函数实例简介本⽂主要介绍如何通过pyplot来绘制函数图。
主要绘制函数如下: - ⼀元⼀次函数 - ⼀元⼆次函数 - 指数函数 - ⾃然对数函数 -正弦函数⼀元⼀次函数代码import matplotlib.pyplot as pltimport numpy as npx = np.arange(0, 10, 0.1)y = x * 2plt.title("⼀元⼀次函数")plt.plot(x, y)plt.show()⼀元⼆次函数代码import matplotlib.pyplot as plt import numpy as npx = np.arange(-10, 10, 0.1)y = x**2 + 2*x + 1plt.title("⼀元⼆次函数") plt.plot(x, y)plt.show()指数函数代码import matplotlib.pyplot as plt import numpy as npimport mathx = np.arange(0, 10, 0.1)y = 2**xplt.title("指数函数")plt.plot(x, y)plt.show()⾃然对数函数代码import matplotlib.pyplot as pltimport numpy as npimport mathx = np.arange(0, 10, 0.1)e = math.ey = e**xplt.title("⾃然对数函数")plt.plot(x, y)plt.show()正弦函数代码import matplotlib.pyplot as pltimport numpy as np# linspace 第⼀个参数序列起始值, 第⼆个参数序列结束值,第三个参数为样本数默认50 x = np.linspace(-np.pi, np.pi, 100)y = np.sin(x)plt.title("正弦函数")plt.plot(x, y)plt.show()源码以上这篇使⽤python的pyplot绘制函数实例就是⼩编分享给⼤家的全部内容了,希望能给⼤家⼀个参考,也希望⼤家多多⽀持。
pymol蛋白相作结果surface

pymol蛋白相作结果surface
下面是使用PyMol渲染蛋白质结构的示例:
●from pymol import cmd
●读取蛋白质结构
●cmd.load(1bcy.pdb)
●创建表面
●cmd.surface(protein,protein)
●设置表面颜色
●cmd.color(gray,protein)
●显示表面
●cmd.show(surface,protein)
这段代码将读取名为1bcy.pdb的蛋白质结构。
然后,它将创建一个表面,并将其命名为protein。
最后,它将设置表面的颜色为灰色,并显示表面。
渲染的表面如下所示:
可以使用PyMol提供的各种工具来调整表面的颜色、透明度和其他属性。
例如,可以使用以下命令来更改表面的颜色:cmd.color(red,protein)
这将将表面的颜色更改为红色。
还可以使用PyMol来渲染蛋白质结构的其他方面,例如残基、接口和氢键。
例如,以下命令将渲染蛋白质结构的所有残基:cmd.show(sticks,protein)这将将蛋白质结构的所有残基以棍棒状显示。
pymol 单体 多聚物结构

pymol 单体多聚物结构
PyMOL是一个开放源码,由使用者赞助的分子三维结构显示软件。
适用于创作高品质的小分子或是生物大分子(特别是蛋白质)的三维结构图像。
在所有正式发表的科学文献中的蛋白质结构图像中,有四分之一是使用PyMOL来制作。
PyMOL是少数可以用在结构生物学领域的开放源代码视觉化工具。
PyMOL的命令可以用来表示单体和多聚物结构,例如:
1. 显示单体:
```python
select monomer, resn [单体残基名称]
color [颜色名称], resn [单体残基名称]
```
2. 显示多聚物:
```python
select polymer, chain [链名称] and resn [残基名称]
color [颜色名称], chain [链名称] and resn [残基名称]
```
以上信息仅供参考,具体使用PyMOL表示单体和多聚物结构时,建议查阅PyMOL软件的使用手册或在线教程,以获取更详细和准确的信息。
python蛋白质可视化案例

python蛋白质可视化案例
蛋白质可视化是生物信息学中一个非常重要的领域,它可以帮助我们更好地理解蛋白质的结构和功能。
在Python中,有许多库可以帮助我们实现蛋白质的可视化,其中最常用的库是PyMOL和VMD。
下面是一个使用PyMOL进行蛋白质可视化的案例:
1. 首先,你需要安装PyMOL。
你可以在PyMOL的官方网站上下载安装包,或者使用pip命令进行安装:
```shell
pip install pymol
```
2. 打开PyMOL,然后加载你想要可视化的蛋白质。
你可以使用PDB文件
格式来加载蛋白质,这是一种常见的蛋白质数据格式。
你可以从Protein Data Bank网站上下载你想要可视化的蛋白质的PDB文件。
3. 在PyMOL中,你可以使用各种命令来查看蛋白质的结构。
例如,你可以使用`show`命令来显示蛋白质的原子,`select`命令来选择你想要查看的原子,等等。
4. 你可以使用`rotate`和`translate`命令来旋转和移动视图,以便更好地查看蛋白质的结构。
你还可以使用`color`命令来改变蛋白质的颜色,以便更好地区分不同的原子。
5. 最后,你可以使用`render`命令将蛋白质可视化的结果保存为图像文件,以便于分享和展示。
下面是一个简单的PyMOL命令示例,用于加载一个名为""的蛋白质文件,并显示其原子:
```python
load
show atoms
```
这只是一个简单的例子,实际上PyMOL有许多其他功能和命令,可以帮助你更好地理解和可视化蛋白质的结构。
最新pymol作图的一个实例

最新pymol作图的⼀个实例Pymol作图的实例这是⼀个只是⽤⿏标操作的初步教程Pdb⽂件3ODU.pdb打开⽂件pymol右侧All指所有的对象,2ODU指刚才打开的⽂件,(sele)是选择的对象按钮A:代表对这个对象的各种action,S:显⽰这个对象的某种样式,H:隐藏某种样式,L:显⽰某种label,C:显⽰的颜⾊下⾯是操作过程:点击all中的H,选择everything,隐藏所有点击3ODU中的S,选择cartoon,以cartoon形式显⽰蛋⽩质点击3ODU中的C,选择by ss,以⼆级结构分配颜⾊,选择点击右下⾓的S,窗⼝上⾯出现蛋⽩质氨基酸序列,找到1164位ITD,是配体点击选择ITD ,此时sele中就包含ITD这个残基,点击(sele)⾏的A,选择rename selection,窗⼝中出现,更改sele为IDT,点击(IDT)⾏的S选择sticks,点击C,选择by element,选择,,调整窗⼝使此分⼦清楚显⽰。
寻找IDT与蛋⽩质相互作⽤的氢键:IDT⾏点击A 选择find,选择polar contacts,再根据需要选择,这⾥选择to other atoms inobject ,分⼦显⽰窗⼝中出现⼏个黄⾊的虚线,IDT⾏下⾯出现了新的⼀⾏,这就是氢键的对象,点击这⼀⾏的C,选择red red,把氢键显⽰为红⾊。
接着再显⽰跟IDT形成氢键的残基点击3ODU⾏的S,选择lines,显⽰出所有残基的侧链,使⽤⿏标转动蛋⽩质寻找与IDT 以红⾊虚线相连的残基,分别点击选择这些残基。
注意此时selecting要是residures。
选择的时候要细⼼。
取消选择可以再次点击已选择的残基。
使⽤上述的⽅法把选择的残基(sele)改名为s1。
点击S1⾏的S选择sticks,C选择by elements,点击L选择residures显⽰出残基名称.在这个例⼦中发现其中有⼀个N含有3个氢键有两个可以找到与其连接的氨基酸残基,另⼀个找不到,这是因为这个氢键可能是与⽔分⼦形成的,⽔分⼦在pdb⽂件中只⽤⼀个O表⽰,sticks显⽰⽅式没有显⽰出来⽔分⼦,点击all⾏S选择nonbonded,此时就看到⼀个⽔与N形成氢键,点击分⼦空⽩处,然后点击选择这个⽔分⼦,更改它的名字为w。
如何用Pymol做出那些美呆的结构图(基础篇)

如何用Pymol做出那些美呆的结构图(基础篇)摘自微信公众号:BioEngX生化工程实验室小伙伴们在阅读文献的时候是不是经常会看到那种美呆了的蛋白质结构图,比如这样的、这样的、还有这样的......认真的小伙伴在这些论文配图的注解里一定看过这么一句话:“The image was generated in Pymol” (Reference 1)今天,小编就简单的介绍下Pymol以及其基础的用法。
Pymol是一个开放源码,由使用者赞助的分子三维结构显示软件,由Warren Lyford DeLano编写,并且由DeLano Scientific LLC负责商业发行。
DeLanoScientific LLC是一个私人的软件公司,它致力于创造让普遍的科学与教育社群都能取得的好用软件工具。
当然部分软件也是收费的。
Pymol被用来创作高品质的分子(特别是生物大分子如蛋白质)三维结构。
据软件作者宣称,在所有正式发表的科学论文中的蛋白质结构图像中,有四分之一是使用Pymol来制作的。
Pymol名字的来源:“Py”表示该软件基于python这个计算机语言,“Mol”则是英文分子(molucule)的缩写,表示该软件用来显示分子结构。
当你打开Pymol后,你将会看到如下图所示的界面:该界面分为2窗口,上面的外部GUI窗口(External GUI)和下面的Viewer Window。
Viewer Window又分为左右两块,左边用来显示结构图像的(Viewer),右边则是一个内部GUI窗口(Internal GUI)。
Viewer自身包含一个命令行(如图中左下方的PyMOL>提示符),可以用来输入Pymol命令;在Inernal GUI中则可以选定一些特定的对象并完成一些操作。
Internal GUI右上角有五个按键,A,S,H,L,C分别是五个单词的缩写Actions,Show,Hide,Label,Color。
Python实现手绘图效果实例分享

Python实现⼿绘图效果实例分享⾸先我们来看看原图:接着我们来看看效果图:通过分析我们不难发现以下特征:主要颜⾊为⿊⽩灰边界线条较重相同或相近⾊趋于⽩⾊略有光源效果需要⽤到的库有:numpyPIL代码实现:import numpy as npfrom PIL import ImagebaseImg = Image.open("./img/myimg2.jpg").convert("L") # 这⾥放置你要⼿绘的图⽚原图a = np.array(baseImg).astype("float")depth = 8.grad = np.gradient(a) # 取图像灰度的梯度值grad_x, grad_y = grad # 模拟图像梯度值grad_x = grad_x*depth/100.grad_y = grad_y*depth/100.A = np.sqrt(grad_x**2+grad_y**2+1)uni_x = grad_x/Auni_y = grad_y/Auni_z = 1./Avec_el = np.pi/2.2 # 光源俯视⾓度,弧度值vec_az = np.pi/4. # 官员的⽅位⾓度,弧度值dx = np.cos(vec_el)*np.cos(vec_az) # 光源对x轴的影响dy = np.sin(vec_el)*np.sin(vec_az) # 光源对y轴的影响dz = np.sin(vec_el) # 光源对z轴的影响b = 255*(dx*uni_x+dy*uni_y+dz*uni_z) # 光源归⼀化b = b.clip(0,255) # 限制img = Image.fromarray(b.astype("uint8")) # 重构图像img.save("./toImg/myImage1.jpg") # 保存图像实例扩展:from PIL import Imageimport numpy as np#为了便于⽂件的导⼊,可以使⽤相对路径,将⽂件和程序放在同⼀个⽂件夹下vec_el=np.pi/2.2vec_az=np.pi/4.depth=10.im=Image.open("HIT2.jpg").convert('L')a=np.asarray(im).astype('float')grad=np.gradient(a)grad_x,grad_y=gradgrad_x=grad_x*depth/100.grad_y=grad_y*depth/100.dx=np.cos(vec_el)*np.cos(vec_az)dy=np.cos(vec_el)*np.sin(vec_az)dz=np.sin(vec_el)A=np.sqrt(grad_x**2+grad_y**2+1.)uni_x=grad_x/Auni_y=grad_y/Auni_z=1./Aa2=255*(dx*uni_x+dy*uni_y+dz*uni_z)a2=a2.clip(0,255)im2=Image.fromarray(a2.astype('uint8'))im2.save('hit2-SH.jpg')到此这篇关于Python实现⼿绘图效果实例分享的⽂章就介绍到这了,更多相关Python实现⼿绘图效果内容请搜索以前的⽂章或继续浏览下⾯的相关⽂章希望⼤家以后多多⽀持!。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
Pymol作图的实例
这是一个只是用鼠标操作的初步教程
Pdb文件3ODU.pdb
打开文件
pymol右侧
All指所有的对象,2ODU指刚才打开的文件,(sele)是选择的对象
按钮A:代表对这个对象的各种action,
S:显示这个对象的某种样式,
H:隐藏某种样式,
L:显示某种label,
C:显示的颜色
下面是操作过程:
点击all中的H,选择everything,隐藏所有
点击3ODU中的S,选择cartoon,以cartoon形式显示蛋白质
点击3ODU中的C,选择by ss,以二级结构分配颜色,选择
点击右下角的S,窗口上面出现蛋白质氨基酸序列,找到1164位ITD,是配体
点击选择ITD ,此时sele中就包含ITD这个残基,点击(sele)行的A,选
择rename selection,窗口中出现,更改sele
为IDT,点击(IDT)行的S选择sticks,点击C,选择by element,选择,,调
整窗口使此分子清楚显示。
寻找IDT与蛋白质相互作用的氢键:
IDT行点击A 选择find,选择polar contacts,再根据需要选择,这里选择to other atoms in
object ,分子显示窗口中出现几个黄色的虚线,IDT行下面出现了新的一行,这就是氢键的对象,点击这一行的C,选择
red red,把氢键显示为红色。
接着再显示跟IDT形成氢键的残基
点击3ODU行的S,选择lines,显示出所有残基的侧链,使用鼠标转动蛋白质寻找与IDT 以红色虚线相连的残基,分别点击选择这些残基。
注意此时selecting要是
residures。
选择的时候要细心。
取消选择可以再次点击已选择的残基。
使用上述的方法把选择的残基(sele)改名为s1。
点击S1行的S选择sticks,C选择by elements,点击L选择residures显示出残基名称.在这个例子中发现其中有一个N含有3个氢键有两个可以找到与其连接的氨基酸残基,另一个找不到,这是因为这个氢键可能是与水分子形成的,水分子在pdb文件中只用一个O表示,sticks显示方式没有显示出来水分子,点击all行S选择nonbonded,此时就看到一个水与N形成氢键
,点击分子空白处,然后点击选择这个水分子,更改它的名字为w。
在all 行点击H,选择lines,在选择nonbonded,把这些显示方式去掉只留下cartoon。
点击w行s 选择nb-spheres.
到目前为止已经差不多了
下面是一些细节的调整
残基名称位置的调整:点击右下角是3-button viewing
转变为3-button viewing editing,这样就可以编辑修改
pdb文件了,咱们修改的是label的位置。
按住ctrl键点击窗口中的残基名称label,鼠标拖拽到适合的部位,是显示更清晰。
然后调整视角方向直到显示满意为止,这时就可以保存图片了,file>>save image as>>png
这样的图片勉强能用,为了得到更高质量和更漂亮得图片在作如下处理
Setting >> cartoon>>highlight color
Setting >> cartoon>>fancy helix
Setting >> transperency>>cartoon>>50% 调节透明度
Setting >>label>>size 可以调节label字体大小
Display>>background color>>white 背景设置为白色
此时窗口中的图像应该不怎么好看,点击右上角的ray
,等待一会,完成后就发现图片好多了,这时千万不要再在图片上点击了,赶快保存图片。
最后再介绍几个命令,还是命令更厉害
set label_size,28 设置字体大小
在最后一步ray的时候,可以输入命令ray,要得到其他分辨率的图片可以打命令ray x,y
如ray 2000,1400就生成2000*1400分辨率的图片。
打包的文件包括3ODU.pdb 3ODU.pse 1.png 2.png打开pse文件可以直接看到制作完成时的样子
飞毛腿2011-4-18 六年级上册语文第一次月考(三)
一、看拼音,写词语。
(5分)
xiǎo xiàng()里传出了yōu yáng()的琴声,是谁在为路人yǎn z òu()?连那qí n miǎn()的蚂蚁也停下了奔忙的脚步。
二、给带点的字选择正确的读音画上“”线。
(3分)
唱和.(héhè)吟诵.(yǒng sòng)刚劲.(jìn jìng)
哈.达(hāhǎ)要挟.(xiá xié)沮丧.(sāng sàng)
三、写出同音字,把词语补充完整。
(4分)
mì静()奥()蜂()()集
yùn 音()()含()船()育
四、请你为“肖”字加上偏旁,组成新的字填在空格里。
(3分)
陡()的悬崖胜利的()息俊()的奶娘
()好的铅笔弥漫的()烟畅()的商品
五、用“和”字组词并填空。
(2分)
中国经济发展需要良好的外部环境,只有国际(),国内(),两岸和解,才能实现中国的和平崛起。
六、把下面的成语补充完整。
(3分)
()崖峭壁行色()()()()讥笑
()()安席()()望重可见一()
七、“俏丽、斑斓、粗壮、优美”是《草虫的村落》中的词语,请让它们走进新的语境。
(4分)
()的大树旁,有一片色彩()的野花,蝴蝶穿着()的彩衣来了,高兴得在花丛中跳起了()的舞蹈。
八、在括号里填上带点词语的反义词。
(3分)
1、我对母亲的怀念之情竟越发增强
..,丝毫没有因为岁月的流逝而()。
2、天空逐渐暗下来,原来清晰
..的景物,一下子都变得()。
3、京张铁路的提前竣工,极大地鼓舞
..了中国人民的士气,给藐视中国人帝国主义者一个沉重的()。
九、按要求写句子。
(7分)
1、我就不相信,这些小精灵会不爱我们祖国的海岛,会不愿在这里安居乐业。
改为肯定句:
2、它们的村子散布在林边缘的小丘上。
缩句:
3、我随手把生日礼物郑重地交给妈妈。
修改病句:
4、根据《詹天佑》课文内容用“虽然……但是……”说一句话:
5、有些战士灰心了,撅着嘴巴,说:“西瓜嫌我们的岛艰苦,不愿在这里安家。
”
改为第三人称转述:
6、仿写句子。
(2分)
你好,清凉的山泉!你捧出一面明镜,是要我重新梳妆吗?
十、学以致用。
(4分)
1、我捡起一朵落花,拾起一片落叶,我看到了它们蕴含的生命的奥秘,这时不禁想起龚
自珍的诗
句:。
2、秋天来了,树叶一片片往下落,唯有野菊花在山中开得正欢,难怪元稹用
“
”来表达自己对它的喜爱之情。
3、母爱是世界上最伟大的爱,做儿女的怎么能够报答得了母亲的爱呢?这使我们想起了
诗
句:。
4、关于爱国的名言,我记住了邓小平爷爷的一句
话:。
十一、回想课文内容填空。
(11分)
1、《草虫的村落》一文中的“游侠”指,村落
指。
(2分)
2、“彩色的翅膀”借指蝴蝶等小昆虫,也象
征。
(1分)
3、《山中访友》:山中的古桥、、等朋友给了“我”好心情,好记忆。
4、詹天佑是我国杰出的爱国工程师。
课文
从、、三个方面叙述詹天佑主持修筑京张铁路的过程,他根据居庸关、八达岭不同的山势创造性地设计
了、两种施工方法。
作为一名中华少年,我想对詹天佑说:。
(6分)
十二、选择题。
(5分)
1、下列说法错误的一项是()。
A、“望”按部首查字法,应查部首“王”部,在“德高望重”中的理解为“名望”。
B、“水”字应按“先中间后两边”的笔顺规则写。
C、“亚”和“凸”两个字笔画数都相同。
D、“甲”按音序查字法,应查音序“J”,“花甲”指六十岁。
