pET-SUMO大肠杆菌表达载体说明
pET表达载体

表达载体pETA.pET系统是有史以来在E.coli中克隆表达重组蛋白的功能最强大的系统。
目的基因被克隆到pET质粒载体上,受噬菌体T7强转录及翻译(可选择)信号控制;表达由宿主细胞提供的T7 RNA聚合酶诱导。
T7 RNA 聚合酶机制十分有效并具选择性:充分诱导时,几乎所有的细胞资源都用于表达目的蛋白;诱导表达后仅几个小时,目的蛋白通常可以占到细胞总蛋白的50%以上。
尽管该系统极为强大,却仍能很容易地通过降低诱导物的浓度来削弱蛋白表达。
降低表达水平可能可以提高某些目的蛋白的可溶部分产量。
该系统的另一个重要优点是在非诱导条件下,可以使目的基因完全处于沉默状态而不转录。
用不含T7 RNA聚合酶的宿主菌克隆目的基因,即可避免因目的蛋白对宿主细胞的可能毒性造成的质粒不稳定(详见I. F.部分)。
如果用非表达型宿主细胞克隆,可以通过两种方法启动目的蛋白的表达:用带有受λpL 和pI 启动子控制的T7 RNA聚合酶的λCE6噬菌体侵染宿主细胞,或者将质粒转入带有受lacUV5 控制的T7 RNA聚合酶基因的表达型细胞。
在第二种情形下,可以通过在细菌培养基中加入IPTG 来启动表达。
尽管有时(例如非毒性目的蛋白) 可以直接将目的基因克隆到表达型宿主细胞中,但这种策略并不是通用做法。
两种T7启动子以及多种拥有不同抑制本底表达水平的宿主细胞共同构成了一个极为灵活而有效的系统,使各种目的蛋白得以最优化表达。
所有pET载体以及相关产品均以试剂盒形式提供,用户可以很方便地进行克隆、表达检测以及纯化目的蛋白的所有操作。
pET 表达系统包括质粒和宿主菌。
您可参考系统组成部分,选择符合具体需要的载体/宿主菌最佳组合。
B.使用许可及协议Novagen的T7表达系统,包括细菌、噬菌体和带有T7 RNA 聚合酶基因的质粒,均依照非商业用户应用声明相应条款有条件提供。
详情请垂询。
C. 系统组成pET表达系统提供目的基因克隆和表达所需的核心试剂。
碧云天SUMO Protease产品说明书

SUMO Protease产品简介:碧云天生产的SUMO Protease (Small Ubiquitin-related Modifier Protease),是一种重组表达的高度特异性识别SUMO 化修饰或SUMO 结构域,并水解SUMO 羧基端(C 端) x-Gly-Gly-x 肽段中Gly-Gly 后的肽键,从而去除SUMO 化修饰或者去除SUMO 融合表达蛋白中SUMO 结构域的蛋白酶。
本产品是一种来自Saccharomyces cerevisiae 的高活性的半胱氨酸蛋白酶(cysteinyl protease) Ulp1 (Ubl-specific protease 1, ubiquitin-like protein-specific protease 1)基因片段的重组表达蛋白。
SUMO 是一种泛素样蛋白(Ubiquitin-like Protein),是一种非常常见蛋白翻译后修饰(post-translation modification, PTM),对于蛋白的稳定性、生物学功能有重要的调节作用。
SUMO 也常被作为一种非常高效和有用的标签用于蛋白的表达纯化,例如碧云天的D2918 pET-N-His-PreScission-SUMO 质粒就是一种在N 端同时表达His 标签和SUMO 标签的原核表达质粒。
将SUMO 作为标签和目的蛋白的氨基端(N 端)进行融合表达时可以改善目的蛋白的折叠,提高目的蛋白的可溶性和产量。
随后使用SUMO Protease 对SUMO 融合蛋白进行酶切,去除SUMO 标签就可以获得完全没有标签蛋白干扰的目的蛋白。
因此SUMO Protease 可以高效且特异性地用于从重组融合蛋白上完全切割去除SUMO 标签,从而最大限度地减少了对目的蛋白结构和功能的影响。
SUMO Protease 进行酶切时的最适pH 值为8.0,最佳酶切温度为30ºC 。
大肠杆菌表达系统

调控方式为控制 T7 RNA 聚合 酶的启动子调控类型
ColE1 ori
AmpR
T7 启动子
目的基因
热诱导
T7 RNA 聚合酶
PL 启动子 T7 RNA 聚合酶基因
cI857
p15A ori
KanR
T7 表达系统存在的问题
T7 表达系统表达目的基因的水平是目前所有表达系统中最高的,但 也不可避免出现在相对较高的本底转录,如果目的基因产物对大肠杆 菌宿主有毒性,会影响细胞的生长。
因此 PL 、 PR 表达系统都选用温度敏感突变体 cI 857(ts) 的基因产物 来调控 PL 、 PR 启动子的转录。
在较低温度(30℃)时以活性形式存在 在较高温度(42℃)时失活脱落
PL 和 PR 表达系统
宿主菌中没有 cI 基因产物,PL、PR 启动子的高强度直接转录,带有PL
或 PR 启动子的表达载体在普通大肠杆菌中相当不稳定。
其他表达系统
pH调控型 采用大肠杆菌赖氨酸脱羧酶基因 cadA 启动子构建表达载体。
cadA 启动子受培养基中 H+ 浓度,即 pH 值调控。(在pH﹥8 时抑 制,pH ﹤ 6时激活,pH = 6时转录活力最高)。
用噬菌体 DE3 的溶源菌作为表 达载体的宿主菌,调控方式为 化学诱导型,类似于 Lac 表达 系统。
E.Coli (DE3)
T7 启动子
目的基因
IPTG 诱导
T7 RNA 聚合酶
lac 启动子 T7 RNA 聚合酶基因
T7 表达系统
转录调控的机理 温度诱导型 PL 启动子控制 T7 RNA 聚合 酶基因,通过热诱导方式激发 T7 噬菌体启动子的转录。
斑马鱼p53基因原核表达载体的构建及其在大肠杆菌中的表达

p53 基 因 在 大 肠 杆 菌 中 成 功 表 达 ,表 达 的 p53 融 合 蛋 白 分 子 量 大 约 为 53kD,透 析 复 性 后 获 得 了 高 纯 度
可溶性的 p53 蛋白。
关键词:p53 基因;斑马鱼;原核表达
中 图 分 类 号 :Q786
文 献 标 识 码 :A
文 章 编 号 :0439-8114 (2010)03-0526-03
胞。 挑取单克隆菌落接种于 LB 培养基中,37℃摇荡 过夜, 转接扩大培养至菌液 OD600 达 0.5~0.8 时,加 入 终 浓 度 为 1 mmol / L 的 IPTG,37℃ 诱 导 1、3、5h 后,分别取样进行 SDS-PAGE 分析,5 h 后收集菌体 溶 于 溶 菌 缓 冲 液 (50 mmol / L NaH2PO4,300 mmol / L NaCl,10 mmol / L 咪唑,pH 值 8.0) 中, 超声破碎菌 体 ,12 000 r / min 离 心 20 min, 分 别 取 上 清 和 沉 淀 进行 SDS-PAGE 分析。 1.2.6 p53 蛋 白 的 变 性 纯 化 及 复 性 参 照 Qiagen 镍柱蛋白纯化操作步骤,在变性条件下纯化以包涵 体形式表达的 p53 蛋白。 采用尿素梯度透析法对纯 化的 p53 蛋白进行复性,尿素浓度梯度分别为 8、6、 4、2,0M。 首先将变性纯化的 p53 蛋白用变性结合缓 冲 液 (8 mol / L 尿 素 ,100 mmol / L NaH2PO4,100mM Tris-HCl, pH 值 8.0)稀释 5 倍后装入透析袋中,放 置在透析液中 (100 mmol / L Tris-HCl,100 mmol / L NaH2PO4,1 mmol / L EDTA,2 mmol / L 还原型谷胱甘 肽,0.2 mmol / L 氧化型谷胱甘肽,20%丙三醇,1%甘 氨酸,pH 值 8.0),4℃磁力搅拌透析 12 h, 每隔 12 h 更换含低浓度尿素的透析液,其他成分不变,最后 将 p53 透析至水中,离心去除沉淀,复性的 p53 蛋 白 冷 冻 干 燥 后 经 12% SDS-PAGE 电 泳 检 测 后 于- 80℃保存。
PET系统_原核表达详细总结

04
应用前景展望
在生物制品领域的应用前景
1 2
疫苗生产
利用PET系统进行原核表达,可实现疫苗抗原的 高效制备,降低生产成本和时间。
抗体药物生产
通过PET系统表达抗体,能够简化抗体药物的生 产流程,提高生产效率和产品质量。
3
生物制品质量控制
PET系统可用于生物制品质量控制,如蛋白质结 构鉴定、功能检测和残留物质检测等。
疾病机制研究
PET技术可用于研究疾病的发 生机制、发展过程和治疗方法 ,有助于深入了解疾病的本质
。
医学影像诊断
PET技术能够提供高灵敏度和特异 性的医学影像,有助于肿瘤、神 经系统疾病等疾病的早期诊断。
生物医学材料研究
通过PET技术对生物医学材料进行 标记和检测,能够提高材料的安全 性和有效性。
05
培养基
提供实验菌种生长和繁殖所需的营 养成分。
试剂和仪器
包括各种分子生物学试剂和细胞培 养设备,用于实验操作。
实验方法
活性检测
通过生物学活性检测,评估目的蛋白的功 能和效果。
目的基因克隆
将目的基因从染色体或质粒上克隆到表达 载体中。
表达载体转化
将克隆好的表达载体转化到实验菌种中。
蛋白纯化
采用各种纯化技术,将目的蛋白从细胞中 分离出来。
诱导表达
通过添加诱导剂或其他方法,启动目的基 因的表达。
02
实验结果
pet系统表达的蛋白质量分析
总结词
在利用PET系统进行原核表达的过程中,需要关注蛋白的质量和产量。
详细描述
通过SDS-PAGE和Western Blot分析,可以检测到目标蛋白的分子量标准,同时利用定量蛋白试剂盒可以测定 其浓度。
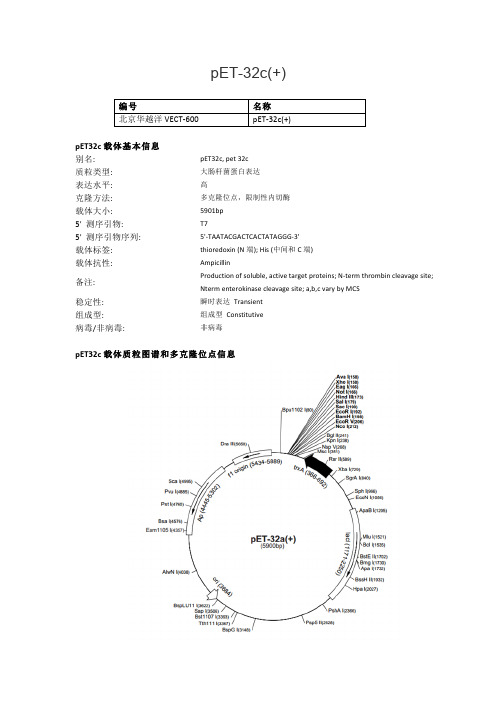
pET-32c(+)大肠杆菌表达载体说明
pET-32c(+)编号 名称北京华越洋VECT-‐600 pET-‐32c(+)pET32c载体基本信息别名: pET32c, p et 32c质粒类型: 大肠杆菌蛋白表达表达水平: 高克隆方法: 多克隆位点,限制性内切酶载体大小: 5901bp5' 测序引物: T75' 测序引物序列: 5'-‐TAATACGACTCACTATAGGG-‐3'载体标签: thioredoxin (N端); H is (中间和C端) 载体抗性: Ampicillin备注: Production o f s oluble, a ctive t arget p roteins; N-‐term t hrombin c leavage s ite; Nterm e nterokinase c leavage s ite; a,b,c v ary b y M CS稳定性: 瞬时表达 Transient 组成型: 组成型 Constitutive 病毒/非病毒: 非病毒pET32c载体质粒图谱和多克隆位点信息pET32c载体简介The pET-‐32 series is designed for cloning and high-‐level expression of peptide sequences fused with t he 109aa T rx•Tag™ thioredoxin p rotein (1). C loning s ites a re a vailable f or p roducing f usion proteins a lso c ontaining c leavable H is•Tag® a nd S•Tag™ sequences f or d etection a nd p urification. Unique sites are shown on the circle map. Note that the sequence is numbered by the pBR322 convention, so the T7 expression region is reversed on the circle map. The cloning/expression region of the coding strand transcribed by T7 RNA polymerase is shown below. The f1 origin is oriented s o t hat i nfection w ith h elper p hage w ill p roduce v irions c ontaining s ingle-‐stranded D NA that corresponds to the coding strand. Therefore, single-‐stranded sequencing should be performed u sing t he T7 t erminator p rimer.pET32c载体序列ORIGIN1 ATCCGGATAT AGTTCCTCCT TTCAGCAAAA AACCCCTCAA GACCCGTTTA GAGGCCCCAA 61 GGGGTTATGC TAGTTATTGC TCAGCGGTGG CAGCAGCCAA CTCAGCTTCC TTTCGGGCTT 121 TGTTAGCAGC CGGATCTCAG TGGTGGTGGT GGTGGTGCTC GAGTGCGGCC GCAAGCTTGT 181 CGACGGAGCT CGAATTCGGA TCCACAGATA TCCCATGGCC TTGTCGTCGT CGTCGGTACC 241 CAGATCTGGG CTGTCCATGT GCTGGCGTTC GAATTTAGCA GCAGCGGTTT CTTTCATACC 301 AGAACCGCGT GGCACCAGAC CAGAAGAATG ATGATGATGA TGGTGCATAT GGCCAGAACC 361 AGAACCGGCC AGGTTAGCGT CGAGGAACTC TTTCAACTGA CCTTTAGACA GTGCACCCAC 421 TTTGGTTGCC GCCACTTCAC CGTTTTTGAA CAGCAGCAGA GTCGGGATAC CACGGATGCC481 ATATTTCGGC GCAGTGCCAG GGTTTTGATC GATGTTCAGT TTTGCAACGG TCAGTTTGCC541 CTGATATTCG TCAGCGATTT CATCCAGAAT CGGGGCGATC ATTTTGCACG GACCGCACCA 601 CTCTGCCCAG AAATCGACGA GGATCGCCCC GTCCGCTTTG AGTACATCCG TGTCAAAACT 661 GTCGTCAGTC AGGTGAATAA TTTTATCGCT CATATGTATA TCTCCTTCTT AAAGTTAAAC 721 AAAATTATTT CTAGAGGGGA ATTGTTATCC GCTCACAATT CCCCTATAGT GAGTCGTATT 781 AATTTCGCGG GATCGAGATC GATCTCGATC CTCTACGCCG GACGCATCGT GGCCGGCATC 841 ACCGGCGCCA CAGGTGCGGT TGCTGGCGCC TATATCGCCG ACATCACCGA TGGGGAAGAT 901 CGGGCTCGCC ACTTCGGGCT CATGAGCGCT TGTTTCGGCG TGGGTATGGT GGCAGGCCCC 961 GTGGCCGGGG GACTGTTGGG CGCCATCTCC TTGCATGCAC CATTCCTTGC GGCGGCGGTG 1021 CTCAACGGCC TCAACCTACT ACTGGGCTGC TTCCTAATGC AGGAGTCGCA TAAGGGAGAG 1081 CGTCGAGATC CCGGACACCA TCGAATGGCG CAAAACCTTT CGCGGTATGG CATGATAGCG 1141 CCCGGAAGAG AGTCAATTCA GGGTGGTGAA TGTGAAACCA GTAACGTTAT ACGATGTCGC 1201 AGAGTATGCC GGTGTCTCTT ATCAGACCGT TTCCCGCGTG GTGAACCAGG CCAGCCACGT 1261 TTCTGCGAAA ACGCGGGAAA AAGTGGAAGC GGCGATGGCG GAGCTGAATT ACATTCCCAA 1321 CCGCGTGGCA CAACAACTGG CGGGCAAACA GTCGTTGCTG ATTGGCGTTG CCACCTCCAG 1381 TCTGGCCCTG CACGCGCCGT CGCAAATTGT CGCGGCGATT AAATCTCGCG CCGATCAACT 1441 GGGTGCCAGC GTGGTGGTGT CGATGGTAGA ACGAAGCGGC GTCGAAGCCT GTAAAGCGGC 1501 GGTGCACAAT CTTCTCGCGC AACGCGTCAG TGGGCTGATC ATTAACTATC CGCTGGATGA 1561 CCAGGATGCC ATTGCTGTGG AAGCTGCCTG CACTAATGTT CCGGCGTTAT TTCTTGATGT 1621 CTCTGACCAG ACACCCATCA ACAGTATTAT TTTCTCCCAT GAAGACGGTA CGCGACTGGG 1681 CGTGGAGCAT CTGGTCGCAT TGGGTCACCA GCAAATCGCG CTGTTAGCGG GCCCATTAAG 1741 TTCTGTCTCG GCGCGTCTGC GTCTGGCTGG CTGGCATAAA TATCTCACTC GCAATCAAAT 1801 TCAGCCGATA GCGGAACGGG AAGGCGACTG GAGTGCCATG TCCGGTTTTC AACAAACCAT 1861 GCAAATGCTG AATGAGGGCA TCGTTCCCAC TGCGATGCTG GTTGCCAACG ATCAGATGGC 1921 GCTGGGCGCA ATGCGCGCCA TTACCGAGTC CGGGCTGCGC GTTGGTGCGG ACATCTCGGT 1981 AGTGGGATAC GACGATACCG AAGACAGCTC ATGTTATATC CCGCCGTTAA CCACCATCAA 2041 ACAGGATTTT CGCCTGCTGG GGCAAACCAG CGTGGACCGC TTGCTGCAAC TCTCTCAGGG 2101 CCAGGCGGTG AAGGGCAATC AGCTGTTGCC CGTCTCACTG GTGAAAAGAA AAACCACCCT 2161 GGCGCCCAAT ACGCAAACCG CCTCTCCCCG CGCGTTGGCC GATTCATTAA TGCAGCTGGC 2221 ACGACAGGTT TCCCGACTGG AAAGCGGGCA GTGAGCGCAA CGCAATTAAT GTAAGTTAGC 2281 TCACTCATTA GGCACCGGGA TCTCGACCGA TGCCCTTGAG AGCCTTCAAC CCAGTCAGCT 2341 CCTTCCGGTG GGCGCGGGGC ATGACTATCG TCGCCGCACT TATGACTGTC TTCTTTATCA 2401 TGCAACTCGT AGGACAGGTG CCGGCAGCGC TCTGGGTCAT TTTCGGCGAG GACCGCTTTC 2461 GCTGGAGCGC GACGATGATC GGCCTGTCGC TTGCGGTATT CGGAATCTTG CACGCCCTCG 2521 CTCAAGCCTT CGTCACTGGT CCCGCCACCA AACGTTTCGG CGAGAAGCAG GCCATTATCG 2581 CCGGCATGGC GGCCCCACGG GTGCGCATGA TCGTGCTCCT GTCGTTGAGG ACCCGGCTAG 2641 GCTGGCGGGG TTGCCTTACT GGTTAGCAGA ATGAATCACC GATACGCGAG CGAACGTGAA 2701 GCGACTGCTG CTGCAAAACG TCTGCGACCT GAGCAACAAC ATGAATGGTC TTCGGTTTCC 2761 GTGTTTCGTA AAGTCTGGAA ACGCGGAAGT CAGCGCCCTG CACCATTATG TTCCGGATCT 2821 GCATCGCAGG ATGCTGCTGG CTACCCTGTG GAACACCTAC ATCTGTATTA ACGAAGCGCT 2881 GGCATTGACC CTGAGTGATT TTTCTCTGGT CCCGCCGCAT CCATACCGCC AGTTGTTTAC 2941 CCTCACAACG TTCCAGTAAC CGGGCATGTT CATCATCAGT AACCCGTATC GTGAGCATCC 3001 TCTCTCGTTT CATCGGTATC ATTACCCCCA TGAACAGAAA TCCCCCTTAC ACGGAGGCAT 3061 CAGTGACCAA ACAGGAAAAA ACCGCCCTTA ACATGGCCCG CTTTATCAGA AGCCAGACAT 3121 TAACGCTTCT GGAGAAACTC AACGAGCTGG ACGCGGATGA ACAGGCAGAC ATCTGTGAAT3181 CGCTTCACGA CCACGCTGAT GAGCTTTACC GCAGCTGCCT CGCGCGTTTC GGTGATGACG 3241 GTGAAAACCT CTGACACATG CAGCTCCCGG AGACGGTCAC AGCTTGTCTG TAAGCGGATG 3301 CCGGGAGCAG ACAAGCCCGT CAGGGCGCGT CAGCGGGTGT TGGCGGGTGT CGGGGCGCAG 3361 CCATGACCCA GTCACGTAGC GATAGCGGAG TGTATACTGG CTTAACTATG CGGCATCAGA 3421 GCAGATTGTA CTGAGAGTGC ACCATATATG CGGTGTGAAA TACCGCACAG ATGCGTAAGG 3481 AGAAAATACC GCATCAGGCG CTCTTCCGCT TCCTCGCTCA CTGACTCGCT GCGCTCGGTC 3541 GTTCGGCTGC GGCGAGCGGT ATCAGCTCAC TCAAAGGCGG TAATACGGTT ATCCACAGAA 3601 TCAGGGGATA ACGCAGGAAA GAACATGTGA GCAAAAGGCC AGCAAAAGGC CAGGAACCGT 3661 AAAAAGGCCG CGTTGCTGGC GTTTTTCCAT AGGCTCCGCC CCCCTGACGA GCATCACAAA 3721 AATCGACGCT CAAGTCAGAG GTGGCGAAAC CCGACAGGAC TATAAAGATA CCAGGCGTTT 3781 CCCCCTGGAA GCTCCCTCGT GCGCTCTCCT GTTCCGACCC TGCCGCTTAC CGGATACCTG 3841 TCCGCCTTTC TCCCTTCGGG AAGCGTGGCG CTTTCTCATA GCTCACGCTG TAGGTATCTC 3901 AGTTCGGTGT AGGTCGTTCG CTCCAAGCTG GGCTGTGTGC ACGAACCCCC CGTTCAGCCC 3961 GACCGCTGCG CCTTATCCGG TAACTATCGT CTTGAGTCCA ACCCGGTAAG ACACGACTTA 4021 TCGCCACTGG CAGCAGCCAC TGGTAACAGG ATTAGCAGAG CGAGGTATGT AGGCGGTGCT 4081 ACAGAGTTCT TGAAGTGGTG GCCTAACTAC GGCTACACTA GAAGGACAGT ATTTGGTATC 4141 TGCGCTCTGC TGAAGCCAGT TACCTTCGGA AAAAGAGTTG GTAGCTCTTG ATCCGGCAAA 4201 CAAACCACCG CTGGTAGCGG TGGTTTTTTT GTTTGCAAGC AGCAGATTAC GCGCAGAAAA 4261 AAAGGATCTC AAGAAGATCC TTTGATCTTT TCTACGGGGT CTGACGCTCA GTGGAACGAA 4321 AACTCACGTT AAGGGATTTT GGTCATGAGA TTATCAAAAA GGATCTTCAC CTAGATCCTT 4381 TTAAATTAAA AATGAAGTTT TAAATCAATC TAAAGTATAT ATGAGTAAAC TTGGTCTGAC 4441 AGTTACCAAT GCTTAATCAG TGAGGCACCT ATCTCAGCGA TCTGTCTATT TCGTTCATCC 4501 ATAGTTGCCT GACTCCCCGT CGTGTAGATA ACTACGATAC GGGAGGGCTT ACCATCTGGC 4561 CCCAGTGCTG CAATGATACC GCGAGACCCA CGCTCACCGG CTCCAGATTT ATCAGCAATA 4621 AACCAGCCAG CCGGAAGGGC CGAGCGCAGA AGTGGTCCTG CAACTTTATC CGCCTCCATC 4681 CAGTCTATTA ATTGTTGCCG GGAAGCTAGA GTAAGTAGTT CGCCAGTTAA TAGTTTGCGC 4741 AACGTTGTTG CCATTGCTGC AGGCATCGTG GTGTCACGCT CGTCGTTTGG TATGGCTTCA 4801 TTCAGCTCCG GTTCCCAACG ATCAAGGCGA GTTACATGAT CCCCCATGTT GTGCAAAAAA 4861 GCGGTTAGCT CCTTCGGTCC TCCGATCGTT GTCAGAAGTA AGTTGGCCGC AGTGTTATCA 4921 CTCATGGTTA TGGCAGCACT GCATAATTCT CTTACTGTCA TGCCATCCGT AAGATGCTTT 4981 TCTGTGACTG GTGAGTACTC AACCAAGTCA TTCTGAGAAT AGTGTATGCG GCGACCGAGT 5041 TGCTCTTGCC CGGCGTCAAT ACGGGATAAT ACCGCGCCAC ATAGCAGAAC TTTAAAAGTG 5101 CTCATCATTG GAAAACGTTC TTCGGGGCGA AAACTCTCAA GGATCTTACC GCTGTTGAGA 5161 TCCAGTTCGA TGTAACCCAC TCGTGCACCC AACTGATCTT CAGCATCTTT TACTTTCACC 5221 AGCGTTTCTG GGTGAGCAAA AACAGGAAGG CAAAATGCCG CAAAAAAGGG AATAAGGGCG 5281 ACACGGAAAT GTTGAATACT CATACTCTTC CTTTTTCAAT ATTATTGAAG CATTTATCAG 5341 GGTTATTGTC TCATGAGCGG ATACATATTT GAATGTATTT AGAAAAATAA ACAAATAGGG 5401 GTTCCGCGCA CATTTCCCCG AAAAGTGCCA CCTGAAATTG TAAACGTTAA TATTTTGTTA 5461 AAATTCGCGT TAAATTTTTG TTAAATCAGC TCATTTTTTA ACCAATAGGC CGAAATCGGC 5521 AAAATCCCTT ATAAATCAAA AGAATAGACC GAGATAGGGT TGAGTGTTGT TCCAGTTTGG 5581 AACAAGAGTC CACTATTAAA GAACGTGGAC TCCAACGTCA AAGGGCGAAA AACCGTCTAT 5641 CAGGGCGATG GCCCACTACG TGAACCATCA CCCTAATCAA GTTTTTTGGG GTCGAGGTGC 5701 CGTAAAGCAC TAAATCGGAA CCCTAAAGGG AGCCCCCGAT TTAGAGCTTG ACGGGGAAAG 5761 CCGGCGAACG TGGCGAGAAA GGAAGGGAAG AAAGCGAAAG GAGCGGGCGC TAGGGCGCTG5821 GCAAGTGTAG CGGTCACGCT GCGCGTAACC ACCACACCCG CCGCGCTTAA TGCGCCGCTA 5881 CAGGGCGCGT CCCATTCGCC A//其他大肠杆菌表达载体:pBV221 ptdTomato pET-‐52b(+) pAmCyanpDsRed-‐Express2 pBV220 pCold-‐GST pColdS-‐SUMOpCold T F pCold I V pCold I II pCold I IpCold I pE-‐SUMO pCold-‐ProS2 pBAD102/D-‐TOPOpBAD202/D-‐TOPO pACYC184 pBAD/Thio-‐TOPO pBad/Myc-‐His CpBad/Myc-‐His B pBad/Myc-‐His A pBad/His C pBad/His BpBad/His A pBAD-‐TOPO pET-‐23b(+) pET-‐23a(+)pET-‐23c(+) pET-‐23(+) pET-‐12b(+) pET-‐12c(+)pET-‐12a(+) pET-‐11b(+) pET-‐11a(+) pET-‐11c(+)pBad24 pQE-‐82L pQE-‐81L pQE-‐80LpQE-‐32 pQE-‐9 pQE-‐16 pQE-‐31pQE-‐60 pQE-‐70 pQE-‐40 pET-‐51b(+)pET-‐50b(+) pET-‐49b(+) pET-‐48b(+) pET-‐47b(+)pET-‐26b(+) pET-‐32a(+) pET-‐21b(+) pET-‐22b(+)pET-‐14b pET-‐16b pET-‐15b pET-‐19bpET-‐20b(+) pET-‐21d(+) pET-‐21c(+) pET-‐21b(+)pET-‐21a(+) pET-‐24a(+) pET-‐24d(+) pET-‐25b(+)pET-‐27b(+) pET-‐28a(+) pET-‐30a(+) pET-‐42a(+)pET-‐43.1c(+) pET-‐43.1b(+) pET-‐43.1a(+) pET-‐44a(+)pET-‐44c(+) pET-‐46 E K/LIC pET-‐37b(+) pTrcHis2 CpTrcHis2 B pTrcHis2 A pET303/CT-‐His pET302/NT-‐HispRSET-‐CFP pRSET-‐EmGFP pRSET-‐BFP pGFPuvpET300/NT-‐DEST pET301/CT-‐DEST pGEM-‐T pBad43pGEX-‐4T-‐3 pGEX-‐5X-‐2 pBlueScript S K(+) pG-‐Tf2pG-‐KJE8 pGro7 pET-‐SUMO pSE380pET-‐17b pET102/D-‐TOPO pCDFDuet-‐1 pMAL-‐p5xpTf16 pET-‐28c(+) pBluescript I I S K(+) pET-‐30b(+)pSUMO pProEX H Tc pProEX H Tb pProEX H TapKD3 pKD13 pKD46 pTYB1pTYB2 pTWIN2 pBluescript I I K S(-‐) pTYB12pMAL-‐p5e pACYCDuet-‐1 pEGM-‐11ZF(+) pEGM-‐7ZF(+)PinPoint X a-‐3 PinPoint X a-‐2 PinPoint X a-‐1 pSP73pSP64 pTWIN1 pTYB11 pTXB1pET-‐5b(+) pBad/gIII C pBad/gIII B pBad/gIII ApET-‐5a(+) pMal-‐p4X pMal-‐p2G pkk223-‐3pkk232-‐8 pCYB1 pEZZ18 pBAD18pMAL-‐c5x pMal-‐p2E pMal-‐p2X pET-‐44 E K/LICpET-‐43.1 E K/LIC pET-‐41 E K/LIC pMal-‐c4X pTrcHis BpET-‐31b(+) pET-‐3b(+) pET-‐41a(+) pGEX-‐3XpGEX-‐4T-‐2 pETDuet-‐1 pGEX-‐4T-‐1 pTrc99a pET-‐28b(+) pET-‐His pALEX a,b,c pACYC177 pBR322 pKD4 pKD20 pMXB10 pEcoli-‐6xHN-‐GFPuv pKJE7 pRSET B pGEX-‐KG pGEX-‐2T pRSFDuet-‐1 pCOLADuet-‐1 pTrcHis C pTrcHis A pET-‐41b(+) pET-‐42b(+) pET-‐3a(+) pGEX-‐6P-‐3 pGEX-‐6P-‐2 pGEX-‐6P-‐1 pGEX-‐5X-‐3 pGEX-‐5X-‐1 pGEX-‐2TK pRSET A pMal-‐c2G pMal-‐c2E pMal-‐c2X pRSET C pQE-‐30pET-‐45b(+) pET-‐44b(+) pET-‐42c(+) pET-‐41c(+) pET-‐40b(+) pET-‐33b(+) pET-‐39b(+) pET-‐32 E K/LIC pET-‐32 X a/LIC pET-‐32c(+) pET-‐32b(+) pET-‐30 X a/LIC pET-‐30 E K/LIC pET-‐30c(+) pET-‐29c(+) pET-‐29b(+) pET-‐29a(+) pET-‐24c(+) pET-‐24b(+) pET-‐24(+) pET-‐23d(+) pET-‐11d(+) pBad33。
小分子泛素相关修饰物SUMO融合外源蛋白表达的研究进展

文章编号:2096-0387 (2018) 03-0152-03第4卷第3期 生物化工Vol.4 No.32018 年 6 月Biological Chemical EngineeringJun. 2018小分子泛素相关修饰物SUM O 融合外源蛋白表达的研究进展荣雅昕,王英超'张耀方,卢顺娇,周倩,陈慧慧(天津农学院基础科学学院,天津300384)摘要:S U M 0 (类泛素蛋白修饰分子)是一类具有高度保守序列的低分子量蛋白。
作为融合标签在蛋白质的融合表达得到 了广泛应用。
本文对SUM0融合表达系统的优点进行概述,并综述了其在原核生物和真核生物外源蛋白融合表达的研究情况。
关键词:融合外源蛋白;SU M 0;研究进展 中图分类号:Q786文献标志码:AResearch Progress on the Fusion of Foreign Protein by Small Molecule Ubiquitin PeptideRong Ya -xin , Wang Ying-chao , Zhang Yao -fang , Lu Shun -jiao , Zhou Qian,Chen H ui-hui(College of Basic Science , Tianjin Agriculture University , Tianjin 300384)Abstract : SUMOwas a class of low molecular weight proteins with highly conserved sequences . As a fusion label , the fusion expression of protein has been widely used . In this paper , the advantages of SUMO fusion expression system were summarized and the progress in the fusion expression of exogenous proteins in prokaryotes and eukaryotes was reviewed .Key words : Fusion of exogenous protein;Small ubiquitin-related modifer;Research progressSUMO 是存在于真核生物中起相关修饰作用的 一类蛋白质,具有调节蛋白转运、相互作用,维持基 因完整性等多种功能[1_3]。
SUMO蛋白酶活性片段的表达、纯化及活性测定

SUMO蛋白酶活性片段的表达、纯化及活性测定
SUMO蛋白酶被认为在多种细胞过程中起着调节作用。
为了
进一步研究SUMO蛋白酶的生物学功能,需要进行其活性片
段的表达、纯化及活性测定。
首先,可以使用质粒构建技术从人类cDNA库中克隆SUMO
蛋白酶的一个片段,如Ulp1,Ulp2或Ulp3。
将其插入到表达
载体中,并进行适当的序列验证。
接下来,需要选择适当的宿主细胞进行表达。
常用的宿主细胞为大肠杆菌,但也存在其他表达系统,如酵母菌表达系统。
通过在表达时添加不同的诱导剂,可以控制蛋白的表达量和时机。
之后,需要对表达的蛋白进行纯化。
可以利用亲和纯化柱进行初步的纯化,然后再使用离子交换、凝胶过滤和透析等方式进行进一步的纯化。
纯化后的蛋白需要经过SDS-PAGE和Western blot验证。
最后,需要对SUMO蛋白酶活性片段进行活性测定。
可以观
察其对SUMO修饰蛋白的去SUMO化作用。
可以通过Western blot、荧光标记或质谱等方法进行定量。
活性测定的
结果可以用于进一步研究SUMO蛋白酶的生物学功能及调节
机制。
在表达、纯化和活性测定这一系列实验中,需要严格控制实验条件和研究方法。
同时,还需要注意跟踪和记录实验结果,以便后续分析和讨论。
大肠杆菌表达系统课件

THANKS
感谢观看
3
使用表达优化后的载体,提高表达效率
改进方案提出与实施效果评估
01
02
03
优化培养条件和诱导策 略
调整诱导剂浓度、诱导 时间和温度等参数
使用分阶段诱导策略, 降低蛋白表达速度,促
进正确折叠
改进方案提出与实施效果评估
01
引入伴侣蛋白和辅助因子
02
共表达伴侣蛋白,促进蛋白正确折叠和组装
03
添加辅助因子,提高蛋白稳定性和溶解度
大肠杆菌表达系统实例分析
成功案例展示及经验分享
案例一:高效表达目 标蛋白
优化诱导条件和培养 参数
选择强启动子和合适 载体
成功案例展示及经验分享
目标蛋白表达量高,纯度高 案例二:实现复杂蛋白表达
利用伴侣蛋白和辅助因子
成功案例展示及经验分享
调整诱导时间和温度 成功表达具有生物活性的复杂蛋白
失败案例剖析及原因探讨
学Hale Waihona Puke 实践操作指点实验前准备指点学生熟悉实验器材、 试剂和操作方法,确保实 验顺利进行。
实践操作注意事项
强调实验过程中的安全、 卫生和规范操作,避免误 操作导致实验失败或影响 实验结果。
数据记录与分析
指点学生记录实验数据, 学会分析和解读实验结果 ,培养科学思维和实验技 能。
问题解答与互动讨论
实验原理解答
优化培养温度和时间
降低培养温度、延长培养时间有助于蛋白正确折叠。
改进纯化流程提高效率
选择合适纯化方法
根据蛋白性质选择亲和层析、离子交换层析、凝胶过滤层析等方 法组合使用。
优化纯化条件
调整洗脱液成分、pH值及盐浓度等条件,提高纯化效果。
sumo酶融合表达质粒

sumo酶融合表达质粒
摘要:
1.引言
2.sumo 酶融合表达质粒的定义和作用
3.sumo 酶融合表达质粒的制作方法
4.sumo 酶融合表达质粒的应用领域
5.总结
正文:
sumo 酶融合表达质粒是一种将sumo 酶基因与表达载体结合的生物制品,通过这种方法,可以在细胞内大量表达sumo 酶。
sumo 酶是一种具有高度保守性的蛋白质,广泛存在于多种生物体中,主要功能是对蛋白质进行sumo 化修饰,从而影响其结构和功能。
制作sumo 酶融合表达质粒的方法通常包括以下几个步骤:首先,需要获取sumo 酶的基因序列,这可以通过基因合成或PCR 扩增得到。
然后,将sumo 酶基因与表达载体连接,形成融合表达质粒。
在这个过程中,需要选择合适的启动子和标签,以确保sumo 酶能够在细胞内得到高效表达。
最后,将融合表达质粒转化到宿主细胞中,进行培养和筛选,得到大量表达sumo 酶的细胞。
sumo 酶融合表达质粒在多个领域都有广泛应用。
在基础科研领域,可以通过研究sumo 酶的表达和分布,深入了解其在细胞内的功能和作用机制。
在生物医药领域,sumo 酶融合表达质粒可以用于生产具有重要治疗价值的
sumo 酶相关药物。
此外,sumo 酶融合表达质粒还可以用于生物传感器的研究和开发,为食品安全和环境监测等领域提供技术支持。
总之,sumo 酶融合表达质粒是一种具有重要研究价值和应用前景的生物制品。
大肠杆菌表达系统使用指导

体系选择
研究基因功能: 研究基因功能: 大肠杆菌, 裂殖酵母,昆虫细胞, CHO细胞 大肠杆菌, 裂殖酵母,昆虫细胞, CHO细胞 多肽药物生产: 多肽药物生产: 大肠杆菌, 毕氏酵母, CHO细胞 细胞, 大肠杆菌, 毕氏酵母, CHO细胞, 乳腺组织 疫苗: 大肠杆菌, 酵母, 疫苗: 大肠杆菌, 酵母, 大多数沿用细胞培养产物进行灭毒 单抗生产: 单抗生产: 杂交瘤细胞 工业酶生产: 工业酶生产: 各种微生物
的优点,包括: 人血清白蛋白融合技术具有明显 的优点,包括:HSA与目 与目 标蛋白在胞内经蛋白翻译系统通过肽键连接, 标蛋白在胞内经蛋白翻译系统通过肽键连接, 不需额外的体 外处理;HSA表达水平较高, 融合后可以提高目的蛋白的表 外处理; 表达水平较高, 融合后可以提高目的蛋白的表 表达水平较高 是一个稳定的“ 达水平; 达水平;HSA是一个稳定的“ 惰性” 蛋白, 融合后可以提高 是一个稳定的 惰性” 蛋白, 融合后可以提高 融合蛋白具有比PEG修饰的蛋白药 融合蛋白具有比 修饰的蛋白药 目的蛋白的稳定性 ; HAS融合蛋白具有比 物更长的半衰期 。
(二)融合蛋白技术的内容
• 构建融合蛋白的基本原则是, 将第一个蛋白的终止密码子删除, 构建融合蛋白的基本原则是, 将第一个蛋白的终止密码子删除 终止密码子删除, 再接上带有终止密码子的第二个蛋白基因,以实现两个基因的共同 再接上带有终止密码子的第二个蛋白基因,以实现两个基因的共同 带有终止密码子的第二个蛋白基因 表达。具体步骤有: 表达。具体步骤有: 1.进行目的基因的克隆:根据基因序列互补原则 ,设计合适的引物序 进行目的基因的克隆: 进行目的基因的克隆 为模板, 技术扩增不同的目的DNA片段。 片段。 列,以cDNA为模板,利用 为模板 利用PCR技术扩增不同的目的 技术扩增不同的目的 片段 2.在载体中进行重组:通过限制内切酶将两个 在载体中进行重组: 在载体中进行重组 通过限制内切酶将两个DNA片段进行酶切并回 片段进行酶切并回 收,然后通过连接酶将两个具有相同末端酶切位点的基因片段进行 体外连接,并克隆到高表达质粒载体中,构建重组质粒。 体外连接,并克隆到高表达质粒载体中,构建重组质粒。
高效表达可溶性重组蛋白表达载体_pHisSUMO

3 讨论构建真核表达载体首先要根据其用途和基本策略进行严密的实验设计,如果将目的片段和载体质粒分别双酶切后进行连接,会因酶切效果不理想导致缺失。
因此本研究采取T 载体克隆的方法将目的片段连入P M D -19T 载体中,然后从P M D -19T 重组载体上酶切下目的片段再与同样经酶切处理的PcDNA3载体进行连接,这样就保证了重组质粒PcDNA3-h LY Z 构建的成功和准确,为研究h LY Z 基因的真核表达奠定了基础[6]。
对上述阳性克隆进行DNA 测序,结果表明扩增的5’和3’调控区与已发表的序列的同源性较高。
该调控序列在牛、人和鼠的相应区域进行比较同源性均为97%~100%,说明该调控序列在人、牛和小白鼠上具有较高的保守性[7]。
对于构建特异表达载体来说,5’和3’UTR 的特异启动子和调控系列尤为重要。
许多研究证实其酪蛋白基因5’端和3’端UTR 及启动子是乳腺特异表达的关键调控原件。
另外,乳蛋白基因UTR 是构建乳腺生物反应器表达载体的核心生物元件[8]。
目前已经用于转基因动物乳腺生物反应器的调控元件主要有:β-乳球蛋白基因、aS1和β-酪蛋白调控序列、乳清酸蛋白基因调控序列和乳清白蛋白基因调控序列[9]。
近二十年的研究证明,以β-乳球蛋白和β-酪蛋白基因调控序列构建的乳腺生物反应器表达载体在绵羊、山羊和奶牛上获得高水平表达,而乳清酸蛋白基因调控序列指导的蛋白则在兔和猪等动物上有较高水平的表达,但各有优缺点[3]。
国外研究报道表明酪蛋白是在乳腺的特异表达的蛋白质,是乳腺的主要蛋白[10]。
pC -h LY Z 载体的成功建立为研究酪蛋白基因5’端和3’端的调控序列的调控机理和调控特性提供了一个平台;并用上述牛乳腺特异表达载体表达药用蛋白,建立具有产业开发价值的牛乳腺高效表达技术体系和进一步制作转基因动物奠定了基础。
参考文献:[1]楼善贤,苗得林.溶菌酶的研究进展[J ].中国肿瘤临床,1994,21(9):709.[2]荣晓花,凌沛学.溶菌酶的研究进展[J ].中国生化药物杂志,1999,20(6):319-320.[3]M aga E A ,Murray J D.M ammary gland expression of transgenes and the po 2tential for altering the properties of m ilk [J ].Biotechnol ,1995,13:1452-1456.[4]朱秋菊,孙怀昌.鸡输卵管暂态表达人溶菌酶的研究[J ].生物技术通讯,2006,6:11-12.[5]孙怀昌,于峰,苏建华,等.人溶菌酶基因治疗奶牛乳腺炎的初步研究[J ].畜牧兽医学报,2004,35(2):227-232.[6]李辉,王希彪.中国动物遗传育种研究进展[M].北京:中国农业科学技术出版社,2005,08:111.[7]史瀛仙,季肖东,李光三,等.转基因小鼠的研究[J ].生物工程学报,1999,6(3):205-211.[8]李宁,昊常信,陈永福.牛aS1酪蛋白基因启动区的克隆和序列分析[J ].遗传,1997,19(1):4-8.[9]Liu J ,Lin A ,Zhou Y,et al.Expression control of beta -lac -toglobulin gene and iapplication[J ].Biotechnol I nform ation ,2001,3:23-27.[10]薛京伦,卢大儒.乳腺生物反应器的研究现状[J ].生物技术通报,1998,(3):17-20.高效表达可溶性重组蛋白表达载体———pHisSUM O李璐1,尹成凯2,李德山23(1.东北农业大学生命科学与生物技术研究中心,黑龙江哈尔滨150030;2.东北农业大学生命科学学院,黑龙江哈尔滨150030)摘要:目的:构建含有I L -1基因的原核表达质粒,并对其原核表达情况进行检测,验证pHisS UM O 表达载体的高效可溶性表达。
SUMO-TAT-Sox2融合蛋白的表达及纯化

SUMO-TAT-Sox2融合蛋白的表达及纯化[摘要] 目的将小鼠Sox2基因、穿膜肽TAT以及小分子泛素样修饰蛋白SUMO融合并转入大肠杆菌中进行表达,最终获得大量具有高效穿膜活性的Sox2蛋白。
方法利用PCR技术扩增得SUMO-TAT融合基因,并插入到pET-3c载体。
再通过扩增获得小鼠Sox2基因,将其与pET3c-SUMO-TAT基础载体连接,构建得重组表达载体pET3c-SUMO-TAT-Sox2,然后转化到Rosseta(DE3)表达菌中,经IPTG诱导表达,使用Ni-NTA亲和层析进行分离纯化。
结果成功构建了pET3c-SUMO-TAT-Sox2原核表达载体,经终浓度为1 mmol /L的IPTG诱导4 h后表达约为57kDa的融合蛋白,以包涵体形式存在,Western Blotting检测显示良好特异性,经300 mmol/L咪唑洗脱可获得纯度较高的SUMO-TAT-Sox2融合蛋白。
结论得到大量带有穿膜肽TAT的融合蛋白Sox2,为今后利用重编程因子融合蛋白诱导体细胞重编程为iPS细胞奠定基础。
[关键词] Sox2基因;小分子泛素样修饰蛋白;TAT;原核表达;蛋白纯化转录因子Sox2属于HMG蛋白家族成员之一,具有维持胚胎干细胞自我更新能力,并能抑制中胚层细胞产生多种组织类型的细胞系的分化[1]。
Sox2可作为成体干细胞的分子标记物[2]。
范祖森等揭示了Sox2是通过招募介导组蛋白去甲基化和去乙酰化的NuRD复合物参与自噬调节,在细胞重编程过程中发挥重要作用[13]。
Sox2是通过转录激活SFRP2这一Wnt信号通路的拮抗因子,同时转录抑制WLS这一Wnt运输蛋白从而达到抑制经典Wnt信号通路的作用来实现从人胚胎干细胞向神经分化[4]。
由此可见,Sox2在在维持干细胞多能性以及细胞重编程中发挥重要作用。
小分子泛素样修饰蛋白(small ubiquitin-like modifier, SUMO)是一种与泛素在结构上十分相似的小分子多肽,作为重组蛋白表达的融合标签和分子伴侣,不仅可以与靶蛋白N端结合来帮助其正确转运和折叠,且SUMO有利于增加蛋白的可溶性,能够大大提高蛋白质在大肠杆菌的表达量[35]。
pet经典质粒pet系统是有史以来在大肠杆菌中表达重组蛋白

pET经典质粒pET系统是有史以来在大肠杆菌中表达重组蛋白的功能最强大的系统,也是现今原核表达方面使用最广泛的系统。
该系统中,目的基因被克隆到pET质粒载体上,受强噬菌体T7转录及翻译信号控制;表达由宿主细胞提供的T7 RNA聚合酶诱导。
T7 RNA聚合酶机制十分有效:充分诱导时,几乎所有的细胞资源都用于表达目的蛋白;诱导表达后仅几个小时,目的蛋白通常可以占到细胞总蛋白的50%以上。
尽管该系统极为强大,却仍能很容易地通过改变诱导物的浓度来降低表达水平。
降低表达水平常用以提高某些目的蛋白的可溶部分产量。
该系统的另一个重要优点是在非诱导条件下,可以使目的基因完全处于沉默状态而不转录。
用不含有T7 RNA聚合酶的宿主菌克隆目的基因,即可解决免因目的蛋白表达对宿主细胞的毒性造成的质粒不稳定难题。
两种T7启动子以及多种拥有不同抑制本底表达水平的宿主细胞共同构成了一个极为灵活而有效的系统,使各种目的蛋白都能得以最优化表达。
可选质粒最经典的pET-28a, pET-30a和pET-32a质粒,应用最广,参考文献最多。
下表列出三个经典系列载体主要特性。
其中命名后带有(+)的载体含有f1复制区,可以制备单链DNA,适合突变及测序等应用。
pET-28a: T7lac启动子,高效及严谨型控制表达水平;N端His.Tag/T7.Tag融合标签,可利用His.Tag进行金属离子螯合层析纯化表达蛋白,也可利用T7.T ag融合标签进行基于抗体结合的亲和纯化;含凝血酶(Thrombin)蛋白酶切位点;pET-30a:T7lac启动子;N端His.Tag/S.Tag融合标签,可利用His.Tag进行金属离子螯合层析纯化表达蛋白,也可利用S.Tag融合标签进行亲和纯化及高灵敏度定量检测;含凝血酶(Thrombin)及肠激酶(Enterokinase)蛋白酶切位点;pET-32a:T7lac启动子;Trx融合蛋白表达载体,帮助表达蛋白形成二硫键,增加蛋白溶解性及活性;His.Tag/S.Tag融合标签。
大肠杆菌表达载体

pGEX-1T—凝血酶
pGEX-2T---凝血酶
pGEX-3T---X因子
位相载体
西南大学生物技术专业 基因工程 14
分泌型融合表达载体----pEZZ18
西南大学生物技术专业 基因工程
15
分泌型表达载体----pINIII-ompA1
西南大学生物技术专业 基因工程
16
四、表达产物的纯化
1 、包涵体 (inclusion body) 的纯化:许多情况下表达产物 在细胞内形成不溶的颗粒状包涵体,可通过机械法、冻融 法、超声波处理等破碎细胞,离心收集包涵体,洗涤去除 杂蛋白,用盐酸胍、尿素和 SDS 溶解包涵体,再通过一定 的法使蛋白质折叠。 有的经上法得到后仍然有活性,有的蛋白一旦形成包涵体后 就没有活性了,但可作为抗原。 2、可溶性蛋白的纯化:表达的蛋白可以细胞内解物的上清部分用于 纯化目标蛋白,甚至可直接作为粗酶液进行生化反应。
西南大学生物技术专业 基因工程 7
3 、内含肽表达载体:如 NEB 公司的 Impact-Twin 系 统,将目的蛋白放在两个可自裂解的内含肽 (intein)中间,在得到融合蛋白以后不通过蛋白 酶消解、只需要调节pH值等条件就将标签蛋白切 除。 4 、分泌表达载体:产物可跨膜分泌至胞周间隙, 可避免受细胞内蛋白酶的降解,或使其正确折叠, 或去除N-端甲硫氨酸,以维护活性。 信号肽(signal peptide) 有碱性磷酸酶信号肽、蛋 白质 A 信号肽(如 Amersham 公司的 pEZZ18 系统)。
西南大学生物技术专业 基因工程
23
2 ) 植 物 Ac-Ds 转 座 子 双 因 子 插 入 突 变 : 玉 米 Ac (activator) 因子是一个转座子,含有完整的转 座酶, Ds (dissociation) 是 Ac 缺失转座酶基因 的缺失体,但具两端的反向重复序列。
pET载体

pET载体pET载体,原核表达金标准对于全世界许多研究者,Novagen 的pET 系统已成为在大肠杆菌中蛋白表达的首选。
该系统成功的一个主要原因是目标基因被克隆到不为大肠杆菌RNA 聚合酶识别的T7 启动子之下,因此在加入T7 RNA 聚合酶之前几乎没有表达发生。
克隆到pET 载体的基因实际上是被关闭的,不会由于产生的蛋白对细胞有毒性而引起质粒不稳定。
重组质粒转移到染色体上含有一拷贝由lacUV5 控制的T7 RNA 聚合酶基因的表达宿主中,并通过加入IPTG 诱导表达;也可通过l CE6 感染原始克隆宿主菌来提供T7 RNA 聚合酶。
使用大肠杆菌启动子系统( 如tac 、lac 、trc 、pL) 有困难的许多基因已经在pET 系统中稳定克隆和表达。
T7 RNA 聚合酶的选择性和活性使得几乎所有细胞资源都用于为目标基因表达。
诱导后几小时目标产物就可超过细胞总蛋白的50% 。
新开发的T7 驱动表达技术以pETBlue TM 系统为代表。
pETBlue 载体包括了pET 用于表达的优点,在目标基因克隆和质粒DNA 操作的方面更为方便。
pETBlue TM 系统:新一代T7 表达载体pETBlue 载体代表了新型表达载体,它具备所有广受欢迎的克隆载体的最理想特点和T7 驱动蛋白表达的完全功能。
目标基因以相对于修饰的大肠杆菌tet 启动子的反义方向插到lacZ a - 肽编码区,因此可进行蓝/ 白斑筛选。
正确定位于目标基因正义方向上游的T7 转录和翻译信号使表达成为可能。
与标准pET 载体一样,通过转化λ DE3 溶原菌并用IPTG 诱导或通过l CE6 感染原始宿主菌生产目标蛋白。
优点·蓝/ 白斑筛选,便于克隆·高拷贝数,质粒DNA 高产·以AccepTor TM 载体或perfectly Blunt a 载体形式提供,便于快速PCR 克隆·目标基因无基础水平表达,消除了毒性基因产物相关的质粒不稳定性·表达水平与经典pET 载体相同·用Tuner TM (DE3)pLacI 宿主菌实现真正的表达水平“变阻器”控制。
利用SUMO技术表达可溶性的拟南芥AtRD22蛋白

利用SUMO技术表达可溶性的拟南芥AtRD22蛋白唐玉林;米子岚;钟活权;江年琼【摘要】为了体外获得可溶性的拟南芥AtRD22蛋白,以拟南芥叶片提取的总核糖核酸(ribonucleic acid,RNA)为模版,反转录获得AtRD22的全长互补脱氧核糖核酸(complementary deoxyribonucleic acid,cDNA),分别构建AtRD22的原核重组表达载体pET32a-RD22和pSUMO-RD22,并转化大肠杆菌BL21(DE3)进行AtRD22蛋白的表达.在0.3 mmol/L的异丙基-β-D-硫代半乳糖苷(isopropyl-β-dithiogalactopyr-anoside,IPTG)诱导下,探索不同培养温度和诱导时间对可溶性蛋白表达的影响.结果表明,在实验条件下,转化pET32a-RD22的重组菌表达AtRD22蛋白的总量高于转化pSUMO-RD22的重组菌,但后者的可溶性AtRD22蛋白表达量明显高于前者.当诱导温度为28或16℃,时间分别为6.0或8.0h以上时,转化pSUMO-RD22的重组菌中可溶性目的蛋白的表达量相对较大.为进一步在体外研究AtRD22蛋白质的结构和功能奠定了基础.【期刊名称】《深圳大学学报(理工版)》【年(卷),期】2015(032)006【总页数】7页(P610-616)【关键词】蛋白质工程;拟南芥AtRD22;融合标签;小泛素相关修饰物;基因克隆;原核表达【作者】唐玉林;米子岚;钟活权;江年琼【作者单位】深圳大学生命与海洋科学学院,深圳518060;深圳市海洋生物资源与生态环境重点实验室,深圳518060;深圳大学生命与海洋科学学院,深圳518060;深圳市微生物基因工程重点实验室,深圳518060;深圳大学生命与海洋科学学院,深圳518060;深圳市微生物基因工程重点实验室,深圳518060【正文语种】中文【中图分类】Q943.2;Q786来自拟南芥的一个脱水应答基因(the Arabidopisis thaliana dehydration-responsive gene RD22,AtRD22)的cDNA中有1 179碱基对(base pair,bp)的核苷酸,编码一个含392个氨基酸残基的蛋白,该蛋白属于BURP蛋白家族.BURP蛋白最初是由Hattori等定义的一类在C-端具有保守的BURP结构域的蛋白,其命名取自于4个具有代表性成员:①油菜花粉粒胚胎发生时表达的一种蛋白(microscope-drived embryo from Brassica napus,BNM2);②蚕豆种子中丰度非贮存蛋白(abundant non-storage seed proteins from Vicia faba,USPs);③拟南芥中的一种受干旱诱导的蛋白(dehydration-responsive protein from Arabidopsis thaliana,RD22)[1];④番茄果实成熟时表达的多聚半乳糖醛酸酶Ⅰ的β亚基(β-subunit of polygalacturonase isozyme I from Lycopersicon esculentum,PG1β)[2].BURP 蛋白家族是植物所特有的一类蛋白,已有研究表明,它们在植物的生殖发育、果实成熟以及植物抵抗生物和非生物胁迫中发挥重要功能[3-6].AtRD22蛋白是BURP蛋白家族的典型成员之一,其基本结构如图1.在AtRD22蛋白的BURP结构域中存在较高比例的半胱氨酸、组氨酸和4个保守的半胱氨酸-组氨酸基序,这些氨基酸可能与二硫键的形成有关,也可能具有与过渡金属离子结合的潜力,推测其对蛋白结构的维持和功能的发挥有重要作用,但目前尚未得到验证,因此,通过体外表达获得具有活性的AtRD22蛋白对于研究该蛋白的功能具有重要意义.然而,外源蛋白在大肠杆菌(Escherichiacoli,E.coli)中高水平表达时,新生肽链的聚集速率一旦超过蛋白正确折叠的速率就会导致包涵体的形成[7].如果重组蛋白含有二硫键,而在E.coli体内,由于还原性的环境不利于正确的二硫键的形成,导致重组蛋白链间的错配,也容易导致包涵体的形成.AtRD22蛋白的氨基酸序列中较高比例的半胱氨酸、组氨酸是与二硫键形成有关的氨基酸残基,这可能是该蛋白在E.coli中表达时容易形成包涵体的原因之一,而通过蛋白可溶性分析软件(/)预测该蛋白的可溶性为 0.因此,如何在体外获得可溶性的AtRD22蛋白是进一步研究该蛋白结构和功能急需解决的技术瓶颈.图1 AtRD22蛋白的基本结构示意图Fig.1 Protein structure diagram ofAtRD22融合标签能够在蛋白质的折叠过程中起作用,从而增加重组蛋白质的可溶性表达[8].pET32a载体是一种原核表达载体,利用该载体在E.coli中表达的融合蛋白含有硫氧原还蛋白A(thioredoxin A,TrxA)标签,有研究认为,TrxA具有提高重组蛋白溶解性的能力[9].pSUMO载体是由pET28a改造而来的原核表达载体,它是将一段小泛素相关修饰物(small ubiquitin-related modifier,SUMO)基因插入在pET28a载体的多克隆位点中,编码一个约100个氨基酸残基的小分子泛素样修饰蛋白.利用pSUMO载体在E.coli中表达的融合蛋白含有SUMO融合标签,该标签作为重组蛋白质表达的融合标签和分子伴侣,具有抗蛋白酶水解、提高重组蛋白质可溶性表达等功能[10-11].为了获得AtRD22蛋白以研究其结构与功能,本研究利用反转录聚合酶链式反应(reverse transcription-polymerase chain reaction,RT-PCR)的方法先从拟南芥总核糖核酸(ribonucleic acid,RNA)中克隆到了AtRD22全长互补脱氧核糖核酸(complementary deoxyribonucleic acid,cDNA),再分别克隆到原核重组表达载体pET32a-RD22及pSUMO-RD22中,转化E.coli进行蛋白表达.通过一系列条件的优化,找出获得可溶性AtRD22蛋白质的最佳条件并鉴定了融合蛋白的表达.1 材料与方法1.1 实验材料1.1.1 菌株与载体质粒pET32a由本实验室保存;pSUMO由美国加州大学陈雪梅院士惠赠;E.coli Top10用于基因克隆;BL21(DE3)为融合蛋白表达宿主.1.1.2 植物材料哥伦比亚型(Columbia,Col.)拟南芥.1.1.3 主要试剂Trizol试剂(Invitrogen公司);逆转录试剂盒、EcoRⅠ、NotⅠ、PremeSTAR、T4 DNA连接酶和DNA分子质量标记(Takara公司);聚合酶链式反应(polymerase chain reaction,PCR)纯化试剂盒、琼脂糖凝胶DNA回收试剂盒和质粒小量提取试剂盒(Omega公司);卡那霉素(北京鼎国公司);蛋白分子质量标记(Fermentas公司);His-tag小鼠单克隆抗体、兔抗小鼠二抗(Abcam公司);WTAB显色试剂盒(上海生工).1.2 实验方法1.2.1 拟南芥总RNA提取与cDNA制备取生长周期为1个月左右的拟南芥叶片2~3片(约100 mg),采用经典 Trizol法[12]提取 RNA,并利用PrimeScriptTMRT-PCR试剂盒反转录合成cDNA. 1.2.2 引物设计与合成根据美国国立生物技术信息中心(National Center for Biotechnology Information,NCBI)登录的AtRD22基因序列,由软件Primer Premier 5.0分析并设计聚合酶链式反应(polymerase chain reaction,PCR)引物(表1).表1 实验引物汇总表Table1 List of primers构建pET32a-RD22载体 F1 CTGGATCCAATGGCGATTCGTCTTCCTC BamHⅠ构建pET32a-RD22载体 R1 ACGAGCTCATCTAGTAGCTGAACCACAC SacⅠ构建pSUMO-RD22载体 F2 CTGGATCCAATGGCGATTCGTCTTCCTC EcoRⅠ构建pSUMO-RD22载体 R2 GATGCGGCCGCCTAGTAGCTGAACCACACAAC NotⅠ1.2.3 载体构建及鉴定1)pET32a-RD22重组载体的构建和鉴定.以拟南芥(Col.)cDNA为模版,以 F1和R1为引物,PCR扩增出AtRD22基因的开放阅读框,将PCR产物和pET32a空载体用BamHⅠ和SacⅠ双酶切后用T4连接酶连接,再转化E.coli Top10,之后在含氨苄青霉素(10 mg/L)的LB平板过夜培养后对阳性单克隆依次进行菌落PCR和双酶切鉴定,最后进行测序验证.2)pSUMO-RD22重组载体的构建和鉴定.以pET32a-RD22重组载体为模版,F2和R2为引物,扩增AtRD22基因的开放阅读框,将PCR扩增产物和pSUMO空载体分别用EcoRⅠ和NotⅠ双酶切后,再用T4连接酶连接,转化E.coli Top 10,之后在含硫酸卡那霉素(10 mg/L)的LB平板培养过夜,对阳性单克隆进行菌液PCR和双酶切鉴定,最后进行测序验证.1.2.4 重组质粒的原核可溶性表达条件筛选将测序成功的重组质粒pET32a-RD22和pSUMO-RD22分别转入E.coliBL21(DE3)感受态细胞中,挑取单克隆,在37℃ 的LB液体培养基中经2次扩大培养,至光密度(optical density,OD)D(600)值约为 0.8时,加入终浓度为 0.3 mmol/L的异丙基-β-D-硫代半乳糖苷(isopropyl-β-Dithiogalactopyranoside,IPTG),分别在以下条件下继续诱导培养,在不同时间点分别测定菌液的D(600)值,并取样检测蛋白的表达.pET32a-RD22的诱导条件:37 ℃ 分别诱导 0、0.5、1.0、1.5 和2.0 h;28 ℃ 分别诱导 2.0、4.0 和8.0 h;16 ℃ 分别诱导 6.0、8.0 和12.0h.pSUMO-RD22的诱导条件:37℃分别诱导1.0、2.0和3.0 h;28℃分别诱导2.0、4.0 和8.0 h;16 ℃ 分别诱导6.0、8.0和12.0 h.检测方法:将菌液离心后,根据菌液的D(600)值加入相应体积的平衡缓冲液(50 mmol/L Tris-HCl,200 mmol/L NaCl, pH=8.0)重悬菌体,使样品的菌体浓度一致,各样品留取20 μL用于总蛋白检测,其余样品进行超声破碎后离心取上清,获得各诱导条件下的可溶性蛋白,进行十二烷基硫酸钠聚丙烯酰胺凝胶电泳(dodecyl sulfate,sodium salt-polyacrylamide gel electrophoresis,SDS-PAGE).1.2.5 利用蛋白质免疫印迹鉴定表达的融合蛋白SDS-PAGE电泳后,采用湿法转移法将蛋白质转移至硝酸纤维素膜上,5%脱脂奶粉室温封闭2.0 h后,用鼠抗6×组氨酸抗体4℃ 孵育过夜,然后用洗膜缓冲液(20 mmol/L Tris-HCl,150 mmol/L NaCl,体积分数为0.05%的Tween20)洗膜3次,每次5 min,加入2 000倍稀释的兔抗鼠免疫球蛋白G(immunoglobulin G,IgG),室温孵育2.0 h,洗膜缓冲液洗涤3次,每次5 min,最后用W-TAB试剂盒显色.1.2.6 质谱鉴定表达的目的蛋白将诱导后的蛋白进行SDS-PAGE电泳,考马斯亮蓝染色脱色后,挖取特异性条带部分经胶内酶解[13]后,采用高分辨质谱仪 AB SCIEX Triple TOFTM 5600检测鉴定.2 结果与分析2.1 拟南芥RNA的提取及AtRD22基因的克隆利用Trizol法提取拟南芥叶RNA后取5 μL进行非甲醛变性核酸电泳,见图2(a).RNA条带完整,无拖尾现象,表明该方法获得的RNA比较完整.提取的RNA 利用核酸分析仪测定得到RNA质量浓度为450 ng/μL,D(260)/D(280)=1.85,说明RNA的质量浓度可满足反转录的要求.将RNA反转录成cDNA后,以cDNA为模版,F1和R1为引物,PCR扩增,经琼脂糖凝胶电泳后可见一条与目的基因片段大小(1 179 bp)相符的特异性条带,见图2(b).将该PCR片段与pET32a载体连接后转化E.coli Top10,在LB平板上培养过夜.对阳性单克隆进行菌液PCR和双酶切鉴定,见图2(c)和图2(d),并经测序验证正确.以pET32a-RD22为模版,F2和R2为引物扩增出AtRD22基因的开放阅读框,将扩增产物与pSUMO空载体连接后转入E.coli Top10感受态细胞中,在LB平板上过夜培养后对阳性单克隆进行菌液PCR和双酶切鉴定,见图2(e)和图2(f),并经测序验证正确.图2 重组表达载体的构建及鉴定Fig.2 Construction and identification of recombinant expression vector2.2 融合蛋白的诱导表达图3 转化pET32a-RD22的菌中融合蛋白的诱导表达情况Fig.3 (Color online)The expression of fusion protein using pET32-RD22为建立适宜的AtRD22蛋白的原核表达条件,研究了诱导温度、诱导时间和诱导剂浓度对目标蛋白可溶性表达的影响.结果表明诱导温度和诱导时间对目标蛋白可溶性表达影响较大,见图3和图4(蛋白质相对分子质量单位:u,1 D=1 u),而当诱导剂浓度分别为0.3、0.5和1.0 mmol/L时,可溶性目标蛋白的量几乎无变化(结果未显示),因此选取0.3 mmol/L作为本实验中的诱导剂浓度.转化了pET32a-RD22和pSUMO-RD22的重组 E.coli,经不同条件培养和诱导后,总蛋白中均分别有与各自目的蛋白分子量大小相等的特异性蛋白出现,且转化了pET32a-RD22的重组菌在被诱导1.5~12.0 h后目的条带清晰而粗厚,显示其目的蛋白的表达量较高.但可溶性蛋白中的特异性蛋白的存在情况不同.在诱导温度为37、28和16℃时,不同诱导时间后的pET32a-RD22重组菌经超声破碎获得的上清液作为可溶性蛋白,经SDS-PAGE后均未见与目的蛋白分子量大小一致的明显条带.而pSUMORD22重组菌在不同的温度和时间下诱导培养后,其表达的可溶性蛋白中均有与目的蛋白分子量大小相等的特异性蛋白条带出现;当培养温度为28℃诱导6.0 h,或16℃诱导8.0 h以上时,可溶性蛋白中的特异性条带蛋白表达量相对较高,见图4(b)和图4(c).图4 转化pSUMO-RD22的菌中融合蛋白诱导表达情况Fig.4 (Color online)The expression of fusion protein using pSUMO-RD222.3 可溶性蛋白的鉴定通过对图4的结果分析,对转化了 pSUMORD22重组载体的E.coli在优化条件下,即在IPTG浓度为0.3 mmol/L时,28℃诱导6 h,进行融合蛋白的表达.分别取总蛋白、包涵体蛋白和可溶性蛋白进行SDS-PAGE电泳后转膜进行蛋白质免疫印迹(western blot,WB)分析,一抗为小鼠抗His抗体,二抗为辣根过氧化物酶标记的兔抗小鼠IgG.结果如图5,由图5可见,在总蛋白及可溶性蛋白中均有一条约60 kD(1 D=1 u)的条带,见图5.进一步对该特异性条带进行质谱鉴定,确认该蛋白条带为拟南芥AtRD22蛋白.证明利用pSUMO-RD22载体成功表达了可溶性的SUMO-RD22融合蛋白.图5 利用WB鉴定重组菌蛋白的表达Fig.5 (Color online)Identification of fusion protein using WB3 讨论利用原核表达系统表达异源蛋白具有简便、价廉、高效等优点,已广泛用于科学研究中.但重组的外源蛋白在E.coli中高水平表达时经常导致蛋白聚集容易形成不溶的、无活性的包涵体,而包涵体蛋白虽然较易纯化,但后续的复性等工作繁琐,且不一定能得到具有功能的蛋白,所以在体外表达可溶性的蛋白将大大简化进一步的蛋白实验[7].目前,许多融合标签系统已作为实现蛋白可溶性表达和纯化常用的方法,但不同的融合标签对于提高不同蛋白溶解性及表达量的能力不同[14].TrxA是还原蛋白二硫键的催化剂,它作为融合标签可以提高蛋白的溶解性[15].AtRD22蛋白中可能含有较多的二硫键,其在原核表达中难以进行可溶性表达的原因之一可能是二硫键未能正确折叠,与TrxA融合表达后的AtRD22蛋白的大部分以包涵体形式存在,可溶性目的蛋白的量很少,这可能是由于TrxA未能促进AtRD22蛋白的正确折叠.SUMO作为N-端融合标签,能够显著提高融合蛋白的表达量[10].AtRD22具有亲水的外表面,内部是一个疏水核心,这种结构可能使它对其他不溶性的蛋白以一个类似去垢剂的物质发挥作用,能够促进蛋白的正确折叠[16],提高目标蛋白的稳定性和可溶性[10].本研究将pET32a-RD22转化E.coli后表达的融合蛋白在不同的诱导温度及诱导时间下,融合蛋白的可溶性均很低,而利用pSUMO-RD22转化E.coli表达出的融合蛋白在不同的温度及诱导时间下,融合蛋白的可溶性均相对提高.由此可知,促溶标签对于提高AtRD22蛋白溶解性的影响较大,SUMO融合标签对AtRD22蛋白的促溶作用明显优于TrxA.利用pSUMO-RD22重组载体可在E.coli中表达出可溶性程度较高的AtRD22融合蛋白.分析不同诱导温度及诱导时间对pSUMO-RD22蛋白可溶性表达的影响,结果发现,当诱导温度为37℃时,随着诱导时间的延长,可溶性目的蛋白的含量在1.0、2.0和3.0 h时没有明显改变;当诱导温度为28℃时,可溶性目的蛋白量在诱导4.0和6.0 h时要明显多于诱导2.0 h时;当诱导温度为16℃时,随着诱导时间的增加,可溶性目的蛋白量增加,在诱导的8.0和12.0 h都表现为有较高含量的可溶性目的蛋白.当诱导温度为28和16℃时,可溶性目的蛋白量均明显高于37℃时的获取量.推测当诱导温度较高时,蛋白的表达速度较快,可能使得蛋白不能够正确折叠,进而造成蛋白的可溶性降低.这些结果说明,低温诱导有利于可溶性AtRD22蛋白的表达和积累,一定的诱导时间对于提高可溶性蛋白的量是有效的.考虑到后续蛋白大量纯化的时间和效率,28℃诱导培养6.0 h将是诱导获得该蛋白的较为合适的条件.体外获得具有活性的AtRD22蛋白将有利于对该蛋白的结构和功能的认识,进而丰富对BURP蛋白家族的了解,而在E.coli中表达出可溶性的AtRD22蛋白则为这一工作奠定了基础.结语本研究构建了拟南芥AtRD22基因的原核表达载体,探索了体外表达可溶性融合蛋白的条件,结果表明,利用SUMO表达系统在诱导温度为28℃,诱导6.0 h,或16℃,诱导8.0 h以上时,可在重组菌中表达出相对含量较多的可溶性目的蛋白.参考文献 /References:[1]Yamaguchi-Shinozaki K,Shinozaki K.The plant hormone abscisic acid mediates the drought-induced expression but not the seed-specific expression of rd22,a gene responsive to dehydration stress in Arabidopsis thaliana[J].Molecular& General Genetics,1993,238(1/2):17-25.[2]Hattori J,Boutilier K A,Campagne M M L,et al.A conserved BURP domain defines a novel group of plant proteins with unusual primary structures[J].Molecular&General Genetics:MGG,1998,259(4):424-428. [3]Tang Yulin,Cao Yan,Ou Zhonghua,et al.Regulatable gene expression controlled by the promoter of Sali3-2 under different abiotic stresses[J].Journal of Shenzhen University Science and Engineering,2012,29(1):73-79.(in Chinese)唐玉林,曹雁,欧忠华,等.非生物胁迫因子对大豆Sali3-2基因的调控作用[J].深圳大学学报理工版,2012,29(1):73-79. [4]Harshavardhan V T,Son L V,Seiler C,et al.AtRD22 and AtUSPL1,members of the plant-specific BURP domain family involved in Arabidopsis thaliana drought tolerance[J].PlOS One,2014,9(10):e110065.[5]Tang Yulin,Cao Yan,Gao Zhan,et al.Expression of a vacuole-localized BURP-domain protein from soybean(SALI3-2)enhances tolerance to cadmium and copper stresses[J].PlOS One,2014,9(6):e98830. [6]Tomas Matus J,Aquea F,Espinoza C,et al.Inspection of the grapevine BURP superfamily highlights an expansion of RD22 genes with distinctive expression features in berry development and ABA-mediated stress responses [J].PLOS One,2014,9(10):e110372.[7]Sorensen H P,Mortensen K K.Soluble expression of recombinant proteins in the cytoplasm of Escherichia coli[J].Microbial Cell Factories,2005,4(1):1-8.[8]Young C L,Britton Z T,Robinson A S.Recombinant protein expression and purification:a comprehensive review of affinity tags and microbial applications[J].Biotechnology Journal,2012,7(5):620-634. [9]Dümmler A,Lawrence A M,De Marco A.Simplified screening for the detection of soluble fusion constructs expressed in E.coli using a modular set of vectors[J].Microbial Cell Factories,2005,4(34):1-10.[10]Malakhov M P,Mattern M R,Malakhova O A,et al.SUMO fusions and SUMO-specific protease for efficient expression and purification of proteins[J].Journal of Structural and Functional Genomics,2004,5(1/2):75-86.[11]Zhang Licong,Li Xiaodan,Wei Dandan,et al.Expression of plectasin in Bacillus subtilis using SUMO technology by a maltose-inducible vector[J].Journal of Industrial Microbiology& Biotechnology,2015,42(10):1369-1376.[12]Logemann J,Schell J,Willmitzer L.Improved method for the isolation of RNA from plant tissues[J].Analytical Biochemistry,1987,163(1):16-20.[13]Hu Wei,Fu Qiang,Zhu Pingchuan,et al.An optimized method of protein in-gel digestion for mass spectrometry identification [J].Journalof Southern Agriculture,2011,42(7):802-805.(in Chinese)胡炜,付强,朱平川,等.用于质谱鉴定蛋白质胶内酶解方法的优化[J].南方农业学报,2011,42(7):802-805.[14]Wu Shanshan,Zhu Yun,Chen Shanshan,et al.Progress in fusion tags and its applications in protein soluble expression [J].Chemical Industry and Engineering Progress,2014,33(4):993-998.(in Chinese)吴珊珊,朱芸,陈珊珊,等.融合标签在蛋白质可溶性表达中的应用进展[J].化工进展,2014,33(4):993-998.[15]Li Juan,Liu Wen,Xiao Lei,et al.Two strategies for efficient expression of soluble recombinant human FGF-21[J].Journal of East China Normal University Natural Science,2012(6):114-121.(in Chinese)李娟,刘雯,肖磊,等.实现人源FGF-21高效可溶性表达的两种策略[J].华东师范大学学报自然科学版,2012(6):114-121.[16]Butt T R,Edavettal S C,Hall J P,et al.SUMO fusion technology for difficult-to-express proteins[J].Protein Expression and Purification,2005,43(1):1-9.。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
pET-SUMO编号 载体名称北京华越洋生物VECT4790 pET-‐SUMOpETsumo载体基本信息载体名称: pET-‐SUMO质粒类型: 大肠杆菌表达载体高拷贝/低拷贝: 低拷贝启动子: T7 和 lacO克隆方法: TA C loning载体大小: 5643bp5' 测序引物及序列: T7 F orward3' 测序引物及序列: -‐-‐载体标签: His T ag(6x);SUMO T ag载体抗性: 卡那霉素筛选标记: -‐-‐备注: 宿主菌株BL21(DE3);IPTG诱导 稳定性: -‐-‐组成型: 诱导型病毒/非病毒: 非病毒pETsumo载体质粒图谱和多克隆位点信息pETsumo载体简介The Champion pET-‐SUMO Expression System produces the highest levels of soluble protein i n E. c oli. I t u tilizes a s mall u biquitin-‐related m odifier (SUMO) f usion, b elonging t o the growing family of ubiquitin-‐related proteins, to enhance the solubility of expressed fusion proteins. In contrast to ubiquitin, SUMO is involved in the stabilization and localization of proteins in vivo. After expression, the 11 kd SUMO moiety can be cleaved by the highly specific and active SUMO (ULP-‐1) protease at the carboxyl terminal, producing a native protein*. The Champion pET SUMO Protein and Peptide Expression System o ffers:Greatly enhanced solubility with an N-‐terminal SUMO fusionHighly efficient cleavage-‐ produces n ative p rotein o f i nterest w ith S UMO (ULP-‐1) p rotease*Highly s pecific c leavage-‐ eliminates the chance of your protein of interest being internally digested, regardless of its a mino a cid s equenceSignificantly i ncreased s tability w ith S UMO f usion-‐can b e u sed f or small peptide productionT7lac promoter for high-‐level protein expressionN-‐terminal 6xHis t ag f or p rotein d etection a nd p urification.pETsumo载体序列CAAGGAGATGGCGCCCAACAGTCCCCCGGCCACGGGGCCTGCCACCATACCCACGCCGAAACAAGCGCTC ATGAGCCCGAAGTGGCGAGCCCGATCTTCCCCATCGGTGATGTCGGCGATATAGGCGCCAGCAACCGCAC CTGTGGCGCCGGTGATGCCGGCCACGATGCGTCCGGCGTAGAGGATCGAGATCTCGATCCCGCGAAATTA ATACGACTCACTATAGGGGAATTGTGAGCGGATAACAATTCCCCTCTAGAAATAATTTTGTTTAACTTTA AGAAGGAGATATACATATGGGCAGCAGCCATCATCATCATCATCACGGCAGCGGCCTGGTGCCGCGCGGC AGCGCTAGCATGTCGGACTCAGAAGTCAATCAAGAAGCTAAGCCAGAGGTCAAGCCAGAAGTCAAGCCTG AGACTCACATCAATTTAAAGGTGTCCGATGGATCTTCAGAGATCTTCTTCAAGATCAAAAAGACCACTCC TTTAAGAAGGCTGATGGAAGCGTTCGCTAAAAGACAGGGTAAGGAAATGGACTCCTTAAGATTCTTGTAC GACGGTATTAGAATTCAAGCTGATCAGACCCCTGAAGATTTGGACATGGAGGATAACGATATTATTGAGG CTCACAGAGAACAGATTGGTGGTAGACAAGCTTAGGTATTTATTCGGCGCAAAGTGCGTCGGGTGATGCT GCCAACTTAGTCGAGCACCACCACCACCACCACTGAGATCCGGCTGCTAACAAAGCCCGAAAGGAAGCTG AGTTGGCTGCTGCCACCGCTGAGCAATAACTAGCATAACCCCTTGGGGCCTCTAAACGGGTCTTGAGGGG TTTTTTGCTGAAAGGAGGAACTATATCCGGATTGGCGAATGGGACGCGCCCTGTAGCGGCGCATTAAGCG CGGCGGGTGTGGTGGTTACGCGCAGCGTGACCGCTACACTTGCCAGCGCCCTAGCGCCCGCTCCTTTCGC TTTCTTCCCTTCCTTTCTCGCCACGTTCGCCGGCTTTCCCCGTCAAGCTCTAAATCGGGGGCTCCCTTTA GGGTTCCGATTTAGTGCTTTACGGCACCTCGACCCCAAAAAACTTGATTAGGGTGATGGTTCACGTAGTG GGCCATCGCCCTGATAGACGGTTTTTCGCCCTTTGACGTTGGAGTCCACGTTCTTTAATAGTGGACTCTT GTTCCAAACTGGAACAACACTCAACCCTATCTCGGTCTATTCTTTTGATTTATAAGGGATTTTGCCGATT TCGGCCTATTGGTTAAAAAATGAGCTGATTTAACAAAAATTTAACGCGAATTTTAACAAAATATTAACGC TTACAATTTAGGTGGCACTTTTCGGGGAAATGTGCGCGGAACCCCTATTTGTTTATTTTTCTAAATACAT TCAAATATGTATCCGCTCATGAATTAATTCTTAGAAAAACTCATCGAGCATCAAATGAAACTGCAATTTA TTCATATCAGGATTATCAATACCATATTTTTGAAAAAGCCGTTTCTGTAATGAAGGAGAAAACTCACCGA GGCAGTTCCATAGGATGGCAAGATCCTGGTATCGGTCTGCGATTCCGACTCGTCCAACATCAATACAACC TATTAATTTCCCCTCGTCAAAAATAAGGTTATCAAGTGAGAAATCACCATGAGTGACGACTGAATCCGGT GAGAATGGCAAAAGTTTATGCATTTCTTTCCAGACTTGTTCAACAGGCCAGCCATTACGCTCGTCATCAA AATCACTCGCATCAACCAAACCGTTATTCATTCGTGATTGCGCCTGAGCGAGACGAAATACGCGATCGCT GTTAAAAGGACAATTACAAACAGGAATCGAATGCAACCGGCGCAGGAACACTGCCAGCGCATCAACAATA TTTTCACCTGAATCAGGATATTCTTCTAATACCTGGAATGCTGTTTTCCCGGGGATCGCAGTGGTGAGTA ACCATGCATCATCAGGAGTACGGATAAAATGCTTGATGGTCGGAAGAGGCATAAATTCCGTCAGCCAGTT TAGTCTGACCATCTCATCTGTAACATCATTGGCAACGCTACCTTTGCCATGTTTCAGAAACAACTCTGGC GCATCGGGCTTCCCATACAATCGATAGATTGTCGCACCTGATTGCCCGACATTATCGCGAGCCCATTTAT ACCCATATAAATCAGCATCCATGTTGGAATTTAATCGCGGCCTAGAGCAAGACGTTTCCCGTTGAATATG GCTCATAACACCCCTTGTATTACTGTTTATGTAAGCAGACAGTTTTATTGTTCATGACCAAAATCCCTTA ACGTGAGTTTTCGTTCCACTGAGCGTCAGACCCCGTAGAAAAGATCAAAGGATCTTCTTGAGATCCTTTT TTTCTGCGCGTAATCTGCTGCTTGCAAACAAAAAAACCACCGCTACCAGCGGTGGTTTGTTTGCCGGATC AAGAGCTACCAACTCTTTTTCCGAAGGTAACTGGCTTCAGCAGAGCGCAGATACCAAATACTGTCCTTCT AGTGTAGCCGTAGTTAGGCCACCACTTCAAGAACTCTGTAGCACCGCCTACATACCTCGCTCTGCTAATC CTGTTACCAGTGGCTGCTGCCAGTGGCGATAAGTCGTGTCTTACCGGGTTGGACTCAAGACGATAGTTAC CGGATAAGGCGCAGCGGTCGGGCTGAACGGGGGGTTCGTGCACACAGCCCAGCTTGGAGCGAACGACCTA CACCGAACTGAGATACCTACAGCGTGAGCTATGAGAAAGCGCCACGCTTCCCGAAGGGAGAAAGGCGGAC AGGTATCCGGTAAGCGGCAGGGTCGGAACAGGAGAGCGCACGAGGGAGCTTCCAGGGGGAAACGCCTGGT ATCTTTATAGTCCTGTCGGGTTTCGCCACCTCTGACTTGAGCGTCGATTTTTGTGATGCTCGTCAGGGGG GCGGAGCCTATGGAAAAACGCCAGCAACGCGGCCTTTTTACGGTTCCTGGCCTTTTGCTGGCCTTTTGCT CACATGTTCTTTCCTGCGTTATCCCCTGATTCTGTGGATAACCGTATTACCGCCTTTGAGTGAGCTGATACCGCTCGCCGCAGCCGAACGACCGAGCGCAGCGAGTCAGTGAGCGAGGAAGCGGAAGAGCGCCTGATGCG GTATTTTCTCCTTACGCATCTGTGCGGTATTTCACACCGCAATGGTGCACTCTCAGTACAATCTGCTCTG ATGCCGCATAGTTAAGCCAGTATACACTCCGCTATCGCTACGTGACTGGGTCATGGCTGCGCCCCGACAC CCGCCAACACCCGCTGACGCGCCCTGACGGGCTTGTCTGCTCCCGGCATCCGCTTACAGACAAGCTGTGA CCGTCTCCGGGAGCTGCATGTGTCAGAGGTTTTCACCGTCATCACCGAAACGCGCGAGGCAGCTGCGGTA AAGCTCATCAGCGTGGTCGTGAAGCGATTCACAGATGTCTGCCTGTTCATCCGCGTCCAGCTCGTTGAGT TTCTCCAGAAGCGTTAATGTCTGGCTTCTGATAAAGCGGGCCATGTTAAGGGCGGTTTTTTCCTGTTTGG TCACTGATGCCTCCGTGTAAGGGGGATTTCTGTTCATGGGGGTAATGATACCGATGAAACGAGAGAGGAT GCTCACGATACGGGTTACTGATGATGAACATGCCCGGTTACTGGAACGTTGTGAGGGTAAACAACTGGCG GTATGGATGCGGCGGGACCAGAGAAAAATCACTCAGGGTCAATGCCAGCGCTTCGTTAATACAGATGTAG GTGTTCCACAGGGTAGCCAGCAGCATCCTGCGATGCAGATCCGGAACATAATGGTGCAGGGCGCTGACTT CCGCGTTTCCAGACTTTACGAAACACGGAAACCGAAGACCATTCATGTTGTTGCTCAGGTCGCAGACGTT TTGCAGCAGCAGTCGCTTCACGTTCGCTCGCGTATCGGTGATTCATTCTGCTAACCAGTAAGGCAACCCC GCCAGCCTAGCCGGGTCCTCAACGACAGGAGCACGATCATGCGCACCCGTGGGGCCGCCATGCCGGCGAT AATGGCCTGCTTCTCGCCGAAACGTTTGGTGGCGGGACCAGTGACGAAGGCTTGAGCGAGGGCGTGCAAG ATTCCGAATACCGCAAGCGACAGGCCGATCATCGTCGCGCTCCAGCGAAAGCGGTCCTCGCCGAAAATGA CCCAGAGCGCTGCCGGCACCTGTCCTACGAGTTGCATGATAAAGAAGACAGTCATAAGTGCGGCGACGAT AGTCATGCCCCGCGCCCACCGGAAGGAGCTGACTGGGTTGAAGGCTCTCAAGGGCATCGGTCGAGATCCC GGTGCCTAATGAGTGAGCTAACTTACATTAATTGCGTTGCGCTCACTGCCCGCTTTCCAGTCGGGAAACC TGTCGTGCCAGCTGCATTAATGAATCGGCCAACGCGCGGGGAGAGGCGGTTTGCGTATTGGGCGCCAGGG TGGTTTTTCTTTTCACCAGTGAGACGGGCAACAGCTGATTGCCCTTCACCGCCTGGCCCTGAGAGAGTTG CAGCAAGCGGTCCACGCTGGTTTGCCCCAGCAGGCGAAAATCCTGTTTGATGGTGGTTAACGGCGGGATA TAACATGAGCTGTCTTCGGTATCGTCGTATCCCACTACCGAGATATCCGCACCAACGCGCAGCCCGGACT CGGTAATGGCGCGCATTGCGCCCAGCGCCATCTGATCGTTGGCAACCAGCATCGCAGTGGGAACGATGCC CTCATTCAGCATTTGCATGGTTTGTTGAAAACCGGACATGGCACTCCAGTCGCCTTCCCGTTCCGCTATC GGCTGAATTTGATTGCGAGTGAGATATTTATGCCAGCCAGCCAGACGCAGACGCGCCGAGACAGAACTTA ATGGGCCCGCTAACAGCGCGATTTGCTGGTGACCCAATGCGACCAGATGCTCCACGCCCAGTCGCGTACC GTCTTCATGGGAGAAAATAATACTGTTGATGGGTGTCTGGTCAGAGACATCAAGAAATAACGCCGGAACA TTAGTGCAGGCAGCTTCCACAGCAATGGCATCCTGGTCATCCAGCGGATAGTTAATGATCAGCCCACTGA CGCGTTGCGCGAGAAGATTGTGCACCGCCGCTTTACAGGCTTCGACGCCGCTTCGTTCTACCATCGACAC CACCACGCTGGCACCCAGTTGATCGGCGCGAGATTTAATCGCCGCGACAATTTGCGACGGCGCGTGCAGG GCCAGACTGGAGGTGGCAACGCCAATCAGCAACGACTGTTTGCCCGCCAGTTGTTGTGCCACGCGGTTGG GAATGTAATTCAGCTCCGCCATCGCCGCTTCCACTTTTTCCCGCGTTTTCGCAGAAACGTGGCTGGCCTG GTTCACCACGCGGGAAACGGTCTGATAAGAGACACCGGCATACTCTGCGACATCGTATAACGTTACTGGT TTCACATTCACCACCCTGAATTGACTCTCTTCCGGGCGCTATCATGCCATACCGCGAAAGGTTTTGCGCC ATTCGATGGTGTCCGGGATCTCGACGCTCTCCCTTATGCGACTCCTGCATTAGGAAGCAGCCCAGTAGTA GGTTGAGGCCGTTGAGCACCGCCGCCGCAAGGAATGGTGCATG其他大肠杆菌表达载体:pBV221 ptdTomato pET-52b(+) pAmCyan pDsRed-Express2 pBV220 pCold-GST pColdS-SUMO pCold TF pCold IV pCold III pCold IIpCold I pE-SUMO pCold-ProS2 pBAD102/D-TOPOpBAD202/D-TOPO pACYC184 pBAD/Thio-TOPO pBad/Myc-His C pBad/Myc-His B pBad/Myc-His A pBad/His C pBad/His B pBad/His A pBAD-TOPO pET-23b(+) pET-23a(+)pET-23c(+) pET-23(+) pET-12b(+) pET-12c(+)pET-12a(+) pET-11b(+) pET-11a(+) pET-11c(+) pBad24 pQE-82L pQE-81L pQE-80LpQE-32 pQE-9 pQE-16 pQE-31pQE-60 pQE-70 pQE-40 pET-51b(+)pET-50b(+) pET-49b(+) pET-48b(+) pET-47b(+)pET-26b(+) pET-32a(+) pET-21b(+) pET-22b(+)pET-14b pET-16b pET-15b pET-19bpET-20b(+) pET-21d(+) pET-21c(+) pET-21b(+)pET-21a(+) pET-24a(+) pET-24d(+) pET-25b(+)pET-27b(+) pET-28a(+) pET-30a(+) pET-42a(+)pET-43.1c(+) pET-43.1b(+) pET-43.1a(+) pET-44a(+)pET-44c(+) pET-46 EK/LIC pET-37b(+) pTrcHis2 C pTrcHis2 B pTrcHis2 A pET303/CT-His pET302/NT-His pRSET-CFP pRSET-EmGFP pRSET-BFP pGFPuvpET300/NT-DEST pET301/CT-DEST pGEM-T pBad43pGEX-4T-3 pGEX-5X-2 pBlueScript SK(+) pG-Tf2pG-KJE8 pGro7 pET-SUMO pSE380pET-17b pET102/D-TOPO pCDFDuet-1 pMAL-p5xpTf16 pET-28c(+) pBluescript II SK(+) pET-30b(+) pSUMO pProEX HTc pProEX HTb pProEX HTa pKD3 pKD13 pKD46 pTYB1pTYB2 pTWIN2 pBluescript II KS(-) pTYB12pMAL-p5e pACYCDuet-1 pEGM-11ZF(+) pEGM-7ZF(+) PinPoint Xa-3 PinPoint Xa-2 PinPoint Xa-1 pSP73pSP64 pTWIN1 pTYB11 pTXB1pET-5b(+) pBad/gIII C pBad/gIII B pBad/gIII A pET-5a(+) pMal-p4X pMal-p2G pkk223-3pkk232-8 pCYB1 pEZZ18 pBAD18pMAL-c5x pMal-p2E pMal-p2X pET-44 EK/LIC pET-43.1 EK/LIC pET-41 EK/LIC pMal-c4X pTrcHis BpET-31b(+) pET-3b(+) pET-41a(+) pGEX-3XpGEX-4T-2 pETDuet-1 pGEX-4T-1 pTrc99apET-28b(+) pET-His pALEX a,b,c pACYC177pBR322 pKD4 pKD20 pMXB10pEcoli-6xHN-GFPuv pKJE7 pRSET B pGEX-KGpGEX-2T pRSFDuet-1 pCOLADuet-1 pTrcHis C pTrcHis A pET-41b(+) pET-42b(+) pET-3a(+) pGEX-6P-3 pGEX-6P-2 pGEX-6P-1 pGEX-5X-3 pGEX-5X-1 pGEX-2TK pRSET A pMal-c2GpMal-c2E pMal-c2X pRSET C pQE-30pET-45b(+) pET-44b(+) pET-42c(+) pET-41c(+) pET-40b(+) pET-33b(+) pET-39b(+) pET-32 EK/LIC pET-32 Xa/LIC pET-32c(+) pET-32b(+) pET-30 Xa/LIC pET-30 EK/LIC pET-30c(+) pET-29c(+) pET-29b(+) pET-29a(+) pET-24c(+) pET-24b(+) pET-24(+)pET-23d(+) pET-11d(+) pBad33。
