全外显子组检测技术参数要求
全外显子测序技术原理

全外显子测序技术原理
全外显子测序技术(Whole Exome Sequencing,WES)是一种
用于测序一个个体的所有外显子的方法,它基于高通量测序技术,可高效地捕获并测序外显子组成的DNA序列。
全外显子测序技术的原理如下:
1. 样本准备:从需要测序的个体中提取DNA样本,并将其进
行适当处理,如切割成较小的片段。
2. 捕获外显子:将DNA样本与一组外显子引物(探针)混合,这些引物特异性地结合到外显子的DNA序列。
这一步是为了
捕获并富集外显子序列。
3. 清洗和纯化:对混合物进行洗涤步骤,以去除未结合的
DNA和杂质。
4. 文库构建:将捕获到的DNA片段进行核酸链扩增,并添加
适当的测序适配器,以便于后续的高通量测序。
5. 测序:使用高通量测序技术(如Illumina HiSeq或NovaSeq
平台)对文库中的DNA进行测序。
通过测序仪器发出的激光,可将测序适配器的序列逐个读取出来。
6. 数据分析:将得到的测序数据处理和分析,包括序列比对、重复序列标记、错配位点纠正等步骤。
然后利用生物信息学软件将测序读取的数据转化为DNA序列信息。
7. 变异检测和注释:对测序数据进行比对分析和变异检测,将测出的突变或基因变异与已知的基因数据库进行比对和注释,以确定哪些基因存在突变。
全外显子测序技术通过仅测序外显子而不测序整个基因组,可以大幅减少测序成本和数据存储需求。
同时,它也可以更集中地关注外显子区域,这些区域通常占据了大部分人类基因的功能变异。
因此,全外显子测序技术被广泛应用于基因变异研究、疾病诊断和个体化医学领域。
DNA的质量监测通常有两个方法

2)DNA的质量监测通常有两个方法:首先OD260/OD280比值应该在1.8左右(1.7-1.9),否则意味着DNA样品中存在大量的蛋白质或RNA污染。
其次,琼脂糖电泳分析时应主要以超螺旋条带为主。
最多不超过三条带(分别为超螺旋DNA,线性化DNA和环状DNA)。
否则意味质粒DNA的质量不高,应该重新制备。
2.限制性内切酶的活性1)限制性内切酶一般需要低温保存,而且反复的升降温过程对酶活性的损害很明显。
因而为了确保在有效期内的限制性内切酶不会失活,限制性内切酶的日常保存和使用应当很小。
2)建议购买具有保温功能的冻存盒保存限制性内切酶(-20度),而且取用限制性内切酶时,也应该使用具有保温功能的冻存盒,尽量防止酶的温度反复出现大的波动。
3.限制性内切酶的用量1)限制性内切酶的单位定义通常为:在合适的温度下,完全消化1ugDNA底物所需的酶量定义为一个单位。
2)在这个单位定义中,有几个不确定因素:首先是底物,不同的酶单位定义是选择的底物可能不同(常用的几个底物DNA包括:Lambda DNA ,AD2 DNA 和一些质粒DNA);第二个不确定因素是限制性内切酶在底物DNA上的酶切位点的个数。
由于单位定义中要求完全消化,因而底物上某个酶的酶切位点的个数的多少,就直接影响了该酶的单位定义。
3)因而,在进行酶切时,用1ul酶(一般10IU/ul)消化1ugDNA的通常做法是很不科学的,这也导致在实际工作中,大家要进行多次预实验才能确定最合适酶切条件。
4)以前,我推荐了一个在线的双酶切设计软件,double digestion designer, 可以精确地计算酶切时的限制性内切酶的用量。
使用中,能够注意到,用来进行双酶切的两个酶的用量有时竟然相差近20倍(EcoRI + NheI),而且发现,小片段PCR产物(100-500bp)进行酶切时,需要的酶量比质粒DNA酶切时用量多10倍以上。
5)该软件目前可以免费使用,用户名和密码都是test。
全外显子测序检验的临床意义与样本要求

全外显子测序检验的临床意义与样本要求在当今医学领域,全外显子测序检验正逐渐成为临床诊断和治疗中不可或缺的重要工具。
全外显子测序是一种高通量的基因组测序技术,能够对所有外显子区域进行全面的检测和分析,从而帮助医生发现患者潜在的遗传变异和突变,为疾病的诊断和治疗提供更精准的信息。
针对全外显子测序检验的临床意义和样本要求,本文将从多个角度进行探讨,并共享个人观点和理解。
一、全外显子测序检验的临床意义1. 诊断和治疗指导:全外显子测序能够为医生提供全面的遗传变异信息,帮助精准诊断疾病类型和确定治疗方案。
尤其对于罕见遗传病、癌症等复杂疾病的诊断和治疗指导具有重要意义。
2. 遗传沟通和家族风险评估:通过全外显子测序检验,可以帮助患者进行遗传沟通,评估患病风险,并为家族成员提供相关遗传信息,帮助他们进行风险评估和健康管理。
3. 个性化医学:全外显子测序检验为个性化医学提供了重要的基础数据,可以根据个体的基因组信息,制定个性化的预防、诊断和治疗方案,实现精准医疗。
二、全外显子测序检验的样本要求1. 样本类型:全外显子测序通常需要采集患者的血液样本,获取其中的DNA进行测序分析。
对于一些特定疾病或研究项目,还可能需要获取肿瘤组织样本等特定样本。
2. 样本质量:样本的质量直接影响着全外显子测序的准确性和可靠性。
在采集和保存样本时,需要注意避免血液凝块和样本污染等情况,保证样本的纯度和完整性。
3. 样本数量:通常情况下,全外显子测序需要一定数量的DNA样本才能进行测序分析。
对于不同的实验项目和测序评台,样本数量的要求可能会有所不同,需要根据具体情况进行调整。
三、个人观点和理解全外显子测序作为一种新型的基因组测序技术,对于临床诊断和治疗具有重要意义。
通过对个体基因组的全面检测,我们能够更好地了解疾病的遗传基础,为精准医学提供数据支持。
然而,在进行全外显子测序检验时,我们也需要考虑样本的要求和质量,以确保测序结果的准确性和可靠性。
全外显子基因检测报告解读

全外显子基因检测报告解读全外显子基因检测是一种最新的基因检测技术,它可以检测出所有外显子(编码蛋白质的区域)的突变情况,从而为疾病的诊断、治疗及防止健康问题提供重要的信息。
以下是一份全外显子基因检测报告的详细解读。
一、基本信息报告的第一页通常会列出基本信息,包括受检者姓名、性别、年龄、样本编号、检测日期等。
另外还会有一个简要的概述,告诉你检测的目的、方法和结论。
如果报告中涉及复杂的技术术语,可以先阅读概述来了解基本情况。
二、基因突变检测结果接下来的几页将列出所有被检测的基因以及它们的突变情况。
可能会有几百个基因需要检测,因此这部分的信息通常会分成多页,并按字母顺序排列。
对于每个基因,报告将列出它的基本信息,比如基因名称、编码的蛋白质名称、突变频率等。
然后,报告将列出每个检测到的突变,并给出它们的类型、位置和临床意义。
突变信息会根据重要性排序,首先展示疾病相关的突变,然后是与药物反应和其他健康问题相关的突变。
对于每个突变,报告还会提供相关文献的参考,你可以根据这些参考了解更多关于该突变的信息。
同时,报告还将给出每个突变的相关遗传学术语的详细解释,帮助你更好地理解突变的含义。
需要注意的是,突变不一定等同于疾病或健康问题。
对于大部分突变,它们仅是个别变异或常见变异。
而仅有少数突变才是真正的病因。
因此,在评估突变的风险时,需要综合考虑突变的类型、位置、临床意义以及与该突变相关的其他因素。
三、全基因组分析全基因组分析通常是报告的最后一个部分。
它会列出可能和受检者背景相关的健康问题和风险。
例如,基于该受检者基因组数据的预测,可能提示有高风险的健康问题,例如糖尿病、心脏病等。
此外,还会给出一些健康建议,例如建议采取特定的饮食、运动和生活方式。
需要注意的是,全基因组分析的准确性和实用性仍需进一步研究。
目前,科学家们尚未完全了解人类全部基因对健康的影响,而全基因组分析只提供了有限的信息。
因此,报告中的这部分内容应被视为参考,其结果和建议应和受检者的医生共同讨论和制定治疗计划。
外显子组测序技术在遗传疾病检测中的应用

外显子组测序技术在遗传疾病检测中的应用随着遗传学研究的深入,越来越多的遗传疾病得到了解决。
而随着科技的不断进步,人们开发出了越来越多的工具来解决遗传疾病的检测问题。
其中,外显子组测序技术已成为一项非常有效的检测手段,被广泛应用于遗传疾病的检测中。
本文将会对外显子组测序技术在遗传疾病检测中的应用进行探究,旨在为大家更好地了解这一技术。
外显子组测序技术的原理和特点外显子组测序技术(Exome Sequencing)是一种高通量测序技术,能够快速而准确地对人类基因组中存在的编码蛋白的外显子进行测序,包含人类基因组中的大约2%的基因部分,是人类基因组测序的有效手段。
外显子组测序技术是一种基于高通量测序技术的分子生物学技术,是通过将人类基因组中的编码蛋白的外显子与大量的引物反复杂交,在使得这些引物与DNA序列结合的过程中,将其扩增并进行序列测序。
拥有比全基因组测序更高的覆盖度、更快的处理速度以及更低的成本,使得外显子组测序技术成为大规模测序的主流工具之一。
外显子组测序技术不仅可以用于人类基因组的测序,同时也可以用来分析外显子,从而更好地了解人类的基因遗传机制。
外显子组测序技术的应用外显子组测序技术具有高精度和高可靠性的特点,使其在遗传疾病的诊断和基础研究领域中广泛应用。
此外,外显子组测序技术可以通过测序结果,实现遗传疾病的基因诊断和基因分型,探究基因底层的遗传机制,便于更好地分析和解决遗传疾病的治疗问题。
同时,外显子组测序技术还可以用来进行遗传病毒病例筛查、幼儿常见疾病筛查、基础分子遗传学研究、基因功能研究和人群遗传学研究等领域,具有广泛的应用前景。
外显子组测序技术的优缺点虽然外显子组测序技术在遗传疾病检测方面具有很大的优势,但是在实际应用中还存在着一些缺点,需要进行全面评估和优化。
其优点主要表现在以下几个方面:首先,外显子组测序技术可以对大量的外显子序列进行高通量测序。
与以往的Sanger测序方式相比,外显子组测序技术可以大大提高测序效率和速度。
外显子组测序技术

外显子组测序技术一、前言外显子组测序技术是一种高通量测序技术,它可以通过对人类基因组的外显子进行测序,来寻找与疾病相关的基因变异。
本文将详细介绍外显子组测序技术的原理、方法和应用。
二、原理外显子组测序技术是一种全基因组测序的变体,它只对基因组中编码蛋白质的区域(即外显子)进行测序。
这种技术可以检测到与疾病相关的单核苷酸多态性(SNP)、插入/缺失(indel)和结构变异等多种类型的突变。
三、方法1. 样品准备首先需要从患者或正常人身上提取DNA样品,并将其分离成片段。
然后使用特定的酶来切割这些片段,使其只包含编码蛋白质的区域。
2. 库制备接下来需要将这些片段连接到适当大小的DNA片段上,并添加适当的标签以便于后续处理。
这个过程称为库制备。
3. 测序完成库制备之后,需要进行高通量测序。
当前可用于外显子组测序的技术包括Illumina、Ion Torrent和Pacific Biosciences等。
4. 数据分析测序完成后,需要对数据进行处理和分析。
这个过程可以使用各种软件来完成,例如BWA、GATK和SAMtools等。
四、应用外显子组测序技术已经被广泛应用于疾病研究和临床诊断。
例如,在肿瘤学中,它可以检测到肿瘤细胞中的突变,并帮助医生选择最佳的治疗方案。
此外,它还可以用于遗传性疾病的诊断和预测。
五、优缺点1. 优点外显子组测序技术具有高通量、高灵敏度和高特异性等优点。
它可以检测到多种类型的基因变异,并且可以同时对多个样品进行分析。
2. 缺点外显子组测序技术的主要缺点是成本较高,并且需要较长的数据处理时间。
此外,由于只对编码蛋白质区域进行测序,因此无法检测到与非编码RNA相关的突变。
六、总结外显子组测序技术是一种重要的高通量测序技术,它可以用于疾病研究和临床诊断。
虽然它有一些缺点,但随着技术的不断发展,相信它将在未来得到更广泛的应用。
外显子组测序数据分析流程

外显子组测序介绍外显子(exon)是真核生物基因的一部分,包含着合成蛋白质所需要的信息。
全部外显子被称为“外显子组”(Exome)。
外显子组测序(Exome sequencing)是指利用序列捕获技术将全基因组外显子区域DNA捕捉并富集后进行高通量测序的基因组分析方法。
由于外显子组测序捕获目标区域只占人类基因组长度的约1%,因此远比进行全基因组序列测序来得更简便、经济,目标区域覆盖度也更高,便于变异检测。
该项技术可用于以下研究1)检测疾病样本中外显子区域内高风险碱基变异位点;2)配合大样本分析,确定孟德尔遗传疾病相关外显子SNP位点和基因;3)在癌症研究过程中,检测癌症样本外显子区域内的体细胞突变位点和潜在的融合基因;4)用于种群遗传学研究的大规模样本基因组分析,检测SNP位点、LD并绘制种群图谱。
我们能提供详尽的全基因组重测序数据的处理和分析服务。
如您没有标准化的数据、只需流程中的局部分析内容或要求特立独行的数据分析思路,我们亦能满足您的要求。
数据处理和分析流程图预期结果示例图示例图1 各类型SNV在样本中的个数统计。
示例图2 不同类型外显子区域上的SNV类型统计。
示例图4 融合基因预测[1]示例图4 大量样本的GWAS分析结果[2]示例图5 肿瘤样本高频率突变基因统计[3]示例图来源文献[1]. Kangaspeska, S., et al., Reanalysis of RNA-sequencing data reveals several additional fusion genes with multiple isoforms. PLoS One, 2012. 7(10): p. e48745.[2]. Craig, J.E., et al., Rapid inexpensive genome-wide association using pooled whole blood. Genome Res, 2009. 19(11): p. 2075-80.[3]. Bea, S., et al., Landscape of somatic mutations and clonal evolution in mantle cell lymphoma. Proc Natl Acad Sci U S A, 2013. 110(45): p. 18250-5.。
41. ACMG全外显子测序指南.

ACMG全外显子测序指南摘要:美国医学遗传学与基因组学学会(ACMG)以前为序列突变的解释提供了指导.1在过去十年中,随着高通量测序的出现,测序技术迅速发展。
通过采用和利用下一代测序,临床实验室正在进行基因分型,单基因,基因组,外显子,基因组,转录组和遗传疾病表观遗传学检测的不断增加的遗传检测目录。
由于复杂性增加,基因检测的这种转变伴随着序列解释的新挑战。
在这方面,ACMG于2013年召集了一个由ACMG,分子病理学协会(AMP)和美国病理学家学会的代表组成的工作组,重新审视和修订了序列突变解释的标准和准则。
该组由临床实验室主任和临床医生组成。
本报告代表ACMG,AMP和美国病理学家利益相关者联盟组成的工作组的专家意见。
这些建议主要适用于临床实验室使用的遗传检测的范围,包括基因分型,单基因,panel,外显子和基因组。
本报告建议使用具体的标准术语- “致病性”,“可能致病性”,“不确定性意义”,“可能良性”和“良性”来描述在导致孟德尔病症的基因中鉴定的突变。
此外,该建议描述了基于使用典型类型的突变证据(例如,群体数据,计算数据,功能数据,分离数据)的标准将突变分类为这五个类别的过程。
由于本报告中描述的临床基因检测的分析和解释的复杂性增加,ACMG强烈建议临床分子遗传学检测应在经过临床实验室改进修订批准的实验室进行,结果由相关职业认证的临床分子遗传学家或分子遗传病理学家或同等学科专家进行解释。
关键词:ACMG实验室指导; 临床遗传检测; 解释;报告; 序列变异术语;突变报告前言临床分子实验室正在不断增加检测的新的序列突变,因为在检测患者标本时不断发现大量与基因疾病相关的基因。
虽然一些表型与单个基因相关,但许多与多个基因相关。
我们对任何给定序列突变的临床意义的理解是循序渐进的,其范围从那些几乎肯定是疾病致病性突变到几乎肯定是良性的突变。
虽然以前的美国医学遗传学和基因组学会(ACMG)的建议提供了序列突变的解释类别和解释算法,但是这些建议没有提供定义的术语或详细的突变分类指南.1。
遗传学分析技术在疾病诊断中的应用

遗传学分析技术在疾病诊断中的应用随着现代医学技术的日益发展,遗传疾病诊断得到了越来越多的重视。
在遗传疾病的诊断中,遗传学分析技术起着至关重要的作用。
本文将介绍遗传学分析技术在疾病诊断中的应用,包括全外显子组测序、SNP芯片、FISH技术、基因芯片、PCR技术等。
一、全外显子组测序全外显子组测序是一种用于检测单个人类基因组中所有外显子的高通量测序技术。
它可以检测与遗传疾病相关的所有基因,并找出这些基因是否突变,从而提供以每个病人为基础的遗传诊断结果。
目前,全外显子组测序已被广泛运用于遗传疾病的诊断和基因修复等方面。
二、SNP芯片SNP(单核苷酸多态性)芯片是一种基于基因多态性的检测技术。
它可以同时检测数万个SNP位点,并对病人的基因组进行分析。
利用这种技术,医生们可以准确地检测出病人遗传疾病的相关基因位点是否存在突变,为遗传疾病的诊断和治疗提供重要依据。
三、FISH技术FISH技术是一种在细胞水平上分析染色体结构和基因突变的技术。
通过FISH技术,可以观察到基因位点数量的变化和染色体的重排,从而检测遗传疾病的相关基因突变。
常见的FISH技术包括Fluorescence in situ Hybridization(原位荧光杂交)和Array CGH(基因组和染色体芯片分析)等。
四、基因芯片基因芯片是由多种基因片段组成的微型芯片,它可以将大量的DNA序列固定到芯片表面上,并进行高通量的检测。
通过基因芯片技术,可以一次性检测大量的基因位点,从而快速、准确地诊断出病人的遗传疾病。
基因芯片技术广泛应用于遗传性肿瘤、遗传性视网膜病变等疾病的检测和分析。
五、PCR技术PCR(聚合酶链式反应)技术是一种检测DNA序列的传统技术。
它可以通过扩增基因片段,检测DNA序列的变化,从而为遗传疾病的诊断和治疗提供基础数据。
PCR技术已被广泛运用于基因突变的检测、DNA序列的分析和修复等方面。
总之,遗传学分析技术在疾病诊断中的应用已经得到了广泛的认可和应用。
wes标准

wes标准WES是什么?WES全称为Whole Exome Sequencing(全外显子测序),是一种高通量基因测序技术,可以快速、高效地测定一个个体的所有外显子基因。
WES的优点1. 全面性:WES可以检测所有外显子基因,涵盖了人体基因的大部分区域,相比之下,传统的基因检测只能检测特定的基因位点。
2. 高效性:WES是一种高通量的基因测序技术,可以在较短的时间内获得大量基因序列数据。
3. 准确性:WES技术在基因测序的准确性上已经得到了很大的提高,对于SNP和Indel的检测准确率已经很高。
4. 适用性:WES适用于不同的研究领域,包括生物医学研究、疾病诊断、个体化医学等。
WES的应用1. 生物医学研究:WES可以用于探索人类遗传基因的变异与疾病发生的关系,帮助人们更好地了解疾病的发生机制,为疾病的治疗提供更好的思路和方法。
2. 疾病诊断:WES可以用于寻找患者疾病的基因突变,尤其是罕见病的诊断,为病人的治疗提供有力的依据。
3. 个体化医学:WES可以对个人的基因信息进行分析,为个体化医疗提供科学的数据支持。
WES的发展趋势1. 增强数据分析的能力:WES产生的大量数据需要进行生物信息学分析,需要开发更高效、更准确的分析算法和软件。
2. 优化高通量测序平台:高通量基因测序平台在产生WES数据方面仍有改进的空间,可以更加精准和高效地测序。
3. 探索未知的生物学问题:随着WES技术的不断发展,可以用来解析更加复杂、不确定的生物学问题,同时也可以应用到更多不同的研究领域。
结语WES技术已经在疾病诊断、生物医学研究和个体化医疗等领域发挥了巨大作用,对于人类健康的研究和保障有着重要的意义。
随着技术的不断成熟和发展,WES将扮演着越来越重要的角色。
外显子组测序

346: 256-259.
[案例三] 癌症研究:外显子测序研究局限性肺腺癌瘤内异质性[14] 本研究采用多区域取样分析瘤内异质性的研究思路,对11位患者的局限性肺腺癌的48
个肿瘤样品进行了外显子测序。共鉴定出7269个体突变,其中21个是已知的与癌症相关的 基因突变,76% 的体突变及21个已知癌症基因突变中的20个都可以在同一肿瘤的所有区域 样品中检测到,表明对肿瘤的某一区域进行单次活检,以适当的深度对其测序,可以鉴别 出绝大多数突变。而前期关于肾透明细胞癌的研究结果表明,肿瘤不同区域样品的共有突 变仅占突变总数的31%~37%,说明肿瘤异质性在不同癌种间存在差异。
应用方向
孟德尔疾病研究
马布里综合症[1]:发现致病基因PIGV; 逆向性痤疮[2]:发现致病基因NCSTN; 眼皮肤白化病[3]:发现致病基因SLC24A5; 先天性肾脏和尿道畸形[4]:发现致病基因DSTYK;
复杂疾病研究
混合型低脂血症[5]:发现致病 基因ANGPTL3; 孤独症[6]:发现11 个新生突变 ……
[9] Rudin C M, Durinck S, Stawiski E W, et al. Comprehensive genomic analysis identifies SOX2 as a frequently amplified gene in small-cell lung cancer[J]. Nature Genetics, 2012, 44(10): 1111-1116.
人类外显子测序技术参数
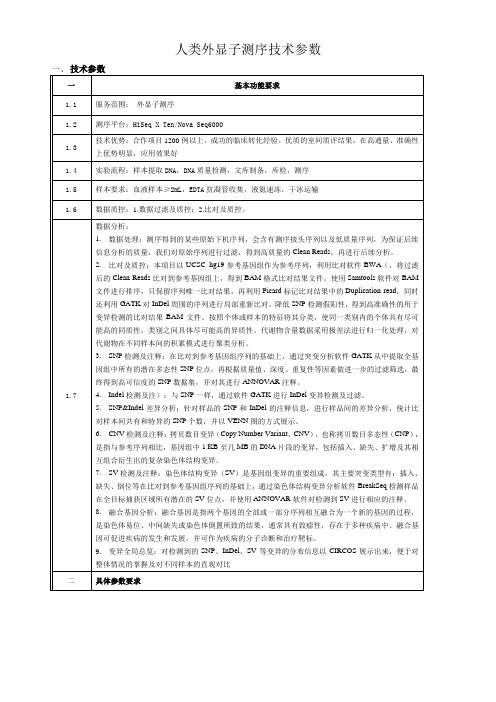
一、技术参数
一
基本功能要求
1.1
服务范围:外显子测序
1.2
测序平台:HiSeqX Ten/Nova Seq6000
1.3
技术优势:合作项目1200例以上,成功的临床转化经验,优质的室间质评结果,在高通量、准确性上优势明显,应用效果好
1.4
实.Clean Reads Rate>80%;2. Adapter polluted rate<10%;3.碱基含量分布,AT,GC不分离;4.Dup率<20%;5.捕获特异性>75%;6.基因组比对率>99%;7.基因组比对率>99%,Q30>85%。
4.数据分析内容:数据处理、比对及质控、SNP检测及注释、Indel检测及注、SNP&Indel差异分析、CNV检测及注释、SV检测及注释、融合基因分析、变异全局总览
9.变异全局总览:对检测到的SNP、InDel、SV等变异的分布信息以CIRCOS展示出来,便于对整体情况的掌握及对不同样本显子捕获系统:AgilentV6 60M
2.质量检测:1%的琼脂糖电泳检测DNA样品是否有降解以及杂质;NanoPhotometer®分光光度计检测样品纯度;Qubit® 3.0 Flurometer检测DNA样品浓度。
2.比对及质控:本项目以UCSC hg19参考基因组作为参考序列,利用比对软件BWA(,将过滤后的Clean Reads比对到参考基因组上,得到BAM格式比对结果文件。使用Samtools软件对BAM文件进行排序,只保留序列唯一比对结果,再利用Picard标记比对结果中的Duplication read,同时还利用GATK对InDel周围的序列进行局部重新比对,降低SNP检测假阳性,得到高准确性的用于变异检测的比对结果BAM文件。按照个体或样本的特征将其分类,使同一类别内的个体具有尽可能高的同质性,类别之间具体尽可能高的异质性。代谢物含量数据采用极差法进行归一化处理,对代谢物在不同样本间的积累模式进行聚类分析。
全外显子组测序的具体方法及步骤

全外显子组测序的具体方法及步骤全外显子组测序(Whole Exome Sequencing,简称WES)是一种高通量测序技术,用于测定一个个体的所有外显子区域的DNA序列。
外显子是编码蛋白质的基因组区域,占据了人类基因组的约1-2%。
WES可以用于寻找致病基因突变,特别是在遗传性疾病的分子诊断中有广泛的应用。
下面将详细介绍WES的具体方法及步骤。
1.样品准备:-提取DNA:从待测个体的外周血或组织样品中提取总DNA。
-细胞裂解:使用特定组织裂解缓冲液将细胞或组织样品裂解,释放DNA。
-纯化DNA:通过离心等步骤,去除杂质,纯化DNA。
2.外显子库建立:- 靶向捕获:使用外显子组富集探针(baits)将DNA中的外显子区域进行加权,并去除非外显子区域的DNA片段。
-杂交反应:将靶向探针与DNA样品进行杂交反应,使探针与待测DNA的外显子区域发生特异性结合。
-洗涤:将未结合的探针洗掉,保留结合的外显子区域DNA片段。
-PCR扩增:对靶向捕获得到的DNA片段进行PCR扩增,以增加样品中外显子区域的DNA原料。
3.高通量测序:-数据库构建:将PCR扩增得到的外显子DNA片段建立一个DNA文库,用于测序。
- 测序反应:使用高通量测序平台(如Illumina HiSeq X)进行DNA文库的测序,得到大量的短序列片段(reads)。
- 数据处理:通过对这些reads进行去除低质量序列、比对到参考基因组等处理,获得高质量的测序数据。
4.数据分析:- 变异检测:使用专门的变异检测软件对样品中的变异进行分析,包括单核苷酸多态性(Single Nucleotide Polymorphisms,简称SNPs)和小片段插入缺失等。
-数据解读:将检测到的变异与公开的数据库进行对比,筛选出可能与疾病相关的变异。
-功能注释:对筛选出的变异进行功能注释,评估其潜在影响,进一步缩小候选基因的范围。
- 候选基因验证:对最终候选基因进行进一步的实验验证,如Sanger测序。
全外显子测序深度计算方法

全外显子测序深度计算方法全外显子测序(Whole-exome sequencing,WES)是一种高通量测序技术,用于测序人类基因组中所有外显子区域的DNA序列。
由于外显子区域占据人类基因组的1%-2%,而包含大部分与蛋白质编码相关的功能位点,因此WES成为了研究人类遗传变异和疾病致病机理的有力工具。
在进行全外显子测序之前,需要对样品进行DNA库构建。
库构建的首要步骤是DNA片段化,将整个基因组或目标区域的DNA随机打断为约150-200 bp的片段,之后使用外显子富集技术,如环境选择性放大法(capturing)或沉降法(solution hybridization),选择片段中的外显子区域进行富集。
完成DNA库构建后,接下来进行高通量测序。
WES通常采用高通量测序平台,如Illumina HiSeq或NovaSeq等,生成数百万到数十亿条短序列(reads),每条短序列通常为100-150 bp。
测序完成后,需要对测序的reads进行质控和数据处理。
测序数据通常以FASTQ格式存储,包含每条reads的序列信息和对应的质量值。
首先,需要使用质控工具(如FastQC)对测序数据进行质量评估。
如果发现有质量较差的reads,可以使用软件工具(如Trimmomatic)进行过滤和修剪,将低质量的reads从数据集中去除。
同时,还需要移除适配体序列,避免影响后续的分析。
经过质控和数据处理后,接下来进行全外显子测序的深度计算。
全外显子测序的深度是指从目标区域得到的平均测序覆盖度,即每个碱基被测序的平均次数。
深度越高,测序数据的准确性和可靠性就越高。
计算全外显子测序的深度可以使用各种软件和工具。
以下是两种常用的深度计算方法:1.简单计数法:此方法将测序覆盖度定义为每个外显子区域中被覆盖的碱基数目的平均值。
通过统计每个外显子区域中的测序片段数目,可以计算得到测序覆盖度。
2.平均深度法:该方法将测序覆盖度定义为每个外显子区域中被测序的平均次数。
全基因组重测序和全外显子重测序技术流程

全基因组重测序和全外显子重测序技术流程全基因组重测序和全外显子重测序技术介绍•全基因组重测序和全外显子重测序是现代基因组学研究中常用的技术。
•这两种技术可以提供大量关于个体基因组的信息,有助于研究遗传变异和疾病相关基因。
全基因组重测序•全基因组重测序是指对个体的全部DNA进行测序。
•流程包括:DNA提取、文库构建、测序、数据分析。
•DNA提取:从样本中提取高质量的基因组DNA。
•文库构建:将提取的DNA进行加工处理,生成可以进行测序的文库。
•测序:采用高通量测序技术,对文库进行测序,获取序列信息。
•数据分析:对获得的序列数据进行质量控制、比对和变异检测等分析。
全外显子重测序•全外显子重测序是指对个体的外显子区域进行测序。
•外显子是编码蛋白质的基因区域。
•流程包括:DNA提取、文库构建、测序、数据分析。
•DNA提取:与全基因组重测序相同,从样本中提取高质量的基因组DNA。
•文库构建:将提取的DNA进行加工处理,生成可以进行测序的文库。
•测序:采用高通量测序技术,对文库进行测序,获取外显子序列信息。
•数据分析:对获得的外显子序列数据进行质量控制、比对和变异检测等分析。
应用领域•全基因组重测序和全外显子重测序广泛应用于人类遗传研究、疾病基因研究、个体基因组学和进化生物学等领域。
•这些技术可以帮助揭示基因组的结构和功能,发现与疾病相关的遗传变异。
结论•全基因组重测序和全外显子重测序技术的发展,为基因组学研究提供了强大的工具。
•这些技术的应用不断拓展,为理解人类和其他生物的基因组差异以及与疾病相关基因的发现提供有力支持。
一文读懂全外显子测序家系突变筛选策略

一文读懂全外显子测序家系突变筛选策略最近老师和同学经常问针对外显子测序的家系遗传病如何进行突变筛选,今天小編就撰稿一篇,希望对老师和同学有所帮助,话不多说,直接看下面的干货。
小编碎语:DNA测序中从测序区域的大小分为全基因组重测序,全外显子测序,靶向区域测序。
全外显子大概占DNA全部碱基对的1%,即大概30M的碱基,目前大部分测序的全外数据量为10G,测序深度大概为100X-150x左右,不同的试剂盒导致不同的捕获效率的不同,不同试剂盒的均一度的不同导致不同区域实际深度不同,由于全外显子测序检测数据量适中(约10G),与全基因组相比(约90G),因为人类疾病有90%在外显子区域,全外显子测序十分具有性价比,可以发现与人类疾病关系密切的外显子部分的相关基因突变。
全外显子的测序应用十分广泛,整体从技术上来说,1)可以检测 SNV 的 germ line 突变;2)也可以在一定程度上检测肿瘤的somatic 突变(深度200X以上);3)可以检测外显子区域的CNV, 融合等突变;从外显子测序技术延伸出的临床应用来说,可以应用于以下的方面:1)确定孟德尔遗传疾病相关基因;2)风险易感基因的发现(与全基因组关联分析类似);3)癌症相关研究(高深度情况下);全外显子测序的高通量分析流程如下:不同试剂盒导致捕获效率的不同,不同试剂盒均一度的区别导致不同区域实际深度差异,下表为目前市场上主流试剂盒的比较。
孟德尔遗传疾病相关研究(家系筛选)通过全外显子生物信息分析,通过初步将得到一些可能的致病突变;如果知道样本家系属于何种致病模式,可以使用不同的筛选模式进行筛选。
筛选模式有:1)常染色体隐性遗传甲、乙:隐性遗传表现为双亲都没病,孩子患病。
患病个体亲代是突变携带者但表型正常,子代患病,如果不存在近亲结婚或生殖隔离等因素,往往患者同一致病基因的不同位点存在致病突变,即患者带有复合杂合突变(compound heterozygous mutations)。
panel全外显子组与全基因组测序的全方位对比

14
捕获探针供应商
国外较大的提供杂交捕获探针库的供应商
Agilent SureSelect
Roche NimbleGen
IDT xGen™
Twist Bioscience Human Core Exome
国内
迪赢生物
15
靶向捕获与冗余数据
液相杂交捕获技术流程图
冗余数据的产生
同源序列
目标序列
同源序列干扰
panel大小约1-2M,约占全外显子的1%-2%左右
2
检测范围与测序深度
全基因组测序 全外显子组测序 不孕不育panel
30-40X 100-150X 500X以上
3
检测方案的选择
WGS
WES panel
panel和WES,临床上如何选择?
panel具有更低廉的价格,更高的测序深度 WES具有更广泛的检测范围
表型遗传异质性较高,疾病 复杂,难以区分,需要对大 量基因进行筛查,更适合 WES或WGS。
PMID: 29398702
5
检测方案的选择
神经系统疾病/发育异常类疾病
精子异常导致的男性不育
智力障碍
小儿癫痫
线粒体病
行为异常 遗传代谢病
神经肌肉病
基因数量多达2000个以上,表型异质性高,症 状难以区分,容易误诊,更适合WES/WGS
WGS包含所有碱基,可以检测 CNV和非编码序列 panel可定制检测范围,可以加 入WES不包含的目标区域
WES测序深度还不足以区分真 假基因,深度更高的panel却有 可能做到。
23
安捷伦各版本WES对比
靶标 大小
设计 大小
V8
35.1 Mb 41.6 Mb
wes全外显子测序原理

wes全外显子测序原理WES(全外显子组测序)是一种高通量测序技术,可以高效地检测人类基因组中的所有编码蛋白质的外显子区域。
WES的基本原理是使用特定的引物(半万通量、数百个碱基对)引导DNA聚合酶在DNA模板上扩增外显子区域,并将扩增片段测序获取DNA序列信息。
WES使用两个主要的步骤:DNA片段化和文库制备、测序和数据分析。
DNA片段化是WES过程中的第一步。
在这个步骤中,科学家将DNA切割成小片段,以便将其放入文库中。
这些DNA片段通常是相对较小(100-300bp)的,这样可以提高测序的覆盖率和可靠性。
在该步骤中,使用不同的酶切割,例如剪刀酶,谷氨酰胺酰化酶等。
文库制备、测序和数据分析是WES过程的第二步。
在这个步骤中,科学家将切割好的DNA片段与引物匹配,每个引物匹配一个外显子。
一些引物刻意设计为靠近外显子边缘,以提高捕获的精密度和覆盖率,使测序产生的数据内容更为丰富。
在测序过程中,DNA片段经过PCR扩增和链分析,得到大量的读取长度为50nt-300nt的测序数据。
随后,这些测序数据经过对比分析,人们可以重建出原始DNA的序列,从而识别具体基因并分析其序列。
数据分析可以使用各种软件和工具进行。
相对于基因组测序,WES具有一些优势。
首先,外显子仅占人类基因组的1%-2%,相对于基因组,WES测序时间相对较短。
其次,外显子通常比内含子更加功能性,这些区域通常是与特定疾病相关的。
WES可以检测的区域是与人类疾病之间的关联更加密切。
最后,WES通常是更经济和有效的,因为需要处理的DNA量较小。
但是,WES方法也有一些限制。
首先,WES仅限于已知的外显子和靶区域,对于一些变异位于外显子间区域或非编码序列区域,他们将无法被检测到。
其次,WES在检测某些类型的变异时,如重复序列的扩增,G-富集区域和GC含量高的区域时可能不是非常敏感。
最后,由于WES是针对外显子进行测序的,不能覆盖整个基因。
在一些研究中,必要时,研究人员需要选择其他技术或方法来检测某些特定的基因区域。
全外显子组检测技术参数要求

附件:全外显子组检测技术参数要求样本量:500个样本一、公司资质:1.拥有先进的高通量二代测序平台和高性能计算平台;2.具有短期处理大量样本,进行全外显子组和全基因组测序的经验;3.实验室具有国内或国外权威机构的资质认证;4.*应标的公司必须通过医学遗传中心选送的样本测试(三个以上生物学重复),并且需交付原始下机数据,以中心提供的标准化流程统一进行质量评估。
二、技术参数:1)污染防控具有独立的实验方法进行样本身份鉴定,可追溯样本间发生的错误2)测序质量1.Q20平均比例在90%以上。
2.Q30平均比例在85%以上。
3.GC content 分布无明显偏移。
3)测序深度、覆盖度统计下文涉及的数据均为经过去接头、比对、排序和去重后的有效数据。
数据统计涉及的相关软件除特别说明外,应使用默认参数。
1.数据质量要求:1)Mapped unique reads相对总reads的比例(PCT_PF_UQ_READS_ALIGNED)不得低于99%2)有效数据总量(PF_UQ_BASES_ALIGNED)不得低于10G3)On targeted bases相对总bases的比例(PCT_USABLE_BASES_ON_BAIT)不得低于50%4)On and near targeted bases相对总bases的比例(PCT_SELECTED_BASES)不得低于80%5)全外显子碱基10X覆盖率(PCT_TARGET_BASES_10X)不得低于95%6)全外显子碱基30X覆盖率(PCT_TARGET_BASES_30X)不得低于80%7)全外显子组各区域覆盖的一致性统计要求:80%以上的target region的normalizedcoverage值不得低于0.3。
200X有效深度肿瘤全外显子组测序

活动要求
样本数量:单个项目≥20个样本 活动价格:¥9900 /样本(建库及测序),¥100/G(基本分析) 项目数量:100个(中国大陆范围)
应用方向
高灵敏扫描瘤内低频突变
图1.高深度外显子组测序高灵敏扫描瘤内异质性
高深度外显子组测序通过高深度reads覆盖, 可以检测瘤内低频(<5%)体细胞变异(Somatic SNVs/CNAs),高灵敏扫描瘤内低频突变(Zhang et al.2014)。
ion sequencing. Science, 2014, 346(6206):256-259.
¥100/Gb
基本信息分析,低价特惠
基于模拟数据及真实数据进行检出率及一 致性评估,诺禾信息分析团队搭建了最佳的 Somatic变异检测分析流程。我们不创造变 异,我们是准确基因组变异的搬运工。
EXPANDS
肿瘤异质性与克隆进化专项 分析工具
EXPANDS等利用Somatic SNVs及CNAs作为 亚克隆区分标记,不仅可以静态绘制单个样本 瘤内亚克隆结构全景,还可以记录转移及复发 灶相比原发灶的亚克隆动态进化过程。
精准解析瘤内异质性
肿瘤内部的Somatic SNVs及CNAs均可以作 为亚克隆区分标记,利用EXPANDS(Andor et al.2013)等软件能够准确解析瘤内亚克隆结构。
描绘肿瘤内亚克隆进化路线
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
附件:全外显子组检测技术参数要求
一、公司资质:
1.拥有先进的高通量二代测序平台和高性能计算平台;
2.具有短期处理大量样本,进行全外显子组和全基因组测序的经验;
3.实验室具有国内或国外权威机构的资质认证;
4.*应标的公司必须通过医学遗传中心选送的样本测试(三个以上生物学重复),并且需交
付原始下机数据,以中心提供的标准化流程统一进行质量评估。
二、技术参数:
1)污染防控
具有独立的实验方法进行样本身份鉴定,可追溯样本间发生的错误
2)测序质量
1.Q20平均比例在90%以上。
2.Q30平均比例在85%以上。
3.GC content 分布无明显偏移。
3)测序深度、覆盖度统计
下文涉及的数据均为经过去接头、比对、排序和去重后的有效数据。
数据统计涉及的相关软件除特别说明外,应使用默认参数。
1.数据质量要求:
1)Mapped unique reads相对总reads的比例(PCT_PF_UQ_READS_ALIGNED)不得低于
99%
2)有效数据总量(PF_UQ_BASES_ALIGNED)不得低于10G
3)On targeted bases相对总bases的比例(PCT_USABLE_BASES_ON_BAIT)不得低于50%
4)On and near targeted bases相对总bases的比例(PCT_SELECTED_BASES)不得低于
80%
5)全外显子碱基10X覆盖率(PCT_TARGET_BASES_10X)不得低于95%
6)全外显子碱基30X覆盖率(PCT_TARGET_BASES_30X)不得低于80%
7)全外显子组各区域覆盖的一致性统计要求:80%以上的target region的normalized
coverage值不得低于0.3
三、项目内容:
500例耳聋患者全外显子组测序技术服务。
