illumina 转录组测序简明实验流程(PE-oligodT NEB)
转录组测序 步骤 流程

转录组测序步骤流程Transcriptome sequencing, also known as RNA-Seq, is a powerful technique used to study the entire set of RNA molecules transcribed by an organism. It provides valuable insights into gene expression levels, alternative splicing, and novel transcript discovery. The process oftranscriptome sequencing involves several steps, which Iwill explain in detail.Firstly, the RNA molecules need to be isolated from the biological sample of interest. This can be done using various methods, such as using a commercial RNA extraction kit or employing a phenol-chloroform extraction protocol. The quality and integrity of the RNA are crucial at this stage to ensure accurate downstream analysis.Once the RNA is extracted, it needs to be convertedinto complementary DNA (cDNA) through reverse transcription. This step involves the use of reverse transcriptase enzyme and random primers or oligo(dT) primers. The cDNA synthesisallows for the amplification of RNA molecules and reduces the presence of ribosomal RNA, which is highly abundant and can hinder sequencing efficiency.Next, the cDNA library preparation is carried out. This involves fragmentation of the cDNA and the addition of sequencing adapters, which contain barcode sequences for sample identification. The fragmented cDNA is then subjected to size selection to obtain the desired insert size range for sequencing. This step ensures that only fragments within the desired range are sequenced, improving the quality of the data obtained.After library preparation, the sequencing step takes place. This can be done using various sequencing platforms, such as Illumina, Ion Torrent, or PacBio. The choice of platform depends on factors like sequencing depth, read length, and cost considerations. During sequencing, the cDNA fragments are amplified on a solid support and undergo cycles of nucleotide incorporation, resulting in the generation of millions of short reads.Once the sequencing is complete, the generated reads need to be processed and aligned to a reference genome or transcriptome. This step involves bioinformatics analysis, where the reads are mapped to the reference using alignment algorithms. This allows for the identification of known transcripts and the discovery of novel transcripts.Following alignment, differential gene expression analysis can be performed to compare gene expression levels between different samples or conditions. This analysis involves statistical methods to identify genes that are significantly upregulated or downregulated. It provides valuable insights into the biological processes and pathways that are affected under different conditions.In conclusion, transcriptome sequencing is a multi-step process that involves RNA extraction, cDNA synthesis, library preparation, sequencing, alignment, and analysis.It provides a comprehensive view of gene expression and transcriptome dynamics. By understanding the steps involved in transcriptome sequencing, researchers can gain valuable insights into gene regulation and biological processes.转录组测序,也被称为RNA-Seq,是一种用于研究生物体转录的所有RNA分子的强大技术。
illumina转录组测序简明实验流程(PE-oligodTNEB)
illumina转录组测序简明实验流程(PE-oligodTNEB)illumina 转录组测序简明实验流程一、实验基本流程图mRNA Library Construction二、mRNA建库流程1.材料准备1.2.1.3.2.样品准备和QC选择质量合格的Total RNA作为mRNA测序的建库起始样品,其质量要求通过Agilent 2100 BioAnalyzer检测结果RIN≥7,28S和18S的RNA 的比值大于或等于1.5:1,起始量的要求范围是0.1∽1ug。
用QUBIT RNA ASSAY KIT对起始的Total RNA进行准确定量。
3.建库实验步骤3.1.mRNA纯化和片段化3.1.1.mRNA纯化纯化原理是用带有Oligod(T)的Beads对Total RNA中mRNA进行纯化。
3.1.2.mRNA片段化3.2.1st Strand cDNA 合成3.3.2nd Strand cDNA 合成根据下表制备反应体系,然后在PCR仪上运行Program3,然后将第2链cDNA合成产物用144uL AMPure XP Beads进行纯化,最后用60μL的Nuclease free water进行重悬,取出55.5μL以备下一步使用;3.4.Perform End Repair/dA-tail3.5.Adaptor Ligation根据下表制备反应体系,然后在PCR仪上运行Program5、Program6,然后100uL AMPure XP Beads进行纯化后用52.5μL的Resuspension Buffer进行重悬,再用50uL AMPure XP Beads3.6.PCR扩增根据下表制备反应体系,然后在PCR仪上运行Program7,然后再45μL用AMPure XP Beads 进行纯化,最后用23μL的Resuspension Buffer进行重悬,取出20μL以备下一步使用;3.7. PCR 产物质控用QUBIT DNA HS ASSAY KIT 对PCR 产物进行准确定量。
安捷伦二代测序操作方法

安捷伦二代测序操作方法安捷伦二代测序(Illumina sequencing)是一种高通量测序技术,也称为第二代测序技术。
它是目前最常用的测序技术之一,具有高效、高精度和经济实惠的特点。
下面将详细介绍安捷伦二代测序的操作方法。
安捷伦二代测序的操作主要包括样品准备、文库构建、测序芯片负载和测序运行、数据分析等步骤。
首先是样品准备。
样品可以是DNA、RNA或其它核酸。
如果是DNA样品,首先需要进行DNA提取和纯化。
对于RNA样品,一般需要进行RNA提取和转录成cDNA。
提取过程需要注意样品的完整性和纯度,以保证后续步骤的准确性和可靠性。
接下来是文库构建。
文库是指将样品DNA或RNA片段连接到测序适配体上的过程。
适配体是一种DNA片段,其中包含引物序列和测序平台特异性序列。
文库构建可以为无法测序的DNA或RNA片段增加引物序列,并通过PCR扩增获得足够的文库量。
文库构建的关键是要控制适配体和样品DNA的比例,以避免过度扩增或不足。
完成文库构建后,可以将样品的文库片段装载到测序芯片上。
测序芯片是一种固定了上千万个DNA克隆簇的玻璃或硅片。
每个克隆簇都含有相同的文库片段,在测序过程中可以通过化学或光学方法进行扩增和测序。
装载完成后,测序芯片被放入测序仪中进行测序运行。
安捷伦二代测序采用的是桥式扩增法(bridge amplification),即在每个克隆簇上通过引物的控制使得DNA扩增成桥状结构,然后进行测序。
测序过程中使用碱基特异性荧光探针,通过测量碱基的荧光强度来确定碱基序列。
测序结束后,得到的原始测序图像数据需要进行图像处理和碱基识别。
图像处理包括去除背景噪音和探针交叉干扰等步骤,以提高测序数据的质量。
碱基识别则是根据荧光信号的强度和位置信息,将测序图像转化为碱基序列。
得到碱基序列后,可以进行测序数据的分析。
常见的分析包括比对参考基因组、寻找变异位点、计算基因表达水平等。
这些分析需要结合相关的生物信息学软件和数据库进行。
转录组测序 步骤 流程

转录组测序步骤流程英文回答:Transcriptome sequencing, also known as RNA sequencing (RNA-seq), is a powerful technique used to study the transcriptome of an organism. The process involves several steps that are essential for obtaining accurate andreliable results.Firstly, the RNA molecules are extracted from the cells or tissues of interest. This step is crucial as it ensures that the RNA represents the gene expression profile of the sample. Various methods can be used for RNA extraction, such as phenol-chloroform extraction or commercial kits.Once the RNA is extracted, it needs to be purified to remove any contaminants, such as genomic DNA or proteins. This purification step is important to ensure that the sequencing reads obtained are specific to the RNA molecules and not from other sources.Next, the purified RNA is converted into complementary DNA (cDNA) through a process called reverse transcription. This step involves the use of reverse transcriptase enzyme to synthesize cDNA from the RNA template. The cDNA represents a copy of the RNA molecules and can be used for sequencing.After obtaining the cDNA, it is then fragmented into smaller pieces to facilitate sequencing. This fragmentation can be achieved through physical methods, such as sonication or enzymatic methods, such as restriction enzyme digestion. The fragmented cDNA is then ready for sequencing library preparation.Library preparation involves adding specific adapters to the fragmented cDNA molecules. These adapters contain sequences that are recognized by the sequencing platform and allow for the attachment of the cDNA fragments to the sequencing flow cell. This step is crucial for the subsequent sequencing process.Once the library is prepared, it is loaded onto the sequencing platform, such as Illumina or PacBio. The sequencing process generates millions of short reads or long reads, depending on the platform used. These reads represent fragments of the cDNA molecules and are used to reconstruct the original RNA sequences.After sequencing, the reads are aligned to a reference genome or transcriptome to determine their origin and quantify gene expression levels. This step involves bioinformatics analysis, where specialized software tools are used to process the sequencing data and generate meaningful results.Finally, the results of the transcriptome sequencing experiment can be interpreted to gain insights into gene expression patterns, alternative splicing events, and other transcriptomic features. This information can be used to study gene function, identify biomarkers, or understand disease mechanisms.中文回答:转录组测序,也被称为RNA测序(RNA-seq),是一种用于研究生物体转录组的强大技术。
简述illumina测序原理和流程

简述illumina测序原理和流程下载温馨提示:该文档是我店铺精心编制而成,希望大家下载以后,能够帮助大家解决实际的问题。
文档下载后可定制随意修改,请根据实际需要进行相应的调整和使用,谢谢!并且,本店铺为大家提供各种各样类型的实用资料,如教育随笔、日记赏析、句子摘抄、古诗大全、经典美文、话题作文、工作总结、词语解析、文案摘录、其他资料等等,如想了解不同资料格式和写法,敬请关注!Download tips: This document is carefully compiled by theeditor. I hope that after you download them,they can help yousolve practical problems. The document can be customized andmodified after downloading,please adjust and use it according toactual needs, thank you! In addition, our shop provides you with various types ofpractical materials,such as educational essays, diaryappreciation,sentence excerpts,ancient poems,classic articles,topic composition,work summary,word parsing,copy excerpts,other materials and so on,want to know different data formats andwriting methods,please pay attention! Illumina测序是一种广泛应用的高通量测序技术,其原理和流程可以概括如下:原理:Illumina测序基于“边合成边测序”(Sequencing by Synthesis, SBS)的原理,利用荧光标记的可逆终止脱氧核苷三磷酸(dNTPs)在DNA合成过程中实现序列的测定。
illumina 转录组测序简明实验流程(PE-oligodT NEB)
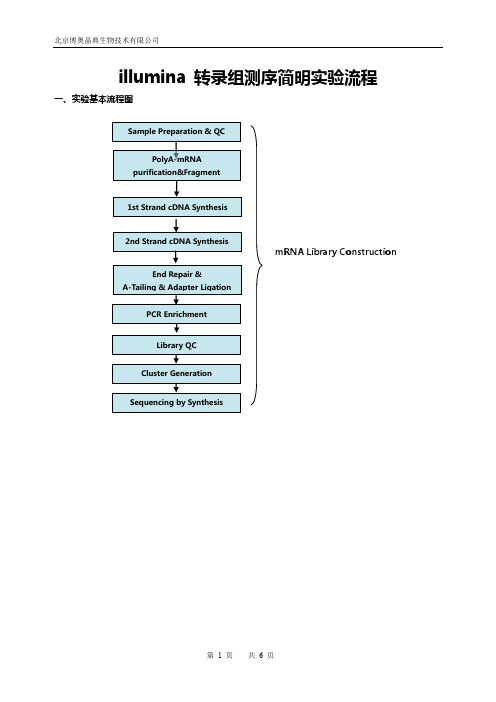
illumina 转录组测序简明实验流程一、实验基本流程图mRNA Library Construction二、mRNA建库流程1.材料准备1.2.1.3.2.样品准备和QC选择质量合格的Total RNA作为mRNA测序的建库起始样品,其质量要求通过Agilent 2100 BioAnalyzer检测结果RIN≥7,28S和18S的RNA 的比值大于或等于1.5:1,起始量的要求范围是0.1∽1ug。
用QUBIT RNA ASSAY KIT对起始的Total RNA进行准确定量。
3.建库实验步骤3.1.mRNA纯化和片段化3.1.1.mRNA纯化纯化原理是用带有Oligod(T)的Beads对Total RNA中mRNA进行纯化。
3.1.2.mRNA片段化3.2.1st Strand cDNA 合成3.3.2nd Strand cDNA 合成根据下表制备反应体系,然后在PCR仪上运行Program3,然后将第2链cDNA合成产物用144uL AMPure XP Beads进行纯化,最后用60µL的Nuclease free water进行重悬,取出55.5µL以备下一步使用;3.4.Perform End Repair/dA-tail3.5.Adaptor Ligation根据下表制备反应体系,然后在PCR仪上运行Program5、Program6,然后100uL AMPure XP Beads进行纯化后用52.5µL的Resuspension Buffer进行重悬,再用50uL AMPure XP Beads3.6.PCR扩增根据下表制备反应体系,然后在PCR仪上运行Program7,然后再45µL用AMPure XP Beads 进行纯化,最后用23µL的Resuspension Buffer进行重悬,取出20µL以备下一步使用;3.7. PCR 产物质控用QUBIT DNA HS ASSAY KIT 对PCR 产物进行准确定量。
转录组测序的流程

转录组测序的流程转录组测序是一种用于研究RNA转录本的高通量测序技术,它可以帮助科研人员了解生物体内部的基因表达情况,从而揭示基因调控、代谢途径等重要生物学过程。
本文将介绍转录组测序的流程,包括样本准备、RNA提取、建库、测序和数据分析等步骤。
1. 样本准备。
转录组测序的第一步是样本准备,样本的选择和处理对后续的实验结果至关重要。
首先需要确定研究的对象,是细胞、组织还是整个生物体,然后采集样本并进行保存。
在采集样本的过程中,需要注意避免RNA的降解和污染,可以使用RNAlater等试剂来稳定RNA。
此外,还需要记录样本的相关信息,如采集时间、处理方法等。
2. RNA提取。
RNA提取是转录组测序的关键步骤,它可以从样本中纯化出RNA,并去除DNA、蛋白质和其他杂质。
常用的RNA提取方法包括酚/氯仿法、硅胶柱法和磁珠法等。
在进行RNA提取时,需要注意保持样本的完整性和纯度,避免外源性RNA的污染。
此外,还需要对提取得到的RNA进行定量和质量检测,确保其可以用于后续的实验。
3. 建库。
建库是将提取得到的RNA转录本转化为可以进行测序的DNA文库的过程。
建库的关键步骤包括RNA的反转录、cDNA合成、末端修复、连接接头、文库扩增和纯化等。
在建库的过程中,需要注意避免外源DNA的污染,确保文库的纯度和完整性。
此外,还需要对建库得到的DNA文库进行定量和质量检测,以确保其可以用于高通量测序。
4. 测序。
建库完成后,就可以进行高通量测序了。
目前常用的转录组测序技术包括RNA-seq和全长转录组测序。
RNA-seq可以对RNA转录本进行定量和差异表达分析,全长转录组测序可以获取RNA的全长序列信息。
在进行测序时,需要选择合适的测序平台和测序深度,确保可以获得足够的数据量用于后续的数据分析。
5. 数据分析。
测序数据的分析是转录组测序的最后一步,它包括数据的质控、比对、表达定量和差异分析等。
在进行数据分析时,需要选择合适的分析软件和算法,对数据进行准确的处理和解释。
转录组测序与单细胞测序的实验流程

转录组测序与单细胞测序的实验流程转录组测序实验流程包括样品提取、RNA提取和纯化、RNA测序文库构建、测序、数据分析和生物信息学分析等步骤。
The experimental process of transcriptome sequencing includes sample extraction, RNA extraction and purification, RNA sequencing library construction, sequencing, data analysis, and bioinformatics analysis.首先,样品提取需要选择合适的组织或细胞,并使用合适的方法来提取RNA。
First, sample extraction requires selecting appropriate tissues or cells and using suitable methods to extract RNA.其次,RNA提取和纯化需要使用RNA提取试剂盒来提取总RNA,然后通过反转录酶将RNA转录成cDNA,最后利用PCR扩增和纯化得到RNA测序文库。
Second, RNA extraction and purification require using RNA extraction kits to extract total RNA, then transcribing RNA into cDNA using reverse transcriptase, and finally obtaining RNA sequencing libraries through PCR amplification and purification.接下来,测序是将RNA测序文库进行高通量测序,通常使用Illumina测序技术。
Next, sequencing is the high-throughput sequencing of RNA sequencing libraries, usually using Illumina sequencing technology.然后,得到的测序数据需要进行质量控制、比对、基因表达量分析、差异表达基因分析等生物信息学分析。
Illumina 测序的原理和应用

8
2021/6/20
8
5. Double-strand bridge is denatured and form two copies of covalently bound single-stranded templates 6. Bridge amplification cycle repeat
18
高通量测序的应用
DNA水平 RNA水平
19
2021/6/20
基因组denovo、重测序 外显子捕获测序 DNA甲基化测序 Chip-Seq 16s、宏基因组
转录组测序 数字表达谱DGE Small RNA lncRNA
19
高通量测序的应用
DNA
Chip-seq
甲基化
RNA
真核 原核
无参 有参
11
2021/6/20
11
Illumina测序流程
eneration 簇生成
序
流
程
Sequencing
测序
Data Analysis 数据分析
12
2021/6/20
12
Two flow cell
13
2021/6/20
Hiseq2500
3‘端
Index primer
17
2021/6/20
5‘端
17
14. Repeat bridge amplification to form reverse strand after index sequencing
15. Sequencing the reverse strands
18
2021/6/20
转录组测序具体详细流程

转录组测序具体详细流程转录组测序是通过分离和分析RNA样本中的所有转录本以及它们的表达量来研究基因表达的方法。
下面将介绍具体详细的转录组测序流程。
1. RNA提取:首先需要从样本中提取RNA,这是转录组测序的重要步骤。
RNA提取的过程包括细胞破碎、RNA释放和RNA纯化等步骤。
常见的RNA提取方法包括TRIzol法、RNAsimple Total RNA kit、RNeasy mini kit等。
2. RNA质量评估:RNA的质量决定了后续测序的准确度和可靠性。
可以使用比色法、电泳法或者生物分析仪等方法评估RNA的质量和纯度。
RNA样品的A260/A280比值通常应在1.8~2.2之间,同时需要检查RNA的完整性。
3. RNA文库制备:RNA文库是指将RNA样品转录成双链DNA,然后通过PCR扩增得到的,包含了所有RNA序列的文库。
RNA文库制备的过程包括反转录、二代DNA文库建立、寡核苷酸连接、PCR扩增、文库质检等步骤。
常见的RNA文库建立方法包括Illumina的TruSeq RNA文库或者NEBNext Ultra RNA文库。
4. 转录组测序:RNA文库制备完成后,可以进行转录组测序。
转录组测序技术一般可以分为两类,即全长测序和短断测序。
全长测序包括PACBio和Nanopore等,具有高准确度和高覆盖度的特点,但是成本较高。
短断测序包括Illumina和IonTorrent等,具有较高的测序深度和适合高通量的优势,但是会产生一些误差。
5. 数据处理与分析:转录组测序得到的数据需要进行清洗、比对和表达定量等数据处理和分析。
常见的数据处理软件包括Trimmomatic、FastQC、STAR、HISAT2等。
表达定量可以使用RSEM、Cufflinks等软件,得到基因表达的数量和差异表达分析的结果。
综上所述,转录组测序是一项复杂的流程,需要经过RNA提取、RNA质量评估、RNA文库制备、转录组测序和数据处理等步骤。
illumina测序方法

illumina测序方法
Illumina测序方法是一种基于边合成边测序(Sequencing-By-Synthesis,SBS)的测序技术。
该方法包括以下步骤:
1. 向反应体系中同时添加DNA聚合酶、接头引物和带有碱基特异荧光标记的4种dNTP。
这些dNTP的3’-OH被化学方法所保护,因而每次只能添加一个dNTP。
2. 在dNTP被添加到合成链上后,所有未使用的游离dNTP和DNA聚合酶会被洗脱掉。
3. 加入激发荧光所需的缓冲液,用激光激发荧光信号,并有光学设备完成荧光信号的记录。
4. 利用计算机分析将光学信号转化为测序碱基。
这样荧光信号记录完成后,再加入化学试剂淬灭荧光信号并去除dNTP 3’-OH保护基团,以便能进行下一轮的测序反应。
Illumina测序方法的特点是每次只添加一个dNTP,这可以很好地解决同聚物长度的准确测量问题(Homopolymer错误)。
其主要的测序错误来源是碱基的替换,目前其测序错误率在1%-%之间。
以上信息仅供参考,如需了解更多信息,建议查阅生物信息学领域的学术文献或咨询相关专家。
Illumina测序介绍 PPT

Denature Double-Stranded Bridge 双链DNA桥式结构变性
Double-stranded bridge is denatured 桥式结构的双链DNA被变性
Result: Two copies of covalently bound single-stranded templates 结果:形成两条与流动槽表面共 价键结合的DNA模板
Hybridized primer is extended by polymerases 在DNA合成酶作用下,杂 交后的引物以单链DNA为 模板进行延伸
3’ extension 3‘延伸反应
12
Bridge Amplification 桥式PCR扩增
Double-stranded bridge is formed 延伸完成后形成双链DNA桥式结构
11
Bridge Amplification 桥式PCR扩增
Single-stranded molecule flips over and forms a bridge by hybridizing to adjacent, complementary primer 共价键结合在流动槽表面的单链DNA 分子与其附近的互补引物杂交,整条 DNA分子折叠后形成一种类似于桥的 结构。
HiSeq HiScan SQ
GA IIx MiSeq NextSeq
ICS/RTA CASAVA
MSR BaseSpace制备是决定测序实验成功与否的关键步骤
① ④②
③②④ ①双端标签① 与流动槽(Flow Cell)结合的区域 ② Read 1和Read2测序引物结合的区域 ③ 插入片段 ④ 标签序列区域(Index)
MSR BaseSpace
转录组测序的流程

转录组测序的流程转录组测序是一种用于研究生物体内所有转录RNA的测序方法,它可以帮助科研人员了解细胞内基因的表达情况,发现新的基因和非编码RNA,识别基因剪切异构体等。
下面将介绍转录组测序的流程。
1. 样品准备。
首先,需要准备好待测的生物样品,如细胞、组织等。
样品的质量和纯度对后续的测序结果有很大的影响,因此在样品准备阶段需要严格控制样品的处理和保存条件,以保证RNA的完整性和稳定性。
2. RNA提取。
接下来,需要从样品中提取总RNA。
RNA提取的方法有很多种,常用的包括酚-氯仿法、硅胶柱纯化法等。
在RNA提取过程中,需要注意避免RNA的降解和污染,保证提取到的RNA质量和纯度。
3. RNA质量检测。
提取到RNA后,需要对RNA的质量进行检测。
常用的方法包括琼脂糖凝胶电泳、分光光度计检测等。
通过检测RNA的完整性和浓度,可以评估RNA的质量,为后续的实验提供保障。
4. RNA样本准备。
在进行转录组测序之前,需要对RNA样本进行准备,包括RNA 的降解、反转录、合成cDNA等。
这些步骤可以帮助将RNA转化为适合测序的文库,为后续的测序实验做好准备。
5. 文库构建。
接下来,需要构建RNA文库。
文库构建的方法有很多种,包括Illumina TruSeq RNA Sample Prep Kit、NEBNext Ultra RNA Library Prep Kit等。
在文库构建过程中,需要注意避免文库的污染和杂交,保证文库的质量和稳定性。
6. 测序。
最后,进行转录组测序。
常用的转录组测序平台包括Illumina HiSeq、Ion Torrent等。
通过测序仪对文库进行测序,可以得到RNA的序列信息,为后续的数据分析提供基础。
总结。
转录组测序是一项复杂的实验技术,需要经过样品准备、RNA 提取、质量检测、RNA样本准备、文库构建和测序等多个步骤。
每个步骤都需要严格控制条件,保证实验的准确性和可靠性。
通过转录组测序,可以深入了解生物体内基因的表达情况,为后续的生物学研究提供重要的数据支持。
转录组测序流程范文

转录组测序流程范文转录组测序是一种用于研究转录物组成和表达水平的技术,可以帮助我们了解基因组中的转录物在特定条件下的表达情况。
在转录组测序流程中,主要包括样本准备、RNA提取、RNA测定、RNA库构建、测序和数据分析等步骤。
下面是对转录组测序流程的详细描述。
一、样本准备样本准备是转录组测序的第一步,需要选择合适的样本来研究感兴趣的生物学问题。
样本可以是生物体的组织、细胞或者培养物,根据需要可能需要进行处理,如处理不同条件下的样本,或者时间序列样本等。
二、RNA提取RNA提取是转录组测序的关键步骤,目的是从样本中纯化出RNA分子。
RNA可以是全长RNA(包括mRNA、rRNA、tRNA等)或者选择性富集一些类型的RNA(如mRNA)。
RNA提取的方法有很多种,常见的方法包括酚酸法、硅胶柱法、磁珠法等。
根据样本的性质和要求选择合适的RNA提取方法。
三、RNA测定RNA测定是对提取的RNA进行质量和纯度评估的步骤,主要目的是确定RNA是否适合后续的转录组测序工作。
RNA测定可以使用紫外光分光光度计测定280nm波长下的吸光度,以评估RNA的纯度。
同时,可以使用比色法或者荧光染料测定RNA的浓度,以评估RNA的含量。
四、RNA库构建RNA库构建是转录组测序的核心步骤,主要包括RNA的反转录、DNA的合成和文库构建等。
首先,通过反转录酶作用将RNA转录为cDNA。
反转录可以使用随机引物或者特异性引物进行。
然后,通过DNA聚合酶将cDNA合成为双链DNA,并在两端加上特定的接头序列。
接头序列可以包含样品索引,以区分不同样品的测序数据。
最后,通过PCR扩增和纯化等步骤得到最终的RNA库。
五、测序测序是转录组测序的重要步骤,可以使用不同的测序技术进行,包括第一代测序技术和第二代测序技术。
其中,第二代测序技术如Illumina HiSeq和Ion Torrent PGM等被广泛应用于转录组测序。
这些技术可以高通量地测序RNA库中的DNA分子,生成数百万的短序列读数。
illuminaSBS测序详解

illuminaSBS测序详解最近回头重新看了illlumina paired end sequence的测序原理视频,发现了以前没有注意的⼀些问题,⽽这些问题也是⼤家平时容易搞错的,因此花了⼏天时间将illumina 的paired end sequence 从构建⽂库到上机测序的整个过程以及原理较为详细的写了出来。
基础知识:illumina测序的核⼼在于利⽤可逆终⽌的、荧光标记的dNTP进⾏边合成边测序Flowcell(流动池)是有着2个或8个lane(泳道)的玻璃板,。
每个lane可以测⼀个样本或者多样本的混合物,且随机布满了能够与⽂库两端接头分别互补配对或⼀致的寡核苷酸(oligos,P7和P5接头)。
⼀个lane包含两列,每⼀列有60个tile,每个tile会种下不同的cluster,每个tile在⼀次循环中会拍照4次(每个碱基⼀次)。
paried-end sequencing⼀、Library Preparation⽂库的构建1. 利⽤转座⼦(transposome)对双链DNA进⾏剪切以及接头(adapter)的连接2. 接头连接成功后,利⽤低循环扩增技术在接头处进⾏修饰,分别在两端添加sequencing primer binding site1/sequencing primer binding site2(即测序引物结合位点)、index1/index2以及我们称之P5和P7的寡核苷酸序列上图并没有将之前的adapter标志出来,下图是维基百科的⽰意图,详细⼀些。
这⾥要注意两点(1)P5和P7是不同的,它们分别和flowcell上的接头互补和相同。
为了⽅便阐述,将与P5互补的接头称为P5’,与P7互补的接头称为P7’。
(2)index1和index2也是不同的,与P5相连的是index2,与P7相连的是index1。
关于index,也叫barcodes,因为⼀个lane可以同时测多个样品,为了避免混淆样品的read products,每种样品的DNA由⼀种index修饰,这样测序得到的reads都是具有index标记的,在测序结果中,依据之前标签与样品的对应关系,就可以获得对应样品的数据。
转录组测序与单细胞测序的实验流程

转录组测序与单细胞测序的实验流程转录组测序的基本步骤包括样本制备、RNA提取、cDNA合成、文库构建、测序以及数据分析。
样本制备是确保实验质量的关键环节,需要控制细胞生长状态、纯化细胞、固定细胞等。
RNA提取通常使用专用试剂盒,确保RNA完整性。
接着通过反转录酶将RNA反向转录为cDNA,之后进行文库构建,包括cDNA片段化、接头连接、PCR扩增等。
文库经测序仪高通量测序,生成大量原始数据。
最后进行生物信息学分析,包括质控、比对、注释、差异分析等,获取基因表达谱。
单细胞测序的流程更为复杂,需要首先对待测细胞进行单细胞分选。
分选方法种类繁多,包括流式细胞分选、激光捕获显微分选、微流体芯片分选、巴氏分装等。
分选后的单细胞需要进行逆转录、cDNA扩增,再进行文库构建和高通量测序。
数据分析部分除了一般分析内容外,还需要对单细胞数据特有的批次效应、缺失值等进行矫正,并整合所有单细胞数据,挖掘细胞亚群信息。
实验室常用的单细胞测序技术包括单细胞转录组测序、单细胞ATAC-seq等。
单细胞转录组测序技术分为全长单细胞转录组测序、5'或3'单细胞转录组测序。
全长单细胞转录组可以探测转录本水平的差异表达,而5'或3'单细胞转录组测序则侧重基因水平表达,成本较低。
单细胞ATAC-seq可全面分析单个细胞的开放染色质区域,揭示转录因子调控网络。
无论是转录组测序还是单细胞测序,关键都在于合理设计实验方案,严格控制实验过程,保证数据质量。
同时,数据分析策略也需要不断优化和完善,才能充分挖掘实验数据的潜在信息。
这些先进的高通量测序技术将极大推动生物医学研究的发展。
2.简述illumina测序的主要流程

2.简述illumina测序的主要流程
Illumina测序是一种高通量的基于 sequencing by synthesis (SBS) 技术的DNA测序技术,可以快速准确地测量细胞内的基因组。
Illumina测序将 RNA 或 DNA 初始物质样品从 DNA 复制和延伸等步骤中获得特定长度的小片段,将每个片段连接到携带反信号的假测序引物上,并且将片段序列以多个反复循环的序列读取步骤完成最终测序结果。
该SBS技术是一种利用一种好链式复制(clonal amplification)在全基因组测序中,利用荧光探测技术,来实现序列信息的高通量检测的算法。
Illumina测序的主要流程如下:
(1) DNA 上机分析。
首先,使用特定的库存酶将母本(比如 DNA 样本)切分成短序列片段,然后将含反信号的假测序引物附着在每个序列片段的双端。
这样的序列片段在碱基子酶中是未知的。
(2) 将 DNA 初步上机分析的结果以温度和酶存在梯度的方法聚类算法进行聚类,得出一个若干列特定双端序列片段的特征向量矩阵/矩阵,这里的温度和酶可根据设计的测序空间大小来调整。
最终,得出若干个基于此矩阵的校正序列片段。
(3) 将聚类得到的特定序列片段与其他包含双端序列标记的片段混合,在启动耦合上机读取,这一步也叫芯片分析或链状化学地图(chain chemical map)。
(4) 将短序列片段在芯片上测定,从而进行链状化学地图,测定的结果再反馈到芯片上,以确定位置。
(5) 最后,利用短序列片段的位置信息,将其重新组装成完整的 DNA 序列,并进行信噪比(Signal- to-Noise Ratio,SNR)分析,从而获得高准确率的基因组序列信息。
Illumina测序介绍

Reverse Strand Cleavage 反链切除
Reverse strands are cleaved and washed away, leaving a cluster with forward strands only 与流动槽表面结合的 DNA反链被 切除并洗去,只留下正链,形成 包含均一单链的 DNA簇
Each lane is randomly coated with a lawn of oligos that are complementary to library adapters 每条通道中都随机植入了能与 接头互补结合的大量短 DNA片段
7 Part # 15045845_Rev.C
FOR RESEARCH USE ONLY
15 Part # 15045845_Rev.C
Fdge Amplification 桥式PCR扩增
Bridge amplification cycle is repeated until multiple bridges are formed
桥式扩增不断重复发生直到形成 数量足够的DNA桥(与PCR反 应类似,区别在于引物不是游离 在溶液中,而是固定在流动槽表 面)
Hybridized primer is extended by polymerases 在DNA 合成酶作用下,杂 交后的引物以单链 DNA为 模板进行延伸
3' extension 3‘延伸反应
12 Part # 15045845_Rev.C
FOR RESEARCH USE ONLY
Bridge Amplification 桥式PCR扩增
Cluster Generation 簇生成
cBot MiSeq NextSeq HiSeq 2500-Rapid
用于高通量宏基因组学的IlluminaDNAPrep文库制备自动化工作流程

用于高通量宏基因组学的IlluminaDNAPrep文库制备自动化工作流程对于微生物实验室而言,尽管NGS在速度、准确性和信息深度方面具备显著优势,但文库制备仍成为高通量实验室的瓶颈(图1)。
为了应对这一挑战,Illumina与PerkinElmer携手合作,为宏基因组学提供了Illumina DNA Prep文库制备自动化工作流程。
这是一个全面的NGS解决方案,支持从DNA提取到DNA分析的全自动化工作流程(图2)。
与手动制备样品相比,在液体处理系统上制备Illumina DNAPrep文库具有显著优势。
主要优势包括通量更高,可扩展性更强,减少人为操作和人为错误,工作流程一致性增强,以及提高速度。
用于宏基因组学的Illumina DNA Prep文库制备自动化工作流程包括使用chemagic 360仪器(PerkinElmer)和chemagic DNA Stool Kit Special(PerkinElmer)从粪便样本中自动提取DNA。
提取DNA后,在Sciclone G3 NGSx液体处理工作站(PerkinElmer)上使用Illumina DNA Prep Library Preparation Kit(Illumina)制备文库。
Illumina DNA Prep Library Prep Kit 具有创新的磁珠固化转座酶(onbead tagmentation)技术,支持从各种生物体和样本中快速简便地制备文库3。
该试剂盒可兼容广泛的DNA起始量范围(100–500 ng),无需对初始DNA样本进行准确定量,节省了文库起始量均一化所需的时间和成本3。
从DNA 提取到最终文库混合池定量,用于宏基因组学的自动化Illumina DNA Prep文库制备工作流程可在不到6小时的时间内,提供多达96个可以进行测序的宏基因组文库。
此应用白皮书使用4名受试者的粪便样本,以比较Illumina DNA Prep文库制备自动化工作流程与标准手动工作流程用于宏基因组学分析的性能。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
illumina 转录组测序简明实验流程一、实验基本流程图
mRNA Library Construction
二、mRNA建库流程
1.材料准备
1.2.
1.3.
2.样品准备和QC
选择质量合格的Total RNA作为mRNA测序的建库起始样品,其质量要求通过Agilent 2100 BioAnalyzer检测结果RIN≥7,28S和18S的RNA 的比值大于或等于1.5:1,起始量的要求范围是0.1∽1ug。
用QUBIT RNA ASSAY KIT对起始的Total RNA进行准确定量。
3.建库实验步骤
3.1.mRNA纯化和片段化
3.1.1.mRNA纯化
纯化原理是用带有Oligod(T)的Beads对Total RNA中mRNA进行纯化。
3.1.2.mRNA片段化
3.2.1st Strand cDNA 合成
3.3.2nd Strand cDNA 合成
根据下表制备反应体系,然后在PCR仪上运行Program3,然后将第2链cDNA合成产物用144uL AMPure XP Beads进行纯化,最后用60µL的Nuclease free water进行重悬,取出
55.5µL以备下一步使用;
3.4.Perform End Repair/dA-tail
3.5.Adaptor Ligation
根据下表制备反应体系,然后在PCR仪上运行Program5、Program6,然后100uL AMPure XP Beads进行纯化后用52.5µL的Resuspension Buffer进行重悬,再用50uL AMPure XP Beads
3.6.PCR扩增
根据下表制备反应体系,然后在PCR仪上运行Program7,然后再45µL用AMPure XP Beads 进行纯化,最后用23µL的Resuspension Buffer进行重悬,取出20µL以备下一步使用;
3.7. PCR 产物质控
用QUBIT DNA HS ASSAY KIT 对PCR 产物进行准确定量。
(1) 2100质控
用2100 Bioanalyzer chip 判断PCR 切胶产物片段大小是否符合后续测序的要求。
(2) 文库摩尔浓度准确定量
通过q-PCR 标准品的摩尔浓度对构建的文库进行摩尔浓度的绝对定量,以保证文库上机用量的准确性,采用Illumina 推荐的KAPA 定量试剂盒(Cat no.KK4602)。
3.8. 混合文库 (Optional)
根据文库的定量及定性结果将每个library 的摩尔浓度统一调整到2nM ,然后根据实验设计选择性的将需要混合的library 进行等量等浓度混合,保证混合后的library 的终浓度也是2nM
,体积大于等于10µL ,然后将文库进行-80℃保存。
三、上机测序 1. 材料准备 1.1.
1.2. 2. 文库上机样品准备
2.1. 准备新鲜配置的NaOH ,将其浓度调整到0.1N ;取0.1N 的NaOH (10µL)与2nM 的文库(10µL)
的进行混合vortex ,离心,室温放置5min ,然后冰上放置;
2.2. 然后将20µL 已经变性成单链DNA 的Library 加入到980µL 预冷的HT1(Hybridization buffer)
中,使文库的终浓度为20pM ,冰上放置;
2.3.
3.Phix control样品准备
3.1.Phix control 2 nM(10µL)的体系,即
3.2.取0.1N的NaOH(10µL)与2nM的Phix control(10µL)的进行混合vortex,离心,室温
放置5min,然后冰上放置;
3.3.然后将20µL已经变性成单链DNA的Phix control加入到980µL预冷的HT1(Hybridization
buffer)中,使Phix control的终浓度为20pM,冰上放置;然后再将Phix control浓度稀释到5pM,即20pM的Phix control(600µL)+HT1(400µL);
4.簇生成
使用TruSeq Rapid PE Cluster Kit将Flowcell和准备好的文库在cBot上进行簇生成,即文库中的分子与Flowcell上固定的引物结合进行桥式PCR扩增,然后才能再HiSeq2500上进行测序。
5.边合成边测序
将完成做好簇生成的Flowcell转移到HiSeq2500仪器上准备测序,根据不同的测序类型以及测序长度,选择正确的Recipe,平均完成1个碱基的测序大概需要9分钟,所以测序周期与测序的长度直接相关。
测序程序正式运行前,首先根据First Base Report判断每条Lane上A,T,C,G碱基信号是否正常,从而判断测序引物结合是否有问题;其次根据First Base Report中cluster数量估测生成的数据量是否满足测序实验要求。
