常见载体的测序引物
真核细胞常见的表达载体及真核细胞表达外源基因的调控(精)

真核细胞常见表达载体1. pCMVp-NEO-BAN载体特点: 该真核细胞表达载体分子量为6600碱基对,主要由CMVp启动子、兔β-球蛋白基因内含子、聚腺嘌呤、氨青霉素抗性基因和抗neo基因以及pBR322骨架构成,在大多数真核细胞内都能高水平稳定地表达外源目的基因。
更重要的是,由于该真核细胞表达载体中抗neo基因存在,转染细胞后,用G418筛选,可建立稳定的、高表达目的基因的细胞株。
插入外源基因的克隆位点包括Sal1、BamH1和EcoR1位点。
注意在此载体中有二个EcoR1位点存在。
2. pEGFP, 增强型绦色荧光蛋白表达载体(Enhanced Fluorecent Protein Vector特点: pEGFP表达载体中含有绿色荧光蛋白,在PCMV启动子驱动下,在真核细胞中高水平表达。
载体骨架中的SV40 origin使该载体在任何表达SV40 T 抗原的真核细胞内进行复制。
Neo抗性盒由SV40早期启动子、Tn5的neomycin/kanamycin抗性基因以及HSV-TK基因的聚腺嘌呤信号组成,能应用G418筛选稳定转染的真核细胞株。
此外,载体中的pUC origin 能保证该载体在大肠杆菌中的复制,而位于此表达盒上游的细菌启动子能驱动kanamycin抗性基因在大肠杆菌中的表达。
用途: 该表达载体EGFP上游有Nde1、Eco47111和Age1克隆位点,将外源基因扦入这些位点,将合成外源基因和EGFP的融合基因。
借此可确定外源基因在细胞内的表达和/或组织中的定位。
亦可用于检测克隆的启动子活性(取代CMV启动子,Acet1-Nhe1。
Excitation maximum = 488 nm; Emission maximum = 507图示为启动子分泌信号肽和多克隆位点区域:Ase1.pCMV…ccg cta gcg cta ccg gtc gcc acc atg- .EGFP…BamH1…SV40 poly A+Nhe1 Age13. pEGFT-Actin, 增强型绿色荧光蛋白/人肌动蛋白表达载体特点: pEGFP-Actin表达载体中含有绿色荧光蛋白和人胞浆β-肌动蛋白基因,在PCMV启动子驱动下,在真核细胞中高水平表达。
pET-20b(+)载体说明
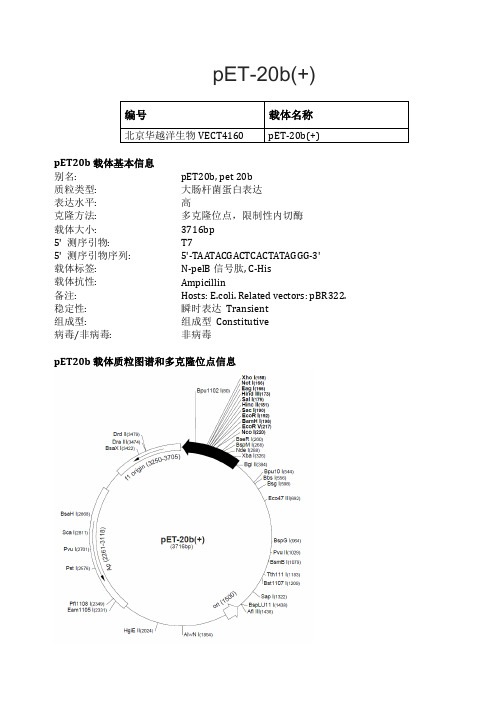
pET-20b(+)pET20b载体基本信息别名: pET20b, pet 20b质粒类型: 大肠杆菌蛋白表达表达水平: 高克隆方法: 多克隆位点,限制性内切酶载体大小: 3716bp5' 测序引物: T75' 测序引物序列: 5'-TAATACGACTCACTATAGGG-3'载体标签: N-pelB信号肽, C-His载体抗性: Ampicillin备注: Hosts: E.coli. Related vectors: pBR322. 稳定性: 瞬时表达Transient组成型: 组成型Constitutive病毒/非病毒: 非病毒pET20b载体质粒图谱和多克隆位点信息pET20b载体简介The pET-20b(+) vector (Cat. No. 69739-3) carries an N-terminal pelB signal sequence for potential periplasmic localization, plus optional C-terminal His•Tag® sequence. Unique sites are shown on the circle map. Note that the sequence is numbered by the pBR322 convention, so the T7 expression region is reversed on the circular map. The cloning/expression region of the coding strand transcribed by T7 RNA polymerase is shown below. The f1 origin is oriented so that infection with helper phage will produce virions containing single-stranded DNA that corresponds to the coding strand. Therefore, single-stranded sequencing should be performed using the T7 terminator primer (Cat. No. 69337-3).pET20b载体序列ORIGIN1 ATCCGGATAT AGTTCCTCCT TTCAGCAAAA AACCCCTCAA GACCCGTTTA GAGGCCCCAA61 GGGGTTATGC TAGTTATTGC TCAGCGGTGG CAGCAGCCAA CTCAGCTTCC TTTCGGGCTT121 TGTTAGCAGC CGGATCTCAG TGGTGGTGGT GGTGGTGCTC GAGTGCGGCC GCAAGCTTGT181 CGACGGAGCT CGAATTCGGA TCCGAATTAA TTCCGATATC CATGGCCATC GCCGGCTGGG241 CAGCGAGGAG CAGCAGACCA GCAGCAGCGG TCGGCAGCAG GTATTTCATA TGTATATCTC301 CTTCTTAAAG TTAAACAAAA TTATTTCTAG AGGGAAACCG TTGTGGTCTC CCTATAGTGA361 GTCGTATTAA TTTCGCGGGA TCGAGATCTC GGGCAGCGTT GGGTCCTGGC CACGGGTGCG421 CATGATCGTG CTCCTGTCGT TGAGGACCCG GCTAGGCTGG CGGGGTTGCC TTACTGGTTA481 GCAGAATGAA TCACCGATAC GCGAGCGAAC GTGAAGCGAC TGCTGCTGCA AAACGTCTGC541 GACCTGAGCA ACAACATGAA TGGTCTTCGG TTTCCGTGTT TCGTAAAGTC TGGAAACGCG 601 GAAGTCAGCG CCCTGCACCA TTATGTTCCG GATCTGCATC GCAGGATGCT GCTGGCTACC 661 CTGTGGAACA CCTACATCTG TATTAACGAA GCGCTGGCAT TGACCCTGAG TGATTTTTCT 721 CTGGTCCCGC CGCATCCATA CCGCCAGTTG TTTACCCTCA CAACGTTCCA GTAACCGGGC 781 ATGTTCATCA TCAGTAACCC GTATCGTGAG CATCCTCTCT CGTTTCATCG GTATCATTAC 841 CCCCATGAAC AGAAATCCCC CTTACACGGA GGCATCAGTG ACCAAACAGG AAAAAACCGC 901 CCTTAACATG GCCCGCTTTA TCAGAAGCCA GACATTAACG CTTCTGGAGA AACTCAACGA 961 GCTGGACGCG GATGAACAGG CAGACATCTG TGAATCGCTT CACGACCACG CTGATGAGCT 1021 TTACCGCAGC TGCCTCGCGC GTTTCGGTGA TGACGGTGAA AACCTCTGAC ACATGCAGCT 1081 CCCGGAGACG GTCACAGCTT GTCTGTAAGC GGATGCCGGG AGCAGACAAG CCCGTCAGGG 1141 CGCGTCAGCG GGTGTTGGCG GGTGTCGGGG CGCAGCCATG ACCCAGTCAC GTAGCGATAG 1201 CGGAGTGTAT ACTGGCTTAA CTATGCGGCA TCAGAGCAGA TTGTACTGAG AGTGCACCAT 1261 ATATGCGGTG TGAAATACCG CACAGATGCG TAAGGAGAAA ATACCGCATC AGGCGCTCTT 1321 CCGCTTCCTC GCTCACTGAC TCGCTGCGCT CGGTCGTTCG GCTGCGGCGA GCGGTATCAG 1381 CTCACTCAAA GGCGGTAATA CGGTTATCCA CAGAATCAGG GGATAACGCA GGAAAGAACA 1441 TGTGAGCAAA AGGCCAGCAA AAGGCCAGGA ACCGTAAAAA GGCCGCGTTG CTGGCGTTTT 1501 TCCATAGGCT CCGCCCCCCT GACGAGCATC ACAAAAATCG ACGCTCAAGT CAGAGGTGGC 1561 GAAACCCGAC AGGACTATAA AGATACCAGG CGTTTCCCCC TGGAAGCTCC CTCGTGCGCT 1621 CTCCTGTTCC GACCCTGCCG CTTACCGGAT ACCTGTCCGC CTTTCTCCCT TCGGGAAGCG 1681 TGGCGCTTTC TCATAGCTCA CGCTGTAGGT ATCTCAGTTC GGTGTAGGTC GTTCGCTCCA 1741 AGCTGGGCTG TGTGCACGAA CCCCCCGTTC AGCCCGACCG CTGCGCCTTA TCCGGTAACT 1801 ATCGTCTTGA GTCCAACCCG GTAAGACACG ACTTATCGCC ACTGGCAGCA GCCACTGGTA 1861 ACAGGATTAG CAGAGCGAGG TATGTAGGCG GTGCTACAGA GTTCTTGAAG TGGTGGCCTA 1921 ACTACGGCTA CACTAGAAGG ACAGTATTTG GTATCTGCGC TCTGCTGAAG CCAGTTACCT 1981 TCGGAAAAAG AGTTGGTAGC TCTTGATCCG GCAAACAAAC CACCGCTGGT AGCGGTGGTT 2041 TTTTTGTTTG CAAGCAGCAG ATTACGCGCA GAAAAAAAGG ATCTCAAGAA GATCCTTTGA 2101 TCTTTTCTAC GGGGTCTGAC GCTCAGTGGA ACGAAAACTC ACGTTAAGGG ATTTTGGTCA 2161 TGAGATTATC AAAAAGGATC TTCACCTAGA TCCTTTTAAA TTAAAAATGA AGTTTTAAAT 2221 CAATCTAAAG TATATATGAG TAAACTTGGT CTGACAGTTA CCAATGCTTA ATCAGTGAGG 2281 CACCTATCTC AGCGATCTGT CTATTTCGTT CATCCATAGT TGCCTGACTC CCCGTCGTGT 2341 AGATAACTAC GATACGGGAG GGCTTACCAT CTGGCCCCAG TGCTGCAATG ATACCGCGAG 2401 ACCCACGCTC ACCGGCTCCA GATTTATCAG CAATAAACCA GCCAGCCGGA AGGGCCGAGC 2461 GCAGAAGTGG TCCTGCAACT TTATCCGCCT CCATCCAGTC TATTAATTGT TGCCGGGAAG 2521 CTAGAGTAAG TAGTTCGCCA GTTAATAGTT TGCGCAACGT TGTTGCCATT GCTGCAGGCA 2581 TCGTGGTGTC ACGCTCGTCG TTTGGTATGG CTTCATTCAG CTCCGGTTCC CAACGATCAA 2641 GGCGAGTTAC ATGATCCCCC ATGTTGTGCA AAAAAGCGGT TAGCTCCTTC GGTCCTCCGA 2701 TCGTTGTCAG AAGTAAGTTG GCCGCAGTGT TATCACTCAT GGTTATGGCA GCACTGCATA 2761 ATTCTCTTAC TGTCATGCCA TCCGTAAGAT GCTTTTCTGT GACTGGTGAG TACTCAACCA 2821 AGTCATTCTG AGAATAGTGT ATGCGGCGAC CGAGTTGCTC TTGCCCGGCG TCAATACGGG 2881 ATAATACCGC GCCACATAGC AGAACTTTAA AAGTGCTCAT CATTGGAAAA CGTTCTTCGG 2941 GGCGAAAACT CTCAAGGATC TTACCGCTGT TGAGATCCAG TTCGATGTAA CCCACTCGTG 3001 CACCCAACTG ATCTTCAGCA TCTTTTACTT TCACCAGCGT TTCTGGGTGA GCAAAAACAG 3061 GAAGGCAAAA TGCCGCAAAA AAGGGAATAA GGGCGACACG GAAATGTTGA ATACTCATAC 3121 TCTTCCTTTT TCAATATTAT TGAAGCATTT ATCAGGGTTA TTGTCTCATG AGCGGATACA3181 TATTTGAATG TATTTAGAAA AATAAACAAA TAGGGGTTCC GCGCACATTT CCCCGAAAAG 3241 TGCCACCTGA AATTGTAAAC GTTAATATTT TGTTAAAATT CGCGTTAAAT TTTTGTTAAA 3301 TCAGCTCATT TTTTAACCAA TAGGCCGAAA TCGGCAAAAT CCCTTATAAA TCAAAAGAAT 3361 AGACCGAGAT AGGGTTGAGT GTTGTTCCAG TTTGGAACAA GAGTCCACTA TTAAAGAACG 3421 TGGACTCCAA CGTCAAAGGG CGAAAAACCG TCTATCAGGG CGATGGCCCA CTACGTGAAC 3481 CATCACCCTA ATCAAGTTTT TTGGGGTCGA GGTGCCGTAA AGCACTAAAT CGGAACCCTA 3541 AAGGGAGCCC CCGATTTAGA GCTTGACGGG GAAAGCCGGC GAACGTGGCG AGAAAGGAAG 3601 GGAAGAAAGC GAAAGGAGCG GGCGCTAGGG CGCTGGCAAG TGTAGCGGTC ACGCTGCGCG 3661 TAACCACCAC ACCCGCCGCG CTTAATGCGC CGCTACAGGG CGCGTCCCAT TCGCCA//其他大肠杆菌表达载体:pBV221 ptdTomato pET-52b(+) pAmCyanpDsRed-Express2 pBV220 pCold-GST pColdS-SUMOpCold TF pCold IV pCold III pCold IIpCold I pE-SUMO pCold-ProS2 pBAD102/D-TOPOpBAD202/D-TOPO pACYC184 pBAD/Thio-TOPO pBad/Myc-His CpBad/Myc-His B pBad/Myc-His A pBad/His C pBad/His BpBad/His A pBAD-TOPO pET-23b(+) pET-23a(+)pET-23c(+) pET-23(+) pET-12b(+) pET-12c(+)pET-12a(+) pET-11b(+) pET-11a(+) pET-11c(+)pBad24 pQE-82L pQE-81L pQE-80LpQE-32 pQE-9 pQE-16 pQE-31pQE-60 pQE-70 pQE-40 pET-51b(+)pET-50b(+) pET-49b(+) pET-48b(+) pET-47b(+)pET-26b(+) pET-32a(+) pET-21b(+) pET-22b(+)pET-14b pET-16b pET-15b pET-19bpET-20b(+) pET-21d(+) pET-21c(+) pET-21b(+)pET-21a(+) pET-24a(+) pET-24d(+) pET-25b(+)pET-27b(+) pET-28a(+) pET-30a(+) pET-42a(+)pET-43.1c(+) pET-43.1b(+) pET-43.1a(+) pET-44a(+)pET-44c(+) pET-46 EK/LIC pET-37b(+) pTrcHis2 CpTrcHis2 B pTrcHis2 A pET303/CT-His pET302/NT-HispRSET-CFP pRSET-EmGFP pRSET-BFP pGFPuvpET300/NT-DEST pET301/CT-DEST pGEM-T pBad43pGEX-4T-3 pGEX-5X-2 pBlueScript SK(+) pG-Tf2pG-KJE8 pGro7 pET-SUMO pSE380pET-17b pET102/D-TOPO pCDFDuet-1 pMAL-p5xpTf16 pET-28c(+) pBluescript II SK(+) pET-30b(+)pSUMO pProEXHTc pProEXHTb pProEXHTapKD3 pKD13 pKD46 pTYB1pTYB2 pTWIN2 pBluescript II KS(-) pTYB12pMAL-p5e pACYCDuet-1 pEGM-11ZF(+) pEGM-7ZF(+)PinPoint Xa-3 PinPoint Xa-2 PinPoint Xa-1 pSP73pSP64 pTWIN1 pTYB11 pTXB1pET-5b(+) pBad/gIII C pBad/gIII B pBad/gIII A pET-5a(+) pMal-p4X pMal-p2G pkk223-3pkk232-8 pCYB1 pEZZ18 pBAD18pMAL-c5x pMal-p2E pMal-p2X pET-44 EK/LIC pET-43.1 EK/LIC pET-41 EK/LIC pMal-c4X pTrcHis BpET-31b(+) pET-3b(+) pET-41a(+) pGEX-3XpGEX-4T-2 pETDuet-1 pGEX-4T-1 pTrc99apET-28b(+) pET-His pALEXa,b,c pACYC177pBR322 pKD4 pKD20 pMXB10pEcoli-6xHN-GFPuv pKJE7 pRSET B pGEX-KGpGEX-2T pRSFDuet-1 pCOLADuet-1 pTrcHis C pTrcHis A pET-41b(+) pET-42b(+) pET-3a(+) pGEX-6P-3 pGEX-6P-2 pGEX-6P-1 pGEX-5X-3 pGEX-5X-1 pGEX-2TK pRSET A pMal-c2GpMal-c2E pMal-c2X pRSET C pQE-30pET-45b(+) pET-44b(+) pET-42c(+) pET-41c(+) pET-40b(+) pET-33b(+) pET-39b(+) pET-32 EK/LIC pET-32 Xa/LIC pET-32c(+) pET-32b(+) pET-30 Xa/LIC pET-30 EK/LIC pET-30c(+) pET-29c(+) pET-29b(+) pET-29a(+) pET-24c(+) pET-24b(+) pET-24(+)pET-23d(+) pET-11d(+) pBad33。
常用pGEX载体图谱

常用pGEX载体图谱Rosetta系列的表达菌株可以提供T7 RNA聚合酶,它能表达PET系列载体上的外源基因。
pGEX系列载体上的外源基因不需要T7 RNA 聚合酶,普通的大肠杆菌经IPTG诱导即可表达Tac启动子是一组由Lac和trp启动子人工构建的杂合启动子,受Lac阻遏蛋白的负调节,它的启动能力比Lac和trp都强。
其中Tac 1是由Trp启动子的-35区加上一个合成的46 bp DNA片段(包括Pribnow 盒)和Lac操纵基因构成,Tac 12是由Trp的启动子-35区和Lac 启动子的-10区,加上Lac操纵子中的操纵基因部分和SD序列融合而成蛋白标签:A myc tag is a polypeptide proteintag derived from the c-myc gene product that can be added to a proteinusing recombinant DNA technology. It can be used for affinity chromatography, then used to separate recombinant, overexpressed protein from wild type protein expressed by the host organism. Itcan also be used in the isolation of protein complexes with multiple subunits.A myc tag can be used in many different assays that require recognition byan antibody. If there is no antibody against the studied protein, adding a myc-tag allows one to follow the protein with an antibody against the Myc epitope. Examples are cellular localization studies by immunofluorescence or detectionby Western blotting.The peptide sequence of the myc-tag is (in 1- and 3-letter codes, respectively):N-EQKLISEEDL-C,N-Glu-Gln-Lys-Leu-Ile-Ser-Glu-Glu-Asp-Leu -C, where N stands for Amino-terminus and C stands for Carboxy terminus. The tag is approximately 1202 Daltons in atomic mass and has 10 amino acids.It can be fused to the C-terminus andthe N-terminus of a protein. It is advisablenot to fuse the tag directly behind the signal peptide of a secretory protein, since it can interfere with translocation intothe secretory pathway.A monoclonal antibody against themyc epitope, named 9E10, is available from the non-commercial Developmental Studies Hybridoma BankpGEX4T1载体基本信息出品公司: GE别名: pGEX-4T-1, pGEX4T1, pGEX 4T 1质粒类型: 大肠杆菌蛋白表达载体表达水平: 高拷贝启动子: Tac克隆方法: 多克隆位点,限制性内切酶载体大小: 4969 bp5' 测序引物及序列: pGEX5': GGGCTGGCAAGCCACGTTTGGTG3' 测序引物及序列: pGEX3': CCGGGAGCTGCATGTGTCAGAGG载体标签: N-GST载体抗性: Ampicillin 氨苄备注: 复制子是pMB1产品目录号: 27-4580-01稳定性: 瞬时表达 Transient组成型: 诱导表达病毒/非病毒: 非病毒pGEX4T1载体质粒图谱和多克隆位点信息原核生物DNA复制起始点,是DNA链上独特的具有起始DNA复制功能的碱基序列。
真核表达载体

真核细胞常见表达载体1、pCMVp-NEO-BAN载体特点: 该真核细胞表达载体分子量为6600碱基对,主要由CMVp启动子、兔β-球蛋白基因内含子、聚腺嘌呤、氨青霉素抗性基因和抗neo基因以及pBR322骨架构成,在大多数真核细胞内都能高水平稳定地表达外源目的基因。
更重要的是,由于该真核细胞表达载体中抗neo基因存在,转染细胞后,用G418筛选,可建立稳定的、高表达目的基因的细胞株。
插入外源基因的克隆位点包括Sal1、BamH1和EcoR1位点。
注意在此载体中有二个EcoR1位点存在。
2、pEGFP, 增强型绦色荧光蛋白表达载体(Enhanced Fluorecent Protein Vector)特点: pEGFP表达载体中含有绿色荧光蛋白,在PCMV启动子驱动下,在真核细胞中高水平表达。
载体骨架中的SV40 origin使该载体在任何表达SV40 T 抗原的真核细胞内进行复制。
Neo抗性盒由SV40早期启动子、Tn5的neomycin/kanamycin抗性基因以及HSV-TK基因的聚腺嘌呤信号组成,能应用G418筛选稳定转染的真核细胞株。
此外,载体中的pUC origin 能保证该载体在大肠杆菌中的复制,而位于此表达盒上游的细菌启动子能驱动kanamycin抗性基因在大肠杆菌中的表达。
用途: 该表达载体EGFP上游有Nde1、Eco47111和Age1克隆位点,将外源基因扦入这些位点,将合成外源基因和EGFP的融合基因。
借此可确定外源基因在细胞内的表达和/或组织中的定位。
亦可用于检测克隆的启动子活性(取代CMV启动子,Acet1-Nhe1)。
3、pEGFT-Actin, 增强型绿色荧光蛋白/人肌动蛋白表达载体特点: pEGFP-Actin表达载体中含有绿色荧光蛋白和人胞浆β-肌动蛋白基因,在PCMV启动子驱动下,在真核细胞中高水平表达。
载体骨架中的SV40 origin使该载体在任何表达SV40 T 抗原的真核细胞内进行复制。
DNA测序模板的制备和测序引物设计中的相关问题

DNA测序模板的制备和测序引物设计中的相关问题王虎王晓健甄一松邹玉宝郑维越张芊核酸序列测定是基因研究的重要手段,本世纪60年代和70年代,科学家们一直致力于研究测定核酸序列的方法。
最初使用的方法只能测定RN A,主要是tRNA,一则因为它的链较短,通常只有74295个核苷酸,二则有可能分离单个tRNA分子。
而D NA的情况却大相径庭,人的染色体有大有小,每条染色体约含5千5百万到2亿5千万个碱基对,远远大于RNA 分子,如何将其切割并分离成500bp以下的片段成了D NA序列测定问题的关键。
基因克隆(gene cloning)和多聚酶链式反应(polymerase chain reaction,PCR)技术使从染色体中分离特定D N A片段的难题迎刃而解,快速高效的测序技术因此产生。
1977年,基于链终止和化学降解的D N A测序法研究成功,略经改善后很快就被推广到世界各国的分子生物学实验室,成为80年代和90年代序列测定革命的基础。
但此种测序法的缺点也是显而易见的,一方面操作步骤繁琐,可测样本量小,另一方面同位素的使用也明显限制了该方法的应用。
八十年代末期,美国加州一位科学家发明了世界上第一台D N A全自动测序仪,随着此后技术的不断成熟与完善,D NA的测序已经实现了高通量、产业化。
由美、英、日、德、法、中六国参与的人类基因组计划(Human Genome Project,HGP),是人类文明史上最伟大的科学创举之一,其核心内容是人类基因组序列图的绘制)))测定人类基因组的全部D NA序列,该计划启动于1990年,原定15年完成,由于技术的成熟与基因组测序的规模化运作,人类基因组工作框架图构建已于2001年6月全部完成。
一、测序的原理D NA测序方法主要有双脱氧链终止法(Sanger法)和化学降解法(M axa m2Gilbert法)。
目前,传统的手工测序方法及仪器自动测序方法均使用双脱氧链终止法。
常用载体测序引物列表

常用载体测序引物列表 载体名称 正向引物 反向引物pACT T7 T3pACT2 GAL4 AD(5’AD) 3'AD/pACT2-R PACYCDUET-1(MCSII) DuetUP2 T7TERpAD/pl-DEST CMV-FpAD-Track pAD-Track-F 无pAS2-1 5’BD M13-26/48 PB42AD PB42ADF PB42ADR PBAD PBAD-F PBAD-R pBABE pBABE5’ pBABE3’ PBACPAK8 BAC1BAC2 pBK-CMV T7/M13-20 T3/M13-26 PBS(SK/KS)/M13- M13F/T7T3/M13R pBV220 PBV220F PBV220R pBI121需根据酶切位点确认 需根据酶切位点确认 pCAMBIA 1301(1300)需根据酶切位点确认 需根据酶切位点确认 pCAMBIA 2300 需根据酶切位点确认 需根据酶切位点确认PCANTB5E S1/M13R S6pCAT3-enhancer pGL3+pcDNA3.0 pEGFP-N5/T7 SP6/BGHpcDNA3.1 pEGFP-N5/T7 BGHpcDNA4 T7/pEGFP-N5 BGHpcDNA6 T7/pEGFP-N5 BGHpcDNAII T7SP6 pCE2.1 M13FM13R pCEP4 pCMV5R/PCEP-F pEGFP-N5/EBV-R pCF-T M13FM13R pCIT7(17Base ) pCMV5R pCI-neo T7(17Base ) T3pCMS-EGFP T7T3 pCMV-3Tag-4A T3 /pFlag-CMV-F T7pCMV5 pCMV5F pCMV5R pCMV5-Flag pCMV5FpCMV5R pCMV-MYC/NUC pCMV5FpCMV5R PCMV-HA pCMV5FpCMV5R pCMV-SportSP6/M13RpCMV-Tag T3T7 pCMV-TNT T7/SP6pCMV5R pCR2.1-TOPO M13F/T7M13R pCR3.1 T7BGH pCS2 SP6T7PDNR-LIB M13F M13RpDonar M13F M13R pDONR201 pDONR-F pDONR-R pDONR221 M13F M13RpDrive T7 SP6 pDRIveR T7 SP6pDsRED1-C1 pDsRED-C1-F pECFP-C-3 pDsRed1-N1 pEGFP-N-5 RFP-NrevpDsRed-Monomer-N1 pEGFP-N-5 pDsRed1-NpDsRED2-C1 pDsRED-ex-C1-F pEGFP-N-3’pEGFP-N-5 pDsRed1-N pDSRED2-N1pDSRED-N1 pEGFP-N-5’ PDSRED-N-R pEASY-BLUNT M13F/T7 M13RTerpEASY-E1 T7 T7pEASY-T1 M13F/T7 M13RpEASY-T3 M13F/T7 M13R/SP6 pECFP-C pEGFP-C-5’ pECFP-C-3' pECFP-N1 pEGFP-N-5’ pEGFP-N-3’pEF/myc/ER pEF-F pCDNA3.1R pEGFP-C pEGFP-C-5’ pEGFP-C-3' pEGFP-N pEGFP-N-5’ pEGFP-N-3’ pENTR/D-TOPO M13F M13RTerpET-*(His) T7 T7pET22(a,b,c) T7 T7TerTerpET28,30,24,49 T7 T7TerpET32(a,b,c) T7/S.tag T7pET-42a (-b, -c)(+) Stag T7 TerpET50 T7/s.tag T7TERpET44 T7/s.tag T7Ter pEYFP-N1 pEGFP-N-5’ pEGFP-N-3’ PETBlue M13-20 PETBlueDOWN PETDUET-1(MCSI) PBRrevBam DuetDOMN1PETDUET-1(MCSII) DuetUP2 T7TERpEYFP-C1 pEGFP-C-5’ pEGFP-C-3' pEYFP-N1 pEGFP-N-5’ pEGFP-N-3’ pFastBac pFastBac-F pFastBac-R pFastBac HT_A,B,C pFastBac-PH pECFP-C-3 pFastBac1 pFastBac-PH pECFP-C-3 pFastBac Dual (MCSI) pFastBac-PH pECFP-C-3 pFLAG-CMV CMV30/pFLAG-CMV-F CMV-24/pFLAG-CMV-Rp3xFlag-CMV-7.1 CMV-F/CMV24 CMV30pGAD424 5’AD 3'ADpGADGH 5’AD 3'ADpGADT7-Rec 5’AD/T7 3'ADpGAPZa-A/B/C a-FACTOR 3'AOXforward 3'AOXpGAPZ-A/B/C pGAPpGBKT7 T7 3'BDpGEM-T(-Easy) M13F/T7 SP6/M13RpGEX-(*)T pGEX-5’ pGEX-3’pGL2 pCMV5R PGL3-Pgl3-BASIC pGL3+ PGL3-pGL3-Enhancer pGL3+ PGL3-PinPoint TM PinPoint Primer SP6pIRES PIRES-F/T7 T3/PIRES-RpIRES2-EGFP pIRES2-EGFP.P5’ pIRES2-EGFP.P3’ pLenti6/v5-Dest pEGFP-N-5’PLEXA PLEXA-F PLEXA-R PLNCX PLNCX-F PLNCX-R pLXSN pLXSN-F pLXSN-RPjc1-tac T7pJet.1.21.Blunt T7primer M13F(-47) pMAL-c2E MalEP3/PMAL-C2X-R pMAL-C2x P5/PMAL-C2X-Fprimer M13F(-47) pMAL-p2X MalEPMD18-T M13R(-48) M13F(-47) PMD19-T M13F(-47) M13R(-48) pMIR-report M13FPPC86 PPC86-F PPC86-R3’AOXpPIC9K 5’AOX/a-factor3'AOX pPICZa 5'AOX/PPICZa-FpPROEXHTA M13R(-48)pQE30or40 pQE30+ pQE30- pRECEIVER CMV-FpRSET T7 T7TER PRSFDUET-1(MCSI) DuetUP1 DuetDOWN1 PRSFDUET-1(MCSII) DuetUP2 T7TERpShuttle-CMV pShuttle-CMV-F pShuttle-CMV-R PSILENCE-1.0-U6 T7 T3pSilencer3.1-H1 hygro M13F(-47) 3.0revpSILENCEV2.0-U6 T7 2.0revpSK01-T M13F M13RpSK-CMV T7 T3Psos H1PSP64/65 SP6pSP72 T7 SP6T7/M13RpSport1 SP6/M13F(-47)pSTBlue-1 T7 SP6pSUPER T7 T3pT7Blue(R) T7 M13F(-47)pTA2 M13F/T7 T3/M13R/T7 M13RpT-Adv M13FpTARGET TM pTarget-F/T7 arget-RPTHIOHISA,B,C TRX-FORWARD TRX-REVERSEpTO-T7 T7 T7TERPBV220-/PTRC99C-R pTRC99a-c PQE30+/PTRC99C-FpTriPLEx2 PT5/5'TriplEx2 T7pTz57r/t M13+ M13-pTWIN-1 T7 T7TERpUC18(19)/118(119) M13F(-47) M13R(-48)M13F(-47)/M13F pUC57 M13R(-48)/M13RpUCm-T M13F/T7 M13RpVAX1 T7 BGHT3/M13R/M13R(-48) pWSK29 T7/M13F/M13F(-47)pXT7 T7 SP6pYES2 T7 pYes2.R。
质粒测序引物

GAL4 ADfor
pGAL4-ADrv
pACT2
p17110
p12584
pACYC184(BamHI-site)
PBRforBam
PBRrevBam
pACYCDuet-1(MCSI)
ACYCDuetUP1 Primer
DuetDOWN1
pACYCDuet-1(MCSII)
DuetUp2
T7-Terminator
M13R
pEASY-T3
M13F/T7
M13R/SP6
pECFP-N(C)
PECFP-N-5.
PECFP-N-3,
pEF/myc/cyto/ER/mito/nuc
pEF-5
Pcdna3.1R/BGH rev
pEF1(4,6/myc-His)
T7/pEF-5
Pcdna3.1R/BGH rev
pEF1(4,6/V5-His)
pDSRED-N1(KAN+)
pEGFP-N-5’
pDSRED-N-R
pEASY-Blunt
M13F
T7/ M13R
pEASY-BluntSimple
M13F
T7/ M13R(距离插入位点合适,优先安排)
pEASY-T1 Simple
M13F
M13R(距离插入位点合适,优先安排)
pEASY-T1
M13F/T7
pBridge(MCSI)
GAL4-Bdfor
3'BD
pBridge(MCSII)
not available
not available
pBS-T II
T3/M13rev
T7/M13for
常用DNA载体

I. 常用DNA载体1. Lambda DNALambda DNA is commonly used as a substrate in restriction enzyme activity assays and for preparation of DNA molecular weight standards. The phage is isolated from a heat-inducible lysogenic E.coliW3110 ( cI 857 S am7) strain. The DNA is isolated from purified phage by standard purification procedure.The molecule is linear double-stranded DNA, 48502 base pairs in length.Molecular Weight: 31.5 x 106 daltons.Storage Buffer: 10mM Tris-HCl (pH 7.6) and 1mM EDTA.Quality Control: Gel analysis for purity and homogeneity of Alw21I, CpoI and HindIII fragmentation2. pBR322 DNApBR322 DNA is a commonly used plasmid cloning vector in E.coli , isolated from E.coli ( dam+, dcm+ ) by ion exchange chromatography. The molecule is a double-stranded circle, 4361 base pairs in length. Molecular Weight: 2.83 x 106 daltons.Storage Buffer: 10mM Tris-HCl (pH 7.6) and 1mM EDTA.Quality Control: ?·More than 90% is in the supercoiled form.·Gel analysis for purity and homogeneity of AluI, BsuRI and HindIII fragmentation patterns.3. pUC18 DNApUC18 DNA is a commonly used plasmid cloning vector in E.coli , isolated from E.coli ( dam+, dcm+ ) by ion exchange chromatography. The molecule is a double-stranded circle, 2686 base pairs in length. Molecular Weight: 1.74 x 10 6 daltons.Storage Buffer: 10mM Tris-HCl (pH 7.6) and 1mM EDTA.Quality Control:·More than 90% is in the supercoiled form.·Gel analysis for purity and homogeneity of EcoR I, HindIII and Msp I fragmentation patterns.4. pUC19 DNApUC19 DNA is a commonly used plasmid cloning vector in E.coli , isolated from E.coli ( dam+, dcm+ ) by ion exchange chromatography. The molecule is a double-stranded circle, 2686 base pairs in length. Molecular Weight: 1.74 x 10 6 daltons.Storage Buffer: 10mM Tris-HCl (pH 7.6) and 1mM EDTA.Quality Control:·More than 90% is in the supercoiled form.·Gel analysis for purity and homogeneity of EcoR I, HindIII and Msp I fragmentation patterns.5. pUC57 DNApUC57 DNA is a commonly used plasmid cloning vector in E.coli , isolated from E.coli ( dam+, dcm+ ) by ion exchange chromatography.The molecule is a double-stranded circle, 2710 base pairs in length.Molecular Weight: 1.76 x 10 6 daltons.Storage Buffer: 10mM Tris-HCl (pH 7.6) and 1mM EDTA.Quality Control:·More than 90% is in the supercoiled form.II. 其他常用载体本系列载体均为抽干状态,每管10ug。
pUC18使用说明

pUC18 pUC18大肠杆菌克隆载体基本信息:启动子: Lac/lac复制子: ColE1 ori质粒分类: 大肠杆菌载体;克隆质粒质粒大小: 2686bp质粒标签: LacZ原核抗性: 氨苄青霉素Amp克隆菌株: DH5α培养条件: 37℃,有氧,LB5'测序引物: M13F:TGTAAAACGACGGCCAGT 3'测序引物: M13R:CAGGAAACAGCTATGACCpUC18载体质粒图谱和多克隆位点信息:pUC18载体简介:pUC18是适合于双脱氧法DNA测序的载体,通常运用于重组dna的分子克隆中,首先我们制备大肠杆菌的感受态细胞(能接受外来重组的dna),这种感受态菌株必须不同外来DNA分子发生遗传重组,通常是rec基因缺陷型的突变体,同时它们必须是限制系统缺陷或限制与修饰系统均缺陷的菌株,即具有这三种缺陷(rk mk rec ),同时对氨苄青霉素敏感(ap)。
puc18载体自身带有抗氨苄青霉素基因,而外源片段不具有,这样只有带有puc18的转化子的菌株才能在氨苄青霉素平板上存活,而外源片段自身环化的不能存活,这称为抗性筛选。
pUC18 上带有β-半乳糖苷酶基因(lacZ)的调控序列和β-半乳糖苷酶N 端146 个氨基酸的编码序列,在这个编码区中插有一个多克隆位点,且不影响正常功能,DH5α感受态菌株带有β-半乳糖苷酶C 端部分序列的编码信息,当puc18载体在正常情况下同感受态菌株融合后,互补表达有活性的β-半乳糖苷酶,称为α-互补现象,当有外源片段插入多克隆位点后,不能产生互补,即细菌不能产生β-半乳糖苷酶活性。
在呈色底物X-gal(5-溴-4-氯-3-吲哚-β-半乳糖苷酶)和诱导物IPTG(异丙基-β-D-硫代半乳糖苷)的存在下,互补产物菌落呈现蓝色,不互补产物菌落呈白色,很容易鉴别,这种筛选方法称为α-互补现象筛选(蓝白斑筛选)。
由上可见,puc18载体除了能重组导入外源片段外,在筛选表达过程中还具有重要的作用pUC18载体序列:ORIGIN1 TCGCGCGTTT CGGTGATGAC GGTGAAAACC TCTGACACAT GCAGCTCCCG GAGACGGTCA61 CAGCTTGTCT GTAAGCGGAT GCCGGGAGCA GACAAGCCCG TCAGGGCGCG TCAGCGGGTG121 TTGGCGGGTG TCGGGGCTGG CTTAACTATG CGGCATCAGA GCAGATTGTA CTGAGAGTGC181 ACCATATGCG GTGTGAAATA CCGCACAGAT GCGTAAGGAG AAAATACCGC ATCAGGCGCC241 ATTCGCCATT CAGGCTGCGC AACTGTTGGG AAGGGCGATC GGTGCGGGCC TCTTCGCTAT301 TACGCCAGCT GGCGAAAGGG GGATGTGCTG CAAGGCGATT AAGTTGGGTA ACGCCAGGGT361 TTTCCCAGTC ACGACGTTGT AAAACGACGG CCAGTGCCAA GCTTGCATGC CTGCAGGTCG421 ACTCTAGAGG ATCCCCGGGT ACCGAGCTCG AATTCGTAAT CATGGTCATA GCTGTTTCCT481 GTGTGAAATT GTTATCCGCT CACAATTCCA CACAACATAC GAGCCGGAAG CATAAAGTGT541 AAAGCCTGGG GTGCCTAATG AGTGAGCTAA CTCACATTAA TTGCGTTGCG CTCACTGCCC601 GCTTTCCAGT CGGGAAACCT GTCGTGCCAG CTGCATTAAT GAATCGGCCA ACGCGCGGGG661 AGAGGCGGTT TGCGTATTGG GCGCTCTTCC GCTTCCTCGC TCACTGACTC GCTGCGCTCG721 GTCGTTCGGC TGCGGCGAGC GGTATCAGCT CACTCAAAGG CGGTAATACG GTTATCCACA781 GAATCAGGGG ATAACGCAGG AAAGAACATG TGAGCAAAAG GCCAGCAAAA GGCCAGGAAC841 CGTAAAAAGG CCGCGTTGCT GGCGTTTTTC CATAGGCTCC GCCCCCCTGA CGAGCATCAC 901 AAAAATCGAC GCTCAAGTCA GAGGTGGCGA AACCCGACAG GACTATAAAG ATACCAGGCG 961 TTTCCCCCTG GAAGCTCCCT CGTGCGCTCT CCTGTTCCGA CCCTGCCGCT TACCGGATAC 1021 CTGTCCGCCT TTCTCCCTTC GGGAAGCGTG GCGCTTTCTC AAAGCTCACG CTGTAGGTAT 1081 CTCAGTTCGG TGTAGGTCGT TCGCTCCAAG CTGGGCTGTG TGCACGAACC CCCCGTTCAG 1141 CCCGACCGCT GCGCCTTATC CGGTAACTAT CGTCTTGAGT CCAACCCGGT AAGACACGAC 1201 TTATCGCCAC TGGCAGCAGC CACTGGTAAC AGGATTAGCA GAGCGAGGTA TGTAGGCGGT 1261 GCTACAGAGT TCTTGAAGTG GTGGCCTAAC TACGGCTACA CTAGAAGAAC AGTATTTGGT 1321 ATCTGCGCTC TGCTGAAGCC AGTTACCTTC GGAAAAAGAG TTGGTAGCTC TTGATCCGGC 1381 AAACAAACCA CCGCTGGTAG CGGTGGTTTT TTTGTTTGCA AGCAGCAGAT TACGCGCAGA 1441 AAAAAAGGAT CTCAAGAAGA TCCTTTGATC TTTTCTACGG GGTCTGACGC TCAGTGGAAC 1501 GAAAACTCAC GTTAAGGGAT TTTGGTCATG AGATTATCAA AAAGGATCTT CACCTAGATC 1561 CTTTTAAATT AAAAATGAAG TTTTAAATCA ATCTAAAGTA TATATGAGTA AACTTGGTCT 1621 GACAGTTACC AATGCTTAAT CAGTGAGGCA CCTATCTCAG CGATCTGTCT ATTTCGTTCA 1681 TCCATAGTTG CCTGACTCCC CGTCGTGTAG ATAACTACGA TACGGGAGGG CTTACCATCT 1741 GGCCCCAGTG CTGCAATGAT ACCGCGAGAC CCACGCTCAC CGGCTCCAGA TTTATCAGCA 1801 ATAAACCAGC CAGCCGGAAG GGCCGAGCGC AGAAGTGGTC CTGCAACTTT ATCCGCCTCC 1861 ATCCAGTCTA TTAATTGTTG CCGGGAAGCT AGAGTAAGTA GTTCGCCAGT TAATAGTTTG 1921 CGCAACGTTG TTGCCATTGC TACAGGCATC GTGGTGTCAC GCTCGTCGTT TGGTATGGCT 1981 TCATTCAGCT CCGGTTCCCA ACGATCAAGG CGAGTTACAT GATCCCCCAT GTTGTGCAAA 2041 AAAGCGGTTA GCTCCTTCGG TCCTCCGATC GTTGTCAGAA GTAAGTTGGC CGCAGTGTTA 2101 TCACTCATGG TTATGGCAGC ACTGCATAAT TCTCTTACTG TCATGCCATC CGTAAGATGC 2161 TTTTCTGTGA CTGGTGAGTA CTCAACCAAG TCATTCTGAG AATAGTGTAT GCGGCGACCG 2221 AGTTGCTCTT GCCCGGCGTC AATACGGGAT AATACCGCGC CACATAGCAG AACTTTAAAA 2281 GTGCTCATCA TTGGAAAACG TTCTTCGGGG CGAAAACTCT CAAGGATCTT ACCGCTGTTG 2341 AGATCCAGTT CGATGTAACC CACTCGTGCA CCCAACTGAT CTTCAGCATC TTTTACTTTC 2401 ACCAGCGTTT CTGGGTGAGC AAAAACAGGA AGGCAAAATG CCGCAAAAAA GGGAATAAGG 2461 GCGACACGGA AATGTTGAAT ACTCATACTC TTCCTTTTTC AATATTATTG AAGCATTTAT 2521 CAGGGTTATT GTCTCATGAG CGGATACATA TTTGAATGTA TTTAGAAAAA TAAACAAATA 2581 GGGGTTCCGC GCACATTTCC CCGAAAAGTG CCACCTGACG TCTAAGAAAC CATTATTATC 2641 ATGACATTAA CCTATAAAAA TAGGCGTATC ACGAGGCCCT TTCGTC//pUC18其他相关大肠杆菌克隆载体:pUC57-KanpUC57-SimplepUC57pUC119pUC118pJET1.2 T RecombinedpMD-19T(Simple)pGEM-3Zf(+)pBluescript II SK(+)pBluescript II KS(+)pACYC184 pSP73 pACYC177 pUC19 pUC18 pBR322 pMD-18T。
常用载体测序引物列表

常⽤载体测序引物列表常⽤载体测序引物列表载体名称正向引物反向引物pACT T7 T3pACT2 GAL4 AD(5’AD) 3'AD/pACT2-R PACYCDUET-1(MCSII) DuetUP2 T7TERpAD/pl-DEST CMV-FpAD-Track pAD-Track-F ⽆pAS2-1 5’BD M13-26/48 PB42AD PB42ADF PB42ADR PBAD PBAD-F PBAD-R pBABE pBABE5’ pBABE3’ PBACPAK8 BAC1BAC2 pBK-CMV T7/M13-20 T3/M13-26 PBS(SK/KS)/M13- M13F/T7T3/M13R pBV220 PBV220F PBV220R pBI121需根据酶切位点确认需根据酶切位点确认 pCAMBIA 1301(1300)需根据酶切位点确认需根据酶切位点确认 pCAMBIA 2300 需根据酶切位点确认需根据酶切位点确认PCANTB5E S1/M13R S6pCAT3-enhancer pGL3+pcDNA3.0 pEGFP-N5/T7 SP6/BGHpcDNA3.1 pEGFP-N5/T7 BGHpcDNA4 T7/pEGFP-N5 BGHpcDNA6 T7/pEGFP-N5 BGHpcDNAII T7SP6 pCE2.1 M13FM13R pCEP4 pCMV5R/PCEP-F pEGFP-N5/EBV-R pCF-T M13FM13R pCIT7(17Base ) pCMV5R pCI-neo T7(17Base ) T3pCMS-EGFP T7T3 pCMV-3Tag-4A T3 /pFlag-CMV-F T7pCMV5 pCMV5F pCMV5R pCMV5-Flag pCMV5FpCMV5R pCMV-MYC/NUC pCMV5FpCMV5R PCMV-HA pCMV5FpCMV5R pCMV-SportSP6/M13RpCMV-Tag T3T7 pCMV-TNT T7/SP6pCMV5R pCR2.1-TOPO M13F/T7M13R pCR3.1 T7BGH pCS2 SP6T7PDNR-LIB M13F M13RpDonar M13F M13R pDONR201 pDONR-F pDONR-R pDONR221 M13F M13RpDrive T7 SP6 pDRIveR T7 SP6pDsRED1-C1 pDsRED-C1-F pECFP-C-3 pDsRed1-N1 pEGFP-N-5 RFP-NrevpDsRed-Monomer-N1 pEGFP-N-5 pDsRed1-NpDsRED2-C1 pDsRED-ex-C1-F pEGFP-N-3’pEGFP-N-5 pDsRed1-N pDSRED2-N1pDSRED-N1 pEGFP-N-5’ PDSRED-N-R pEASY-BLUNT M13F/T7 M13RTerpEASY-E1 T7 T7pEASY-T1 M13F/T7 M13RpEASY-T3 M13F/T7 M13R/SP6 pECFP-C pEGFP-C-5’ pECFP-C-3' pECFP-N1 pEGFP-N-5’ pEGFP-N-3’pEF/myc/ER pEF-F pCDNA3.1R pEGFP-C pEGFP-C-5’ pEGFP-C-3' pEGFP-N pEGFP-N-5’ pEGFP-N-3’ pENTR/D-TOPO M13F M13RTerpET-*(His) T7 T7pET22(a,b,c) T7 T7TerTerpET28,30,24,49 T7 T7TerpET32(a,b,c) T7/S.tag T7pET-42a (-b, -c)(+) Stag T7 TerpET50 T7/s.tag T7TERpET44 T7/s.tag T7Ter pEYFP-N1 pEGFP-N-5’ pEGFP-N-3’ PETBlue M13-20 PETBlueDOWN PETDUET-1(MCSI) PBRrevBam DuetDOMN1 PETDUET-1(MCSII) DuetUP2 T7TERpEYFP-C1 pEGFP-C-5’ pEGFP-C-3' pEYFP-N1 pEGFP-N-5’ pEGFP-N-3’ pFastBac pFastBac-F pFastBac-R pFastBac HT_A,B,C pFastBac-PH pECFP-C-3 pFastBac1 pFastBac-PH pECFP-C-3 pFastBac Dual (MCSI) pFastBac-PH pECFP-C-3 pFLAG-CMV CMV30/pFLAG-CMV-F CMV-24/pFLAG-CMV-Rp3xFlag-CMV-7.1 CMV-F/CMV24 CMV30pGAD424 5’AD 3'ADpGADGH 5’AD 3'ADpGADT7-Rec 5’AD/T7 3'ADpGAPZa-A/B/C a-FACTOR 3'AOXforward 3'AOXpGAPZ-A/B/C pGAPpGBKT7 T7 3'BDpGEM-T(-Easy) M13F/T7 SP6/M13RpGEX-(*)T pGEX-5’ pGEX-3’pGL2 pCMV5R PGL3-Pgl3-BASIC pGL3+ PGL3-pGL3-Enhancer pGL3+ PGL3-PinPoint TM PinPoint Primer SP6pIRES PIRES-F/T7 T3/PIRES-RpIRES2-EGFP pIRES2-EGFP.P5’ pIRES2-EGFP.P3’ pLenti6/v5-Dest pEGFP-N-5’PLEXA PLEXA-F PLEXA-R PLNCX PLNCX-F PLNCX-R pLXSN pLXSN-F pLXSN-RPjc1-tac T7pJet.1.21.Blunt T7primer M13F(-47) pMAL-c2E MalEP3/PMAL-C2X-R pMAL-C2x P5/PMAL-C2X-Fprimer M13F(-47) pMAL-p2X MalEPMD18-T M13R(-48) M13F(-47) PMD19-T M13F(-47) M13R(-48) pMIR-report M13FPPC86 PPC86-F PPC86-R3’AOXpPIC9K 5’AOX/a-factor3'AOX pPICZa 5'AOX/PPICZa-FpPROEXHTA M13R(-48)pQE30or40 pQE30+ pQE30- pRECEIVER CMV-FpRSET T7 T7TER PRSFDUET-1(MCSI) DuetUP1 DuetDOWN1 PRSFDUET-1(MCSII) DuetUP2 T7TER pShuttle-CMV pShuttle-CMV-F pShuttle-CMV-R PSILENCE-1.0-U6 T7 T3pSilencer3.1-H1 hygro M13F(-47) 3.0revpSILENCEV2.0-U6 T7 2.0revpSK01-T M13F M13RpSK-CMV T7 T3Psos H1PSP64/65 SP6pSP72 T7 SP6T7/M13RpSport1 SP6/M13F(-47)pSTBlue-1 T7 SP6pSUPER T7 T3pT7Blue(R) T7 M13F(-47)pTA2 M13F/T7 T3/M13R/T7 M13RpT-Adv M13FpTARGET TM pTarget-F/T7 arget-RPTHIOHISA,B,C TRX-FORWARD TRX-REVERSE pTO-T7 T7 T7TERPBV220-/PTRC99C-R pTRC99a-c PQE30+/PTRC99C-F pTriPLEx2 PT5/5'TriplEx2 T7pTz57r/t M13+ M13-pTWIN-1 T7 T7TERpUC18(19)/118(119) M13F(-47) M13R(-48)M13F(-47)/M13F pUC57 M13R(-48)/M13RpUCm-T M13F/T7 M13RpVAX1 T7 BGHT3/M13R/M13R(-48) pWSK29 T7/M13F/M13F(-47)pXT7 T7 SP6pYES2 T7 pYes2.R。
引物合成、基因合成、测序和亚克隆

引物合成、基因合成、测序和亚克隆在现代分子生物学中,进行分子克隆和基因编辑技术时,引物合成、基因合成、测序和亚克隆是必不可少的技术手段。
引物合成引物是指在PCR(聚合酶链式反应)等反应中用来特异性引导DNA扩增的短小寡核苷酸序列。
引物一般由20-30个碱基组成,必须与目标DNA片段的5'和3'端配对,以确保扩增的特异性。
引物的合成可以通过化学合成、PCR法等手段实现。
基因合成基因合成是指通过化学合成的方式合成完整的DNA序列,然后将其插入到表达载体中,用于在细胞中大量表达蛋白质。
目前商业化的基因合成技术已经能够合成几百万个碱基对的DNA序列,从而实现大规模人工定制的基因构建。
测序测序是指通过不同的技术手段确定DNA或RNA的序列,包括Sanger测序、高通量测序等方法。
测序技术不仅可以用于研究基因及其表达机制,还可以用于研究细菌、病毒等微生物及其基因组学,以及研究整个生物系统中基因与环境相互作用的规律。
亚克隆亚克隆是指将DNA分子从一个表达载体转移到另一个表达载体的过程。
亚克隆技术广泛应用于分子克隆、基因编辑及生物合成等领域。
在分子克隆中,亚克隆技术可用于将目标DNA插入到表达载体中,以大量表达目标蛋白质。
在基因编辑中,亚克隆技术可用于替换或删除目标基因。
在生物合成中,亚克隆技术可用于将多个基因组合在一起,以实现对生物物质的定制合成。
总的来说,引物合成、基因合成、测序和亚克隆等分子技术,不仅提高了人们对生命现象的理解,而且有着广泛的应用前景,包括新药研发、生物质能源生产、环境修复等多个领域。
常用载体构建说明书

常用载体构建说明书步骤:一、基本耗材准备(抗生素、LB液体培养基、LB固体培养基、离心管、枪头、三角瓶)二、制备感受态大肠杆菌三、设计引物四、Pcr扩增五、Pcr产物检测六、Pcr产物回收七、双酶切pcr产物和质粒八、酶切产物回收九、目的基因与载体连接十、转化感受态大肠杆菌十一、单克隆检测十二、测序比对十三、提质粒十四、酶切验证十五、转化感受态农杆菌十六、单克隆检测十七、侵染液配制一、基本耗材准备1、抗生素的制备(抗生素为索来宝公司)常用抗生素Kan(卡那)Amp(氨苄)Rif(利福平)母液浓度:Kan 50mg/ml Amp 100 mg/ml Rif 100 mg/ml工作浓度:Kan 50ng/ul Amp 100 ng/ul Rif 100 ng/ul举例:称取kan固体1g 于注射器中,加入20ml ddH2O,溶解后用过滤器注入灭过菌的离心管中,-20度保存,使用比例1:1000注意:Rif溶解时加入DMSO2、培养基的配制LB液体培养基1000ml 200ml牛肉膏5g 1g蛋白胨10g 2g氯化钠10g 2gLB固体培养基1000ml 200ml牛肉膏蛋白胨氯化钠琼脂粉5g10g10g15g1g2g2g3g120℃高温高压灭菌15-20min,固体培养基冷却后加入相应抗性,加入比例1:1000,然后把培养基倒90cc的培养皿中,一般情况下一个培养皿可倒20ml培养基。
3、离心管、枪头、三角瓶0.2ml、1.5ml、2.0ml离心管各200-500个,50ml离心管2个10ul、200ul、1000ul 枪头各2盒50ml、100ml、250ml 、500ml三角瓶各2个去离子水或双蒸水500ml二、制备感受态大肠杆菌(所用超级感受态细胞制备试剂盒购于上海生工)BT Media 培养基制备:1支BT Media加50 ml蒸馏水配制,放入250ml三角瓶,高压灭菌即可准备工作:将BT Buffer A和BT Buffer B 放冰上遇冷,遇冷低温离心机至4℃1、用超低温冰箱中保存的菌种在LB平板上进行划线,置37℃培养箱中静置培养12-16h待菌落生长到1-2mm大小。
常用载体及相关通用引物

常用载体及相关通用引物默认分类2008-10-06 13:16:31 阅读512 评论0 字号:大中小载体一端另一端pBlueScriptSK(+) M13F(-77)/M13F(-47)/M13F/T7 M13R(-88)/M13R(-48)/M13R/T3 pcDNA3 H1.3F/pCMV-F /pEGFP-N-5/T7 BGHpcDNA3.1(+) H1.3F/pCMV-F /pEGFP-N-5/T7 pCDNA3.1R/BGHpcDNA3.0(+)/myc-His C/A/B H1.3F/pCMV-F /pEGFP-N-5/T7 BGHpcDNA3.1(+)/myc-His C/A/B H1.3F/pCMV-F /pEGFP-N-5/T7 pCDNA3.1R/BGHpcDNA3.1(+)/myc-His/LacZ H1.3F/pCMV-F /pEGFP-N-5/T7 pCDNA3.1R/BGHpcDNA3.1/His C/A/B H1.3F/pCMV-F /pEGFP-N-5/T7 pCDNA3.1R/BGHpcDNA3.1/His/LacZ H1.3F/pCMV-F /pEGFP-N-5/T7 pCDNA3.1R/BGHpcDNA3.1/Hygro(-) H1.3F/pCMV-F /pEGFP-N-5/T7 pCDNA3.1R/BGHpcDNA3.1/Hygro(+) H1.3F/pCMV-F /pEGFP-N-5/T7 pCDNA3.1R/BGHpcDNA3.1/Hygro/LacZ H1.3F/pCMV-F /pEGFP-N-5/T7 pCDNA3.1R/BGHpcDNA3.1/V5-His C/A/B H1.3F/pCMV-F /pEGFP-N-5/T7 pCDNA3.1R/BGHpcDNA3.1/V5-His/LacZ H1.3F/pCMV-F /pEGFP-N-5/T7 pCDNA3.1R/BGHpcDNA3.1/V5-His-TOPO H1.3F/pCMV-F /pEGFP-N-5/T7 pCDNA3.1R/BGHpcDNA3.1/V5-His-TOPO/LacZ H1.3F/pCMV-F /pEGFP-N-5/T7 pCDNA3.1R/BGHpcDNA3.1/Zeo(-) H1.3F/pCMV-F /pEGFP-N-5/T7 pCDNA3.1R/BGHpcDNA3.1/Zeo(+) H1.3F/pCMV-F /pEGFP-N-5/T7 pCDNA3.1R/BGHpcDNA3.1/Zeo/CAT H1.3F/pCMV-F /pEGFP-N-5/T7 pCDNA3.1R/BGHpColdI DNA p-GEX3’pColdII DNA p-GEX3’pColdIII DNA p-GEX3’pColdIV DNA p-GEX3’pDream2.1 H1.3F/pCMV-F /pEGFP-N-5/T7 SP6pEGFP-N1,2,3 H1.3F/pCMV-F /pEGFP-N-5 pEGFP-N-3’/pEGFP-C-5’/pEGFP-C-3' pET15b T7 T7terpET20b T7 T7terpET22b T7 T7terpET28a T7 T7terpET28b T7 T7terpET30b T7 T7terpFastBac Dual pEGFP-C-3'pFastBac HT A,B,C pEGFP-C-3'pFastBac1 pEGFP-C-3'pGEM-T Easy M13F(-77)/M13F(-47)/M13F/T7 M13R(-88)/M13R(-48)/M13RpGEX-4T-1/3 p-GEX5’ p-GEX3’pGS-21a p-GEX5’ T7terpLenti6/V5-D-TOPO M13F(-47)/M13F/T7 M13R(-48)/M13R/T3pLenti6/V5-GW/lacZ M13F(-47)/M13F/T7 M13R(-48)/M13R/T3pMAL-p2x M13F(-47)/M13F/PBV220R M13R(-48)pMD18-T M13F(-77)/M13F(-47)/M13F M13R(-88)/M13R(-48)/M13RpMD19-T M13F(-77)/M13F(-47)/M13F M13R(-88)/M13R(-48)/M13RpMTBiPV5-His GFP p-GEX3’/ M13F(-47)/M13F M13R(-88)/M13R(-48)/M13R /pEGFP-C-3'/BGH pPCR script M13F(-77)/M13F(-47)/M13F/T7 M13R(-88)/M13R(-48)/M13R/T3pPIC3.5 5’AOX 3’AOXpPIC9 5’AOX 3’AOXpPICZ C/A/B 5’AOX 3’AOXpPICZalpha C/A/B 5’AOX/α-Factor 3’AOXpSecTag2 A,B,C H1.3F/pCMV-F /pEGFP-N-5/T7 BGHpTWIN T7 T7terpUC57 M13F(-77)/M13F(-47)/M13F M13R(-88)/M13R(-48)/M13RpUC18 M13F(-47)/M13F M13R(-48)/M13RpUC19 M13F(-47)/M13F M13R(-48)/M13R其他…… ……注:1)排列顺序为:有左到右==(离插入为点)由远到近;2)若有多个引物,红色的为首选引物:3)选择测序通用引物时,以距离插入位点约50碱基为佳;4)同时有T7,T7ter的载体,单反应首选T7ter;5)同时有T7,pCDNA3.1R的载体,单反应首选pCDNA3.1R ;6)同时有T7,BGH的载体,单反应首选BGH;7)同时有pCMV-F,BGH的载体,单反应首选BGH;。
水稻突变体介绍及鉴定(很详细)

RMD水稻突变体信息及基因型鉴定1.背景介绍:突变体对于遗传学研究有着重要作用,随着拟南芥和水稻等物种全基因组测序的开展,人类积累了前所未有的基因序列信息,为了弄清这些基因序列的生物学信息,寻找该基因区段序列发生变异的突变体是阐释基因功能最直接最有效的方法。
植物在自然的环境条件下也会产生突变性状,早期普通正向遗传学研究往往通过寻找与某种生物学特性相关的突变体来发掘或定位某个特定基因。
为配合植物功能基因组研究高通量的策略,构建水稻等物种的大型突变体库已成为必然,借助水稻全基因组测序信息、通过反向遗传学的手段大规模地筛选突变体库,理论上可以获得基因组中任一基因的突变体,最终实现阐释基因功能的目的。
2.原理:2.1农杆菌介导的T-DNA 插入农杆菌是寄主范围非常广泛的土壤杆菌,它能通过伤口侵染植物导致冠瘿瘤和毛状根的发生。
1974从根癌农杆菌中分离出一种与肿瘤诱导相关的质粒,称为致瘤质粒(Tumor-inducing plasmid),简称Ti 质粒。
Ti 质粒上存在一段DNA,能够转移并整合到植物基因组中,称为Transferred DNA,简称T-DNA。
研究发现,T-DNA 两端存在非常保守的同向重复的25bp 序列,分别称为左边界(LB)和右边界(RB)。
T-DNA 的转移只与边界序列相关,尤其是RB,而与T-DNA区段的其它基因或序列无关。
我们将T-DNA 区段上的致瘤基因和其它无关序列去掉,利用其转移的特性,实现农杆菌介导的T-DNA 转入水稻愈伤,从而构建水稻突变体库。
大量研究表明,农杆菌T-DNA 整合到植物基因组中的位置是随机的,并且整合到植物基因组中的T-DNA 能稳定遗传。
由于插入到植物基因组中的T-DNA 区段序列已知,这样随机插入到植物基因组中的T-DNA类似于给植物基因“贴”了一个序列标签。
我们利用这个标签,通过各类PCR技术最终可以获取其插入的位点。
2.2 水稻Tos17 反转录转座子创造水稻突变体的另一种方法是利用植物的反转录转座子,它们是以DNA→RNA→DNA 的方式进行转座,在水稻上已发现大约40 种长未端重复的反转录转座子,它们是Tos1-Tos32,RIRE1-RIRE8,其中5 类被证明是有转座活性的,分别是Tos10、Tos17、Tos19、Tos25 和Tos27。
DNA的化学合成及应用

DNA化学合成基因是由具有特定的核苷酸顺序的核酸组成的功能单位,它携有特定的遗传信息,在染色体上按一定的顺序排列。
基因的基本单位为核苷酸,每个核苷酸包含三种成分:碱基、戊糖和磷酸。
DNA的单体单位为脱氧核苷酸,其中戊糖是D-2-脱氧核糖。
四种碱基分别为腺嘌呤(A) 、鸟嘌呤(G)、胞嘧啶(C)和胸腺嘧啶(T)。
在核苷酸中两类杂环化合物嘧啶和嘧呤具有很明显的芳香族特性。
嘌呤本身可以认为是嘧啶的1种衍生物,它是1个嘧啶环和咪唑环耦合在一起组成的。
在大多数细胞中,碱基只有少数呈游离或不结合形式存在,而且它们通常是酶水解核苷酸的产物。
游离嘧啶和嘌呤碱基在水中是相对难溶解的,是弱碱性化合物,随pH不同能以两种或多种互变异构的形式存在。
嘧呤碱基的第九氮原子或嘧啶碱基的第一氮原子与戊糖的第一碳原子形成β-N糖苷酸通称核苷。
核苷按所含糖的不同分为:D-核糖核苷类和2-脱氧-D-核糖核苷类。
在大多数细胞中只有微量的游离核苷存在。
核苷比相应的碱基易溶于水,在不同pH条件下存在与碱基类似的互变异构,N-糖苷键对酸不稳定。
核苷的磷酸酯称核苷酸。
核糖有3个游离羟基,所以核糖核苷酸有2′、3′和5′核苷酸。
脱氧核糖只有2个羟基可以酯化,故脱氧核糖核苷酸只有3′和5′核苷酸。
核酸分子是由核苷酸通过3′→5′磷酸二酯键连接而成。
DNA的化学合成研究始于50年代。
1952年阐明核酸大分子是由许多核苷酸通过3′-5′磷酸二酯链键连接起来的这个基本结构以后,化学家们便立即开始尝试核酸的人工合成。
英国剑桥大学Todd实验室于1958年首先合成了具有3′→5′磷酸二酯键结构的TpT和pTpT,此后,Khorana等人对基因的人工合成作出了划时代的贡献,不仅创建了基因合成的磷酸二酯法,而且发展了一系列有关核苷酸的糖上羟基、碱基的氨基和磷酸基的保护基及缩合剂和合成产物的分离、纯化方法。
到目前为此,使用的DNA合成方法有磷酸三酯法、亚磷酸酯法及亚磷酸酰胺法。
测序通用引物序列

测序通用引物序列常见载体的测序引物:Primer of Vector:Vector:Primer(F);Primer(R)pACT T7 T3pACT2 GAL4 AD pACT2-RpAS2-1 GAL4 BD pAS2-1.RpB42AD pB42ADF pB42ADRpBACPAK8 BAC1 BAC2pBK-CMV T7 T3PBS(SK/KS)/M13- M13F/T7 T3/M13RpBV220 PBV220F PBV220RpCAMBIA 1301(1300) P1 P2pCAMBIA 2300 M13R(-48) M13F(-47)PCANTB5E S1/M13R S6pCAT3-enhancer RVP3 此载体无反向引物pcDNA3.0 CMV-F/T7 SP6/BGHpcDNA3.1 T7 BGHpcDNA4 T7/CMV-F BGHpcDNA6 T7/CMV-F(J21025在T7前面)BGHpcDNAII T7 SP6pCE2.1 M13F M13RpCEP4 pCEP-F EBV-RpCF-T M13F M13RpCI T7(17Base)此载体无反向引物pCI-neo T7(17Base)T3pCMS-EGFP T7 T3pCMV-3Tag-4A T3 /PFLAG-CMV-F T7pCMV5 pCMV5F pCMV5RpCMV5-Flag pCMV5F pCMV5RpCMV-MYC/HA pCMV-F pCMV-RpCMV-Sport M13F/T7 SP6/M13RpCMV-Tag(KAN+) T3 T7pCR2.1-TOPO M13F/T7 M13RpCR3.1 T7 BGHpCS2 SP6 T7pDonar M13F M13RpDONR221 M13F M13RpDrive T7 SP6pDRIveR(KAN+) T7 SP6pDsRED1-C1 pDsRED-ex-C1-F pEGFP-N-3’(距离很近,一般不用) pDsRED2-C1(KAN+) pDsRED-ex-C1-F pEGFP-N-3’pDSRED-N1(KAN+) pEGFP-N-5’ PDSRED-N-RpECFP-C pEGFP-C-5’ pECFP-C-3′pECFP-N1 pEGFP-N-5’ pEGFP-N-3’pEF/myc/ER pEF-F pCDNA3.1RpEGFP-C(KAN+) pEGFP-C-5’ pEGFP-C-3′pEGFP-N(KAN+) pEGFP-N-5’ pEGFP-N-3’pENTR/D-TOPO(KAN+) M13F M13RpET-*(His)(KAN+) T7 T7 TerpET22(a,b,c)(AMP+) T7 T7 TerpET28,30,24,49(KAN+) T7 T7 TerpET32(a,b,c)(AMP+) T7/S.tag T7 TerpET50(amp+) T7/s.tag T7TERpET44(AMP+) T7/s.tag T7 TerpEYFP-N1 pEGFP-N-5’ pEGFP-N-3’pEYFP-C1 pEGFP-C-5’ pEGFP-C-3′pFLAG-CMV pFLAG-CMV -F pFLAG-CMV -R pGAD424 GAL4 AD 3′ADpGADGH GAL4 AD 3′ADpGADT7-Rec GAL4 AD/T7 3′ADpGAPZa-A a-FACTOR 3′AOXpGBKT7(Kana+) T7 3′BDpGEM-T(-Easy) M13F/T7 SP6/M13RpGEX-(*)T pGEX-4T-5’ pGEX-4T-3’pGL2 GLP1 GLP2pGL3 RVP3 GLP2(RVP4)(没有特别说明用GLP2) PinpointTM Vector Pinpoint Primer SP6pIRES PIRES-F/T7 T3/PIRES-R (J40720)pIRES2-EGFP pIRES2-EGFP.P5’ pIRES2-EGFP.P3’pLXSN pLXSN-F pLXSN-RpMAL-c2E MalE primer M13F(-47)pMAL-C2x P5’ P3’pMAL-p2X MalE primer M13F(-47)PMD18-T M13R(-48) M13F(-47)PMD19-T M13F(-47) M13R(-48)pPIC9K(AMP+、ZEO+)5’AOX/a-factor 3’AOX pPICZa 5′AOX/PPICZa-F 3′AOXpPROEXHTA M13R(-48) 无反向引物pQE30or40 pQE30-F pQE-R:pRSET T7 T7TERpSilencer3.1-H1 hygro M13F(-47) 3.0REV pSILENCEV2.0-U6 T7 2.0REVpSK01-T M13F M13RpSK-CMV(KAN+) T7 T3pSP72 T7 SP6pSport1 SP6/M13F(-47) T7/M13RpSUPER T7 T3pTA2 M13F/T7(优先使用M13F)T3/M13RpT-Adv M13F /T7 M13RpTARGET TM pTarget.F(在T7前面)/T7 pTarget.R pTO-T7 T7 T7TERpTRC99a-c pTRC99C-F pTRC99C-RpTriPLEx2 5′pTriPLEx2 T7pTWIN-1 T7 T7TER(客户确认再用)pUC18(19)/118(119) M13F(-47) M13R(-48)pUCm-T M13F/T7 M13RpVAX1 T7 BGHpWSK29 T7/M13F/M13F(-47) T3/M13R/M13R(-48)pXT7 T7 SP6pYES2 T7 pYes2.RpLEXA pLEXA-F pLEXA-RpLNCX pLNCX-F pLNCX-RpRECEIVER CMV-F 此载体无反向引物pSHUTTLE-CMV PSHUTTLE-CMV-F PSHUTTLE-CMV-R pSP64/65 SP6 此载体无反向引物pSTBlue-1 T7 SP6pT7Blue(R) T7 M13F(-47)引物名称序列(5′-3′):M13R:CAG GAA ACA GCT ATG ACCM13F:TGT AAA ACG ACG GCC AGTM13F(-47):CGC CAG GGT TTT CCC AGT CAC GACM13R(-48):AGC GGA TAA CAA TTT CAC ACA GGA M13(-96):CCC TCA TAG TTA GCG TAA CGSP6:ATT TAG GTG ACA CTA TAGT7:TAA TAC GAC TCA CTA TAG GGT7 terminator:TGC TAG TTA TTG CTC AGC GGT3:ATT AAC CCT CAC TAA AGG GApGEX-4T-5′:GGG CTG GCA AGC CAC GTT TGG TG pGEX-4T-3′:CCG GGA GCT GCA TGT GTC AGA GG GLp1:TGT ATC TTA TGG TAC TGT AAC TGGLp2:CTT TAT GTT TTT GGC GTC TTC CARVp3:CTA GCA AAA TAG GCT GTC CCRVp4:GAC GAT AGT CAT GCC CCG CGpcDNA3.1R:TAG AAG GCA CAG TCG AGGPinPoint primer:CGT GAC GCG GTG CAG GGC G pCMV-F:TCT AAA AGC TGC GGA ATT GTpCMV-R:TCCAAACTCATCAATGTATCpTRC99C-F: TTG CGC CGA CAT CAT AACpTRC99C-R: CTGCGTTCTGATTTAATCTGpCEP-F: AGA GCT CGT TTA GTG AAC CGEBV-R : GTG GTT TGT CCA AAC TCA TCpIRES2-EGFP.P5’: GTA GGC GTG TAC GGT GGG AG pIRES2-EGFP.P3’: AAC GCA CAC CGG CCT TAT TC3′AD: AGA TGG TGC ACG ATG CAC AGCMV -F:CGC AAA TGG GCG GTA GGC GTGS1:CAA CGT GAA AAA ATT ATT ATT CGCS6:GTA AAT GAA TTT TCT GTA GTA GG5`AOX1:GAC TGG TTC CAA TTG ACA AGC3`AOX1:GCA AAT GGC ATT CTG ACA TCCα-Factor:TAC TAT TGC CAG CAT TGC TGCGAL4 AD:TAC CAC TAC AAT GGA TGpACT2-R:GTGCACGATGCACAGTTGAApB42ADF:CCA GCC TCT TGC TGA GTG GAG ATGpB42ADR:AAG CCG ACA ACC TTG ATT GGA GpEGFP-N-5’:TGG GAG GTC TAT ATA AGC AGA G pEGFP-N-3’:CGT CGC CGT CCA GCT CGA CCA G pEGFP-C-5’:CAT GGT CCT GCT GGA GTT CGT G pEGFP-C-3′ :TAT GGC TGA TTA TGA TCA GTPBV220F:AAG AAG GGC AGC ATT CAA AGPBV220R:CTG CGT TCT GAT TTA ATC TGU6:ATG GAC TAT CAT ATG CTT ACC GTA2.0rev primer:AGG CGA TTA AGT TGG GTA3.0rev:GAG TTA GCT CAC TCA TTA GGCS.tag:GAA CGC CAG CAC ATG GAC5′TriplEx2:CTC CGA GAT CTG GAC GAG CRVP4:GAC GAT AGT CAT GCC CCG CGRVP3:CTA GCA AAA TAG GCT GTC CCpQE30-R:GTT CTG AGG TCA TTA CTG GpQE30-F:TGA GCG GAT AAC AAT TTC ACpEF-F:TCA AGC CTC AGA CAG TGG TTCpDSRED-N1-R:TGA AGC GCA TGA ACT CCT TG pDsRED-express-C1-F:TCC CAC AAC GAG GAC TAC AC pCMV5R:ATT ATA GAG GAC ACC TAG TCpCMV5F:TTC CAA AAT GTC GTA ATA ACP5′:TGC GTA CTG CGG TGA TCA ACP3′:CTG CAA GGC GAT TAA GTT GGBAC2:ACG CAC AGA ATC TAG CGC TTBAC1:AAC CAT CTC GCA AAT AAA TA3′BD:TAA GAG TCA CTT TAA AAT TTG TAT AC pTarget.F:CCA GGA TTT TCC CAG TCA CpTarget.R:GGC TTT ACA CTT TAT GCT TCT7(17base):ACA TCC ACT TTG CCT TTC TCpYes2.R:TCG GTT AGA GCG GAT GTGGAL4 BD:TCA TCG GAA GAG AGT AGpAS2-1.R:AAG AGT TAC TCA AGA ACA AGA ApFlag-CMV-F: GGT AGG CGT GTA CGG TGGpFlag-CMV-R: GCA CTG GAG TGG CAA CTTpIRES-F: CTT ACT GAC ATC CAC TTT GCpIRES.R: CAC TGC ATT CTA GTT GTG GTP1:CCA GGC TTT ACA CTT TAT GCP2:GCG ATT AAG TTG GGT AAC GCpLEXA-F:CGT CAG CAG AGC TTC ACC ATpLEXA-R:TAA AAC CTA AGA GTC ACT TTpLNCX-F:AGC TCG TTT AGT GAA CCG TCA GAT CG pLNCX-R:ACC TAC AGG TGG GGT CTT TCA TTC CC pLXSN-F:CTT GAA CCT CCT CGT TCG ACpLXSN-R:GTT GCT GAC TAA TTG AGA TGpSHUTTLE-CMV-F:GGT CTA TAT AAG CAG AGG TG pSHUTTLE-CMV-R:GTG GTA TGG CTG ATT ATG ATC AG MalE Primer:GGT CGT CAG ACT GTC GAT GAA GCC。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
常见载体的测序引物:Primer of Vector:Vector:Primer(F);Primer(R)pACT T7 T3pACT2 GAL4 AD pACT2-RpAS2-1 GAL4 BD pAS2-1.RpB42AD pB42ADF pB42ADRpBACPAK8 BAC1 BAC2pBK-CMV T7 T3PBS(SK/KS)/M13- M13F/T7 T3/M13RpBV220 PBV220F PBV220RpCAMBIA 1301(1300) P1 P2pCAMBIA 2300 M13R(-48) M13F(-47)PCANTB5E S1/M13R S6pCAT3-enhancer RVP3 此载体无反向引物pcDNA3.0 CMV-F/T7 SP6/BGHpcDNA3.1 T7 BGHpcDNA4 T7/CMV-F BGHpcDNA6 T7/CMV-F(J21025在T7前面)BGHpcDNAII T7 SP6pCE2.1 M13F M13RpCEP4 pCEP-F EBV-RpCF-T M13F M13RpCI T7(17Base)此载体无反向引物pCI-neo T7(17Base)T3pCMS-EGFP T7 T3pCMV-3Tag-4A T3 /PFLAG-CMV-F T7pCMV5 pCMV5F pCMV5RpCMV5-Flag pCMV5F pCMV5RpCMV-MYC/HA pCMV-F pCMV-RpCMV-Sport M13F/T7 SP6/M13RpCMV-Tag(KAN+) T3 T7pCR2.1-TOPO M13F/T7 M13RpCR3.1 T7 BGHpCS2 SP6 T7pDonar M13F M13RpDONR221 M13F M13RpDrive T7 SP6pDRIveR(KAN+) T7 SP6pDsRED1-C1 pDsRED-ex-C1-F pEGFP-N-3’(距离很近,一般不用) pDsRED2-C1(KAN+) pDsRED-ex-C1-F pEGFP-N-3’pDSRED-N1(KAN+) pEGFP-N-5’ PDSRED-N-RpECFP-C pEGFP-C-5’ pECFP-C-3′pECFP-N1 pEGFP-N-5’ pEGFP-N-3’pEF/myc/ER pEF-F pCDNA3.1RpEGFP-C(KAN+) pEGFP-C-5’ pEGFP-C-3′pEGFP-N(KAN+) pEGFP-N-5’ pEGFP-N-3’pENTR/D-TOPO(KAN+) M13F M13RpET-*(His)(KAN+) T7 T7 TerpET22(a,b,c)(AMP+) T7 T7 TerpET28,30,24,49(KAN+) T7 T7 TerpET32(a,b,c)(AMP+) T7/S.tag T7 TerpET50(amp+) T7/s.tag T7TERpET44(AMP+) T7/s.tag T7 TerpEYFP-N1 pEGFP-N-5’ pEGFP-N-3’pEYFP-C1 pEGFP-C-5’ pEGFP-C-3′pFLAG-CMV pFLAG-CMV -F pFLAG-CMV -R pGAD424 GAL4 AD 3′ADpGADGH GAL4 AD 3′ADpGADT7-Rec GAL4 AD/T7 3′ADpGAPZa-A a-FACTOR 3′AOXpGBKT7(Kana+) T7 3′BDpGEM-T(-Easy) M13F/T7 SP6/M13RpGEX-(*)T pGEX-4T-5’ pGEX-4T-3’pGL2 GLP1 GLP2pGL3 RVP3 GLP2(RVP4)(没有特别说明用GLP2) PinpointTM Vector Pinpoint Primer SP6pIRES PIRES-F/T7 T3/PIRES-R (J40720)pIRES2-EGFP pIRES2-EGFP.P5’ pIRES2-EGFP.P3’pLXSN pLXSN-F pLXSN-RpMAL-c2E MalE primer M13F(-47)pMAL-C2x P5’ P3’pMAL-p2X MalE primer M13F(-47)PMD18-T M13R(-48) M13F(-47)PMD19-T M13F(-47) M13R(-48)pPIC9K(AMP+、ZEO+)5’AOX/a-factor 3’AOX pPICZa 5′AOX/PPICZa-F 3′AOXpPROEXHTA M13R(-48) 无反向引物pQE30or40 pQE30-F pQE-R:pRSET T7 T7TERpSilencer3.1-H1 hygro M13F(-47) 3.0REV pSILENCEV2.0-U6 T7 2.0REVpSK01-T M13F M13RpSK-CMV(KAN+) T7 T3pSP72 T7 SP6pSport1 SP6/M13F(-47) T7/M13RpSUPER T7 T3pTA2 M13F/T7(优先使用M13F)T3/M13RpT-Adv M13F /T7 M13RpTARGET TM pTarget.F(在T7前面)/T7 pTarget.R pTO-T7 T7 T7TERpTRC99a-c pTRC99C-F pTRC99C-RpTriPLEx2 5′pTriPLEx2 T7pTWIN-1 T7 T7TER(客户确认再用)pUC18(19)/118(119) M13F(-47) M13R(-48)pUCm-T M13F/T7 M13RpVAX1 T7 BGHpWSK29 T7/M13F/M13F(-47) T3/M13R/M13R(-48)pXT7 T7 SP6pYES2 T7 pYes2.RpLEXA pLEXA-F pLEXA-RpLNCX pLNCX-F pLNCX-RpRECEIVER CMV-F 此载体无反向引物pSHUTTLE-CMV PSHUTTLE-CMV-F PSHUTTLE-CMV-R pSP64/65 SP6 此载体无反向引物pSTBlue-1 T7 SP6pT7Blue(R) T7 M13F(-47)引物名称序列(5′-3′):M13R:CAG GAA ACA GCT ATG ACCM13F:TGT AAA ACG ACG GCC AGTM13F(-47):CGC CAG GGT TTT CCC AGT CAC GACM13R(-48):AGC GGA TAA CAA TTT CAC ACA GGAM13(-96):CCC TCA TAG TTA GCG TAA CGSP6:ATT TAG GTG ACA CTA TAGT7:TAA TAC GAC TCA CTA TAG GGT7 terminator:TGC TAG TTA TTG CTC AGC GGT3:ATT AAC CCT CAC TAA AGG GApGEX-4T-5′:GGG CTG GCA AGC CAC GTT TGG TG pGEX-4T-3′:CCG GGA GCT GCA TGT GTC AGA GG GLp1:TGT ATC TTA TGG TAC TGT AAC TGGLp2:CTT TAT GTT TTT GGC GTC TTC CARVp3:CTA GCA AAA TAG GCT GTC CCRVp4:GAC GAT AGT CAT GCC CCG CGpcDNA3.1R:TAG AAG GCA CAG TCG AGGPinPoint primer:CGT GAC GCG GTG CAG GGC G pCMV-F:TCT AAA AGC TGC GGA ATT GTpCMV-R:TCCAAACTCATCAATGTATCpTRC99C-F: TTG CGC CGA CAT CAT AACpTRC99C-R: CTGCGTTCTGATTTAATCTGpCEP-F: AGA GCT CGT TTA GTG AAC CGEBV-R : GTG GTT TGT CCA AAC TCA TCpIRES2-EGFP.P5’: GTA GGC GTG TAC GGT GGG AG pIRES2-EGFP.P3’: AAC GCA CAC CGG CCT TAT TC3′AD: AGA TGG TGC ACG ATG CAC AGCMV -F:CGC AAA TGG GCG GTA GGC GTGS1:CAA CGT GAA AAA ATT ATT ATT CGCS6:GTA AAT GAA TTT TCT GTA GTA GG5`AOX1:GAC TGG TTC CAA TTG ACA AGC3`AOX1:GCA AAT GGC ATT CTG ACA TCCα-Factor:TAC TAT TGC CAG CAT TGC TGCGAL4 AD:TAC CAC TAC AAT GGA TGpACT2-R:GTGCACGATGCACAGTTGAApB42ADF:CCA GCC TCT TGC TGA GTG GAG ATGpB42ADR:AAG CCG ACA ACC TTG ATT GGA GpEGFP-N-5’:TGG GAG GTC TAT ATA AGC AGA G pEGFP-N-3’:CGT CGC CGT CCA GCT CGA CCA G pEGFP-C-5’:CAT GGT CCT GCT GGA GTT CGT G pEGFP-C-3′ :TAT GGC TGA TTA TGA TCA GTPBV220F:AAG AAG GGC AGC ATT CAA AGPBV220R:CTG CGT TCT GAT TTA ATC TGU6:ATG GAC TAT CAT ATG CTT ACC GTA2.0rev primer:AGG CGA TTA AGT TGG GTA3.0rev:GAG TTA GCT CAC TCA TTA GGCS.tag:GAA CGC CAG CAC ATG GAC5′TriplEx2:CTC CGA GAT CTG GAC GAG CRVP4:GAC GAT AGT CAT GCC CCG CGRVP3:CTA GCA AAA TAG GCT GTC CCpQE30-R:GTT CTG AGG TCA TTA CTG GpQE30-F:TGA GCG GAT AAC AAT TTC ACpEF-F:TCA AGC CTC AGA CAG TGG TTCpDSRED-N1-R:TGA AGC GCA TGA ACT CCT TG pDsRED-express-C1-F:TCC CAC AAC GAG GAC TAC AC pCMV5R:ATT ATA GAG GAC ACC TAG TCpCMV5F:TTC CAA AAT GTC GTA ATA ACP5′:TGC GTA CTG CGG TGA TCA ACP3′:CTG CAA GGC GAT TAA GTT GGBAC2:ACG CAC AGA ATC TAG CGC TTBAC1:AAC CAT CTC GCA AAT AAA TA3′B D:TAA GAG TCA CTT TAA AAT TTG TAT AC pTarget.F:CCA GGA TTT TCC CAG TCA CpTarget.R:GGC TTT ACA CTT TAT GCT TCT7(17base):ACA TCC ACT TTG CCT TTC TCpYes2.R:TCG GTT AGA GCG GAT GTGGAL4 BD:TCA TCG GAA GAG AGT AGpAS2-1.R:AAG AGT TAC TCA AGA ACA AGA ApFlag-CMV-F: GGT AGG CGT GTA CGG TGGpFlag-CMV-R: GCA CTG GAG TGG CAA CTTpIRES-F: CTT ACT GAC ATC CAC TTT GCpIRES.R: CAC TGC ATT CTA GTT GTG GTP1:CCA GGC TTT ACA CTT TAT GCP2:GCG ATT AAG TTG GGT AAC GCpLEXA-F:CGT CAG CAG AGC TTC ACC ATpLEXA-R:TAA AAC CTA AGA GTC ACT TTpLNCX-F:AGC TCG TTT AGT GAA CCG TCA GAT CG pLNCX-R:ACC TAC AGG TGG GGT CTT TCA TTC CC pLXSN-F:CTT GAA CCT CCT CGT TCG ACpLXSN-R:GTT GCT GAC TAA TTG AGA TGpSHUTTLE-CMV-F:GGT CTA TAT AAG CAG AGG TG pSHUTTLE-CMV-R:GTG GTA TGG CTG ATT ATG ATC AG MalE Primer:GGT CGT CAG ACT GTC GAT GAA GCC。
