pLVX-IRES-mCherry慢病毒载体使用说明
慢病毒使用操作指南

慢使用操作指南慢使用操作指南1.慢简介1.1 定义:慢是一种特殊的,能够在细胞中长期存活并进行复制。
1.2 用途:慢广泛应用于基因转导、基因敲除和基因表达等实验研究中。
2.慢使用前准备工作2.1 实验室准备工作2.1.1 实验室空间准备:确保实验室内有足够的操作空间和合适的消毒设备。
2.1.2 材料准备:准备好所需的培养基、细胞培养物、慢载体等。
2.1.3 设备准备:确保离心机、冰箱等设备正常工作,并准备好相关的仪器和器材。
2.2 人员准备工作2.2.1 人员培训:对参与慢操作的人员进行培训,了解操作步骤和安全风险。
2.2.2 个人防护:提供必要的防护装备,如实验服、手套、面罩等。
3.慢操作步骤3.1 细胞培养3.1.1 细胞培养物准备:根据实验需求选择合适的细胞培养物,并进行细胞的预处理和培养。
3.1.2 细胞密度调整:根据实验要求,调整细胞培养物的密度,以保证细胞的正常生长。
3.2 慢感染3.2.1 慢载体注射:将慢载体注射到培养好的细胞中。
3.2.2 感染条件控制:根据实验需求,控制慢感染的时间、浓度和温度等条件。
3.3 细胞培养和检测3.3.1 细胞培养:将感染好的细胞进行培养,并观察细胞的生长状态。
3.3.2 细胞检测:使用相关实验方法,对感染细胞进行检测和分析。
4.实验安全措施4.1 操作环境控制:确保实验室内通风良好,避免慢的扩散和污染。
4.2 废液处理:将产生的废液经过正确处理,避免对环境和人体造成污染和伤害。
4.3 事故应急处理:在发生事故或意外情况时,立即采取应急措施,并及时报告相关人员。
5.附件本文档所涉及的附件,包括但不限于实验记录表格、实验数据文件等。
6.法律名词及注释6.1 慢:指一种具有长周期和潜伏期的。
7.结束语感谢您阅读本文档,如有任何疑问或意见,请随时与我们联系。
慢病毒(过表达)包装步骤

慢病毒(过表达)包装步骤秦超1.转染复苏293T细胞,传2-3代进行转染,转染推荐使用合元公司的慢病毒转染试剂。
转染步骤:(以10cm培养皿为例)⑴最好在铺细胞后20h左右进行转染,控制转染前细胞密度70%—90%,保证细胞处于良好的状态,转染前一小时把一半培养基(约5ml)换成新的(含血清,因为此转染试剂不需换液)。
⑵加psin 10ug,pspax2 10ug,pmd2.g 5ug于800ul opti—mem,混匀⑶加40ul慢病毒转染试剂于800ul opti—mem,混匀,室温静置5min⑷将⑶所得的转染试剂稀释液滴加到⑵所得到的质粒稀释液中,边加边轻轻混匀,室温放置20min⑸取出细胞培养皿,将⑷得到的质粒转染试剂复合体加入到细胞培养基中,前后轻轻推摇使混合均匀,放回培养箱。
2.收毒(36—48h)收毒前如果质粒带有荧光标签可先看一下转染效率,一般达到60%即可.⑴将培养皿中的病毒上清液吸出到15cm离心管中,然后2000rpm离心10min,以沉淀细胞碎片。
⑵取上清用0.22um滤清过滤到浓缩管(用蛋白质浓缩管即可)中。
4000rpm离心至所需体积。
⑶浓缩完毕后,吸出浓缩后的病毒液,按每次的接毒量分装,-80℃冻存。
由于反复冻融会降低慢病毒滴度,因此避免反复冻融。
3.接毒接毒前12-20h铺细胞,使接毒时细胞密度约为40%—50%,务必使用生长状态良好的细胞。
将分装好的慢病毒滴加到细胞中,加polybrene使其终浓度为8ug/ml细胞密度60%—70%时可以再接毒一次。
4.检测及培养细胞系(48h)如果带有荧光标签可直接显微镜看一下感染效率,如需用药杀用puromycin杀三天(对照组完全杀死),剩下的即为基因整合进去的细胞。
如需培养成细胞系,可继续培养。
如果剩下的细胞较少可用高浓度血清,待细胞聚团时用胰酶消化一下,使细胞铺匀。
慢病毒生产及使用操作手册

3
地址:上海市徐汇区斜土路 1175 号景泰大厦 1503 实验室:上海市张江高科技园区蔡伦路 150 号 1 号楼 2 楼 邮箱:service@
400-092-0065 021-54121689
慢病毒生产及使用操作手册
当细胞生长到汇合率达到 80%~90%时需要对细胞进行传代操作,以扩大细 胞数量,维持细胞良好的生长状态。
(三)做脂转 complex DMEM 需在 37 度水浴中预热,LipFiterTM 转染试剂需恢复至室温方可使用,
使用前需摇匀。
转染每瓶 T75 的 complex 成分如下:
pspax
10μg
PMD2G pHBLVTM 系列载体
10μg 10μg
转染后 6h 换新鲜培液。
注:LipoFiterTM 转染试剂为汉恒生物产品,使用说明参考 LipoFiterTM 说明书。 LipoFiterTM 转染最适的细胞密度为 50%-70%。
慢病毒生产及使用操作手册
慢病毒生产及使用操作手册
一、实验流程 制备慢病毒穿梭质粒及其辅助包装原件载体质粒,三种质粒载体 分别进行高
纯度无内毒素抽提,共转染 293T 细胞,转染后 6 h 更换为完全培养基,培养 48 和 72h 后,分别收集富含慢病毒颗粒的细胞上清液,病毒上清液通过超离心浓缩 病毒。
2) 包装质粒信息如下: PSPAX2 及 PMD2G 载体图谱和序列信息 (购自 addgene)
2、细胞株 293T,慢病毒的包装细胞,为贴壁依赖型成上皮样细胞,生长培养 基为 DMEM(含 10% FBS)。贴壁细胞经培养生长增殖形成单层细胞。 3、菌株 大肠杆菌菌株 DH5α。用于扩增慢病毒载体和辅助包装载体质粒。
(二)传 293T 细胞
慢病毒使用操作指南

慢病毒使用操作指南一、慢病毒的储存与稀释:1. 病毒的储存:用户收到病毒液后在很短时间内即使用慢病毒进行实验,可以将病毒暂时放置于4 C保存;如需长期保存请放置于-80C(病毒置于冻存管,并使用封口膜封口)注:A.病毒可以存放于-80°C 6个月以上;但如果病毒储存时间超过6个月, 我们建议在使用前需要重新滴定病毒滴度。
B.反复冻融会降低病毒滴度:每次冻融会降低病毒滴度10%因此在病毒使用过程中应尽量避免反复冻融,为避免反复冻融我们强烈建议客户收到病毒后按照每次的使用量进行分装。
2. 病毒的稀释:用户需要稀释病毒时,请将病毒取出置于冰浴融解后,使用培养目的细胞用PBS或无血清培养基(含血清或含双抗不影响病毒感染)混匀分装后4C保存(请尽量在三天内用完),分装后使用。
、慢病毒用于体外(in vitro )实验:感染培养原代细胞和建系细胞1. 慢病毒对各种细胞和组织的亲嗜性不同,用户使用Invabio 提供的慢病毒之前可以通过查阅相关文献,了解慢病毒对您的目的细胞的亲嗜性,感染复数(MOI 值)以及在体内(in vivo )注射所需要的病毒量。
如果没有相关文献支持,可以通过感染预实验得到合适的感染复数(MOI 值)(使用24孔板检测病毒对目的细胞的亲嗜性)2. 慢病毒感染目的细胞预实验① 慢病毒感染目的细胞预实验注意事项A. 测定慢病毒对目的细胞的亲嗜性时,需要同时设置对慢病毒亲嗜性较高的(HEK293T,Hela)细胞作为平行实验的对照细胞。
B.在进行慢病毒感染实验时,可以用完全培养基(培养目的细胞用)稀释;理论上,含有血清、双抗或者其他营养因子的完全培养基不影响慢病毒的感染效率。
C. Invabio提供的病毒单位为TU/ml,即每毫升中含有具有生物活性的病毒颗粒数。
如:病毒滴度为>1X108 TU/ml即每毫升病毒液中至少含有1X108个具有生物活性的慢病毒颗粒。
② 以24 孔培养板为例,进行目的细胞和HEK293T 细胞的感染预实验实验前按照不同的MOI设置不同的感染孔,并根据MOI和细胞数量计算所需要的病毒量,如有必要可以使用PBS溶液或者无血清培养基稀释病毒原液第一天,准备细胞:在24孔培养板接种若干孔,每个孔内接种3~5X104个目的细胞,铺板时细胞的融合率为50%左右,每孔培养基体积为100卩l ;进行病毒感染时细胞的融合度约为70%左右。
慢病毒转染操作

一、慢病毒转染贴壁细胞实验方法
1、慢病毒转染前18-24小时,将贴壁细胞以1×10^5/孔铺到24孔板中。
使细胞在慢病毒转染时的数量为2×10^5/孔左右。
2、第二天,用含有6 μg/ml polybrene的2ml新鲜培养基替换原培养基,加入适量病毒悬液。
37℃孵育。
3、(对polybrene毒性敏感的细胞选作此步骤)4小时后加入2ml新鲜培养基以稀释polybrene。
4、继续培养24小时,用新鲜培养基替换含有病毒的培养基。
5、继续培养。
如果慢病毒含有荧光蛋白,一般转染48小时后可见明显荧光表达,72小时后更加明显。
如需FACS检测转染效率,可在转染后72-96小时进行。
如果慢病毒含有抗性基因并且需要加药筛选,可以在转染3-4天后开始加药。
二、慢病毒转染悬浮细胞实验方法
1、在2×10^5/ml悬浮细胞中加入polybrene至6 μg/ml和适量病毒,充分混匀。
37℃孵育。
或者150g室温离心4小时(选作,部分难转染的细胞系采用此步骤可以提高转染效率)。
2、(对polybrene毒性敏感的细胞选作此步骤)4小时后(或离心结束后)加入等体积新鲜培养基以稀释polybrene。
3、继续培养3-4天。
中间视细胞生长情况可传代或换液。
慢病毒载体构建和包装操作手册

慢病毒载体构建及包装操作手册目录慢病毒收到后的注意事项一、整体实验流程二、实验材料三、慢病毒包装和浓缩四、感染目的细胞附1. 汉恒生物慢病毒质粒列表附2. 慢病毒滴度测定方法简介附3. 慢病毒MOI感染参数附4. 汉恒生物各病毒载体感染目的细胞比较慢病毒安全使用和注意事项➢慢病毒安全使用注意事项(*非常重要!!!*)1)慢病毒相关实验请在生物安全柜(BL-2级别)内操作。
2)操作病毒时请穿实验服,佩戴口罩和手套,尽量不要裸露双手及手臂的皮肤。
3)操作病毒时特别小心病毒溅出。
如果操作时超净工作台有病毒污染,请立即用70%乙醇加1%的SDS溶液擦拭干净。
接触过病毒的枪头,离心管,培养板,培养液请于84消毒液浸泡后统一处理。
4)如需要离心,应使用密封性好的离心管,如有必要请用封口膜封口后离心。
5)病毒相关的废弃物需要特殊收集,统一经高温灭菌处理。
6)实验完毕用香皂清洗双手。
➢慢病毒收到后的注意事项1)慢病毒的储存用户收到病毒液后在短期内即使用慢病毒进行实验,可以将病毒暂时放置于4 ℃保存(尽量一周内用完);如需长期保存请分装后放置于-80℃。
注:a.反复冻融会降低病毒滴度(每次冻融会降低病毒滴度10%-50%);在病毒使用过程中应尽量避免反复冻融,所以我们前期对病毒进行了分装(200 l/tube),收到后直接放置-80℃保存即可。
b.如果病毒储存时间超过6个月,我们建议在使用前重新测定病毒滴度。
2)慢病毒的稀释用户需要稀释病毒时,请将病毒取出置于冰浴融解后,使用培养目的细胞用PBS或无血清培养基(含血清或含双抗不影响病毒感染)混匀分装后置于4℃保存(请尽量一周内用完)。
一、整体实验流程二、实验材料(一)慢病毒载体、包装细胞和菌株该病毒包装系统为三质粒系统,组成为psPAX2, pMD2.G, pHBLV TM系列质粒。
1、载体信息(见附表1)2、细胞株:我们采用293T作为慢病毒的包装细胞。
该细胞系为贴壁依赖型成上皮样细胞,生长培养基为DMEM+10% FBS+双抗。
pLVX-IRES-Neo慢病毒载体使用说明

pLVX-IRES-Neo pLVX-IRES-Neo载体基本信息:载体名称:pLVX-IRES-Neo, pLVX IRES Neo质粒类型: 慢病毒载体;哺乳动物细胞表达载体;双顺反子载体高拷贝/低拷贝: 高拷贝启动子: CMV克隆方法: 多克隆位点,限制性内切酶载体大小: 8269 bp5' 测序引物及序列: CMV-F: CGCAAATGGGCGGTAGGCGTG (Invitrogen)3' 测序引物及序列: IRES-R: CCTCACATTGCCAAAAGACG载体标签: 无标签载体抗性: 氨苄青霉素筛选标记: 新霉素Neomycin克隆菌株: Stbl3 E.coli备注: pLVX-IRES-Neo载体以双顺反子的形式同时表达Neo抗性基因和目的基因;CMV启动子驱动目的基因的过表达。
稳定性: 稳表达组成型: 组成型病毒/非病毒: 慢病毒pLVX-IRES-Neo载体质粒图谱和多克隆位点信息:pLVX-IRES-Neo载体序列:ORIGIN1 TGGAAGGGCT AATTCACTCC CAAAGAAGAC AAGATATCCT TGATCTGTGG ATCTACCACA61 CACAAGGCTA CTTCCCTGAT TAGCAGAACT ACACACCAGG GCCAGGGGTC AGATATCCAC121 TGACCTTTGG ATGGTGCTAC AAGCTAGTAC CAGTTGAGCC AGATAAGGTA GAAGAGGCCA 181 ATAAAGGAGA GAACACCAGC TTGTTACACC CTGTGAGCCT GCATGGGATG GATGACCCGG 241 AGAGAGAAGT GTTAGAGTGG AGGTTTGACA GCCGCCTAGC ATTTCATCAC GTGGCCCGAG 301 AGCTGCATCC GGAGTACTTC AAGAACTGCT GATATCGAGC TTGCTACAAG GGACTTTCCG 361 CTGGGGACTT TCCAGGGAGG CGTGGCCTGG GCGGGACTGG GGAGTGGCGA GCCCTCAGAT 421 CCTGCATATA AGCAGCTGCT TTTTGCCTGT ACTGGGTCTC TCTGGTTAGA CCAGATCTGA481 GCCTGGGAGC TCTCTGGCTA ACTAGGGAAC CCACTGCTTA AGCCTCAATA AAGCTTGCCT 541 TGAGTGCTTC AAGTAGTGTG TGCCCGTCTG TTGTGTGACT CTGGTAACTA GAGATCCCTC601 AGACCCTTTT AGTCAGTGTG GAAAATCTCT AGCAGTGGCG CCCGAACAGG GACTTGAAAG 661 CGAAAGGGAA ACCAGAGGAG CTCTCTCGAC GCAGGACTCG GCTTGCTGAA GCGCGCACGG 721 CAAGAGGCGA GGGGCGGCGA CTGGTGAGTA CGCCAAAAAT TTTGACTAGC GGAGGCTAGA 781 AGGAGAGAGA TGGGTGCGAG AGCGTCAGTA TTAAGCGGGG GAGAATTAGA TCGCGATGGG 841 AAAAAATTCG GTTAAGGCCA GGGGGAAAGA AAAAATATAA ATTAAAACAT ATAGTATGGG 901 CAAGCAGGGA GCTAGAACGA TTCGCAGTTA ATCCTGGCCT GTTAGAAACA TCAGAAGGCT 961 GTAGACAAAT ACTGGGACAG CTACAACCAT CCCTTCAGAC AGGATCAGAA GAACTTAGAT 1021 CATTATATAA TACAGTAGCA ACCCTCTATT GTGTGCATCA AAGGATAGAG ATAAAAGACA 1081 CCAAGGAAGC TTTAGACAAG ATAGAGGAAG AGCAAAACAA AAGTAAGACC ACCGCACAGC 1141 AAGCGGCCGG CCGCTGATCT TCAGACCTGG AGGAGGAGAT ATGAGGGACA ATTGGAGAAG 1201 TGAATTATAT AAATATAAAG TAGTAAAAAT TGAACCATTA GGAGTAGCAC CCACCAAGGC 1261 AAAGAGAAGA GTGGTGCAGA GAGAAAAAAG AGCAGTGGGA ATAGGAGCTT TGTTCCTTGG 1321 GTTCTTGGGA GCAGCAGGAA GCACTATGGG CGCAGCGTCA ATGACGCTGA CGGTACAGGC 1381 CAGACAATTA TTGTCTGGTA TAGTGCAGCA GCAGAACAAT TTGCTGAGGG CTATTGAGGC 1441 GCAACAGCAT CTGTTGCAAC TCACAGTCTG GGGCATCAAG CAGCTCCAGG CAAGAATCCT1501 GGCTGTGGAA AGATACCTAA AGGATCAACA GCTCCTGGGG ATTTGGGGTT GCTCTGGAAA 1561 ACTCATTTGC ACCACTGCTG TGCCTTGGAA TGCTAGTTGG AGTAATAAAT CTCTGGAACA 1621 GATTTGGAAT CACACGACCT GGATGGAGTG GGACAGAGAA ATTAACAATT ACACAAGCTT 1681 AATACACTCC TTAATTGAAG AATCGCAAAA CCAGCAAGAA AAGAATGAAC AAGAATTATT 1741 GGAATTAGAT AAATGGGCAA GTTTGTGGAA TTGGTTTAAC ATAACAAATT GGCTGTGGTA 1801 TATAAAATTA TTCATAATGA TAGTAGGAGG CTTGGTAGGT TTAAGAATAG TTTTTGCTGT 1861 ACTTTCTATA GTGAATAGAG TTAGGCAGGG ATATTCACCA TTATCGTTTC AGACCCACCT 1921 CCCAACCCCG AGGGGACCCG ACAGGCCCGA AGGAATAGAA GAAGAAGGTG GAGAGAGAGA 1981 CAGAGACAGA TCCATTCGAT TAGTGAACGG ATCTCGACGG TATCGCCTTT AAAAGAAAAG 2041 GGGGGATTGG GGGGTACAGT GCAGGGGAAA GAATAGTAGA CATAATAGCA ACAGACATAC 2101 AAACTAAAGA ATTACAAAAA CAAATTACAA AAATTCAAAA TTTTCGGGTT TATTACAGGG 2161 ACAGCAGAGA TCCAGTTTAT CGATAAGCTT GGGAGTTCCG CGTTACATAA CTTACGGTAA 2221 ATGGCCCGCC TGGCTGACCG CCCAACGACC CCCGCCCATT GACGTCAATA ATGACGTATG 2281 TTCCCATAGT AACGCCAATA GGGACTTTCC ATTGACGTCA ATGGGTGGAG TATTTACGGT 2341 AAACTGCCCA CTTGGCAGTA CATCAAGTGT ATCATATGCC AAGTACGCCC CCTATTGACG 2401 TCAATGACGG TAAATGGCCC GCCTGGCATT ATGCCCAGTA CATGACCTTA TGGGACTTTC 2461 CTACTTGGCA GTACATCTAC GTATTAGTCA TCGCTATTAC CATGGTGATG CGGTTTTGGC 2521 AGTACATCAA TGGGCGTGGA TAGCGGTTTG ACTCACGGGG ATTTCCAAGT CTCCACCCCA 2581 TTGACGTCAA TGGGAGTTTG TTTTGGCACC AAAATCAACG GGACTTTCCA AAATGTCGTA 2641 ACAACTCCGC CCCATTGACG CAAATGGGCG GTAGGCGTGT ACGGTGGGAG GTCTATATAA 2701 GCAGAGCTCG TTTAGTGAAC CGTCAGATCG CCTGGAGACG CCATCCACGC TGTTTTGACC 2761 TCCATAGAAG ACACCGACTC TACTAGAGGA TCTATTTCCG GTGAATTCCT CGAGACTAGT 2821 TCTAGAGCGG CCGCGGATCC CGCCCCTCTC CCTCCCCCCC CCCTAACGTT ACTGGCCGAA 2881 GCCGCTTGGA ATAAGGCCGG TGTGCGTTTG TCTATATGTT ATTTTCCACC ATATTGCCGT 2941 CTTTTGGCAA TGTGAGGGCC CGGAAACCTG GCCCTGTCTT CTTGACGAGC ATTCCTAGGG 3001 GTCTTTCCCC TCTCGCCAAA GGAATGCAAG GTCTGTTGAA TGTCGTGAAG GAAGCAGTTC 3061 CTCTGGAAGC TTCTTGAAGA CAAACAACGT CTGTAGCGAC CCTTTGCAGG CAGCGGAACC 3121 CCCCACCTGG CGACAGGTGC CTCTGCGGCC AAAAGCCACG TGTATAAGAT ACACCTGCAA 3181 AGGCGGCACA ACCCCAGTGC CACGTTGTGA GTTGGATAGT TGTGGAAAGA GTCAAATGGC 3241 TCTCCTCAAG CGTATTCAAC AAGGGGCTGA AGGATGCCCA GAAGGTACCC CATTGTATGG 3301 GATCTGATCT GGGGCCTCGG TGCACATGCT TTACATGTGT TTAGTCGAGG TTAAAAAAAC 3361 GTCTAGGCCC CCCGAACCAC GGGGACGTGG TTTTCCTTTG AAAAACACGA TGATAAGCTT 3421 GCCACAACCA TGGCTGAACA AGATGGATTG CACGCAGGTT CTCCGGCCGC TTGGGTGGAG 3481 AGGCTATTCG GCTATGACTG GGCACAACAG ACAATCGGCT GCTCTGATGC CGCCGTGTTC 3541 CGGCTGTCAG CGCAGGGGCG CCCGGTTCTT TTTGTCAAGA CCGACCTGTC CGGTGCCCTG 3601 AATGAACTGC AGGACGAGGC AGCGCGGCTA TCGTGGCTGG CCACGACGGG CGTTCCTTGC 3661 GCAGCTGTGC TCGACGTTGT CACTGAAGCG GGAAGGGACT GGCTGCTATT GGGCGAAGTG 3721 CCGGGGCAGG ATCTCCTGTC ATCTCACCTT GCTCCTGCCG AGAAAGTATC CATCATGGCT 3781 GATGCAATGC GGCGGCTGCA TACGCTTGAT CCGGCTACCT GCCCATTCGA CCACCAAGCG 3841 AAACATCGCA TCGAGCGAGC ACGTACTCGG ATGGAAGCCG GTCTTGTCGA TCAGGATGAT 3901 CTGGACGAAG AGCATCAGGG GCTCGCGCCA GCCGAACTGT TCGCCAGGCT CAAGGCGCGC 3961 ATGCCCGACG GCGAGGATCT CGTCGTGACC CATGGCGATG CCTGCTTGCC GAATATCATG 4021 GTGGAAAATG GCCGCTTTTC TGGATTCATC GACTGTGGCC GGCTGGGTGT GGCGGACCGC 4081 TATCAGGACA TAGCGTTGGC TACCCGTGAT ATTGCTGAAG AGCTTGGCGG CGAATGGGCT4141 GACCGCTTCC TCGTGCTTTA CGGTATCGCC GCTCCCGATT CGCAGCGCAT CGCCTTCTAT 4201 CGCCTTCTTG ACGAGTTCTT CTGAACGCGT CTGGAACAAT CAACCTCTGG ATTACAAAAT 4261 TTGTGAAAGA TTGACTGGTA TTCTTAACTA TGTTGCTCCT TTTACGCTAT GTGGATACGC 4321 TGCTTTAATG CCTTTGTATC ATGCTATTGC TTCCCGTATG GCTTTCATTT TCTCCTCCTT4381 GTATAAATCC TGGTTGCTGT CTCTTTATGA GGAGTTGTGG CCCGTTGTCA GGCAACGTGG 4441 CGTGGTGTGC ACTGTGTTTG CTGACGCAAC CCCCACTGGT TGGGGCATTG CCACCACCTG 4501 TCAGCTCCTT TCCGGGACTT TCGCTTTCCC CCTCCCTATT GCCACGGCGG AACTCATCGC 4561 CGCCTGCCTT GCCCGCTGCT GGACAGGGGC TCGGCTGTTG GGCACTGACA ATTCCGTGGT 4621 GTTGTCGGGG AAGCTGACGT CCTTTCCATG GCTGCTCGCC TGTGTTGCCA CCTGGATTCT 4681 GCGCGGGACG TCCTTCTGCT ACGTCCCTTC GGCCCTCAAT CCAGCGGACC TTCCTTCCCG 4741 CGGCCTGCTG CCGGCTCTGC GGCCTCTTCC GCGTCTTCGC CTTCGCCCTC AGACGAGTCG 4801 GATCTCCCTT TGGGCCGCCT CCCCGCCTGG AATTAATTCT GCAGTCGAGA CCTAGAAAAA 4861 CATGGAGCAA TCACAAGTAG CAATACAGCA GCTACCAATG CTGATTGTGC CTGGCTAGAA 4921 GCACAAGAGG AGGAGGAGGT GGGTTTTCCA GTCACACCTC AGGTACCTTT AAGACCAATG 4981 ACTTACAAGG CAGCTGTAGA TCTTAGCCAC TTTTTAAAAG AAAAGAGGGG ACTGGAAGGG 5041 CTAATTCACT CCCAACGAAG ACAAGATATC CTTGATCTGT GGATCTACCA CACACAAGGC 5101 TACTTCCCTG ATTAGCAGAA CTACACACCA GGGCCAGGGG TCAGATATCC ACTGACCTTT 5161 GGATGGTGCT ACAAGCTAGT ACCAGTTGAG CCAGATAAGG TAGAAGAGGC CAATAAAGGA 5221 GAGAACACCA GCTTGTTACA CCCTGTGAGC CTGCATGGGA TGGATGACCC GGAGAGAGAA 5281 GTGTTAGAGT GGAGGTTTGA CAGCCGCCTA GCATTTCATC ACGTGGCCCG AGAGCTGCAT 5341 CCGGAGTACT TCAAGAACTG CTGATATCGA GCTTGCTACA AGGGACTTTC CGCTGGGGAC 5401 TTTCCAGGGA GGCGTGGCCT GGGCGGGACT GGGGAGTGGC GAGCCCTCAG ATCCTGCATA 5461 TAAGCAGCTG CTTTTTGCCT GTACTGGGTC TCTCTGGTTA GACCAGATCT GAGCCTGGGA 5521 GCTCTCTGGC TAACTAGGGA ACCCACTGCT TAAGCCTCAA TAAAGCTTGC CTTGAGTGCT 5581 TCAAGTAGTG TGTGCCCGTC TGTTGTGTGA CTCTGGTAAC TAGAGATCCC TCAGACCCTT 5641 TTAGTCAGTG TGGAAAATCT CTAGCAGTAG TAGTTCATGT CATCTTATTA TTCAGTATTT 5701 ATAACTTGCA AAGAAATGAA TATCAGAGAG TGAGAGGCCT TGACATTGCT AGCGTTTACC 5761 GTCGACCTCT AGCTAGAGCT TGGCGTAATC ATGGTCATAG CTGTTTCCTG TGTGAAATTG 5821 TTATCCGCTC ACAATTCCAC ACAACATACG AGCCGGAAGC ATAAAGTGTA AAGCCTGGGG 5881 TGCCTAATGA GTGAGCTAAC TCACATTAAT TGCGTTGCGC TCACTGCCCG CTTTCCAGTC 5941 GGGAAACCTG TCGTGCCAGC TGCATTAATG AATCGGCCAA CGCGCGGGGA GAGGCGGTTT 6001 GCGTATTGGG CGCTCTTCCG CTTCCTCGCT CACTGACTCG CTGCGCTCGG TCGTTCGGCT 6061 GCGGCGAGCG GTATCAGCTC ACTCAAAGGC GGTAATACGG TTATCCACAG AATCAGGGGA 6121 TAACGCAGGA AAGAACATGT GAGCAAAAGG CCAGCAAAAG GCCAGGAACC GTAAAAAGGC 6181 CGCGTTGCTG GCGTTTTTCC ATAGGCTCCG CCCCCCTGAC GAGCATCACA AAAATCGACG 6241 CTCAAGTCAG AGGTGGCGAA ACCCGACAGG ACTATAAAGA TACCAGGCGT TTCCCCCTGG 6301 AAGCTCCCTC GTGCGCTCTC CTGTTCCGAC CCTGCCGCTT ACCGGATACC TGTCCGCCTT 6361 TCTCCCTTCG GGAAGCGTGG CGCTTTCTCA TAGCTCACGC TGTAGGTATC TCAGTTCGGT 6421 GTAGGTCGTT CGCTCCAAGC TGGGCTGTGT GCACGAACCC CCCGTTCAGC CCGACCGCTG 6481 CGCCTTATCC GGTAACTATC GTCTTGAGTC CAACCCGGTA AGACACGACT TATCGCCACT 6541 GGCAGCAGCC ACTGGTAACA GGATTAGCAG AGCGAGGTAT GTAGGCGGTG CTACAGAGTT 6601 CTTGAAGTGG TGGCCTAACT ACGGCTACAC TAGAAGAACA GTATTTGGTA TCTGCGCTCT 6661 GCTGAAGCCA GTTACCTTCG GAAAAAGAGT TGGTAGCTCT TGATCCGGCA AACAAACCAC 6721 CGCTGGTAGC GGTGGTTTTT TTGTTTGCAA GCAGCAGATT ACGCGCAGAA AAAAAGGATC6781 TCAAGAAGAT CCTTTGATCT TTTCTACGGG GTCTGACGCT CAGTGGAACG AAAACTCACG6841 TTAAGGGATT TTGGTCATGA GATTATCAAA AAGGATCTTC ACCTAGATCC TTTTAAATTA6901 AAAATGAAGT TTTAAATCAA TCTAAAGTAT ATATGAGTAA ACTTGGTCTG ACAGTTACCA6961 ATGCTTAATC AGTGAGGCAC CTATCTCAGC GATCTGTCTA TTTCGTTCAT CCATAGTTGC7021 CTGACTCCCC GTCGTGTAGA TAACTACGAT ACGGGAGGGC TTACCATCTG GCCCCAGTGC7081 TGCAATGATA CCGCGAGACC CACGCTCACC GGCTCCAGAT TTATCAGCAA TAAACCAGCC7141 AGCCGGAAGG GCCGAGCGCA GAAGTGGTCC TGCAACTTTA TCCGCCTCCA TCCAGTCTAT7201 TAATTGTTGC CGGGAAGCTA GAGTAAGTAG TTCGCCAGTT AATAGTTTGC GCAACGTTGT7261 TGCCATTGCT ACAGGCATCG TGGTGTCACG CTCGTCGTTT GGTATGGCTT CATTCAGCTC7321 CGGTTCCCAA CGATCAAGGC GAGTTACATG ATCCCCCATG TTGTGCAAAA AAGCGGTTAG7381 CTCCTTCGGT CCTCCGATCG TTGTCAGAAG TAAGTTGGCC GCAGTGTTAT CACTCATGGT7441 TATGGCAGCA CTGCATAATT CTCTTACTGT CATGCCATCC GTAAGATGCT TTTCTGTGAC7501 TGGTGAGTAC TCAACCAAGT CATTCTGAGA ATAGTGTATG CGGCGACCGA GTTGCTCTTG7561 CCCGGCGTCA ATACGGGATA ATACCGCGCC ACATAGCAGA ACTTTAAAAG TGCTCATCAT7621 TGGAAAACGT TCTTCGGGGC GAAAACTCTC AAGGATCTTA CCGCTGTTGA GATCCAGTTC7681 GATGTAACCC ACTCGTGCAC CCAACTGATC TTCAGCATCT TTTACTTTCA CCAGCGTTTC7741 TGGGTGAGCA AAAACAGGAA GGCAAAATGC CGCAAAAAAG GGAATAAGGG CGACACGGAA7801 ATGTTGAATA CTCATACTCT TCCTTTTTCA ATATTATTGA AGCATTTATC AGGGTTATTG7861 TCTCATGAGC GGATACATAT TTGAATGTAT TTAGAAAAAT AAACAAATAG GGGTTCCGCG7921 CACATTTCCC CGAAAAGTGC CACCTGACGT CGACGGATCG GGAGATCAAC TTGTTTATTG7981 CAGCTTATAA TGGTTACAAA TAAAGCAATA GCATCACAAA TTTCACAAAT AAAGCATTTT8041 TTTCACTGCA TTCTAGTTGT GGTTTGTCCA AACTCATCAA TGTATCTTAT CATGTCTGGA8101 TCAACTGGAT AACTCAAGCT AACCAAAATC ATCCCAAACT TCCCACCCCA TACCCTATTA8161 CCACTGCCAA TTACCTGTGG TTTCATTTAC TCTAAACCTG TGATTCCTCT GAATTATTTT8221 CATTTTAAAG AAATTGTATT TGTTAAATAT GTACTACAAA CTTAGTAGT//pLVX-IRES-Neo其他相关慢病毒载体:Tet-pLKO-neo Tet-pLKO-puro pPACKH1-GAGpMD2.G pCMV-dR8.2-dvpr pLKO.1-GFP-shRNA pLKO.1-TRC control pLKO.1-hygro pLKO.1-TRCpCDH-MSCV-MCS-EF1-copGFP pCDH-MSCV-MCS-EF1-copGFP-T2A-Puro FUW-tetO-hOKMSFUW-tetO-hOCT4 FUW-tetO-hSOX2 FUW-tetO-hKLF4FUW pLVX-AcGFP1-N1 pLVX-AcGFP1-C1pLVX-AmCyan1-N1 pLVX-DsRed-Express2-C1 pLVX-DsRed-Express2-N1 pLVX-DsRed-Monomer-N1 pLVX-PAmCherry-C1 pLVX-PAmCherry-N1 pLVX-ZsGreen1-N1 pLVX-IRES-ZsGreen1 pLVX-IRES-mCherry pLVX-mCherry-C1 pLVX-mCherry-N1 pLVX-tdTomato-C1 pLKO.1-puro pLentilox 3.7 pLVX-Tet-On-Advanced pLVX-IRES-Puro pLVX-IRES-Neo pLVX-IRES-HygpLVX-EF1α-DsRed-Monomer-C1 pLVX-EF1α-AcGFP1-N1 pLVX-EF1α-AcGFP1-C1 pLVX-EF1α-mCherry-C1 pLVX-EF1α-IRES-mCherry pLVX-EF1α-IRES-ZsGreen1 pLVX-MetLuc Control pLVX-MetLuc pLVX-Hom-Mem1pLVX-Het-2 pLVX-DD-AcGFP1-Actin pPRIME-TET-GFP-FF3pSIH1-H1-CopGFP pCDH-EF1-MCS-T2A-Puro pCDH-CMV-MCS-EF1-Puro pCDF1-MCS2-EF1-copGFP pLOX-CWBmi1 pLOX-CW-CREpRSV-rev pMDLg-pRRE pLL3.7pLVX-DD-AmCyan1 Control pLVX-DD-AmCyan1 Reporter pLVX-DD-tdTomato Reporter pLVX-DD-tdTomato Control pLVX-PTuner-Green pLVX-CherryPicker2pLVX-TetOne-Puro-Luc pLVX-TetOne pLVX-TetOne-PuropLVX-TetOne-Luc pLVX-rHom-Nuc1 pLVX-rHom-Sec1pLVX-rHom-1 pLVX-Hom-Nuc1 pLVX-Het-Nuc1pLVX-PTuner pLVX-PTuner2 pLVX-DD-ZsGreen1 Reporter pLVX-Het-1 pLVX-CherryPicker Control pLVX-Tet3GpCDH-CMV-MCS-EF1-RFP-T2A-Puro pCDH-CMV-MCS-EF1-Hygro pCDH-CMV-MCS-EF1-Neo pCDH-MCS-T2A-Puro-MSCV pCDH1-MCS2-EF1-copGFP pCDF1-MCS2-EF1-Puro pCDH-EF1-MCS-T2A-copGFP pWPXL pLVX-TRE3G-ZsGreen1 pLVX-TRE3G-mCherry pLenti6.3-EmGFP-BveI miR pLenti6/V5-GW/lacZpLenti6.3/V5-GW/EmGFP pLenti6.3-MCS pLenti6.3-DsRed2-BveI miR pLenti6.3-MCS-IRES2-EGFP pLVX-shRNA2 psPAX2VSV-G pSico PGK Puro pcDNA6.2-DsRed2-MCS1 miR pcDNA6.3-EmGFP-NC- II pcDNA6.2-EmGFP-NC- I pcDNA6.2-EmGFP-BsaI miR pLenti6.3-BveI miR pLenti6.3-MCS-IRES2-DsRed2 pLEX-MCSpGIPZ pLP2 pLP1FUGW pFUGW pLOX-Ttag-iresTKpMDLg/pRRE pLentG-KOSM pCMV-dR8.91pLVX-TRE3G-Luc Control pLVX-TRE3G-IRES pCgpvpSico pSicoR pLVTHMpGensil-1 pLVX-EF1α-IRES-Puro pCDF1-MCS2-EF1-copGFP pPACKH1-REV pLVX-Het-Mem1 pLVX-shRNA1pLKO.1-puro-GFP-siRNA pPRIME-TREX-GFP-FF3 pcDNA6.2-DsRed2-BsmBI miR pCDH-MSCV-MCS-EF1-Puro pCDH-CMV-MCS-EF1-copGFP pLVX-TRE3GFUW-tetO-hMYC pLOX-TERT-iresTK pLP/VSVGFUW-M2rtTA pCDH-EF1-MCS-(PGK-Puro) pcDNA6.2-EmGFP-MCS1 miR pLVX-AmCyan1-C1 pLVX-Hom-1 pcDNA6.2-BsaI miRpLVX-DsRed-Monomer-C1 pLVX-mCherry-Actin pTRIPZpLVX-ZsGreen1-C1 pLVX-CherryPicker1 LeGO-iC2pLVX-IRES-tdTomato pCDH-CMV-MCS-EF1-copGFP-T2A-Puro pLKO.3GpLVX-tdTomato-N1 pLVX-PTuner2-C pLVX-PuropLVX-Tight-Puro pLVX-DD-ZsGreen1 Control pSicoR PGK PuropLVX-EF1α-DsRed-Monomer-N1 pCDH-UbC-MCS-EF1-Hygro pLVTHpLVX-EF1α-mCherry-N1 pCDH-CMV-MCS-EF1-RFP。
慢病毒使用操作指南

慢病毒使用操作指南慢病毒是一种常用的实验工具,广泛应用于细胞和分子生物学研究。
本文档旨在提供慢病毒使用的操作指南,帮助研究人员更好地利用慢病毒进行实验研究。
第一部分:慢病毒基本知识1. 什么是慢病毒?慢病毒是一种具有RNA基因组的病毒,属于反转录病毒。
它能够将自身的RNA基因组逆转录成DNA,再与宿主细胞的基因组融合,长期稳定地存在于宿主细胞中。
2. 利用慢病毒进行基因转染的优势相比其他常用的基因转染方法,慢病毒具有以下优势:- 高效性:慢病毒能够高效地转染多种类型的细胞,包括非分裂细胞。
- 长期稳定性:慢病毒转染的基因能够长期稳定地存在于宿主细胞中。
- 遗传稳定性:慢病毒转染的基因可以遗传给后代细胞,因此适用于长期实验研究。
第二部分:慢病毒使用的关键步骤1. 选择合适的慢病毒载体慢病毒载体是慢病毒的基础构建单元,其中包含转录启动子、报告基因等必要的元件。
根据实验需要选择合适的载体。
2. 包装慢病毒慢病毒的包装是将慢病毒载体与包装载体共转染至特定细胞株,通过包装载体中的包装酶,将慢病毒的RNA基因组逆转录成DNA,形成可复制的慢病毒。
3. 提取慢病毒经包装后的细胞培养基中含有包装好的慢病毒。
可通过超速离心等方法,将细胞培养物离心,提取慢病毒。
4. 病毒滴定病毒滴定是用来确定病毒滴度的重要步骤。
通过适当稀释提取的慢病毒,将其感染指定细胞株,计算出感染单位的浓度。
5. 慢病毒感染及筛选将提取的慢病毒添加至目标细胞中,通过细胞培养和筛选,筛选出具有所需基因的细胞株。
第三部分:慢病毒使用的注意事项1. 安全操作慢病毒具有一定的传染性,进行实验时需要遵守生物安全操作规范,佩戴一次性手套、口罩等个人防护装备,避免直接接触慢病毒。
2. 避免交叉感染实验室中使用慢病毒时,应尽可能避免交叉感染。
定期对实验室环境进行消毒,使用一次性材料,避免共享器械等措施可以减少交叉感染的风险。
3. 合理控制病毒滴度对于不同类型的细胞,需要确定合适的病毒滴度,以避免过度感染或感染不足的情况。
pLEX-MCS慢病毒载体使用说明

pLEX-MCS pLEX-MCS载体基本信息:载体名称:pLEXMCS, pLEX MCS, pLEX-MCS质粒类型: 哺乳动物细胞慢病毒表达载体高拷贝/低拷贝: 高拷贝启动子: CMV克隆方法: 多克隆位点,限制性内切酶载体大小: 10682 bp5' 测序引物及序列: pLEX-MCS-FWD: CACCAAAATCAACGGGACTT3' 测序引物及序列: pLEX-MCS-REV: ATATAGACAAACGCACACCGGCCT 载体标签: --载体抗性: 氨苄/博来霉素(Zeocin)筛选标记: 嘌呤霉素(Puromycin)备注:稳定性: 稳定组成型: 诱导型病毒/非病毒: 慢病毒pLEX-MCS载体质粒图谱和多克隆位点信息:pLEX-MCS载体简介:pLEX-MCS载体是慢病毒载体,带有一个多克隆位点。
pLEXMCS载体骨架是设计用来瞬转或稳转表达目的慢病毒,进而感染目的细胞系,导致你的目的基因过表达的载体。
pLEX-MCS载体拥有多个载体特征(具体见上文),这些载体特征使得载体能够有效进行过表达基因研究。
1.具有使用复制的非竞争性慢病毒转染或者转导的能力。
2.在体内和体外使用时都比较容易控制。
3.在进构建稳转细胞系的时候,可以使用嘌呤霉素进行筛选。
4.可以方便将任何目的基因导入到载体的多克隆位点处。
pLEX-MCS载体序列pLEX-MCS其他相关慢病毒载体:Tet-pLKO-neo Tet-pLKO-puro pPACKH1-GAGpMD2.G pCMV-dR8.2-dvpr pLKO.1-GFP-shRNA pLKO.1-TRC control pLKO.1-hygro pLKO.1-TRCpCDH-MSCV-MCS-EF1-copGFP pCDH-MSCV-MCS-EF1-copGFP-T2A-Puro FUW-tetO-hOKMSFUW-tetO-hOCT4 FUW-tetO-hSOX2 FUW-tetO-hKLF4FUW pLVX-AcGFP1-N1 pLVX-AcGFP1-C1pLVX-AmCyan1-N1 pLVX-DsRed-Express2-C1 pLVX-DsRed-Express2-N1 pLVX-DsRed-Monomer-N1 pLVX-PAmCherry-C1 pLVX-PAmCherry-N1pLVX-ZsGreen1-N1 pLVX-IRES-ZsGreen1 pLVX-IRES-mCherrypLVX-mCherry-C1 pLVX-mCherry-N1 pLVX-tdTomato-C1 pLKO.1-puro pLentilox 3.7 pLVX-Tet-On-Advanced pLVX-IRES-Puro pLVX-IRES-Neo pLVX-IRES-HygpLVX-EF1α-DsRed-Monomer-C1 pLVX-EF1α-AcGFP1-N1 pLVX-EF1α-AcGFP1-C1 pLVX-EF1α-mCherry-C1 pLVX-EF1α-IRES-mCherry pLVX-EF1α-IRES-ZsGreen1 pLVX-MetLuc Control pLVX-MetLuc pLVX-Hom-Mem1pLVX-Het-2 pLVX-DD-AcGFP1-Actin pPRIME-TET-GFP-FF3pSIH1-H1-CopGFP pCDH-EF1-MCS-T2A-Puro pCDH-CMV-MCS-EF1-Puro pCDF1-MCS2-EF1-copGFP pLOX-CWBmi1 pLOX-CW-CREpRSV-rev pMDLg-pRRE pLL3.7pLVX-DD-AmCyan1 Control pLVX-DD-AmCyan1 Reporter pLVX-DD-tdTomato Reporter pLVX-DD-tdTomato Control pLVX-PTuner-Green pLVX-CherryPicker2pLVX-TetOne-Puro-Luc pLVX-TetOne pLVX-TetOne-PuropLVX-TetOne-Luc pLVX-rHom-Nuc1 pLVX-rHom-Sec1pLVX-rHom-1 pLVX-Hom-Nuc1 pLVX-Het-Nuc1pLVX-PTuner pLVX-PTuner2 pLVX-DD-ZsGreen1 Reporter pLVX-Het-1 pLVX-CherryPicker Control pLVX-Tet3GpCDH-CMV-MCS-EF1-RFP-T2A-Puro pCDH-CMV-MCS-EF1-Hygro pCDH-CMV-MCS-EF1-Neo pCDH-MCS-T2A-Puro-MSCV pCDH1-MCS2-EF1-copGFP pCDF1-MCS2-EF1-Puro pCDH-EF1-MCS-T2A-copGFP pWPXL pLVX-TRE3G-ZsGreen1 pLVX-TRE3G-mCherry pLenti6.3-EmGFP-BveI miR pLenti6/V5-GW/lacZpLenti6.3/V5-GW/EmGFP pLenti6.3-MCS pLenti6.3-DsRed2-BveI miR pLenti6.3-MCS-IRES2-EGFP pLVX-shRNA2 psPAX2VSV-G pSico PGK Puro pcDNA6.2-DsRed2-MCS1 miR pcDNA6.3-EmGFP-NC- II pcDNA6.2-EmGFP-NC- I pcDNA6.2-EmGFP-BsaI miR pLenti6.3-BveI miR pLenti6.3-MCS-IRES2-DsRed2 pLEX-MCSpGIPZ pLP2 pLP1FUGW pFUGW pLOX-Ttag-iresTKpMDLg/pRRE pLentG-KOSM pCMV-dR8.91pLVX-TRE3G-Luc Control pLVX-TRE3G-IRES pCgpvpSico pSicoR pLVTHMpGensil-1 pLVX-EF1α-IRES-Puro pCDF1-MCS2-EF1-copGFP pPACKH1-REV pLVX-Het-Mem1 pLVX-shRNA1pLKO.1-puro-GFP-siRNA pPRIME-TREX-GFP-FF3 pcDNA6.2-DsRed2-BsmBI miR pCDH-MSCV-MCS-EF1-Puro pCDH-CMV-MCS-EF1-copGFP pLVX-TRE3GFUW-tetO-hMYC pLOX-TERT-iresTK pLP/VSVGFUW-M2rtTA pCDH-EF1-MCS-(PGK-Puro) pcDNA6.2-EmGFP-MCS1 miR pLVX-AmCyan1-C1 pLVX-Hom-1 pcDNA6.2-BsaI miRpLVX-DsRed-Monomer-C1 pLVX-mCherry-Actin pTRIPZpLVX-ZsGreen1-C1 pLVX-CherryPicker1 LeGO-iC2pLVX-IRES-tdTomato pCDH-CMV-MCS-EF1-copGFP-T2A-Puro pLKO.3GpLVX-tdTomato-N1 pLVX-PTuner2-C pLVX-PuropLVX-Tight-Puro pLVX-DD-ZsGreen1 Control pSicoR PGK PuropLVX-EF1α-DsRed-Monomer-N1 pCDH-UbC-MCS-EF1-Hygro pLVTHpLVX-EF1α-mCherry-N1 pCDH-CMV-MCS-EF1-RFP。
慢病毒使用操作手册

慢病毒使用操作手册:1 慢病毒使用操作2 慢病毒安全使用规范3 悬浮细胞感染方法概要4 相关专业术语(详情可参考公司网站FAQ)5 细胞培养器皿的相关参数1、慢病毒使用操作手册1.1 慢病毒的储存与稀释:1.1.1 病毒的储存:用户收到病毒液后在很短时间内即使用慢病毒进行实验,可以将病毒暂时放置于4 ℃保存;如需长期保存请放置于-80℃(病毒置于冻存管,并使用封口膜封口)A.病毒可以存放于-80℃ 6个月以上;但如果病毒储存时间超过6个月,我们建议在使用前需要重新滴定病毒滴度B.反复冻融会降低病毒滴度:每次冻融会降低病毒滴度10%;因此在病毒使用过程中应仅尽量避免反复冻融,为避免反复冻融我们强烈建议客户收到病毒后按照每次的使用量进行分装。
1.1.2 病毒的稀释:用户需要稀释病毒时,请将病毒取出置于冰浴融解后,使用培养目的细胞用PBS或无血清培养基(含血清或含双抗不影响病毒感染)混匀分装后4℃保存(请尽量在三天内用完) 分装后使用。
1.2 慢病毒用于体外(In Vitro)实验:感染培养原代细胞和建系细胞1.2.1 慢病毒对各种细胞和组织的亲嗜性不同,用户使用Invabio提供的慢病毒之前可以通过查阅相关文献,了解慢病毒对您的目的细胞的亲嗜性,感染复数(MOI 值)以及在体(In Vivo)注射所需要的病毒量。
如果没有相关文献支持,可以通过感染预实验得到合适的感染复数(MOI 值)(使用24孔板检测病毒对目的细胞的亲嗜性)1.2.2 慢病毒感染目的细胞预实验1.2.2.1 慢病毒感染目的细胞预实验注意事项A.测定慢病毒对目的细胞的亲嗜性时,需要同时设置对慢病毒亲嗜性较高的细胞(HEK293T,Hela)作为平行实验的对照细胞。
B.在进行慢病毒感染实验时,可以用完全培养基(培养目的细胞用)稀释;理论上,含有血清,双抗或者其他营养因子的完全培养基不影响慢病毒的感染效率。
C. Invabio提供的病毒单位为TU/ml, 即每毫升中含有具有生物活性的病毒颗粒数。
pLVX-Myc-MCS-Strep-IRES-Puro使用说明

pRSV-Rev pMDLg/pRRE pLVX-shRNA2-Puro pLKO.1 GFP shRNA pHIV-H2BmRFP pGreen-Puro pUMVC pOTENT-CMV-Flag-His-Puro pCDF1-MCS2-EF1-Puro pCDH-EF1-luc2-T2A-Tdtomato pCDH-EF1-MCS-T2A-copGFP pCDH-EF1-MCS-T2A-Puro pCDH-CMV-MCS-EF1-copGFP pGenesil-1 pCMV-VSV-G pLVX-Tet3G pLVX-TRE3G-Zsgreen1 pLVX-TRE3G pLP/VSVG pLP2 pLP1 pCMV-dR8.91 psPAX2 pLJM1 pLKO.1-Puro pLKO.1-TRC pLKO.1 pLVX-DsRed-Monomer-N1 pLVX-AcGFP1-N1 pLVX-EF1α-IRES-puro pLVX-EF1α-IRES-mCherry pLVX-IRES-mCherry pLVX-IRES-Neo pLVX-IRES-ZsGreen1 pLVX-IRES-Puro pLVX-shRNA2 pLVX-shRNA1 pLVX-Puro
pLVX-Myc-MCS-Strep-IRES-Puro 载体其他相关的逆病毒载体: pLVX-Tight-Puro pMD2.G pCDH-CMV-MCS-EF1-CopGFP-T2A-Puro pLVX-IRES-tdTomato pRRLSIN.CPPT.PGK-GFP.WPRE pLenti-Bi-cistronic pLKO.1-EGFP pLVX-Tet-On-Advanced pLVX-Myc-MCS-Strep-IRES-Puro pLVTHM pTRIPZ pLKO.1-Hygro TRC2-pLKO.5-Puro
ips操作指南第五版-慢病毒版本

IPS操作指南第四版IPS简介:iPS细胞是通过基因转染技术(gene transfection)将某些转录因子导入动物或入的体细胞使,体细胞直接重构成为胚胎干细胞(embryonic stemcell,ESC)细胞样的多潜能细胞。
现在大多是通过病毒携带特定的转录因子进入体细胞内,来获得多能性干细胞。
2006年日本京都大学Shinya Yamanaka在世界著名学术杂志《细胞》上率先报道了诱导多能干细胞的研究。
他们把Oct3/4,Sox2、c-Myc和Klf4这四种转录因子基因克隆入病毒载体,然后引入小鼠成纤维细胞,发现可诱导其发生转化,产生的iPS细胞在形态、基因和蛋白表达、表观遗传修饰状态、细胞倍增能力、类胚体和畸形瘤生成能力、分化能力等方面都与胚胎干细胞相似。
现分别介绍一下小鼠的和人的iPS的诱导步骤。
一、实验材料慢病毒包装体系:慢病毒表达质粒plvx-OCT4、plvx-SOX2、plvx-KLF4和plvx-c-MYC;辅助质粒PSPAX2和PMD2;病毒包装细胞:293T细胞;ES Feeder细胞:MEF(C57)。
二、实验仪器和耗材(一)实验仪器二级安全柜、37度培养箱、4度冰箱、液氮储存器、倒置显微镜、低温离心机。
(二)实验耗材PBS、无菌水、明胶粉、DMEM培养基、FBS、非必须氨基酸、β-巯基乙醇、胰酶、血清替代物(SR)、mTESR1、T75培养瓶、无菌水、15ml离心管、50ml离心管,巴氏管等。
三、ips诱导方法(一)Feeder细胞1、Feeder细胞的分离(MEF)取妊娠13.5-14.5d的孕鼠,在无菌情况下取出胎鼠,去除鼠头,尾,四肢及内脏,然后用PBS进行冲洗。
将胎体剪碎成小于1mm2的组织块,1000rpm,3min,去掉上层PBS,加入0.25%胰酶-0.04%EDTA2~3ml浸泡组织块,常温消化2~3min。
轻微吹打,待较多细胞溢出后,加入等量的10%FBS DMEM终止消化,通过100目滤网过滤后,将获得的液体离心,1000rpm,5min。
慢病毒包装体系使用说明

慢病毒包装体系使用说明本说明书适用于以下产品:名称货号慢病毒包装体系KLV3501慢病毒包装体系(含293V细胞)KLV3502慢病毒包装体系(含转染试剂)KLV3503慢病毒包装体系(含293V细胞、转染试剂)KLV3504北京英茂盛业生物科技有限公司Web site:1北京英茂盛业生物科技有限公司/产品内容KLV3501KLV3502KLV3503KLV3504慢病毒载体(过表达或RNA 干扰载体任选一种) 3333辅助载体pH1 3 3 3 3 辅助载体pH2 3 3 3 3 HEK293V 细胞 3 3 Polyfect-V 转染试剂33载体采用质粒形式发货,请在收到质粒后放-20℃冻存,也可以直接转化大肠杆菌感受态进行质粒扩增。
HEK293V 细胞采用干冰或培养瓶发货。
请在收到细胞后根据附带说明书进行复苏或传代。
慢病毒载体慢病毒载体中含有病毒整合和表达所需原件及表达外源目的基因的元件。
外源基因通过载体中的多克隆位点插入慢病毒载体中进行表达。
pLV-EGFP-C 的载体图谱见下,本公司的其它慢病毒载体结构与之基本相似。
其它载体信息见本公司网站 或本说明书后面的附表。
慢病毒包装载体慢病毒包装载体包括pH1和pH2,表达生产病毒颗粒所需的病毒蛋白。
载体图谱见下:HEK293V细胞包装细胞的状态对病毒包装效果有直接影响。
我公司保存的293V细胞为低次代293V细胞,细胞性状稳定。
在高密度下生长3天仍可保持贴壁状态,持续产生病毒颗粒,因此可多次收获病毒,降低病毒包装实验成本。
Polyfect‐V转染试剂Polyfect-V转染试剂专为293V细胞转染及慢病毒包装研制,可以在细胞铺板同时进行转染,缩短病毒包装时间;无需要求细胞处于生长对数期,细胞转染时密度可以很高;细胞毒性极低;质粒和转染试剂用量是普通转染试剂的1/3到1/2等显著优点;包装病毒时转染效率接近100%,能提高病毒产量3-5倍。
3北京英茂盛业生物科技有限公司/慢病毒包装概述:病毒包装前需制备转染级慢病毒载体及两种包装载体。
pLVX-DD-AmCyan1Control慢病毒载体使用说明

pLVX-DD-AmCyan1Control慢病毒载体使⽤说明pLVX-DD-AmCyan1 ControlpLVX-DD-AmCyan1 Control载体基本信息:载体名称: pLVX-DD-AmCyan1 Control质粒类型: 慢病毒载体;荧光报告载体⾼拷贝/低拷贝: ⾼拷贝克隆⽅法: 限制性内切酶,多克隆位点启动⼦: CMV IE载体⼤⼩: 9128 bp5' 测序引物及序列: T7 Forward: 5’-TAATACGACTCACTATAGGG-3’3' 测序引物及序列: TK polyA Reverse: 5’-CTTCCGTGTTTCAGTTAGC-3’载体标签: --载体抗性: 氨苄青霉素筛选标记: 嘌呤霉素(Puromycin)克隆菌株: Stbl3宿主细胞(系): 常规细胞系,293、CV-1、CHO等备注: pLVX-DD-AmCyan1 Control 载体是阳性对照载体,需要与pLVX-DD-AmCyan1 Reporter载体共同使⽤;组成型表达去稳定的青⾊荧光蛋⽩AmCyan1;DD-AmCyan1与AmCyan1相⽐,N端融合了ProteoTuner destabilization domain (DD)去稳定结构域,导致AmCyan1在细胞内很快被蛋⽩酶降解,降低了背景荧光,是研究启动⼦活性的理想报告基因。
稳定性: 稳表达组成型/诱导型: 组成型病毒/⾮病毒: 慢病毒pLVX-DD-AmCyan1 Control载体质粒图谱和多克隆位点信息:pLVX-DD-AmCyan1 Control载体简介:pLVX-DD-AmCyan1 Control 载体描述pLVX-DD-AmCyan1 Control constitutively expresses the destabilized cyan fluorescent protein DD-AmCyan1. The vector can be used as a control to monitor ligand-dependent stabilization of DD-AmCyan1 in your cell-type of interest.The Lenti-X DD-AmCyan1 Vector Set (available as part of the Lenti-X DD Cyan Reporter System; Cat. No. 631748) includes two HIV-1-based, lentiviral expression vectors that can efficiently transduce both dividing and nondividing mammalian cells. This reporter set can be used to monitor promoter activity in live cells and in vivo. pLVX-DD-AmCyan1 Reporter is a promoterless vector that can be used to monitor transcription from different promoters and promoter/enhancer combinations inserted into the multiple cloning site (MCS). The gene downstream of the MCS encodes the cyan fluorescent protein DD-AmCyan1, amodified version of AmCyan1 that is tagged on its N-terminus with the ProteoTuner? destabilization domain (DD; 1). In the absence of the Shield1 ligand, the DD tag induces rapid degradation of the fluorescent reporter, minimizing any background caused by leaky promoters; but upon addition of Shield1 at the time of promoter activation, the DD-tagged reporter molecules are stabilized, increasing the signal-to-noise ratio.pLVX-DD-AmCyan1 Control drives reporter expression via a constitutive promoter, and thereby serves as a positive control. DD-AmCyan1 ReporterAmCyan1 (excitation and emission maxima: 453 and 486, respectively) is a humancodon-optimized variant of the wildtype Anemonia majano cyan fluorescent protein (AmCyan) that exhibits enhanced emission characteristics (2, 3). DDAmCyan1 is tagged on its N-terminus with the ProteoTuner DD, which causes rapid, proteasomal degradation of DDAmCyan1. However, when the membrane-permeant, stabilizing ligand Shield1 is added to themedium, it binds to the DD and prevents degradation of the DD-AmCyan1 reporter protein, thereby causing it to accumulate inside the cell.In the absence of the stabilizing ligand Shield1, the DD causes the degradation of anyDD-AmCyan1 reporter protein produced prior to promoter activation, thus minimizing background fluorescence caused by leaky promoters. To analyze promoter activity, the inducer of choice is added to the medium along with Shield1, which effectively stabilizes the reporter protein, allowing it to accumulate. As a result, only the reporter molecules expressed during promoter induction will contribute to the fluorescence signal, providing a considerably higher signal-to-noise ratio than that obtained with non-destabilized or constitutively destabilized reporter systems. For both vectors, the promoter’s activity level can be directly correlated to the fluorescence level.Lentiviral ElementsThe reporter and control vectors each contain all of the viral processing elements necessary for the production of replication-incompetent lentivirus, as well as elements to improve viral titer, transgene expression, and overall vector function. The woodchuck hepatitis virus posttranscriptional regulatory element (WPRE) promotes RNA processing events and enhances nuclear export of viral and transgene RNA (4), leading to increased viral titers from packaging cells, and enhanced expression of your gene of interest in target cells. In addition, each vector includes a Rev-response element (RRE), which further increases viral titers by enhancing the transport of unspliced viral RNA out of the nucleus (5). Finally, each vector also contains a central polypurine tract/central termination sequence element (cPPT/CTS). During target cell infection, this element creates a central DNA flap that increases nuclear import of the viral genome, resulting in improved vector integration and more efficient transduction (6). Lentiviral particles derived from the vectors allow you to monitor your promoter of interest in virtually any cell type, even primary cells.Antibiotic SelectionIn addition to lentiviral elements, the reporter and control vectors each contain a puromycin resistance gene (Puror) under the control of the murine phosphoglycerate kinase promoter (PPGK) for the selection of stable transductants. The vectors also contain pUC origins of replication and E. coli ampicillin resistance genes (Ampr) for propagation and selection in bacteriaPropagation in E. coliRecommended host strains: DH5α and other general purpose strains.Selectable marker: plasmid confers resistance to ampicillin (100 µg/ml) in E. coli hosts.E. coli replication origin: pUCCopy number: highExcitation and Emission Maxima of AmCyan1Excitation: 453 nmEmission: 486 nmReferences1. Banaszynski, L. et al. (2006) Cell 126(5):995–1004.2. Matz, M. V. et al. (1999) Nature Biotech. 17(10):969–973.3. Haas, J. et al. (1996) Curr. Biol. 6(3):315-324.4. Zufferey, R. et al. (1999) J. Virol. 73(4):2886–28925. Cochrane, A. W. et al. (1990) Proc. Natl. Acad. Sci. USA 87(3):1198–1202.6. Zennou, V. et al. (2000) Cell 101(2):173–185.pLVX-DD-AmCyan1 Control载体序列pLVX-DD-AmCyan1 Control其他相关慢病毒载体:Tet-pLKO-neo Tet-pLKO-puro pPACKH1-GAGpMD2.G pCMV-dR8.2-dvpr pLKO.1-GFP-shRNA pLKO.1-TRC control pLKO.1-hygro pLKO.1-TRCpCDH-MSCV-MCS-EF1-copGFP pCDH-MSCV-MCS-EF1-copGFP-T2A-Puro FUW-tetO-hOKMSFUW-tetO-hOCT4 FUW-tetO-hSOX2 FUW-tetO-hKLF4FUW pLVX-AcGFP1-N1 pLVX-AcGFP1-C1pLVX-AmCyan1-N1 pLVX-DsRed-Express2-C1 pLVX-DsRed-Express2-N1 pLVX-DsRed-Monomer-N1 pLVX-PAmCherry-C1 pLVX-PAmCherry-N1pLVX-ZsGreen1-N1 pLVX-IRES-ZsGreen1 pLVX-IRES-mCherrypLVX-mCherry-C1 pLVX-mCherry-N1 pLVX-tdTomato-C1 pLKO.1-puro pLentilox 3.7 pLVX-Tet-On-Advanced pLVX-IRES-Puro pLVX-IRES-Neo pLVX-IRES-HygpLVX-EF1α-DsRed-Monomer-C1 pLVX-EF1α-AcGFP1-N1 pLVX-EF1α-AcGFP1-C1 pLVX-EF1α-mCherry-C1 pLVX-EF1α-IRES-mCherry pLVX-EF1α-IRES-ZsGreen1 pLVX-MetLuc Control pLVX-MetLuc pLVX-Hom-Mem1pLVX-Het-2 pLVX-DD-AcGFP1-Actin pPRIME-TET-GFP-FF3pSIH1-H1-CopGFP pCDH-EF1-MCS-T2A-Puro pCDH-CMV-MCS-EF1-Puro pCDF1-MCS2-EF1-copGFP pLOX-CWBmi1 pLOX-CW-CREpRSV-rev pMDLg-pRRE pLL3.7pLVX-DD-AmCyan1 Control pLVX-DD-AmCyan1 Reporter pLVX-DD-tdTomato Reporter pLVX-DD-tdTomato Control pLVX-PTuner-Green pLVX-CherryPicker2pLVX-TetOne-Puro-Luc pLVX-TetOne pLVX-TetOne-PuropLVX-TetOne-Luc pLVX-rHom-Nuc1 pLVX-rHom-Sec1pLVX-rHom-1 pLVX-Hom-Nuc1 pLVX-Het-Nuc1pLVX-PTuner pLVX-PTuner2 pLVX-DD-ZsGreen1 ReporterpLVX-Het-1 pLVX-CherryPicker Control pLVX-Tet3GpCDH-CMV-MCS-EF1-RFP-T2A-Puro pCDH-CMV-MCS-EF1-Hygro pCDH-CMV-MCS-EF1-Neo pCDH-MCS-T2A-Puro-MSCV pCDH1-MCS2-EF1-copGFP pCDF1-MCS2-EF1-Puro pCDH-EF1-MCS-T2A-copGFP pWPXL pLVX-TRE3G-ZsGreen1 pLVX-TRE3G-mCherry pLenti6.3-EmGFP-BveI miR pLenti6/V5-GW/lacZpLenti6.3/V5-GW/EmGFP pLenti6.3-MCS pLenti6.3-DsRed2-BveI miR pLenti6.3-MCS-IRES2-EGFP pLVX-shRNA2 psPAX2 VSV-G pSico PGK Puro pcDNA6.2-DsRed2-MCS1 miR pcDNA6.3-EmGFP-NC- II pcDNA6.2-EmGFP-NC- I pcDNA6.2-EmGFP-BsaI miR pLenti6.3-BveI miR pLenti6.3-MCS-IRES2-DsRed2 pLEX-MCSpGIPZ pLP2 pLP1FUGW pFUGW pLOX-Ttag-iresTKpMDLg/pRRE pLentG-KOSM pCMV-dR8.91pLVX-TRE3G-Luc Control pLVX-TRE3G-IRES pCgpvpSico pSicoR pLVTHMpGensil-1 pLVX-EF1α-IRES-Puro pCDF1-MCS2-EF1-copGFP pPACKH1-REV pLVX-Het-Mem1 pLVX-shRNA1pLKO.1-puro-GFP-siRNA pPRIME-TREX-GFP-FF3 pcDNA6.2-DsRed2-BsmBI miR pCDH-MSCV-MCS-EF1-Puro pCDH-CMV-MCS-EF1-copGFP pLVX-TRE3GFUW-tetO-hMYC pLOX-TERT-iresTK pLP/VSVGFUW-M2rtTA pCDH-EF1-MCS-(PGK-Puro) pcDNA6.2-EmGFP-MCS1 miR pLVX-AmCyan1-C1 pLVX-Hom-1 pcDNA6.2-BsaI miRpLVX-DsRed-Monomer-C1 pLVX-mCherry-Actin pTRIPZpLVX-ZsGreen1-C1 pLVX-CherryPicker1 LeGO-iC2pLVX-IRES-tdTomato pCDH-CMV-MCS-EF1-copGFP-T2A-Puro pLKO.3G pLVX-tdTomato-N1 pLVX-PTuner2-C pLVX-PuropLVX-Tight-Puro pLVX-DD-ZsGreen1 Control pSicoR PGK PuropLVX-EF1α-DsRed-Monomer-N1 pCDH-UbC-MCS-EF1-Hygro pLVTH pLVX-EF1α-mCherry-N1 pCDH-CMV-MCS-EF1-RFP。
慢病毒使用操作指南

慢病毒使用操作指南1. 引言1.1 目的本文档旨在提供关于慢病毒使用的详细操作指南,以确保实验室人员能够正确、安全地处理和利用该种类病毒。
2. 概述2.1 定义与特点- 慢病毒是一类具有复制缓激活机制并且感染后可长期存在于寄主体内的RNA或DNA 症候群相关性(SAR)等多个领域中发挥重要作用。
3. 实验前准备工作在进行任何涉及到潜在危险生物材料如此类型之前, 快速评估风险,并采取适当措施来最小化这些风险。
4 . 材料清单:下面列出了完成所需任务时可能需要的基本设备和试剂: - 生物安全柜级别II (BSL-2) 或更高级别环境下进行所有步骤;- 防护手套、防护眼镜/面罩和实验服;...5 . 实验流程:进行以下步骤以成功执行您对操纵蠕虫样品库存的需求:5.1 准备工作- 确保实验室环境符合慢病毒操作所需要的生物安全级别;- 检查所有设备和试剂是否齐全并处于良好状态。
5.2 培养基准备:...6 . 安全注意事项与风险评估7 . 应急措施及处理方法8 . 相关法律名词及注释:在本文档中,以下是一些常见使用到的法律名词以及其相应解释说明。
a) 生物安全柜 (Biosafety Cabinet, BSC):是用来提供对人员、产品和环境进行防护,并且在其中进行微生物学或细胞培养等活动时,提供无菌条件下能够有效地限制有害因素扩散传播而又不影响正常运行过程。
9. 结束语本文档涵盖了详尽的慢病毒使用操作指南,旨在确保实验室人员正确理解并遵循相关规定以最大程度上降低任何可能存在的危险性。
10. 文档涉及附件:请参考随信发送之文件列表。
慢病毒载体包装构建过程
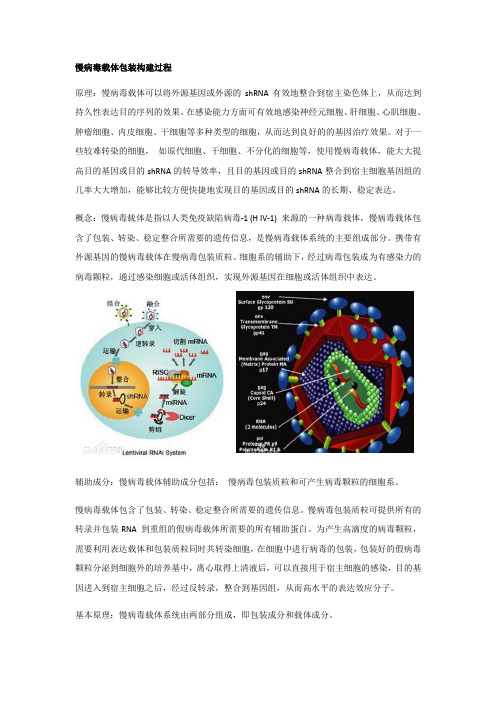
慢病毒载体包装构建过程原理:慢病毒载体可以将外源基因或外源的shRNA有效地整合到宿主染色体上,从而达到持久性表达目的序列的效果。
在感染能力方面可有效地感染神经元细胞、肝细胞、心肌细胞、肿瘤细胞、内皮细胞、干细胞等多种类型的细胞,从而达到良好的的基因治疗效果。
对于一些较难转染的细胞,如原代细胞、干细胞、不分化的细胞等,使用慢病毒载体,能大大提高目的基因或目的shRNA的转导效率,且目的基因或目的shRNA整合到宿主细胞基因组的几率大大增加,能够比较方便快捷地实现目的基因或目的shRNA的长期、稳定表达。
概念:慢病毒载体是指以人类免疫缺陷病毒-1 (H IV-1) 来源的一种病毒载体,慢病毒载体包含了包装、转染、稳定整合所需要的遗传信息,是慢病毒载体系统的主要组成部分。
携带有外源基因的慢病毒载体在慢病毒包装质粒、细胞系的辅助下,经过病毒包装成为有感染力的病毒颗粒,通过感染细胞或活体组织,实现外源基因在细胞或活体组织中表达。
辅助成分:慢病毒载体辅助成分包括:慢病毒包装质粒和可产生病毒颗粒的细胞系。
慢病毒载体包含了包装、转染、稳定整合所需要的遗传信息。
慢病毒包装质粒可提供所有的转录并包装RNA 到重组的假病毒载体所需要的所有辅助蛋白。
为产生高滴度的病毒颗粒,需要利用表达载体和包装质粒同时共转染细胞,在细胞中进行病毒的包装,包装好的假病毒颗粒分泌到细胞外的培养基中,离心取得上清液后,可以直接用于宿主细胞的感染,目的基因进入到宿主细胞之后,经过反转录,整合到基因组,从而高水平的表达效应分子。
基本原理:慢病毒载体系统由两部分组成,即包装成分和载体成分。
包装成分:由HIV-1基因组去除了包装、逆转录和整合所需的顺式作用序列而构建,能够反式提供产生病毒颗粒所必需的蛋白。
包装成分通常被分开构建到两个质粒上,一个质粒表达Gag和Pol蛋白,另一个质粒表达Env蛋白,其目的也是降低恢复成野生型病毒的可能。
将包装成分与载体成分的3个质粒共转染细胞(如人肾293T细胞),即可在细胞上清中收获只有一次性感染能力而无复制能力的、携带目的基因的HIV-1载体颗粒。
慢病毒包装试剂盒说明书

YRGene 慢病毒包装试剂盒说明书产品编号:LPK010 产品规格:10个10cm 皿 产品简介:赢润生物的慢病毒包装试剂盒包括如下成分:(1) 优化配比的慢病毒包装辅助质粒混合物,可兼容大多数慢病毒表达载体。
(2) 高效转染试剂(Invitrogen Lip2000原装产品分装,293T 细胞转染效率接近 100%)。
(3 )高效率的慢病毒浓缩液,无需超速离心,也不需要价格昂贵的过滤柱,快速富集病毒粒子,其优 势在于操作安全简单,对设备要求低,产毒效率高,能够快速、高效地收获高滴度病毒。
产品组成:1. 转染前,传代 293FT 细胞于10cm 培养皿中(例如,接种 1 x 107细胞于10cm 培养皿中,使用完全 培养基DMEM+10%FBS培养),当细胞密度能够达到 90-95%即可进行转染。
Tips :培养基里面不要添加抗生素。
2. 转染前1-3小时,更换培养基,加入7ml 新鲜的完全培养基(DMEM+10%FBS ),注意不要添加抗生素。
3. 准备转染。
在5ml 离心管中,分别配制 A 管与B 管试剂(Tube A and Tube B )配好后,放置5min ,然后将A 管缓慢加入B 管,混合均匀。
室温放置20min ,使得脂质体-DNA 混合物形成。
Tips :混合后可能会出现淡淡的乳白状,不会影响转染。
然后将混合液逐滴均匀加入 10cm 培养皿,轻微混匀。
置于37C, 5% CO ?培养箱中培养过夜。
4. 第三天,更换培养基,加入 10ml 的完全培养基,同样注意不要加抗生素。
5. 转染后48-72h 后收取上清,转移至 15ml 离心管。
Tips :上清里面含有病毒,请小心操作。
6. 3000rpm 在4C 离心15min ,去除沉淀。
7. 上清液用0.45卩m 滤器过滤后转移到新的离心管中。
慢病毒浓缩:1. 每10ml 过滤后的病毒初始液,加入 Concen Solution 3ml ,每20-30min 混合一次,共进行 3-5次。
慢病毒操作资料说明

慢病毒操作资料说明慢病毒操作资料说明1、Hexadimethrine bromide.pdf:这是Hexadimethrine bromide(别名Polybrene)的说明书,该试剂用于提高慢病毒的感染效率,在病毒滴度测定和病毒感染实验中均需使用;2、Lenti-X™ Concentrator.pdf:这是Lenti-X Concentrator的说明书,该试剂用于病毒粒子的浓缩纯化;3、Lenti-X™ Lentiviral Expression Systems User Manual.pdf:这是过表达慢病毒(用于基因过表达)的操作说明书4、Lenti-X™ shRNA Expression Systems User Manual.pdf:这是干扰慢病毒(用于基因干扰)的操作说明书5、pLVX-shRNA1 Vector Information.pdf:pLVX-shRNA1慢病毒干扰载体说明书6、pLVX-IRES-Neo Vector Information.pdf:pLVX-IRES-Neo 慢病毒过表达载体说明书7、慢病毒(Lentivirus)载体.pdf:慢病毒载体介绍8、Lenti-X™ Lentiviral Packaging Systems FAQs.pdf:慢病包装系统常见问题介绍9、病毒纯化-PEG6000.doc:病毒纯化方法10、病毒滴度测定-针对没有绿色荧光蛋白标记的病毒.doc:病毒滴度测定-针对没有绿色荧光蛋白标记的病毒;11、病毒滴度测定-针对有绿色荧光蛋白标记的病毒.doc:病毒滴度测定-针对有绿色荧光蛋白标记的病毒;12、Production, concentration and titration of pseudotyped HIV-1-based lentiviral vectors.pdf:慢病毒包装、纯化、滴度测定操作指南,供理论学习;13、C0508磷酸钙法细胞转染试剂盒.pdf:磷酸钙转染方法;14、慢病毒实验方法.doc:慢病毒包装报告单;15、慢病毒包装、纯化、滴度测定及感染.doc:慢病毒包装、纯化、滴度测定及感染操作指南;。
pLVX-DD-AmCyan1 Reporter慢病毒载体使用说明

pLVX-DD-AmCyan1 Reporter pLVX-DD-AmCyan1 Reporter载体基本信息:载体名称:pLVX-DD-AmCyan1 Reporter质粒类型: 慢病毒载体;荧光报告载体高拷贝/低拷贝: 高拷贝克隆方法: 限制性内切酶,多克隆位点启动子: 无载体大小: 8494 bp5' 测序引物及序列: --3' 测序引物及序列: --载体标签: 青色荧光蛋白DD-AmCyan1载体抗性: 氨苄青霉素筛选标记: 嘌呤霉素(Puromycin)克隆菌株: Stbl3宿主细胞(系): 常规细胞系,293、CV-1、CHO等备注: pLVX-DD-AmCyan1 Reporter载体没有启动子;DD-AmCyan1与AmCyan1相比,N端融合了ProteoTuner destabilization domain (DD)去稳定结构域,导致AmCyan1在细胞内很快被蛋白酶降解,降低了背景荧光,是研究启动子活性的理想报告基因。
稳定性: 稳表达组成型/诱导型: --病毒/非病毒: 慢病毒pLVX-DD-AmCyan1 Reporter载体质粒图谱和多克隆位点信息:pLVX-DD-AmCyan1 Reporter载体简介:pLVX-DD-AmCyan1 Reporter is a promoterless reporter vector that allows the functional analysis of different promotersand promoter/enhancer combinations inserted into its multiple cloning site (MCS). The vector encodes the reporter proteinDD-AmCyan1, a ligand-dependent, destabilized cyan fluorescent protein that minimizes background fluorescence fromleaky promoters. A promoter must be cloned into the MCS, located upstream of the DD-AmCyan1 coding sequence.Without the addition of a functional promoter, the vector will not expressDD-AmCyan1.Location of Features5' LTR: 1–635PBS (primer binding site): 636–653Ψ (packaging signal): 685–822RRE (Rev-response element): 1303–1536cPPT/CTS (central polypurine tract/central termination sequence): 2028–2151MCS (multiple cloning site): 2195–2234DD (FKBP-L106P destabilization domain): 2247–2570AmCyan1 (Anemonia majano cyan fluorescent protein): 2577–3263PPGK (phosphoglycerate kinase promoter): 3274–3782Puror(puromycin resistance gene): 3803–4402WPRE (woodchuck hepatitis virus posttranscriptional regulatory element): 4416–5007 3' LTR: 5211–5847pUC origin of replication: 6317–6987 (complementary)Ampr(am picillin resistance gene; β-lactamase): 7132–8128 (complementary)pLVX-DD-AmCyan1 Reporter载体序列pLVX-DD-AmCyan1 Reporter其他相关慢病毒载体:Tet-pLKO-neo Tet-pLKO-puro pPACKH1-GAGpMD2.G pCMV-dR8.2-dvpr pLKO.1-GFP-shRNA pLKO.1-TRC control pLKO.1-hygro pLKO.1-TRCpCDH-MSCV-MCS-EF1-copGFP pCDH-MSCV-MCS-EF1-copGFP-T2A-Puro FUW-tetO-hOKMSFUW-tetO-hOCT4 FUW-tetO-hSOX2 FUW-tetO-hKLF4FUW pLVX-AcGFP1-N1 pLVX-AcGFP1-C1pLVX-AmCyan1-N1 pLVX-DsRed-Express2-C1 pLVX-DsRed-Express2-N1 pLVX-DsRed-Monomer-N1 pLVX-PAmCherry-C1 pLVX-PAmCherry-N1pLVX-ZsGreen1-N1 pLVX-IRES-ZsGreen1 pLVX-IRES-mCherrypLVX-mCherry-C1 pLVX-mCherry-N1 pLVX-tdTomato-C1pLKO.1-puro pLentilox 3.7 pLVX-Tet-On-Advanced pLVX-IRES-Puro pLVX-IRES-Neo pLVX-IRES-HygpLVX-EF1α-DsRed-Monomer-C1 pLVX-EF1α-AcGFP1-N1 pLVX-EF1α-AcGFP1-C1 pLVX-EF1α-mCherry-C1 pLVX-EF1α-IRES-mCherry pLVX-EF1α-IRES-ZsGreen1 pLVX-MetLuc Control pLVX-MetLuc pLVX-Hom-Mem1pLVX-Het-2 pLVX-DD-AcGFP1-Actin pPRIME-TET-GFP-FF3pSIH1-H1-CopGFP pCDH-EF1-MCS-T2A-Puro pCDH-CMV-MCS-EF1-Puro pCDF1-MCS2-EF1-copGFP pLOX-CWBmi1 pLOX-CW-CREpRSV-rev pMDLg-pRRE pLL3.7pLVX-DD-AmCyan1 Control pLVX-DD-AmCyan1 Reporter pLVX-DD-tdTomato Reporter pLVX-DD-tdTomato Control pLVX-PTuner-Green pLVX-CherryPicker2pLVX-TetOne-Puro-Luc pLVX-TetOne pLVX-TetOne-PuropLVX-TetOne-Luc pLVX-rHom-Nuc1 pLVX-rHom-Sec1pLVX-rHom-1 pLVX-Hom-Nuc1 pLVX-Het-Nuc1pLVX-PTuner pLVX-PTuner2 pLVX-DD-ZsGreen1 Reporter pLVX-Het-1 pLVX-CherryPicker Control pLVX-Tet3GpCDH-CMV-MCS-EF1-RFP-T2A-Puro pCDH-CMV-MCS-EF1-Hygro pCDH-CMV-MCS-EF1-Neo pCDH-MCS-T2A-Puro-MSCV pCDH1-MCS2-EF1-copGFP pCDF1-MCS2-EF1-Puro pCDH-EF1-MCS-T2A-copGFP pWPXL pLVX-TRE3G-ZsGreen1 pLVX-TRE3G-mCherry pLenti6.3-EmGFP-BveI miR pLenti6/V5-GW/lacZpLenti6.3/V5-GW/EmGFP pLenti6.3-MCS pLenti6.3-DsRed2-BveI miR pLenti6.3-MCS-IRES2-EGFP pLVX-shRNA2 psPAX2VSV-G pSico PGK Puro pcDNA6.2-DsRed2-MCS1 miR pcDNA6.3-EmGFP-NC- II pcDNA6.2-EmGFP-NC- I pcDNA6.2-EmGFP-BsaI miR pLenti6.3-BveI miR pLenti6.3-MCS-IRES2-DsRed2 pLEX-MCSpGIPZ pLP2 pLP1FUGW pFUGW pLOX-Ttag-iresTKpMDLg/pRRE pLentG-KOSM pCMV-dR8.91pLVX-TRE3G-Luc Control pLVX-TRE3G-IRES pCgpvpSico pSicoR pLVTHMpGensil-1 pLVX-EF1α-IRES-Puro pCDF1-MCS2-EF1-copGFP pPACKH1-REV pLVX-Het-Mem1 pLVX-shRNA1pLKO.1-puro-GFP-siRNA pPRIME-TREX-GFP-FF3 pcDNA6.2-DsRed2-BsmBI miRpCDH-MSCV-MCS-EF1-Puro pCDH-CMV-MCS-EF1-copGFP pLVX-TRE3GFUW-tetO-hMYC pLOX-TERT-iresTK pLP/VSVGFUW-M2rtTA pCDH-EF1-MCS-(PGK-Puro) pcDNA6.2-EmGFP-MCS1 miR pLVX-AmCyan1-C1 pLVX-Hom-1 pcDNA6.2-BsaI miRpLVX-DsRed-Monomer-C1 pLVX-mCherry-Actin pTRIPZpLVX-ZsGreen1-C1 pLVX-CherryPicker1 LeGO-iC2pLVX-IRES-tdTomato pCDH-CMV-MCS-EF1-copGFP-T2A-Puro pLKO.3GpLVX-tdTomato-N1 pLVX-PTuner2-C pLVX-PuropLVX-Tight-Puro pLVX-DD-ZsGreen1 Control pSicoR PGK PuropLVX-EF1α-DsRed-Monomer-N1 pCDH-UbC-MCS-EF1-Hygro pLVTHpLVX-EF1α-mCherry-N1 pCDH-CMV-MCS-EF1-RFP。
慢病毒使用操作手册

慢病毒使用操作手册简介:慢病毒(Lentivirus)是一种常用于生命科学研究中的病毒载体,其具有较强的基因转染能力和稳定的基因表达特性。
本操作手册旨在向研究者提供一个详细的慢病毒使用指南,帮助他们顺利进行慢病毒基因转染实验并获得准确可靠的研究结果。
一、慢病毒基本原理慢病毒属于反转录病毒,其基因组为单链RNA。
慢病毒在寄主细胞内通过逆转录过程将RNA转录为DNA,随后将DNA插入宿主基因组中。
这使得慢病毒成为将外源基因稳定集成到宿主基因组中的理想工具。
二、慢病毒使用前准备1. 实验室条件准备:确保工作台面干净整洁,并准备好所需的培养物、培养器具和试剂。
2. 慢病毒载体制备:根据实验需要,选择合适的慢病毒载体,并通过慢病毒包装系统将目标基因插入载体中。
三、慢病毒转染实验步骤1. 细胞培养:将目标细胞接种在培养皿中,并选择合适的培养基进行细胞培养。
2. 慢病毒感染:将预制的慢病毒悬液加入培养皿中,控制感染浓度和时间,以实现最佳的感染效果。
3. 筛选标记:根据实验需要,在感染后适当的时间点添加筛选标记物,如抗生素或荧光标记剂。
4. 选择和扩增:将受筛选标记影响的细胞单克隆分离,扩增和保存。
5. 验证表达:使用合适的实验方法,如western blot或PCR等,来确认目标基因的表达情况。
6. 结果分析:对实验结果进行统计学分析,并绘制适当的图表。
四、注意事项和常见问题解决方案1. 实验前应认真阅读文献,了解慢病毒的基本原理和实验操作流程。
2. 制备慢病毒载体时,应仔细验证目标基因的序列和正确插入。
3. 慢病毒感染时应注意控制感染浓度和时间,避免细胞毒性和非特异性感染。
4. 筛选标记物的选择应根据实验需要和细胞类型进行合理选择。
5. 实验过程中,注意严格遵守实验室安全和生物安全操作规范。
6. 常见问题解决方案:如遇到感染效率低或细胞毒性问题,可以尝试优化感染条件或调整细胞培养条件。
如果基因表达不稳定,可以尝试选择合适的筛选标记物或优化基因载体。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
ORIGIN 1 TGGAAGGGCT AATTCACTCC CAAAGAAGAC AAGATATCCT TGATCTGTGG ATCTACCACA 61 CACAAGGCTA CTTCCCTGAT TAGCAGAACT ACACACCAGG GCCAGGGGTC AGATATCCAC
The presence of mCherry allows transductants to be visualized by fluorescence microscopy and sorted by flow cytometry with standard FITC filter sets (mCherry has an excitation maximum of 587 nm and an emission maximum of 610 nm).
pLVX-IRES-mCherry 载体以双顺反子的形式同时表达
备注:
mCherry 荧光蛋白和目的基因; CMV 启动子驱动目的基因的过表达;
mCherry 是第三代荧光蛋白,亮度高,适用于流式细胞仪。
稳定性:
稳表达
组成型:
诱导型
病毒/非病毒:
慢病毒
pLVX-IRES-mCherry 载体质粒图谱和多克隆位点信息:
pLVX-IRES-mCherry contains all of the viral processing elements necessary for the production of replication-incompetent lentivirus, as well as elements to improve viral titer, transgene expression, and overall vector function. The woodchuck hepatitis virus posttranscriptional regulatory element (WPRE) promotes RNA processing events and enhances nuclear export of viral RNA (2), leading to increased viral titers from packaging cells. In addition, the vector includes a Rev-response element (RRE), which further increases viral titers by enhancing the transport of unspliced viral RNA out of the nucleus (3). Finally, pLVX-IRES-mCherry also contains a central polypurine tract/central termination sequence element (cPPT/CTS). During target cell infection, this element creates a central DNA flap that increases nuclear import of the viral genome, resulting in improved vector integration and more efficient transduction (4). The vector also contains a pUC origin of replication and an E. coli ampicillin resistance gene (Ampr) for propagation and selection in bacteria.
pLVX-IRES-mCherry 载体简介:
Description pLVX-IRES-mCherry is an HIV-1-based, lentiviral expression vector that allows the simultaneous expression of your protein of interest and mCherry in virtually any mammalian cell type, including primary cells. mCherry is a mutant fluorescent protein derived from the tetrameric Discosoma sp. red fluorescent protein, DsRed (1). The vector expresses the two proteins from a bicistronic mRNA transcript, allowing mCherry to be used as an indicator of transduction efficiency and a marker for selection by flow cytometry.
Expression of the bicistronic transcript is driven by the constitutively active human cytomegalovirus immediate early promoter (PCMV IE) located just upstream of the MCS. An encephalomyocarditis virus (EMCV) internal ribosome entry site (IRES), positioned between the MCS and mCherry, facilitates cap-independent translation of mCherry from an internal start site at the IRES/mCherry junction (1).
pLVX-IRES-mCherry
编号 北京华越洋 VECT231241
载体名称 pLVX-IRES-mCherry
Байду номын сангаас
pLVX-IRES-mCherry 载体基本信息:
载体名称:
pLVX-IRES-mCherry
质粒类型:
慢病毒载体;哺乳动物细胞表达载体;双顺反子载体; 荧光报告载体
高拷贝/低拷贝:
高拷贝
121 TGACCTTTGG ATGGTGCTAC AAGCTAGTAC CAGTTGAGCC AGATAAGGTA GAAGAGGCCA 181 ATAAAGGAGA GAACACCAGC TTGTTACACC CTGTGAGCCT GCATGGGATG GATGACCCGG 241 AGAGAGAAGT GTTAGAGTGG AGGTTTGACA GCCGCCTAGC ATTTCATCAC GTGGCCCGAG 301 AGCTGCATCC GGAGTACTTC AAGAACTGCT GATATCGAGC TTGCTACAAG GGACTTTCCG 361 CTGGGGACTT TCCAGGGAGG CGTGGCCTGG GCGGGACTGG GGAGTGGCGA GCCCTCAGAT 421 CCTGCATATA AGCAGCTGCT TTTTGCCTGT ACTGGGTCTC TCTGGTTAGA CCAGATCTGA 481 GCCTGGGAGC TCTCTGGCTA ACTAGGGAAC CCACTGCTTA AGCCTCAATA AAGCTTGCCT 541 TGAGTGCTTC AAGTAGTGTG TGCCCGTCTG TTGTGTGACT CTGGTAACTA GAGATCCCTC 601 AGACCCTTTT AGTCAGTGTG GAAAATCTCT AGCAGTGGCG CCCGAACAGG GACTTGAAAG 661 CGAAAGGGAA ACCAGAGGAG CTCTCTCGAC GCAGGACTCG GCTTGCTGAA GCGCGCACGG 721 CAAGAGGCGA GGGGCGGCGA CTGGTGAGTA CGCCAAAAAT TTTGACTAGC GGAGGCTAGA 781 AGGAGAGAGA TGGGTGCGAG AGCGTCAGTA TTAAGCGGGG GAGAATTAGA TCGCGATGGG 841 AAAAAATTCG GTTAAGGCCA GGGGGAAAGA AAAAATATAA ATTAAAACAT ATAGTATGGG 901 CAAGCAGGGA GCTAGAACGA TTCGCAGTTA ATCCTGGCCT GTTAGAAACA TCAGAAGGCT 961 GTAGACAAAT ACTGGGACAG CTACAACCAT CCCTTCAGAC AGGATCAGAA GAACTTAGAT 1021 CATTATATAA TACAGTAGCA ACCCTCTATT GTGTGCATCA AAGGATAGAG ATAAAAGACA 1081 CCAAGGAAGC TTTAGACAAG ATAGAGGAAG AGCAAAACAA AAGTAAGACC ACCGCACAGC 1141 AAGCGGCCGG CCGCTGATCT TCAGACCTGG AGGAGGAGAT ATGAGGGACA ATTGGAGAAG 1201 TGAATTATAT AAATATAAAG TAGTAAAAAT TGAACCATTA GGAGTAGCAC CCACCAAGGC 1261 AAAGAGAAGA GTGGTGCAGA GAGAAAAAAG AGCAGTGGGA ATAGGAGCTT TGTTCCTTGG 1321 GTTCTTGGGA GCAGCAGGAA GCACTATGGG CGCAGCGTCA ATGACGCTGA CGGTACAGGC 1381 CAGACAATTA TTGTCTGGTA TAGTGCAGCA GCAGAACAAT TTGCTGAGGG CTATTGAGGC 1441 GCAACAGCAT CTGTTGCAAC TCACAGTCTG GGGCATCAAG CAGCTCCAGG CAAGAATCCT 1501 GGCTGTGGAA AGATACCTAA AGGATCAACA GCTCCTGGGG ATTTGGGGTT GCTCTGGAAA 1561 ACTCATTTGC ACCACTGCTG TGCCTTGGAA TGCTAGTTGG AGTAATAAAT CTCTGGAACA 1621 GATTTGGAAT CACACGACCT GGATGGAGTG GGACAGAGAA ATTAACAATT ACACAAGCTT 1681 AATACACTCC TTAATTGAAG AATCGCAAAA CCAGCAAGAA AAGAATGAAC AAGAATTATT
