_SNaPshot技术检测茶树SNP研究
利用SNP标记构建漳州南部茶树种质资源的分子身份证

利用SNP标记构建漳州南部茶树种质资源的分子身份证王泽涵;于文涛;樊晓静;刘财国;房婉萍;蔡春平;叶乃兴
【期刊名称】《江苏农业科学》
【年(卷),期】2022(50)18
【摘要】为了对漳州南部茶树种质资源进行有效地标识和保护,利用SNP分子标记技术对该地区茶树种质资源的指纹图谱和分子身份证进行构建。
以福建省漳州市云霄县、诏安县的74份茶树种质为材料,利用SNP分子标记技术对茶树种质资源进行基因分型,再用筛选出的24个最佳位点进行茶树品种资源分子身份证的构建。
本研究构建了74份茶树种质的DNA指纹图谱;以“4位数字的茶树基本信息+24个基因型”为身份证编码标准,构建茶树品种资源的分子身份证,进而生成相应的条形码和二维码信息,可以快速进行扫码识别。
本研究构建了漳州南部茶树种质资源的分子身份证,使得每一个茶树种质具有唯一的分子身份证,对于地方特色茶树种质资源的鉴定、保护和推广具有重要意义。
【总页数】6页(P284-289)
【作者】王泽涵;于文涛;樊晓静;刘财国;房婉萍;蔡春平;叶乃兴
【作者单位】福建农林大学园艺学院;福州海关技术中心/福建省检验检疫技术研究重点实验室;南京农业大学园艺学院
【正文语种】中文
【中图分类】S571.102.4
【相关文献】
1.利用ISSR和RAPD标记构建红麻种质资源分子身份证
2.利用SSR标记构建沿阶草种质资源分子身份证
3.利用SSR标记构建沿阶草种质资源分子身份证
4.利用SNP标记构建茶树品种资源分子身份证
5.利用SSR荧光标记构建山东地方梨种质资源分子身份证
因版权原因,仅展示原文概要,查看原文内容请购买。
大吉岭茶树品种的全基因组SNP发现-它们的功能影响及其在种群结构和性状关联中的应用

大吉岭茶树品种的全基因组SNP发现-它们的功能影响及其在
种群结构和性状关联中的应用
Genome-wide SNP discovery from Darjeeling tea cultivars - their functional impacts and application toward population structure and trait associations
大吉岭茶树品种的全基因组SNP发现-它们的功能影响及其在种群结构和性状关联中的应用
*Genomics
通过测序和鉴定作物种质中功能相关核苷酸变异进行基因分型是揭示所需性状遗传控制的关键步骤。
通过双消化限制性位点相关的DNA测序方法对大吉岭茶的优良品种进行了SNP基因分型。
这项研究报告了从〜10.4GB序列数据中发现的一组54206个高质量SNP标记,涵盖了参考茶基因组的15条染色体。
这些种之间的遗传相关性符合贝叶斯聚类,UPGMA和PCoA方法的分析。
发现的SNP的基因组位置及其对带注释基因的推定作用,使人对其在茶系统生物学中的功能方面有了深刻的了解。
鉴定出一组95个基因受到高影响力变体的影响。
全基因组关联分析21种农艺和生化表型导致性状相关的多态性位点具有很高的置信度(p <0.05和0.001)。
茶树叶绿体基因组SNP分子标记的初步研究

茶树叶绿体基因组SNP分子标记的初步研究
罗祥宗;胡云飞;吴淋慧;赵雅琦;郑伟铭;黎巷汝;李力
【期刊名称】《茶叶科学》
【年(卷),期】2022(42)6
【摘要】传统的叶绿体基因分子标记在茶组植物分类系统研究中的应用价值相对有限,为了筛选可用于茶树鉴别与母系溯源的SNP(单核苷酸多态性)位点组合,将18个已报道的茶组植物叶绿体全基因组序列进行比对,通过设计通用引物,在169个茶树品种/品系中进行候选分子标记扩增与一代测序分析,筛选出16对引物,共含25个SNP位点可用于茶树品种母系溯源与鉴别分析。
另外,将SNP位点组成的DNA 指纹图谱结合茶树品种资源基本信息进行数字编码,最终形成由30位数字组成的茶树品种资源分子身份证,并构建相应的条形码和二维码用于品种识别。
本研究数据为茶树品种的母本溯源与鉴别提供新的思路。
【总页数】11页(P768-778)
【作者】罗祥宗;胡云飞;吴淋慧;赵雅琦;郑伟铭;黎巷汝;李力
【作者单位】武夷学院茶与食品学院;安徽农业大学茶与食品科技学院;福建农林大学园艺学院
【正文语种】中文
【中图分类】S571.1;S324
【相关文献】
1.利用SNP标记构建茶树品种资源分子身份证
2.郏县红牛全基因组测序分析及关键基因SNP分子标记在育种的应用研究
3.菝葜叶绿体基因组的组装分析与分子标记研究
4.利用SNP标记构建漳州南部茶树种质资源的分子身份证
5.住院病人耐甲氧西林金黄色葡萄球菌耐药情况及感染危险因素分析
因版权原因,仅展示原文概要,查看原文内容请购买。
Snapshot技术平台中文

S n a p s h o t技术平台中文文档编制序号:[KKIDT-LLE0828-LLETD298-POI08]Snapshot技术平台SnaPshot技术平台是Applied Biosystems,ABI公司推出了专为检测 SNP 设计的分析软件和试剂盒可对多个 SNP 位点同时进行基因分型 ,也被称为 minisequencing 。
该方法针对不同突变位点设计不同长度的引物SNaPshot 反应后 ,产物通过电泳分离、五色荧光检测、Gene mapper 分析 ,可在一次电泳胶内检测多个 SNP位点。
这个平台是建立在3730,3130等PCR测序仪上的技术。
3730XL型DNA序列检测仪一. SnaPshot工作原理应用 SNaPshot 进行定点的序列分析 ,其基本原理遵循了DNA 直接测序中的双脱氧终止法 ,所不同的是 PCR 反应中只有不同荧光标记的ddNTP。
由于每个 SNP 位点的引物 3′端都紧靠SNP点 ,因此每一种引物在聚合酶作用下 ,根据模板的的序列 ,只延伸一个核苷酸。
然后用先进的荧光检测系统 ,检测延伸的那个核苷酸的种类。
1.多重SNaPshot反应的工作原理:在一个SNaPshot反应体系中,针对每个待测SNP 位点在其上游或下游设计一条单向的寡核苷酸引物(正向引物或反向引物),引物的Tm 值要求在50度以上,在 AmpliTaq聚合酶和 4种不同荧光标.记的ddNTP存在的情况下,各条引物与各自互补的DNA 模板结合, 聚合酶在引物的3’末端延伸单个碱基反应即告终止,产物的长度为引物长度+1bp。
延伸的碱基就是该样本在该位点上的基因型,其中纯合子表现为单峰,杂合子表现为双峰。
为了能够分辨不同SNP的不同基因型,可在引物的5’末端加上不同长度的Poly C 或Poly T,使各条引物以长度区分。
经电泳将其分开。
最短的引物一般设定为20bp, 相邻两个SNP的引物之间长度一般相差 4-6个核苷酸,以便区分。
(完整版)Snapshot技术平台(中文)

精心整理Snapshot技术平台SnaPshot技术平台是Applied Biosystems,ABI公司推出了专为检测SNP 设计的分析软件和试剂盒可对多个SNP 位点同时进行基因分型,也被称为minisequencing 。
该方法针对不同突变位点设计不同长度的引物SNaPshot 反应后,产物通过电泳分离、五色荧光检测、Gene mapper 分析,可在一次电泳胶内检测多个SNP位点。
这个平台是建立在3730,3130等PCR测序仪上的技术。
3730XL型DNA序列检测仪一.??? SnaPshot工作原理应用SNaPshot 进行定点的序列分析,其基本原理遵循了DNA 直接测序中的双脱氧终止法,所不同的是PCR 反应中只有不同荧光标记的ddNTP。
由于每个SNP位点的引物3′端都紧靠SNP点,因此每一种引物在聚合酶作用下,根据模板的的序列,只延伸一个核苷酸。
然后用先进的荧光检测系统,检测延伸的那个核苷酸的种类。
1.多重SNaPshot反应的工作原理:在一个SNaPshot反应体系中,针对每个待测SNP 位点在其上游或下游设计一条单向的寡核苷酸引物(正向引物或反向引物),ddNTP4-64, 是图2工作的进度取决于多重PCR和EXTENSION的条件优化过程。
还有测序仪的通量也是很重要的,3730每天可以做16次96道电泳,如果每次出4个位点,那每天的通量就是6000个GENOTYPING,是中等通量的检测。
二.??? SnaPshot工作流程1.引物的设计SNAPSHOT可以检测多重的SNP,我们一般选择4-5个位点做一个组合。
现对这些目的SNP进行引物的设计。
每个SNP设计3条引物,其中2条是目的片段的扩增,长度在200-500bp左右,Tm值在60度左右。
第3条的引物的是延伸ddNTP,设计在SNP位点的上游或者是反向的下游。
引物设计注意事项:1.同一反应管中,不同位点的引物长度必须不同,最好能相差4-6个碱基。
基于GBS测序的全基因组SNP揭示贵州地方茶组植物资源的亲缘关系
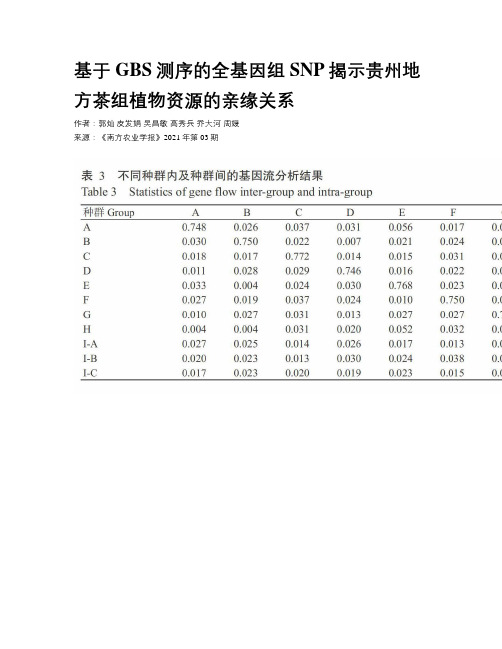
基于GBS测序的全基因组SNP揭示贵州地方茶组植物资源的亲缘关系作者:郭灿皮发娟吴昌敏高秀兵乔大河周媛来源:《南方农业学报》2021年第03期摘要:【目的】分析贵州地方茶组植物资源的亲缘关系,为明确贵州地方茶组植物资源的遗传关系及其保护、利用提供科学依据。
【方法】以从贵州省内不同区域收集的41份茶组植物资源及贵州省茶叶研究所茶树种质资源圃保存的18份省内外育成茶树品种为材料,利用基于GBS(Genotyping by sequencing)的简化基因组测序技术对其基因组SNP位点进行检测,基于获得的高质量SNP位点对这些材料进行遗传特征分析。
【结果】从59份茶组植物材料获得45.84 Gb高质量序列(Clean reads)数据,平均每个材料为795.6 Mb,约占改良版茶树基因组大小(2.93 Gb)的26.5%,平均比对率为72.62%,经过滤后得到248772个高质量SNP位点,其中83.98%的高质量SNP位点分布在基因间区,16.02%分布于基因区;有22614个SNP 位点分布在内含子,15038个SNP位点分布在外显子区,2203个SNP位点分布在非翻译区(UTR)。
59份茶组植物材料的观察杂合度(Ho)为0.016~0.081,期望杂合度(He)为0.006~0.064,F为-0.331~0.737。
主成分分析结果、系统发育进化树构建情况及遗传结构分析结果均显示59份茶组植物材料可分为3个类群,其中全部茶种(Camellia sinensis)材料归在一个类群、疏齿茶(C. remotiserrata)和大厂茶(C. tachangensis)归在一个类群、9份突肋茶(C. costata)单独归在一个类群,但疏齿茶与大厂茶及两个区域的大厂茶均处于独立的亚类群,此外茶种中的阿萨姆变种(C. sinensis var. assamica,CSA)和中国变种(C. sinensis var. sinensis,CSS)也处于不同的进化分支;突肋茶与疏齿茶和大厂茶的亲缘关系较其与茶种的亲缘关系更近。
采用SNaPshot方法对中国老年黄斑变性与C2和C3基因单核苷酸多态性进行相关性研究

( 1 . Co l l e g e o f ma t e r i a l s s c i e n c e a n d En gi n e e r i n g, S o u t h we s t J i a o t o n g Un i v e r s i t y, C h e n g d u, S i c h u a n 6 1 0 0 3 1 , C h i n a ;
a g e r e l a t e d ma c u l a r d e g e n e r a t i o n( AM D)i n a Ha n Ch i n e s e p o p u l a t i o n 。 Me t h o d s Two h u n d r e d p a t i e n t s wi t h we t AM D, 1 2 0 p a —
刘 小 琦
( 1 . 西南 交通 大学材料 科 学与 工程 学 院 , 四 川成都 6 1 0 0 3 1 ; 2 . 四川 省人 民 医院检验 科 , 四川成 都 6 1 0 0 3 1 )
摘 要 : 目的 通 过 对 中 国 大 陆 汉 族 人 C 2 、 C 3基 因的 多 态性 进 行 病 例 对 照研 究 。探 寻 补 体 旁路 途 径 中相 关 基 因 变异 与 老 年 黄斑 变性( AMD ) 的 关 系。 方 法 收 集 2 0 0例 湿 性 AMD 患 者 , 1 2 0例 干 性 AMD 患 者 , 5 0 0例 匹配 的健 康 对 照 , 采 用 单 碱 基 末 端 延 伸( S Na P s h o t ) 方法对其 C 2 、 C 3基 因上 的各 2个单 核 苷 酸 多态性 位 点 ( s NP ) 进 行 基 因型检 测 , 并 进 行 关联 分 析 。 结 果
基于简化基因组测序技术的油茶SNP标记开发及指纹图谱构建

基于简化基因组测序技术的油茶SNP标记开发及指纹图谱构建廖宏泽;孙曼曼;黄小娟;郝丙青;孙佳星;江泽鹏;王东雪;刘凯【期刊名称】《中南林业科技大学学报》【年(卷),期】2024(44)4【摘要】【目的】基于简化基因组,挖掘油茶单核苷酸多态性(SNP)位点,筛选可用于油茶种质鉴定的简化SNP组合位点,构建SNP指纹图谱,建立一种快速准确鉴定广西油茶主栽良种苗木的SNP分子标记方法。
【方法】以12个油茶无性系种质的两个重复共24份油茶标准样本为材料,采用ddRADseq流程进行文库构建,使用BWA将过滤后的测序数据比对到已发布的南荣油茶Camellia oleiferavar.“Nanyongensis”参考基因组上,利用GATK进行SNP位点筛选,ANNOVAR 软件进行SNP位点注释,STRUCTURE软件进行群体结果分析,使用PLINK进行主成分分析;利用R语言,使用条件随机筛选法(CRS),筛选能够区分出油茶种质的最简SNP组合,绘制指纹图谱。
【结果】测序数据质量良好,可用于SNP分子标记位点的开发筛选。
与参考基因组比对后,共获得622064个为SNP标记位点,其中无基因型缺失的SNP位点40147个,多态性信息含量(PIC)大于0.35的SNP位点2094个,前后60 bp碱基保守无变异的SNP位点共184个。
最终筛选出可以将12个油茶无性系种质区分开的15个核心SNP标记位点组合,并以此绘制出SNP 指纹图谱。
【结论】基于简化基因组,建立了广西主要栽培油茶种质的SNP标记开发和指纹图谱绘制的方法,为苗期油茶种质的快速准确鉴定提供了理论依据和技术指导,助力规范油茶苗木市场,促进油茶产业健康发展。
【总页数】10页(P128-137)【作者】廖宏泽;孙曼曼;黄小娟;郝丙青;孙佳星;江泽鹏;王东雪;刘凯【作者单位】广西民族大学海洋与生物技术学院;广西壮族自治区林业科学研究院广西特色经济林培育与利用重点实验室【正文语种】中文【中图分类】S794.4【相关文献】1.甘蓝SNP标记开发及主要品种的DNA指纹图谱构建2.基于地黄转录组数据的SNP标记开发与地黄指纹图谱构建3.基于简化基因组测序技术的甘薯HRM分子标记开发及其应用4.基于全基因组SNP构建甘蓝型油菜指纹图谱5.基于KASP技术的SNP标记用于西瓜品种指纹图谱构建和种子纯度检测因版权原因,仅展示原文概要,查看原文内容请购买。
茶树春梢萌发早晚关联基因_CsAL1_的CAPS_分子标记开发

茶叶科学 2024,44(2):207~218Journal of Tea Science 茶树春梢萌发早晚关联基因CsAL1的CAPS分子标记开发黄梦迪1,陈兰1,苏芹1,胡锦瑜1,刘桂芝1,谭月萍3,刘硕谦1,2*,田娜1,2*1. 茶学教育部重点实验室,湖南长沙 410128;2. 湖南农业大学园艺学院茶学系,湖南长沙 410128;3. 湖南省茶业集团股份有限公司,湖南长沙 410126摘要:茶树(Camellia sinensis)作为一种叶用型经济植物,春梢萌发时间是关系茶叶经济价值的重要生物学性状。
因此,选育茶树早生品种,对提升茶叶品质和经济效益具有重要的现实意义。
以铁观音为参考基因组,通过全基因组关联分析,筛选到一个与茶树春梢萌发早晚高度相关的基因CsAL1(Auxilin-like 1,TGY040711)。
运用SNP calling获得CsAL1各样本的SNPs,将SNPs与其春梢萌发表型进行关联分析,获得与早生性状显著相关的SNP位点。
分析各SNPs的酶切位点,选择合适位点开发茶树早生性状相关CAPS分子标记。
利用标记对12份茶树材料基因组DNA进行PCR扩增和酶切检测,随后在72份茶树材料中进一步验证,以期借助CAPS 分子标记技术为探究CsAL1中单碱基位点突变与茶树早生性状的联系提供参考,为茶树早生品种的选育提供理论支撑。
关键词:茶树;SNP;CAPS分子标记;早生;分子标记育种中图分类号:S571.1;S326 文献标识码:A 文章编号:1000-369X(2024)02-207-12The Development of CAPS Molecular Markers for CsAL1, A Gene Associated with Early and Late SpringTip Emergence in Tea PlantsHUANG Mengdi1, CHEN Lan1, SU Qin1, HU Jinyu1, LIU Guizhi1,TAN Yueping3, LIU Shuoqian1,2*, TIAN Na1,2*1. Key Laboratory of Tea Science of Ministry of Education, Changsha 410128, China;2. Department of Tea Science, College ofHorticulture, Hunan Agricultural University, Changsha 410128, China; 3. Hunan Tea Group Co., Ltd., Changsha 410126, ChinaAbstract:Camellia sinensis is an economically important foliar plant. The time of spring sprouting is a crucial biological trait that affects the economic value of tea. Therefore, selecting and breeding early sprouting tea cultivars are of great practical significance for improving the quality and economic benefits of tea. The study used ‘Tieguanyin’ as the reference genome and screened a gene, CsAL1 (Auxilin-like 1, TGY040711), which was highly significantly correlated with the time of spring sprouting based on genome-wide association analysis. SNP calling was used to obtain SNPs of CsAL1 in each sample. Correlation analysis of the SNPs and spring sprouting phenotypes was performed to obtain the key SNP loci that associating with spring sprouting traits. Suitable enzyme cleavage sites were analyzed for each SNP to develop CAPS molecular markers related to early-sprouting traits in tea plants. PCR was used to amplify the CAPS molecular markers, which were then digested in the genomic DNA of 12 tea收稿日期:2024-01-26 修订日期:2024-03-25基金项目:湖南省种业创新项目(2021NK1008)、国家自然科学基金(U22A20500、32172629)、全国茶树育种联合攻关项目(GJCSYZLHGG-12)作者简介:黄梦迪,女,硕士研究生,从事茶树栽培育种及分子生物学方面研究。
SNaPshot法

SNaPshot法(小测序法)SNaPshot实验原理:该技术由美国应用生物公司(ABI)开发,基于荧光标记单碱基延伸原理的分型技术,也称小测序,主要针对中等通量的SNP分型项目。
在一个含有测序酶,四种荧光标记的ddNTP,紧挨多态位点5’端的不同长度延伸引物和PCR产物模板的反应体系中,引物延伸一个碱基即终止,经ABI测序仪电泳后,根据峰的颜色可知掺入的碱基种类,从而确定该样本的基因型,根据峰移动的胶位置确定该延伸产物对应的SNP位点。
对于PCR产物模板可通过多重PCR反应体系来获得。
通常用于5-30个SNP位点分析。
图1 SNaPshot方法检测SNP工作原理SNaPshot实验流程:优点:1、通量高,一次反应可扩增最多10-15个位点;2、数据完整性高,相对于MassArray高通量分析的时候经常发生个别样本失败或者个别位点丢失的情况,MassArra方法丢失的数据进行补充造价很高,一般不会补,而该方法可以以较低成本补充实验;3、适应性强,实验结果可靠,该方法是ABI公司在10多年前开发成功的,历经了很多验证,发表了大量文章。
送样要求:1、组织样本:-20℃或-80℃低温冰箱中保存,液氮或者干冰运输;市内或短途运输可冰袋运输。
若提供的材料为新鲜组织、血液细胞等生物材料,请提供足够提取2ug以上基因DNA的材料量。
2、DNA样本:体积≥50ul,浓度≥50ng/ul,DNA总量≥2ug,纯度OD260/280 在1.7~1.9之间,不含PCR抑制剂,须注明准确浓度。
样品须低温冰袋运输。
3、血液样本:要求保存于抗凝管中或冻存管中,体积大于2ml,请用干冰运送,保证DNA不降解。
4、样品为石蜡包埋组织切片的,要求提供10张组织切片光片(面积>10mm ×10mm,厚度约5-10um)。
服务说明:1、需要提供的材料包括:组织、细胞、血液等样品材料或纯化好的DNA;2、需要提供目的SNP位点的rs号或该位点上下游各200bp的序列以及突变的类型;3、某些SNP由于上下游序列中含有其他SNP位点或特殊结构,需要重复设计引物和摸索实验,会导致实验周期加长,但是不会增加额外费用。
snptrace基因分型

snptrace基因分型
SNaPshot®是一种基于PCR扩增和限制性片段长度多态性(RFLP)分析的SNP分型技术。
SNaPshot®最初由Illumina公司开发,现在已成为一种广泛使用的SNP分型技术。
SNaPshot®技术的基本原理是,首先使用PCR扩增目标区域的DNA,然后使用限制性内切酶切割PCR产物,产生具有不同大小的片段。
这些片段可以通过电泳分离和成像来确定它们的数量和大小,进而确定SNP位点的等位基因。
SNaPshot®技术的优点是灵敏度高、分辨率好、操作简单、成本低,适用于多种类型的DNA样本,如血液、唾液、口腔拭子等。
同时,SNaPshot®技术还可以与其他分子标记技术相结合,如微卫星不稳定性(MSI)分析、单核苷酸多态性(SNP)芯片等,以提高SNP分型的准确性和可靠性。
SNaPshot®技术的应用范围广泛,包括疾病诊断、药物代谢、人类遗传学、生物医学研究等领域。
例如,在疾病诊断方面,SNaPshot®技术可用于检测某些遗传性疾病,如囊性纤维化、遗传性乳腺癌等。
在药物代谢方面,SNaPshot®技术可用于检测个体对某些药物的代谢差异,以指导药物治疗和剂量调整。
在人类遗传学方面,SNaPshot®技
术可用于研究家族性疾病的遗传机制,以及人类进化和群体分化等方面的研究。
SNP检测方法

SNP检测方法—SNapShot SNP(Single Nucleotide Polymorphisms),即单核苷酸多态性,是指在基因组水平上由单个核苷酸的变异所引起的DNA序列多态性,它是人类中可遗传的变异最常见的一种,并作为第三代遗传标志。
人体许多表型差异、对药物或疾病的易感性等等都可能与SNP有关,因此被广泛用于群体遗传学研究和疾病相关基因研究。
目前,SNP检测技术已经很成熟,主要有以下几种检测方法:TaqMan 探针法、HRM法、SNapShot法、dHPLC、illumina BeadXpress法等,本文主要针对SNapShot法进行阐述。
SNapShot法是一种新兴的SNP检测技术,该技术有美国应用生物公司(ABI)开发的,是基于荧光标记单碱基延伸原理的分型技术,也称小测序,其原理是先根据基因序列,设计特异性的扩增引物及延伸引物,在一个含有测序酶、四种荧光标记ddNTP、紧邻多态位点5’端的不同长度的延伸引物和PCR产物模板的反应体系中,引物延伸一个碱基即终止,经过测序仪检测后,根据峰的移动位置确定该延伸产物对应的SNP位点,根据峰的颜色可得知渗入的碱基种类,从而确定该样本的基因型。
SNapShot法操作简单、经济,可以在多种遗传分析仪上进行快速、准确地基因分型,实现了SNP分析的自动化,可以方便高效地用于高通量的SNP 验证及中通量的SNP筛选。
目前SNapShot技术广受科研人员的认可,国内耳聋基因SNP筛查、结合易感基因SNP位点筛查等用到的检测技术均为SNapShot技术,此外在nature、SCI等多个顶级杂志中也发表了多篇SNapShot技术相关的文献。
由此说明SNapShot技术在SNP研究领域中的地位在不断的提升,已成为SNP 研究中不可或缺的一部分。
北京阅微基因技术有限公司现拥有成熟的SNapShot技术平台,配备ABI3730xl测序仪及国内的杰出人才,实验结果准确、质量高,实验数据符合发表顶级杂志的要求,并提供相应的技术指导及疑难问题解答。
一套基于SnaPshot技术开发的豌豆耐热相关SNP标记及应用[发明专利]
![一套基于SnaPshot技术开发的豌豆耐热相关SNP标记及应用[发明专利]](https://img.taocdn.com/s3/m/27760c1b86c24028915f804d2b160b4e777f8144.png)
专利名称:一套基于SnaPshot技术开发的豌豆耐热相关SNP 标记及应用
专利类型:发明专利
发明人:王栋,杨涛,丁汉凤,宗绪晓,李娜娜,刘荣,田茜
申请号:CN202210439663.8
申请日:20220425
公开号:CN114574626A
公开日:
20220603
专利内容由知识产权出版社提供
摘要:本发明公开了一套基于SnaPshot技术开发的豌豆耐热相关SNP标记及应用。
本发明采用分期播种的方式,对2358份来自世界各地的豌豆种质进行耐热筛选实验,通过产量相关性状鉴定,制定出合理的豌豆耐热分级标准,获得了包含耐热和热敏感种质的豌豆种质群体,并通过一套利用SNaPshot技术开发的豌豆耐热相关SNP标记(20个标记),对其进行遗传多样性和群体遗传结构分析,为将来的豌豆耐热遗传机理及耐热育种研究提供了理论和现实依据。
申请人:山东省农业科学院,中国农业科学院作物科学研究所
地址:250100 山东省济南市历城区工业北路202号
国籍:CN
代理机构:济南诚智商标专利事务所有限公司
代理人:韩百翠
更多信息请下载全文后查看。
首款茶树高密度SNP芯片开发成功

首款茶树高密度SNP芯片开发成功
佚名
【期刊名称】《农业科技与信息》
【年(卷),期】2022()7
【摘要】中国农业科学院茶叶研究所茶树遗传育种团队基于“龙井43”基因组参考序列和茶树重测序数据,开发出一款200K茶树SNP芯片。
相关研究成果在《植物生物技术杂志》上发表。
实验验证表明,该芯片可以有效定位关键QTL位点,发掘茶树重要功能基因。
利用其对不同茶树品种的SNP分型研究,发现茶树大叶种和小叶种之间的两个强选择位点,对揭示茶树品质调控关键基因和两个变种间的进化关系具有重要意义。
【总页数】1页(P46-46)
【正文语种】中文
【中图分类】S57
【相关文献】
1.全球首款4G终端芯片开发成功
2.新思科技完整实施流程助力英飞凌在成功实现首款40hm 3G基带处理器芯片设计和一次流片成功
3.三星发布首款4Gb DDR3高密度内存芯片
4.博通推出业内首款针对云级网络的高密度25/100G以太网交换机芯片
5.水科院完成首个鲤鱼高密度SNP分型芯片开发
因版权原因,仅展示原文概要,查看原文内容请购买。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
编号 Code 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 名称 Name 龙井 43 白毫早 福鼎大白茶 铁观音 黄棪 乌牛早 名山白毫 长叶白毫 龙井 43 (重复) F1 代 -1804 F1 代 -1813 F1 代 -1814 F1 代 -1820 F1 代 -1907 F1 代 -1920 云抗 14 号 云选 9 号 云抗 43 号 福安大白茶 中茶 108 来源 Origin 浙江 湖南 福建 福建 福建 浙江 四川 云南 浙江 浙江 浙江 浙江 浙江 浙江 浙江 云南 云南 云南 福建 浙江
茶叶科学 2014 , 34 ( 2 ) : 180~187 Journal of Tea Science
投稿平台:
SNaPshot 技术检测茶树 SNP 研究
张成才 1,谭礼强
1,2
,王丽鸳 1,韦康 1,成浩
1*
1. 中国农业科学院茶叶研究所,国家茶树改良中心, 浙江 杭州 310008; 2. 四川农业大学园艺学院,四川 雅安 614025
摘要: 为了提高茶树 SNPs 分型效率,促进 SNPs 在茶树遗传育种中的应用,研究了 SNaPshot 技术进行茶树 SNPs 分型的可行性。从实验室前期确证的 SNPs 中,选择 10 个作为目标 SNPs;使用 SNaPshot 技术在不同的 茶树品种中进行分型;然后,对分型结果进行比对和统计,以验证 SNaPshot 技术检测茶树 SNPs 的准确性、 重复性以及在茶树遗传多样性分析等方面的可用性。结果发现, 6 个 SNPs 分型结果与测序结果一致,准确率 为 60%;目标 SNPs 在龙井 43 及其重复实验中的分型结果完全一致; 6 个 SNPs 的等位基因数都是 2 ,期望杂 合度( He)介于 0.37~0.52 ,观测杂合度( Ho)介于 0.32~0.74 ,多态性信息含量( PIC )介于 0.36~0.50 ;结果 表明, SNaPshot 技术对茶树 SNPs 的分型准确性高、重复性好,可以用于茶树遗传多样性分析以及茶树遗传图 谱构建等方面的研究。 关键词: 茶树; SNPs; SNaPshot ;遗传多样性;遗传图谱 中图分类号: S571.1 ;Q52 DOI:10.13305/ki.jts.2014.02.014 文献标识码: A 文章编号: 1000-369X( 2014 )02-180-08
Abstract: In order to increase the genotyping efficiency of tea SNPs and promote th e application of SNPs in tea genetic breeding investigation, the feasibility of SNaPshot in SNPs genotyping of tea plant was investigated. Ten SNPs were selected from previous experiment as target SNPs. Then, these SNPs were detected in different tea cultivars by SNaPshot technology. Six among 10 SNPs were successful detected, with an accuracy of 60%. The polymorphism diversity of these SNPs was also analyzed. The value of NA was 2, He ranged from 0.37 to 0.52, Ho ranged from 0.32 to 0.74, PIC ranged from 0.36 to 0.50. All these markers were shown coincide between `Longjing43` and its repeated test. The markers reported here will be useful for tea genetic linkage map construction and genetic diversity study. The SNaPshot technology will promote the genetic study and accelerate the breeding process in tea plant. Keywords: tea plant, SNPs, SNaPshot, genetic diversity, genetic linkage map
根据 SNPs 类型差异或上下游序列结构的 差异,从实验室前期在 8 个品种(表 1)中确
182
茶 叶 科 学34 卷 Nhomakorabea证的 SNPs [18]中,选择了 10 个 SNPs 作为目标 位点,使用 SNaPshot 方法在不同茶树个体中 进行检测。 SNPs 位点信息见表 2。 实验中,SNaPshot Multiplex Kit 购于 ABI 公司、 Platinum Taq DNA 聚合酶购于 Invitrogen 公司、SAP 和 ExoI 酶购于 Fermentas 公司。
- 20℃保存备用。
使用
该技术成功检测了 77 个甜瓜的 EST-SNPs, 并 用于遗传图谱的构建, 展示了该技术在植物遗 传图谱构建研究中拥有的广阔前景。 茶树是多年生木本植物, 其遗传学研究远 远落后于其他作物。通过探索适合茶树的 SNPs 检测技术,促进 SNP 技术在茶树遗传育 种研究中的应用, 对促进茶树高密度遗传图谱
2期
张成才,等:SNaPshot 技术检测茶树 SNP 研究
181
量丰富、分布广泛、突变率低、可以实现自动 化高通量检测等优点 ,在动植物的高密度遗 传图谱构建 [3-4] 、分子标记辅助育种 [5] 和遗传 多 样 性分 析 等 方 面 发挥 了 重要 的作 用。 目 前,多种检测手段已经用于 SNPs 的分型,如 以凝胶电泳为基础的单链构象多态性标记 ( PCR-SSCP ) 、酶切扩增多态性序列标记 (Cleaved Amplified Polymorphic Sequence , CAPS) 和 等 位 基 因 特 异 性 PCR 标 记 (Allele-specific PCR , AS-PCR) 等;以及自动 化程度较高,可实现中、高通量 SNP 分型的 DNA 测序技术、飞行质谱检测技术和微测序 技术( Minisequencing )等 。 SNaPshot 技术也称微测序技术,是由美 国应用生物系统公司( Applied Biosystems , ABI)开发的一种 SNPs 多重分析技术,可实 现中通量的 SNPs 分型 。其基本原理是:首 先,使用引物扩增目标 SNPs 所在片段,在扩 增产物中加入核酸外切酶 I( Exo I)和碱性磷 酸酶( Shrimp Alkaline Phosphatase , SAP)消 化掉反应体系中的引物序列和剩余的 dNTPs; 然后,以纯化后的扩增产物为模板,使用测序 酶、 双脱氧核苷酸 ( ddNTPs) 和 5′-端紧靠 SNP 位点的延伸引物进行 PCR 反应;最后,使用 测序仪和 GeneScan 等软件进行基因分型和数 据分析
[14]
1 材料与方法
1.1 实验材料和试剂 选取龙井 43 等 13 个种植于杭州国家茶树 种质资源圃的茶树品种, 以及 6 个龙井 43× 白毫 早杂交 F1 代单株作为实验材料 (表 1) 。 分别采 摘一芽二叶新梢,使用改良的 CTAB 法提取基 因组 DNA[19],用 1%琼脂糖凝胶电泳检测基因 组 DNA 质量, 使用 NANODROP 2000C 型分光 光度计(Thermo Fisher Scientific, USA )检测 DNA 浓度,将基因组 DNA 稀释到 30 ng· µL-1,
[7,9] [8] [7] [6] [2]
构建、精确地 QTL 定位,加快茶树育种进程 具有重要意义。目前,黄建安等 [15] 使用单链 构象多态性技术( Single Strand Conformation Polymorphism , PCR-SSCP )方法研究了茶树 多酚氧化酶基因中的 SNPs 分布; 王丽鸳等 [16] 建 立 了 基 于 茶 树 表 达 序 列 标 签 ( Expressed Sequence Tag ,EST)数据库的 EST-SNP 开发 体系,筛选出 818 个候选 EST-SNPs,并使用 重测序技术成功确证了其中的 33 个 SNPs; 张 成才等 [17-18]建立了茶树 SNPs 的酶切扩增多态 性 标 记 ( Cleaved Amplified Polymorphic Sequence, CAPS)和衍生酶切扩增多态性标记 ( Derived Cleaved Amplified Polymorphic sequence, dCAPS)转化体系,并分别对标记 的可用性进行了验证。然而,测序技术成本较 高 , PCR-SSCP 技术 难以 获得位 点的准确 信 息,CAPS 和 dCAPS 技术检测效率较低 [7]。因 此,以上方法在进行大样本、多位点的 SNPs 分型时则表现出明显不足。而 SNaPshot 技术 检测成本低于测序技术,检测效率大大高于 CAPS/dCAPS 技术, 恰好能够弥补以上检测方 法的不足。本文探讨了使用 SNaPshot 技术检 测茶树 SNPs 的可行性,拟为促进 SNPs 技术 在构建高密度的茶树遗传图谱中的应用、 加快 茶树育种进程提供技术支持。
。该技术可同时进行多达几十个
SNPs 的检测,准确性高,检测速度快,成本 低廉。目前,已经广泛地用于人类疾病相关性 分析和法医学领域中的 SNPs 鉴定 [10-12]。在植 物遗传学领域中的应用相对较少。 Torjek 等 [13] 使用 18 组 SNaPshot 反应检测了拟南芥的 112 个 SNPs,指出该技术能够有效实现植物基因 组中多个 SNPs 的同时检测; Deleu 等
