常用大肠杆菌及其基因型
常用大肠杆菌 (K-12株来源) 的基因型

3[bTbZb KRX[NLQWXUXP_ LJ[JUXP XWURWN4grrnI>>sss=rbibpb=cmk=cl
2/01
?twv~~ ?twv~* ?twv*v
பைடு நூலகம்
bm[c;@4@9@jd]^efhZ_l n M6-* GP M,9yxx TGQF trs o N GKPDOQDCu
X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X \ ` g iX
wr 6@KG@QGPs ;us 0OGQPFs /u 0u @KC :@JAOLLHs 3u qw*~*r 5I`]WUf]Sc A]`_[_Y5 ? HST`cSe`ci IS_fS] xKCu DCu xr ?LRKEs 9u +u @KC .@SGPs 9u =u qw*~yr LU[W_UW AAAs }}~t}~xu yr ?@KGPBFt8DOOLKs -us <GDGO@s 3u @KC 6DPPGKEs 3u qw*~{r EW_W BBs wvytww*u
批注本地保存成功开通会员云端永久保存去开通
. u*wvy{ .3012,|+/ xz~}
:QO@GK ,,z ,5xw q./yr ,6x{u{ ,62}wtw~uyxq ,=ywy -tI@ -|vv -3xy| .2w .2{ .2{l .8{vwyvp /.~|{z /.~}|} /9w|z} 2,wvw 26:w}z 36~y 36wvw 36wv{ 36wv| 36wv} 36wv~ 36wv* 36wwv 4~vx 4~vy 5/y*x 6-wv|w 6<ww~z 6<ww*y 7LS@,IRD 99w ;+8*v ;1w ;1x ;2x >5wt,IRD
大肠杆菌基因型及遗传符号说明系列一DXY

大肠杆菌基因型及遗传符号说明系列一点击次数:982 作者:佚名发表于:2009-09-27 00:00转载请注明来自丁香园来源:丁香园实验室的一般大肠杆菌拥有4288条基因,每条基因的长度约为950bp,基因间的平均间隔为118bp (基因Ⅷ)。
E.coli基因组中还包含有许多插入序列,如λ-噬菌体片段和一些其他特殊组份的片段,这些插入的片段都是由基因的水平转移和基因重组而形成的,由此表明了基因组具有它的可塑造性。
利用大肠杆菌基因组的这种特性对其进行改造,使其中的某些基因发生突变或缺失,从而给大肠杆菌带来可以观察到的变化,这种能观察到的特征叫做大肠杆菌的表现型(Phenotype),把引起这种变化的基因构成叫做大肠杆菌的基因型(Genotype)。
具有不同基因型的菌株表现出不同的特性。
分子克隆中常用的大肠杆菌及其遗传标记按Demerec等1966年提出的命名原则,采用的菌株所有的基因都假定处于野生型状态,除非在基因型上另外注明。
大肠杆菌基因型的表示方法(Demerec, et, al. 1966):一、一般规则:1、根据基因产物或其作用产物的英文名称的第一个字母缩写成3个小写斜体字母来表示。
例如:D NA Adenine Methylase→dam。
2、不同的基因座,其中任何一个突变所产生的表型变化可能相同,其表示方法是在3个小写斜体字母后加上一个斜体大写字母来表示区别。
例如:Recombination→recA、recB、recC。
3、突变位点应通过在突变基因符号后加不同数字表示。
如supE44(sup基因座E的44位突变)。
如果不知道几个等位基因中哪一/几个发生了功能性突变,则用连字符“ -”代替大写字母,如trp-31。
4、细菌的基因型中应该包含关于其携带的质粒或附加体的的信息。
这些符号包括菌株携带的质粒或附加体、质粒或附加体上的突变基因座和突变位点。
其基因符号应与基因座的表示符号明显区别,符号的第一个字母大写、不斜体并位于括号内;质粒或附加体上的突变基因座和突变位点的基因符号的表示方法与染色体上突变基因座、突变位点的符号相同。
常用大肠杆菌 (K-12株来源) 的基因型
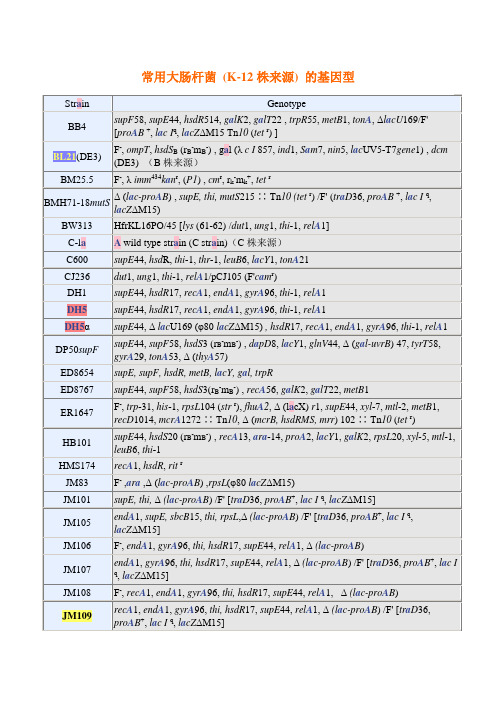
supE44,hsdR17,recA1,endA1,gyrA96,thi-1,relA1
DH5α
supE44, ΔlacU169 (φ80lacZΔM15), hsdR17,recA1,endA1,gyrA96,thi-1,relA1
DP50supF
supE44,supF58,hsdS3 (rB-mB-) ,dapD8,lacY1,glnV44, Δ (gal-uvrB) 47,tyrT58,gyrA29,tonA53, Δ (thyA57)
常用大肠杆菌(K-12株来源)的基因型
Strain
Genotype
BB4
supF58,supE44,hsdR514,galK2,galT22 ,trpR55,metB1,tonA, ΔlacU169/F' [proAB+,lac Iq,lacZΔM15 Tn10(tetr) ]
BL21(DE3)
F-,ompT,hsdSB(rB-mB-) , gal (λc I857,ind1,Sam7,nin5,lacUV5-T7gene1) ,dcm(DE3)(B株来源)
K803
F-,lacY1 or Δ (lac Iq-Y) 6 (lac-3) ,supE44,galK2,galT22,mcrA, rfbD,metB1,mcrB1,hsdR3
LE392
supE44,supF58,hsdR514,galK2,galT22,metB1,trpR55,lacY1
MC1061
hsdR, mcrB,araD139, Δ (araABCห้องสมุดไป่ตู้leu) 7679, ΔlacX74,galU, galK, rpsL, thi
MV1184
bl21de3基因型 -回复

bl21de3基因型-回复BL21(DE3)基因型是一种被广泛应用于分子生物学研究中的大肠杆菌基因型。
BL21(DE3)是指一株大肠杆菌菌株,DE3则表示其内含有一个重组T7 RNA聚合酶基因。
在本文中,我将逐步解释BL21(DE3)基因型的含义、应用以及工作原理。
首先,我们来介绍一下BL21(DE3)基因型的含义。
BL21(DE3)基因型是指大肠杆菌一株具有特定基因组合的菌株。
其中BL21表示该菌株的基本型号,DE3则表示该菌株具有一个重组T7 RNA聚合酶基因。
T7 RNA聚合酶是一种能够特异性地识别T7启动子的聚合酶,因此在融合了该基因的大肠杆菌中,T7启动子能够高效地驱动外源基因的转录和表达。
BL21(DE3)基因型在分子生物学研究中得到了广泛应用。
这主要归功于其高效的外源基因表达能力。
BL21(DE3)菌株中的T7 RNA聚合酶能够识别T7启动子,在转录过程中高效地驱动外源基因的转录和表达。
这种高效的表达系统可以大大提高外源蛋白的产量,便于后续的纯化和功能研究。
在利用BL21(DE3)基因型进行外源基因表达时,通常需要构建一个含有目标基因的质粒,并利用化学方法或电穿孔法将质粒导入到大肠杆菌中。
接着,将转化后的菌株进行筛选,选择包含目标质粒的细菌克隆。
最后,通过T7 RNA聚合酶的识别和驱动,使转录和表达目标基因。
BL21(DE3)基因型还可以与其他辅助的工具和系统一起使用,以进一步提高外源基因的表达效率。
例如,可以利用大肠杆菌中的分泌系统来帮助将产生的蛋白在菌体内向外分泌,从而便于纯化和功能研究。
此外,还可以通过调控T7 RNA聚合酶基因的表达,实现目标基因的适时和高效表达。
总结起来,BL21(DE3)基因型是一种被广泛应用于分子生物学研究中的大肠杆菌基因型。
其具有高效的外源基因表达能力,能够提高外源蛋白的产量,并便于后续的纯化和功能研究。
通过将目标质粒转化到BL21(DE3)菌株中,并利用T7 RNA聚合酶的驱动,可以实现目标基因的高效转录和表达。
大肠杆菌基因工程菌常用类型

1、大肠杆菌DH5a菌株DH5a是世界上最常用的基因工程菌株之一。
由于DH5α是DNA酶缺陷型菌株,有利于基因克隆,保存质粒,但该菌株的蛋白酶没有缺陷,表达的蛋白容易被降解,因此通常不作为表达菌株。
E.coli DH5a在使用pUC系列质粒载体转化时,可与载体编码的β-半乳糖苷酶氨基端实现α-互补。
可用于蓝白斑筛选鉴别重组菌株。
基因型:F-,φ80dlacZΔM15,Δ(lacZYA-argF)U169,deoR,recA1,endA1,hsdR17(rk-,mk+),phoA,supE44,λ-,thi-1,gyrA96,relA12、大肠杆菌BL21(DE3) 菌株该菌株用于高效表达克隆于含有噬菌体T7启动子的表达载体(如pET系列)的基因。
T7噬菌体RNA聚合酶位于λ噬菌体DE3区,该区整合于BL21的染色体上。
该菌适合表达非毒性蛋白。
基因型:F-,ompT,hsdS(rBB-mB-),gal,dcm(DE3)3、大肠杆菌BL21(DE3) pLysS菌株该菌株含有质粒pLysS,因此具有氯霉素抗性。
PLysS含有表达T7溶菌酶的基因,能够降低目的基因的背景表达水平,但不干扰目的蛋白的表达。
该菌适合表达毒性蛋白和非毒性蛋白。
基因型:F-,ompT hsdS(rBB-mB-),gal,dcm(DE3,pLysS ,Camr4、大肠杆菌JM109菌株该菌株在使用pUC系列质粒载体进行DNA转化或用M13 phage载体进行转染时,由于载体DNA产生的LacZa多肽和JM09编码的LacZΔM15进行α-互补,从而显示β-半乳糖苷酶活性,由此很容易鉴别重组体菌株。
基因型:recA1,endA1,gyrA96,thi-1,hsdR17,supE44,relA1,Δ(lac-proAB)/F’[traD36,proAB+,lacIq,lacZΔM15]5、大肠杆菌TOP10菌株该菌株适用于高效的DNA克隆和质粒扩增,能保证高拷贝质粒的稳定遗传。
细菌菌株基因型及基因符号说明

大肠杆菌基因型及遗传符号说明前言:实验室的一般大肠杆菌拥有4288条基因,每条基因的长度约为950bp,基因间的平均间隔为118bp(基因Ⅷ)。
E.coli基因组中还包含有许多插入序列,如λ-噬菌体片段和一些其他特殊组份的片段,这些插入的片段都是由基因的水平转移和基因重组而形成的,由此表明了基因组具有它的可塑造性。
利用大肠杆菌基因组的这种特性对其进行改造,使其中的某些基因发生突变或缺失,从而给大肠杆菌带来可以观察到的变化,这种能观察到的特征叫做大肠杆菌的表现型(Phenotype),把引起这种变化的基因构成叫做大肠杆菌的基因型(Genotype)。
具有不同基因型的菌株表现出不同的特性。
分子克隆中常用的大肠杆菌及其遗传标记按Demerec等1966年提出的命名原则,采用的菌株所有的基因都假定处于野生型状态,除非在基因型上另外注明。
大肠杆菌基因型的表示方法(Demerec, et, al. 1966):一、一般规则:1、根据基因产物或其作用产物的英文名称的第一个字母缩写成3个小写斜体字母来表示。
例如:DNA Adenine Methylase→dam。
2、不同的基因座,其中任何一个突变所产生的表型变化可能相同,其表示方法是在3个小写斜体字母后加上一个斜体大写字母来表示区别。
例如:Recombination→recA、recB、recC。
3、突变位点应通过在突变基因符号后加不同数字表示。
如supE44(sup基因座E的44位突变)。
如果不知道几个等位基因中哪一/几个发生了功能性突变,则用连字符“-”代替大写字母,如trp-31。
4、细菌的基因型中应该包含关于其携带的质粒或附加体的的信息。
这些符号包括菌株携带的质粒或附加体、质粒或附加体上的突变基因座和突变位点。
其基因符号应与基因座的表示符号明显区别,符号的第一个字母大写、不斜体并位于括号内;质粒或附加体上的突变基因座和突变位点的基因符号的表示方法与染色体上突变基因座、突变位点的符号相同。
大肠杆菌菌株类型

大肠杆菌菌株类型
大肠杆菌有多种菌株类型,其中一些是常用于科研和工业应用的典型菌株,比如DH5a和BL21(DE3)。
此外,大肠杆菌也有一些特殊的病型,这些病型主要根据大肠杆菌的感染性状进行分类,包括肠道致病性的大肠杆菌(EPEC)、肠道产毒素性的大肠杆菌(ETEC)、肠道侵袭性的大肠杆菌(EIEC)、肠道出血性的大肠杆菌(EHEC)和肠集聚性的大肠杆菌(EAEC)。
此外,还有一些新近发现的菌株,如肠产志贺样毒素同时具有一定侵袭力的大肠杆菌(ESIES)。
以上信息仅供参考,如有需要,建议查阅专业生物书籍或文献。
各株大肠杆菌区别

JM109,DH5a,BL21这些感受态有何区别已有1人参与转自生物病毒(凯歌的空间)JM109,DH5a,BL21这些感受态有何区别1:DH5a菌株DH5a是一种常用于质粒克隆的菌株。
E.coli DH5a在使用pUC系列质粒载体转化时,可与载体编码的β-半乳糖苷酶氨基端实现α-互补。
可用于蓝白斑筛选鉴别重组菌株。
基因型:F-,φ80dlacZΔM15,Δ(lacZYA-argF)U169,deoR,recA1,endA1,hsdR17(rk-,mk+),phoA,supE44,λ-,thi-1,gyrA96,relA12:BL21(DE3) 菌株该菌株用于高效表达克隆于含有噬菌体T7启动子的表达载体(如pET系列)的基因。
T7噬菌体RNA聚合酶位于λ 噬菌体DE3区,该区整合于BL21的染色体上。
该菌适合表达非毒性蛋白。
基因型:F-,ompT,hsdS(rBB-mB-),gal,dcm(DE3)3:BL21(DE3) pLysS菌株该菌株含有质粒pLysS,因此具有氯霉素抗性。
PLysS含有表达T7溶菌酶的基因,能够降低目的基因的背景表达水平,但不干扰目的蛋白的表达。
该菌适合表达毒性蛋白和非毒性蛋白。
基因型:F-,ompT hsdS(rBB-mB-),gal,dcm(DE3,pLysS ,Camr4:JM109菌株该菌株在使用pUC系列质粒载体进行DNA转化或用M13 phage载体进行转染时,由于载体DNA产生的LacZa多肽和JM09编码的LacZΔM15进行α-互补,从而显示β-半乳糖苷酶活性,由此很容易鉴别重组体菌株基因型:recA1,endA1,gyrA96,thi-1,hsdR17,supE44,relA1,Δ(lac-proAB)/F’*traD36,proAB+,lacIq,lacZΔM15+5:TOP10菌株该菌株适用于高效的DNA克隆和质粒扩增,能保证高拷贝质粒的稳定遗传。
大肠杆菌的基因型-概述说明以及解释

大肠杆菌的基因型-概述说明以及解释1.引言1.1 概述大肠杆菌是一种常见的革兰氏阴性杆菌,属于肠道菌群中的重要成员。
它在自然界和人体内广泛存在,并且具有广泛的基因型多样性。
这使得大肠杆菌成为了微生物遗传学和进化生物学领域的研究模型。
在大肠杆菌中,基因型是指该菌株拥有的基因组合和基因的分布情况。
大肠杆菌的基因型可以通过不同的方法进行分类和鉴定。
目前主要的分类方法包括单核苷酸多态性分析、基因片段分析和全基因组测序等。
通过这些方法,我们可以更全面地了解大肠杆菌的基因型组成和种群结构。
大肠杆菌的基因型在其功能和特点方面具有重要意义。
大肠杆菌是一种典型的益生菌,它在人体内具有多种有益作用,包括帮助消化吸收、维持肠道稳定性和参与免疫调节等。
不同基因型的大肠杆菌可能具有不同的功能特点,比如某些基因型可能携带耐药基因或致病因子,导致感染和疾病的发生。
因此,对大肠杆菌基因型的研究有助于我们深入了解其功能机制和生态适应能力。
总之,大肠杆菌作为一种常见的菌株,其基因型具有多样性和重要性。
通过研究大肠杆菌的基因型,我们可以深入探索其功能特点和生态适应能力,进一步促进微生物遗传学和进化生物学的研究。
未来,我们可以通过结合多样的研究方法和技术,进一步挖掘和解析大肠杆菌基因型的奥秘,并探索其在人体健康和疾病中的作用。
文章结构是指文章部分之间的逻辑关系和组织,它有助于读者理解文章的内容和思路。
本文的结构如下:1. 引言1.1 概述1.2 文章结构1.3 目的2. 正文2.1 大肠杆菌的基因型分类2.2 大肠杆菌基因型的功能和特点3. 结论3.1 大肠杆菌基因型的重要性3.2 未来研究的方向文章结构部分是为了描述本文的组织结构,它有助于读者了解文章的内容安排和逻辑关系。
在本文中,我们首先介绍引言部分,包括概述、文章结构和目的。
在概述中,我们简要介绍了大肠杆菌的基因型。
在文章结构中,我们明确了本文的结构和章节安排,帮助读者理解文章的整体框架。
细菌基因型及其基因符号1102

大肠杆菌基因型及遗传符号说明前言:实验室的一般大肠杆菌拥有4288条基因,每条基因的长度约为950bp,基因间的平均间隔为118bp(基因Ⅷ)。
E.coli基因组中还包含有许多插入序列,如λ-噬菌体片段和一些其他特殊组份的片段,这些插入的片段都是由基因的水平转移和基因重组而形成的,由此表明了基因组具有它的可塑造性。
利用大肠杆菌基因组的这种特性对其进行改造,使其中的某些基因发生突变或缺失,从而给大肠杆菌带来可以观察到的变化,这种能观察到的特征叫做大肠杆菌的表现型(Phenotype),把引起这种变化的基因构成叫做大肠杆菌的基因型(Genotype)。
具有不同基因型的菌株表现出不同的特性。
分子克隆中常用的大肠杆菌及其遗传标记按Demerec等1966年提出的命名原则,采用的菌株所有的基因都假定处于野生型状态,除非在基因型上另外注明。
大肠杆菌基因型的表示方法(Demerec, et, al. 1966):一、一般规则:1、根据基因产物或其作用产物的英文名称的第一个字母缩写成3个小写斜体字母来表示。
例如:DNA Adenine Methylase→dam。
2、不同的基因座,其中任何一个突变所产生的表型变化可能相同,其表示方法是在3个小写斜体字母后加上一个斜体大写字母来表示区别。
例如:Recombination→recA、recB、recC。
3、突变位点应通过在突变基因符号后加不同数字表示。
如supE44(sup基因座E的44位突变)。
如果不知道几个等位基因中哪一/几个发生了功能性突变,则用连字符“-”代替大写字母,如trp-31。
4、细菌的基因型中应该包含关于其携带的质粒或附加体的的信息。
这些符号包括菌株携带的质粒或附加体、质粒或附加体上的突变基因座和突变位点。
其基因符号应与基因座的表示符号明显区别,符号的第一个字母大写、不斜体并位于括号内;质粒或附加体上的突变基因座和突变位点的基因符号的表示方法与染色体上突变基因座、突变位点的符号相同。
大肠杆菌克隆菌株TOP10和DH5α的比较

大肠杆菌克隆菌株TOP10和DH5α的比较大肠杆菌克隆菌株是一种经过基因工程改造的细菌,可以用于转化和扩增外源DNA,如质粒、文库或表达载体。
不同的克隆菌株具有不同的特性,如转化效率、抗性、突变、表达水平等。
本文将比较两种常见的大肠杆菌克隆菌株:TOP10和DH5α,分析它们的优缺点和价格。
TOP10的优缺点TOP10是一种来源于大肠杆菌K12的菌株,具有mcr/mrr突变,可以克隆甲基化的DNA,如真核基因组DNA。
它的基因型如下:•Top10:F- mcrA Δ(mrr-hsdRMS-mcrBC) φ80lacZΔM15 ΔlacX74 recA1 araD139 Δ(ara-leu)7697 galU galK rpsL (StrR) endA1 nupGTOP10的优点主要有以下几个方面:•高转化效率:TOP10具有高达10^9 cfu/µg DNA的转化效率,这意味着它可以获得更多的重组菌株,提高克隆的成功率。
•低突变率:TOP10具有recA1突变,可以抑制同源重组,保持质粒的序列完整性。
•高稳定性:TOP10具有endA1突变,可以阻断内切核酸酶的表达,减少质粒DNA的降解。
它还可以在不含抗生素的培养基中维持质粒的复制。
•广泛的耐药性:TOP10具有多种抗性基因,如rpsL (StrR)、nupG等,可以使用多种抗生素进行筛选。
•高数据一致性:TOP10具有高度的基因组稳定性和质粒保留性,可以保证实验的可重复性和可靠性。
TOP10的缺点主要有以下几个方面:•高成本:TOP10的价格相对较高,可能超出一些实验室的预算。
•低表达水平:TOP10不适合用于蛋白表达,因为它缺乏一些必要的表达因子,如DE3溶原菌、T7 RNA聚合酶等。
•低兼容性:TOP10不适合用于一些特殊的载体,如含有重复序列的慢病毒载体和其它逆转录病毒载体,因为它可能会发生质粒的重组或缺失。
DH5α的优缺点DH5α是一种由Messing在1975年从大肠杆菌K12菌株中构建而成的菌株,具有mcr/mrr突变,可以克隆甲基化的DNA,如真核基因组DNA。
bl21de3基因型 -回复

bl21de3基因型-回复BL21DE3基因型是一种常见的细菌基因型,它在生物学和基因工程领域具有广泛的应用。
本文将一步一步解释BL21DE3基因型的含义、特点以及其在科研和工业应用中的作用。
首先,我们来了解BL21DE3基因型的含义和起源。
BL21DE3是大肠杆菌(Escherichia coli)的一种特殊基因型,它是由原始BL21菌株经过基因重组而得到的。
BL21DE3基因型是通过插入DE3(由pET 系列质粒携带的T7 RNA聚合酶基因)来构建的。
这样一来,BL21DE3基因型就具备了T7 RNA聚合酶的表达能力,可以高效地产生大量的目标蛋白。
BL21DE3基因型的特点主要体现在两个方面:高效的蛋白表达和容易操作。
首先,由于BL21DE3基因型携带T7 RNA聚合酶基因,它可以利用T7促进子启动子驱动的高效表达系统来表达目标蛋白。
这个系统可以在感染T7 RNA聚合酶的大肠杆菌中产生大量的目标蛋白,其产量通常远远高于其他常见的表达系统。
因此,BL21DE3基因型在大规模蛋白表达和纯化方面具有巨大的优势。
其次,BL21DE3基因型的操作相对简单。
在基因工程实验中,只需将目标基因序列克隆到适当的pET质粒中,然后将质粒转化到BL21DE3细胞中。
这个过程通常可以通过化学法或电穿孔法实现。
由于BL21DE3基因型的广泛应用,研究人员已经开发了各种工具和试剂盒,使得BL21DE3基因型的操作更加简便和高效。
BL21DE3基因型在科研和工业中都发挥着重要的作用。
首先,它被广泛应用于蛋白表达和纯化领域。
由于BL21DE3基因型的高效表达能力,它成为了研究人员首选的蛋白表达系统。
不仅如此,BL21DE3基因型还被广泛用于表达溶酶酶原,用于蛋白酶活性研究和酶工程领域。
此外,BL21DE3基因型还被用作包括降解有害物质、产生重要药物和生化品等在内的工业生产菌株。
例如,一些重要药物如胰岛素、生长激素等就是通过BL21DE3基因型进行大规模工业生产的。
大肠杆菌基因型及遗传符号说明

大肠杆菌基因型及遗传符号说明前言:实验室的一般大肠杆菌拥有4288条基因,每条基因的长度约为950bp,基因间的平均间隔为118bp(基因Ⅷ)。
E.coli基因组中还包含有许多插入序列,如λ-噬菌体片段和一些其他特殊组份的片段,这些插入的片段都是由基因的水平转移和基因重组而形成的,由此表明了基因组具有它的可塑造性。
利用大肠杆菌基因组的这种特性对其进行改造,使其中的某些基因发生突变或缺失,从而给大肠杆菌带来可以观察到的变化,这种能观察到的特征叫做大肠杆菌的表现型(Phenotype),把引起这种变化的基因构成叫做大肠杆菌的基因型(Genotype)。
具有不同基因型的菌株表现出不同的特性。
分子克隆中常用的大肠杆菌及其遗传标记按Demerec等1966年提出的命名原则,采用的菌株所有的基因都假定处于野生型状态,除非在基因型上另外注明。
大肠杆菌基因型的表示方法(Demerec, et, al. 1966):一、一般规则:1、根据基因产物或其作用产物的英文名称的第一个字母缩写成3个小写斜体字母来表示。
例如:DNA Adenine Methylase→dam。
2、不同的基因座,其中任何一个突变所产生的表型变化可能相同,其表示方法是在3个小写斜体字母后加上一个斜体大写字母来表示区别。
例如:Recombination→recA、recB、recC。
3、突变位点应通过在突变基因符号后加不同数字表示。
如supE44(sup基因座E的44位突变)。
如果不知道几个等位基因中哪一/几个发生了功能性突变,则用连字符“-”代替大写字母,如trp-31。
4、细菌的基因型中应该包含关于其携带的质粒或附加体的的信息。
这些符号包括菌株携带的质粒或附加体、质粒或附加体上的突变基因座和突变位点。
其基因符号应与基因座的表示符号明显区别,符号的第一个字母大写、不斜体并位于括号内;质粒或附加体上的突变基因座和突变位点的基因符号的表示方法与染色体上突变基因座、突变位点的符号相同。
大肠杆菌不同感受态特点

JM109,DH5a,BL21感受态有何区别1、DH5a菌株DH5a是一种常用于质粒克隆的菌株。
E.coli DH5a在使用pUC系列质粒载体转化时,可与载体编码的β-半乳糖苷酶氨基端实现α-互补。
可用于蓝白斑筛选鉴别重组菌株。
基因型:F-,φ80dlacZΔM15,Δ(lacZYA-argF)U169,deoR,recA1,endA1,hsdR17(rk-,mk+),phoA,supE44,λ-,thi-1,gyrA96,relA1。
2、BL21(DE3) 菌株该菌株用于高效表达克隆于含有噬菌体T7启动子的表达载体(如pET系列)的基因。
T7噬菌体RNA聚合酶位于λ噬菌体DE3区,该区整合于BL21的染色体上。
该菌适合表达非毒性蛋白。
基因型:F-,ompT,hsdS(rBB-mB-),gal,dcm(DE3)3、BL21(DE3) pLysS菌株该菌株含有质粒pLysS,因此具有氯霉素抗性。
PLysS含有表达T7溶菌酶的基因,能够降低目的基因的背景表达水平,但不干扰目的蛋白的表达。
该菌适合表达毒性蛋白和非毒性蛋白。
基因型:F-,ompT hsdS(rBB-mB-),gal,dcm(DE3,pLysS ,Camr。
4:JM109菌株该菌株在使用pUC系列质粒载体进行DNA转化或用M13 phage载体进行转染时,由于载体DNA产生的LacZa多肽和JM09编码的LacZΔM15进行α-互补,从而显示β-半乳糖苷酶活性,由此很容易鉴别重组体菌株。
基因型:recA1,endA1,gyrA96,thi-1,hsdR17,supE44,relA1,Δ(lac-proAB)/F’[traD36,proAB+,lacIq,lacZΔM15]5:TOP10菌株该菌株适用于高效的DNA克隆和质粒扩增,能保证高拷贝质粒的稳定遗传。
基因型:F- ,mcrAΔ(mrr-hsd RMS-mcrBC),φ80 ,lacZΔM15,△lacⅩ74,recA1 ,ara Δ139Δ(ara-leu)7697,galU ,galK ,rps,(Strr) endA1,nupG6、TOP10F'菌株带lacIq,需加IPTG诱导表达克隆于lac启动子后的外源基因,用于蓝白斑筛选时,需加入IPTG和X-Gal。
常见大肠杆菌菌株的基因型

常见大肠杆菌菌株的基因型1:DH5a菌株常用于质粒克隆的菌株。
使用pUC系列质粒载体转化时,可与载体编码的β-半乳糖苷酶氨基端实现α-互补。
可用于蓝白斑筛选鉴别重组菌株。
基因型:F-,φ80dlacZΔM15,Δ(lacZYA-argF)U169,deoR,recA1,endA1,hsdR17(rk-,mk ),phoA,supE44,λ-,thi-1,gyrA96,relA12:BL21(DE3) 菌株用于高效表达克隆于含有噬菌体T7启动子的表达载体(如pET系列)的基因。
T7噬菌体RNA聚合酶位于λ噬菌体DE3区,该区整合于BL21的染色体上。
该菌适合表达非毒性蛋白。
基因型:F-,ompT,hsdS(rBB-mB-),gal,dcm(DE3)3:BL21(DE3) pLysS菌株该菌株含有质粒pLysS,因此具有氯霉素抗性。
PLysS含有表达T7溶菌酶的基因,能够降低目的基因的背景表达水平,但不干扰目的蛋白的表达。
该菌适合表达毒性蛋白和非毒性蛋白。
基因型:F-,ompT hsdS(rBB-mB-),gal,dcm(DE3,pLysS ,Camr4:JM109菌株该菌株在使用pUC系列质粒载体进行DNA转化或用M13 phage载体进行转染时,由于载体DNA产生的LacZa多肽和JM09编码的LacZΔM15进行α-互补,从而显示β-半乳糖苷酶活性,由此很容易鉴别重组体菌株基因型:recA1,endA1,gyrA96,thi-1,hsdR17,supE44,relA1,Δ(lac-proAB)/F’[traD36,proAB ,lacIq,lacZΔM15]5:TOP10菌株该菌株适用于高效的DNA克隆和质粒扩增,能保证高拷贝质粒的稳定遗传。
基因型:F- ,mcrAΔ(mrr-hsd RMS-mcrBC),φ80 ,lacZΔM15,△lacⅩ74,recA1 ,araΔ139Δ(ara-leu)7697,galU ,galK ,rps,(Strr) endA1,nupG6:HB101菌株该菌株遗传性能稳定,使用方便,适用于各种基因重组实验基因型:supE44,hsdS20(rB-mB-),recA13,ara-14,proA2,lacY1,galK2,rpsL20,xyl-5,mtl-1,leuB6,thi-17:Top10F'菌株带lacIq,需加IPTG诱导表达克隆于lac启动子后的外源基因,用于蓝白斑筛选时,需加入IPTG和X-Gal。
大肠杆菌表达宿主的特点

大肠杆菌表达宿主的特点1:DH5a菌株DH5a是一种常用于质粒克隆的菌株。
E.coli DH5a在使用pUC系列质粒载体转化时,可与载体编码的β-半乳糖苷酶氨基端实现α-互补。
可用于蓝白斑筛选鉴别重组菌株。
基因型:F-,φ80dlacZΔM15,Δ(lacZYA-argF)U169,deoR,recA1,endA1,hsdR17(rk-,mk+),phoA,supE44,λ-,thi-1,gyrA96,relA12:BL21(DE3) 菌株该菌株用于高效表达克隆于含有噬菌体T7启动子的表达载体(如pET 系列)的基因。
T7噬菌体RNA聚合酶位于λ 噬菌体DE3区,该区整合于BL21的染色体上。
该菌适合表达非毒性蛋白。
基因型:F-,ompT,hsdS(rBB-mB-),gal,dcm(DE3)3:BL21(DE3) pLysS菌株该菌株含有质粒pLysS,因此具有氯霉素抗性。
PLysS含有表达T7溶菌酶的基因,能够降低目的基因的背景表达水平,但不干扰目的蛋白的表达。
该菌适合表达毒性蛋白和非毒性蛋白。
基因型:F-,ompT hsdS(rBB-mB-),gal,dcm(DE3,pLysS ,Camr 4:JM109菌株该菌株在使用pUC系列质粒载体进行DNA转化或用M13 phage 载体进行转染时,由于载体DNA产生的LacZa多肽和JM09编码的LacZΔM15进行α-互补,从而显示β-半乳糖苷酶活性,由此很容易鉴别重组体菌株基因型:recA1,endA1,gyrA96,thi-1,hsdR17,supE44,relA1,Δ(lac-proAB)/F’[traD36,proAB+,lacIq,lacZΔM15]5:TOP10菌株该菌株适用于高效的DNA克隆和质粒扩增,能保证高拷贝质粒的稳定遗传。
基因型:F- ,mcrAΔ(mrr-hsd RMS-mcrBC),φ80 ,lacZΔM15,△lac Ⅹ74,recA1 ,araΔ139Δ(ara-leu)7697,galU ,galK ,rps,(Strr) endA1,nupG 6:HB101菌株该菌株遗传性能稳定,使用方便,适用于各种基因重组实验基因型:supE44,hsdS20(rB-mB-),recA13,ara-14,proA2,lacY1,galK2,rpsL20,xyl-5,mtl-1,leuB6,thi-1 7:M110或SCS110大多数大肠杆菌菌株中含有Dam甲基化酶和Dcm甲基化酶,前者可以在GATC序列中腺嘌呤N-6位上引入甲基,后者在CCA/TGC 序列的第一个胞嘧啶C-5位置上引入甲基。
常见大肠杆菌感受态的特点和用途解析

1:DH5a 菌株DH5a 是一种常用于质粒克隆的菌株。
E.coli DH5a在使用 pUC 系列质粒载体转化时,可与载体编码的β-半乳糖苷酶氨基端实现α-互补。
可用于蓝白斑筛选鉴别重组菌株。
基因型:F-, φ80dlacZΔM15, Δ(lacZYA-argFU169, deoR , recA1, endA1,hsdR17(rk-, mk+, phoA , supE44, λ-, thi-1, gyrA96, relA12:BL21(DE3 菌株该菌株用于高效表达克隆于含有噬菌体 T7启动子的表达载体(如 pET 系列的基因。
T7噬菌体 RNA 聚合酶位于λ 噬菌体 DE3区,该区整合于 BL21的染色体上。
该菌适合表达非毒性蛋白。
基因型:F-, ompT , hsdS (rBB-mB -, gal , dcm (DE33:BL21(DE3 pLysS菌株该菌株含有质粒 pLysS ,因此具有氯霉素抗性。
PLysS 含有表达 T7溶菌酶的基因,能够降低目的基因的背景表达水平,但不干扰目的蛋白的表达。
该菌适合表达毒性蛋白和非毒性蛋白。
基因型:F-, ompThsdS (rBB-mB -, gal , dcm (DE3, pLysS , Camr4:JM109菌株该菌株在使用 pUC 系列质粒载体进行 DNA 转化或用 M13 phage载体进行转染时,由于载体 DNA 产生的 LacZa 多肽和 JM09编码的LacZΔM15进行α-互补 ,从而显示β-半乳糖苷酶活性,由此很容易鉴别重组体菌株基因型 :recA1, endA1, gyrA96, thi -1, hsdR17, supE44, relA1, Δ(lac -proAB/F’[traD36, proAB+, lacIq , lacZΔM15]5:TOP10菌株该菌株适用于高效的 DNA 克隆和质粒扩增 ,能保证高拷贝质粒的稳定遗传。
大肠杆菌基因型说明

大肠杆菌基因型及遗传符号说明前言:实验室的一般大肠杆菌拥有4288条基因,每条基因的长度约为950bp,基因间的平均间隔为118bp(基因Ⅷ)。
E.coli基因组中还包含有许多插入序列,如λ-噬菌体片段和一些其他特殊组份的片段,这些插入的片段都是由基因的水平转移和基因重组而形成的,由此表明了基因组具有它的可塑造性。
利用大肠杆菌基因组的这种特性对其进行改造,使其中的某些基因发生突变或缺失,从而给大肠杆菌带来可以观察到的变化,这种能观察到的特征叫做大肠杆菌的表现型(Phenotype),把引起这种变化的基因构成叫做大肠杆菌的基因型(Genotype)。
具有不同基因型的菌株表现出不同的特性。
分子克隆中常用的大肠杆菌及其遗传标记按Demerec等1966年提出的命名原则,采用的菌株所有的基因都假定处于野生型状态,除非在基因型上另外注明。
大肠杆菌基因型的表示方法(Demerec, et, al. 1966):一、一般规则:1、根据基因产物或其作用产物的英文名称的第一个字母缩写成3个小写斜体字母来表示。
例如:DNA Adenine Methylase→dam。
2、不同的基因座,其中任何一个突变所产生的表型变化可能相同,其表示方法是在3个小写斜体字母后加上一个斜体大写字母来表示区别。
例如:Recombination→recA、recB、recC。
3、突变位点应通过在突变基因符号后加不同数字表示。
如supE44(sup基因座E的44位突变)。
如果不知道几个等位基因中哪一/几个发生了功能性突变,则用连字符“-”代替大写字母,如trp-31。
4、细菌的基因型中应该包含关于其携带的质粒或附加体的的信息。
这些符号包括菌株携带的质粒或附加体、质粒或附加体上的突变基因座和突变位点。
其基因符号应与基因座的表示符号明显区别,符号的第一个字母大写、不斜体并位于括号内;质粒或附加体上的突变基因座和突变位点的基因符号的表示方法与染色体上突变基因座、突变位点的符号相同。
jm109基因型

jm109基因型jm109基因型是一种常见的大肠杆菌(Escherichia coli)基因型,它是由大肠杆菌JM109菌株所携带的基因型。
JM109菌株是一种常用的实验室菌株,常用于重组DNA的制备、质粒转化和蛋白表达等研究中。
本文将介绍jm109基因型的特点、应用以及相关的研究进展。
jm109基因型具有多个重要特点,使其成为常用的实验室菌株之一。
首先,它具有较高的转化效率,能够高效地接受外源DNA的转化。
其次,jm109基因型还具有多种抗性基因,包括抗生素抗性基因和选择标记基因,这使得研究人员能够通过选择性培养来筛选携带目标基因的菌落。
此外,jm109基因型还具有较好的生长特性,能够在常规培养条件下快速生长和繁殖。
jm109基因型在分子生物学研究中有着广泛的应用。
首先,它常被用于质粒的构建和制备。
质粒是一种环状DNA分子,可以携带外源基因并在宿主细胞中复制和表达。
通过将目标基因插入到适当的质粒载体中,并将质粒转化到jm109细胞中,研究人员可以获得大量携带目标基因的细胞。
其次,jm109基因型还被广泛用于蛋白表达研究。
研究人员可以将目标蛋白的编码序列插入到适当的表达载体中,并将载体转化到jm109细胞中,从而实现目标蛋白的高效表达。
此外,jm109基因型还被用于遗传工程和基因编辑等研究领域。
近年来,关于jm109基因型的研究也在不断深入。
研究人员通过对jm109基因型的基因组测序和比较分析,揭示了其基因型的特点和变异情况。
此外,还有研究报道了jm109基因型的转化机制、质粒复制和表达调控等方面的研究成果。
这些研究为进一步理解jm109基因型的特点和应用提供了重要的科学依据。
尽管jm109基因型在分子生物学研究中有着广泛的应用,但也存在一些局限性。
首先,由于jm109基因型是一种大肠杆菌菌株,其生长和繁殖需要一定的培养条件和培养基,这增加了实验的复杂性和成本。
其次,由于jm109基因型是一种大肠杆菌菌株,其表达系统和代谢途径可能与其他细菌有所差异,这可能会对研究结果的解释和应用带来一定的局限性。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
Commonly used strains /wiki/E._coli_genotypes 1.AG1endA1 recA1 gyrA96 thi-1 relA1 glnV44 hsdR17(rK - mK+)2.AB1157thr-1, araC14, leuB6(Am), Δ(gpt-proA)62, lacY1, tsx-33, qsr'-0,glnV44(AS), galK2(Oc), LAM-, Rac-0, hisG4(Oc), rfbC1, mgl-51,rpoS396(Am), rpsL31(strR), kdgK51, xylA5, mtl-1, argE3(Oc), thi-1∙Bachmann BJ: Derivation and genotypes of some mutant derivatives of Escherichia coli K-12.Escherichia coli and Salmonella typhimurium. Cellular and Molecular Biology (Edited by: F C Neidhardt J L Ingraham KB Low B Magasanik M Schaechter H E Umbarger). Washington, D.C., American Society for Microbiology 1987, 2:1190-1219.See CGSC#11573.BL21E. coli B F- dcm ompT hsdS(rB - mB-) gal [malB+]K-12(λS)∙The "malB region" was transduced in from the K-12 strain W3110 to make the strain Mal+λS. See Studier et al. (2009) J. Mol. Biol.394(4), 653 for a discussion of the extent of the transfer.∙Stratagene E. coli Genotype Strains4.BL21(AI)F– ompT gal dcm lon hsdSB (rB- mB-) araB::T7RNAP-tetA∙an E. coli B strain carrying the T7 RNA polymerase gene in the araB locus of the araBAD operon q.∙Transformed plasmids containing T7 promoter driven expression are repressed until L-arabinose induction of T7 RNA polymerase.∙Derived from BL21.∙See the product page for more information.∙Brian Caliendo (Voigt lab) reported trouble getting the Datsenko and Wanner (2000) plasmid pCP20 to transform into this strain, when other strains transformed fine. Cause is unknown.5.BL21(DE3)F–ompT gal dcm lon hsdSB (rB-mB-) λ(DE3 [lacI lacUV5-T7 gene 1 ind1 sam7nin5])∙an E. coli B strain with DE3, a λ prophage carrying the T7 RNA polymerase gene and lacI q∙Transformed plasmids containing T7 promoter driven expression are repressed until IPTG induction of T7 RNA polymerase from a lacpromoter.∙Derived from B834 (Wood, 1966) by transducing to Met+.∙See the original Studier paper or the summary in Methods in Enzymology for more details.∙Whole genome sequence available [1]6.BL21 (DE3) pLysSF- ompT gal dcm lon hsdSB (rB- mB-) λ(DE3) pLysS(cm R)∙pLysS plasmid chloramphenicol resistant; grow with chloramphenicol to retain plasmid∙Chloramphenicol resistant∙The pLysS plasmid encodes T7 phage lysozyme, an inhibitor for T7 polymerase which reduces and almost eliminates expression fromtransformed T7 promoter containing plasmids when not induced.∙see Moffatt87 for details of pLysS and pLysE plasmids7.BNN93F- tonA21 thi-1 thr-1 leuB6 lacY1 glnV44 rfbC1 fhuA1 mcrB e14-(mcrA-)hsdR(rK -mK+) λ-∙Some C600 strains are really BNN93 8.BNN97∙BNN93 (λgt11)o A λgt11 lysogen producing phage at 42C9.BW26434, CGSC Strain # 7658Δ(araD-araB)567, Δ(lacA-lacZ)514(::kan), lacIp-4000(lacI q), λ-, rpoS396(Am)?, rph-1, Δ(rhaD-rhaB)568, hsdR514∙This information is from a printout sent by the E. coli Genetic Stock Center with the strain.∙ B.L. Wanner strain∙rph-1 is a 1bp deletion that results in a frameshift over last 15 codons and has a polar effect on pyrE leading to suboptimalpyrimidine levels on minimal medium. (Jensen 1993 J Bact. 175:3401) ∙Δ(araD-araB)567 was formerly called ΔaraBAD AH33 by Datsenko and Wanner∙Am = amber(UAG) mutation∙Reference: Datsenko and Wanner, 2000, PNAS, 97:6640NOTE:∙This promoter driving the expression of lacI was sequenced in this strain using a primer in mhpR (upstream of lacI) and a primer in the opposite orientation in lacI. The lac promoter was found to be identical to wildtype. Thus, the -35 sequence was GCGCAA not GTGCAA as expected with lacI q. Therefore this strain (or at least theversion obtained from the E. coli Genetic Stock Center) does NOT appear to be lacI q. According to Barry Wanner, this is an unexpected result. -Reshma 13:19, 5 May 2005 (EDT)∙"We have now confirmed that BW25113, BW25141, and BW26434 are all lacI+, and not lacI q. We thank you for alerting us to the error with respect to BW26434. Apparently, the lacI region was restored to wild-type in a predecessor of BW25113." (from Barry Wanner November 18, 2005)∙The genotype has been corrected at the CGSC10.C600F- tonA21 thi-1 thr-1 leuB6 lacY1 glnV44 rfbC1 fhuA1 λ-∙There are strains circulating with both e14+(mcrA+) and e14-(mcrA-) ∙General purpose host∙See CGSC#3004∙References: Appleyard, R.K. (1954) Genetics 39, 440; Hanahan, D.(1983) J. Mol. Biol. 166, 577.11.C600 hflA150 (Y1073, BNN102)F- thi-1 thr-1 leuB6 lacY1 tonA21 glnV44 λ- hflA150(chr::Tn10) ∙host for repressing plaques of λgt10 when establishing cDNA libraries∙Reference Young R.A. and Davis, R. (1983) Proc. Natl. Acad. Sci.USA 80, 1194.∙Tetracycline resistance from the Tn10 insertion12.CSH50F- λ- ara Δ(lac-pro) rpsL thi fimE::IS1∙See CGSC#8085∙References: Miller, J.H. 1972. Expts.in Molec.Genetics, CSH 0:14-0;Blomfeld et al., J.Bact. 173: 5298-5307, 1991.13.D1210HB101 lacI q lacY+14.DB3.1F- gyrA462 endA1 glnV44 Δ(sr1-recA) mcrB mrr hsdS20(rB -, mB-) ara14 galK2lacY1 proA2 rpsL20(Sm r) xyl5 Δleu mtl1∙useful for propagating plasmids containing the ccdB operon.∙gyrA462 enables ccdB containing plasmid propagation∙streptomycin resistant∙appears to NOT contain lacI (based on a colony PCR) --Austin Che 16:16, 18 June 2007 (EDT)<biblio>1.Bernard-JMolBiol-1992 pmid=13243242.Miki-JMolBiol-1992 pmid=1316444</biblio>15.DH1endA1 recA1 gyrA96 thi-1 glnV44 relA1 hsdR17(rK - mK+) λ-∙parent of DH5α∙An Hoffman-Berling 1100 strain derivative (Meselson68)∙more efficient at transforming large (40-60Kb) plasmids∙nalidixic acid resistant∙Reference: Meselson M. and Yuan R. (1968) Nature 217:1110 PMID 4868368.16.DH5αF- endA1 glnV44 thi-1 recA1 relA1 gyrA96 deoR nupG Φ80d lacZΔM15Δ(lacZYA-argF)U169, hsdR17(r K- m K+), λ–∙An Hoffman-Berling 1100 strain derivative (Meselson68)∙Promega also lists phoA∙nalidixic acid resistant∙References:o FOCUS (1986) 8:2, 9.o Hanahan, D. (1985) in DNA Cloning: A Practical Approach (Glover, D.M., ed.), Vol. 1, p. 109, IRL Press, McLean,Virginia.o Grant, S.G.N. et al. (1990) Proc. Natl. Acad. Sci. USA 87: 4645-4649 PMID 2162051.o Meselson M. and Yuan R. (1968) Nature 217:1110 PMID 4868368.17.DH10B (Invitrogen)F- endA1 recA1 galE15 galK16 nupG rpsL ΔlacX74 Φ80lacZΔM15 araD139 Δ(ara,leu)7697 mcrA Δ(mrr-hsdRMS-mcrBC) λ-∙suitable for cloning methylated cytosine or adenine containing DNA ∙an MC1061 derivative (Casadaban80). Prepare cells for chemical transformation with CCMB80 buffer∙blue/white selection∙While DH10B has been classically reported to be galU galK, the preliminary genome sequence for DH10B indicates that DH10B (and by their lineage also TOP10 and any other MC1061 derivatives) isactually galE galK galU+. Dcekiert 16:37, 23 January 2008 (CST)∙Genome sequence indicates that DH10B is actually deoR+. Presumably TOP10 and MC1061 are also deoR+.∙Streptomycin resistant∙References:o Casdaban, M. and Cohen, S. (1980) J Mol Biol 138:179 PMID 6997493.o Grant, S.G.N. et al. (1990) Proc. Natl. Acad. Sci. USA 87: 4645-4649 PMID 2162051.o E. coli Genetic Stock Center, MC1061 Recordo DH10B Genome Sequencing Project, Baylor College of Medicineo Complete sequence is available, see Durfee08, PMID 18245285.12.DH12S (Invitrogen)mcrA Δ(mrr-hsdRMS-mcrBC) φ80d lacZΔM15 ΔlacX74 recA1 deoR Δ(ara, leu)7697 araD139 galU galK rpsL F' [proAB+ lacI q ZΔM15]∙host for phagemid and M13 vectors∙useful for generating genomic libraries containing methylated cytosine or adenine residues∙streptomycin resistant∙References: Lin, J.J., Smith, M., Jessee, J., and Bloom, F. (1991) FOCUS 13, 96.; Lin, J.J., Smith, M., Jessee, J., and Bloom, F. (1992) BioTechniques 12, 718.19.DM1 (Invitrogen)F- dam-13::Tn9(Cm R) dcm- mcrB hsdR-M+ gal1 gal2 ara- lac- thr- leu- tonR tsxR Su0∙Host for pBR322 and other non-pUC19 plasmids; useful for generating plasmids that can be cleaved with dam and dcm sensitive enzymes ∙Chloramphenicol resistant∙Promega lists as F' not F-∙Reference: Lorow-Murray D and Bloom F (1991) Focus 13:2020.E. cloni(r) 5alpha (Lucigen)fhuA2Δ(argF-lacZ)U169 phoA glnV44 Φ80 Δ(lacZ)M15 gyrA96 recA1 relA1 endA1 thi-1 hsdR17∙Common cloning strain.21.E. cloni(r) 10G (Lucigen)F- mcrAΔ(mrr-hsd RMS-mcr BC) end A1 rec A1 Φ80dlac ZΔM15 Δlac X74 ara D139 Δ(ara,leu)7697 gal U gal K rps L nup G λ- ton A∙Common cloning strain.∙Resistant to phage T1.22.E. cloni(r) 10GF' (Lucigen)[F´ pro A+B+ lac IqZΔM15::Tn10 (TetR)] /mcr A Δ(mrr-hsd RMS-mcr BC) end A1 rec A1 Φ80d lac ZΔM15 Δlac X74 ara D139 Δ(ara, leu)7697 gal U gal K rps L nup Gλ ton A∙Strain for cloning and single-strand DNA production.23.E. coli K12 ER2738 (NEB)F´proA+B+ lacIq Δ(lacZ)M15 zzf::Tn10(TetR)/ fhuA2 glnV Δ(lac-proAB) thi-1 Δ(hsdS-mcrB)5∙Phage propagation strain∙Also available from Lucigen Corporation.24.ER2566 (NEB)F- λ- fhuA2 [lon] ompT lacZ::T7 gene 1 gal sulA11 Δ(mcrC-mrr)114::IS10 R(mcr-73::miniTn10-TetS)2 R(zgb-210::Tn10)(TetS) endA1 [dcm]∙Host strain for the expression of a target gene cloned in the pTYB vectors.∙Carry a chromosomal copy of the T7 RNA polymerase gene inserted into lacZ gene and thus under the control of the lac promoter. In the absence of IPTG induction expression of T7 RNA polymerase issuppressed by the binding of lac I repressor to the lac promoter.∙Deficient in both lon and ompT proteases.25.ER2267 (NEB)F´ proA+B+ lacIq Δ(lacZ)M15 zzf::mini-Tn10 (KanR)/ Δ(argF-lacZ)U169 glnV44 e14-(McrA-) rfbD1? recA1 relA1? endA1 spoT1? thi-1Δ(mcrC-mrr)114::IS10∙Commonly used for titering M13 phage because of the strain's F' plasmid, which carries KanR, and its slow growth, which promotes easy visualization of plaques.26.HB101F- mcrB mrr hsdS20(rB - mB-) recA13 leuB6 ara-14 proA2 lacY1 galK2 xyl-5mtl-1 rpsL20(Sm R) glnV44 λ-Please note that different sources have different genotypes so treat this information with caution.∙From a GIBCO BRL list of competent cells.∙Hybrid of E. coli K12 and E. coli B (but 98% K strain AB266 according to Smith et al.)∙Host for pBR322 and many plasmids∙Sigma lists the deletion Δ(gpt,proA). Check this.∙Promega does not list F-, mcrB, or mrr∙Streptomycin resistant∙References:o Boyer, H.W. and Roulland-Dussoix, D. (1969) J. Mol. Biol. 41, 459.o Smith, M., Lorow, D., and Jessee, J. (1989) FOCUS 11, 56 - pdf version from Invitrogeno Lacks S and Greenberg JR (1977) J Mol Biol 114:153.27.HMS174(DE3)F- recA1 hsdR(rK12- mK12+) (DE3) (Rif R)∙HMS174 strains provide the recA mutation in a K-12 background. Like BLR, these strains may stabilize certain target genes whoseproducts may cause the loss of the DE3 prophage.∙DE3 indicates that the host is a lysogen of lDE3, and therefore carries a chromosomal copy of the T7 RNA polymerase gene undercontrol of the lacUV5 promoter. Such strains are suitable forproduction of protein from target genes cloned in pET vectors by induction with IPTG.28.High-Control(tm) BL21(DE3) (Lucigen)F– ompT gal dcm hsdSB (rB- mB-) (DE3)/Mini-F lacI q1(Gent r)∙The HI-Control BL21(DE3) cells contain a single-copy BAC plasmid harboring a specially engineered version of the lacI q1 repressor allele. The lacI q1 allele expresses ~170-fold more lac repressor protein than the wild-type lacI gene.∙The increased pool of lac repressor in HI-Control BL21(DE3) cells maintains tight control over the expression of T7 RNA polymerase from the lacUV5 promoter, reducing leaky expression of genes cloned under a T7 promoter.∙an E. coli B strain with DE3, a λ prophage carrying the T7 RNA polymerase gene and lacI q∙Transformed plasmids containing T7 promoter driven expression are repressed until IPTG induction of T7 RNA polymerase from a lacpromoter.29.High-Control(tm) 10G (Lucigen)F- mcrAΔ(mrr-hsd RMS-mcr BC) end A1 rec A1 Φ80dlac ZΔM15 Δlac X74 ara D139 Δ(ara,leu)7697 gal U gal K rps L nup G λ- ton A/Mini-F lacI q1(Gent r) ∙The HI-Control 10G cells contain a single-copy BAC plasmid harboring a specially engineered version of the lacI q1 repressor allele. The lacI q1 allele expresses ~170-fold more lac repressor protein than the wild-type lacI gene.∙For stable cloning of T7 protein expression plasmids.∙Resistant to phage T1.30.IJ1126E. coli K-12 recB21 recC22 sbcA5 endA gal thi Su+ Δ(mcrC-mrr)102::Tn10 See Endy:IJ112631.IJ1127IJ1126 lacUV5 lacZ::T7 gene1-KnrSee Endy:IJ112732.JM83rpsL ara Δ(lac-proAB) Φ80dlacZΔM15∙Sigma lists thi. Check this.∙streptomycin resistant33.JM101glnV44 thi-1 Δ(lac-proAB) F'[lacI q ZΔM15 traD36 proAB+] ∙host for M13mp vectors∙recA+, r K+∙original blue/white cloning strain∙has all wt restriction systems∙References: Messing, J. et al. (1981) Nucleic Acids Res. 9, 309;Yanisch-Perron, C., Vieira, J., and Messing, J. (1985) Gene 33, 103.34.JM103endA1 glnV44 sbcBC rpsL thi-1 Δ(lac-proAB) F'[traD36 proAB+ lacI qlacZΔM15]∙streptomycin resistant∙References: Hanahan, D. (1983) J. Mol. Biol. 166:557-80.∙NEB says this strain encodes a prophage encoded EcoP1 endonuclease.∙Sigma lists (P1) (r K-m K+ rP1+ mP1+)35.JM105endA1 glnV44 sbcB15 rpsL thi-1 Δ(lac-proAB) [F' traD36 proAB+ lacI qlacZΔM15] hsdR4(rK -mK+)∙Sigma lists sbcC∙streptomycin resistant∙References: Yanisch-Perron, C., Vieira, J., and Messing, J. (1985) Gene 33, 103.36.JM106endA1 glnV44 thi-1 relA1 gyrA96 Δ(lac-proAB) F- hsdR17(rK -mK+)∙References: Yanisch-Perron, C., Vieira, J., and Messing, J. (1985) Gene 33, 103.37.JM107endA1 glnV44 thi-1 relA1 gyrA96 Δ(lac-proAB) [F' traD36 proAB+ lacI qlacZΔM15] hsdR17(RK - mK+) λ-∙host for M13mp vectors∙recA+, r K+∙Sigma lists e14- (McrA-)∙nalidixic acid resistant∙References: Yanisch-Perron, C., Vieira, J., and Messing, J. (1985) Gene 33, 103.38.JM108endA1 recA1 gyrA96 thi-1 relA1 glnV44 Δ(lac-proAB) hsdR17 (rK - mK+)∙nalidixic acid resistant∙deficient in expression of the lon protease due to IS186 transposon insertion -- J Mairhofer 18:59, 24 March 2010 (CET)<biblio>1.Reference pmid=20138928</biblio>39.JM109endA1 glnV44 thi-1 relA1 gyrA96 recA1 mcrB+Δ(lac-proAB) e14- [F' traD36proAB+ lacI q lacZΔM15] hsdR17(rK -mK+)∙From NEB∙Partly restriction-deficient; good strain for cloning repetitive DNA (RecA–).∙Suppresses many amber mutations when glutamine is acceptable butnot the S100 or S7mutations of λ, e.g., λgt11.∙Can also be used for M13 cloning/sequencing and blue/white screening.∙Sigma lists e14-∙nalidixic acid resistant∙deficient in expression of the lon protease due to IS186 transposon insertion -- J Mairhofer 18:59, 24 March 2010 (CET) ∙From C. Yanisch-Perron, J. Vieira, and J. Messing. Improved M13 phage cloning vectors and host strains: nucleotide sequences of the M13mp18 and pUC19 vectors. Gene, 33(1):103–19, 1985.∙Some information from Mary Berlyn at the E. coli Genetic Stock Center: One of the reasons the original curator of this collection did not accession the JM109, JM103, etc. strains was because she found it impossible to be sure of the derivation and therefore the details of the genotype. But I think it's safe to assume that the F' in this strain is derived from or similar to F128 which extends from the proBA region through the lac operon. It thus carries the wildtype genes for all loci in that region except those indicated as mutant for the genotype of the F'. So it carries the lacZ(alpha-complementation) deletion lacZ58(M150 and the lacI mutation lacIq, but it has the lacY+ gene also on the F-prime. On thechromosome it lacks all the lac operon genes.NOTE: The promoter driving the expression of lacI was sequenced in this strain using a primer in mhpR (upstream of lacI) and a primer in the opposite orientation in lacI. The lac promoter was found to be identical to wildtype. Thus, the -35 sequence was GCGCAA not GTGCAA as expected with lacI Q. Therefore this strain (or at least the version we have) does NOT appear to be lacI Q unless there is another copy of lacI elsewhere. This result is somewhat confirmed by the fact that a lacI regulated promoter driving expression of YFP on a medium copy vector does not repress completely. -Reshma 13:48, 5 May 2005 (EDT)40.JM109(DE3)JM109 + λ(DE3)∙DE3 prophage carrying T7 polymerase expression cassette∙Same cassette as BL21(DE3) carrying a lac inducible T7 RNA polymerase and lacI q∙nalidixic acid resistant41.JM110rpsL thr leu thi lacY galK galT ara tonA tsx dam dcm glnV44 Δ(lac-proAB)e14- [F' traD36 proAB+ lacI q lacZΔM15] hsdR17(rK -mK+)∙Sigma fails to list tonA tsx e14 fhuA hsdR17∙(e14-) status uncertain∙streptomycin resistant42.JM2.300lacI22, LAM-, e14-, rpsL135(strR), malT1(LamR), xyl-7, mtl-1, thi-1 ∙Some folks have been using this strain (i.e., Elowitz, Gardner) and it took me too long to find the CGSC#.∙This strain is no longer available from the CGSC43.LE392glnV44 supF58 (lacY1 or ΔlacZY) galK2 galT22 metB1 trpR55 hsdR514(rK -mK+)∙Sigma lists F- e14- 44.Mach1ΔrecA1398 endA1 tonA Φ80ΔlacM15 ΔlacX74 hsdR(rK - mK+)∙From Invitrogen∙Doubling time approx. 50 min and supposedly fastest growing chemically competent cloning strain available∙Mach1 cells are derivatives of E. coli W strains (ATCC 9637, S. A.Waksman), rather than E. coli K-12. This may have implications for BL-1 status for some facilities (apparently not for MIT).∙See Bloom04 patent for details on the construction and properties of this strain.45.MC1061F-Δ(ara-leu)7697 [araD139]B/rΔ(codB-lacI)3 galK16 galE15 λ-e14-mcrA0 relA1 rpsL150(strR) spoT1 mcrB1 hsdR2(r-m+)∙Streptomycin resistant∙The thr-leu region was transduced from an E. coli B/r strain (SB3118) in early steps of strain construction.∙Parent of DH10B/TOP10 and derived strains∙References:o E. coli Genetic Stock Center, MC1061 Recordo Casdaban, M. and Cohen, S. (1980) J Mol Biol 138:179 PMID 6997493.o Complete DH10B sequence is available, see Durfee08, PMID 18245285.46.MC4100Δ(argF-lac)169* &lambda- e14- flhD5301 Δ(fruK-yeiR)725 F- [araD139]B/r(fruA25)‡ relA1 rpsL150(strR) rbsR22 Δ(fimB-fimE)632(::IS1) deoC1∙The thr-leu region was transduced from an E. coli B/r strain (SB3118) in early steps of strain construction.∙This paper compares MC4100 to MG1655 and describes the significant deletions.∙*The paper referenced above showed that this deletion was larger than previously known. The deletion now covers ykfD-b0350.∙‡The fruA25 allele is attributed to the deletion of fruK-yeiR. This means fruA is present but its promoter has been deleted.∙The paper also shows that the e14 element is deleted in MC4100. One of the genes removed by this deletion is mcrA, which encodes anenzyme that restricts DNA containing methylcytosine. However,other E. coli K-12 restriction/modification systems are stillpresent in MC4100. MC4100 still encodes the McrBC5-methylcytosine=specific restriction enzyme and theHsdR/HsdS/HsdM type I restriction-modification complex.∙Table three of the paper lists all genes believed to be deleted in MC4100. The methods used in the paper can detect deletions but not loss of function mutations.∙See CGSC#615247.MG1655F- λ- ilvG- rfb-50 rph-1This is the "wild type" K-12 strain which was sequenced, and should be used when PCRing genes from the sequenced genome. It also looks very healthy under the microscope -- a dramatic difference from most of the cloning strains, which appear sick.∙See CGSC#6300∙See ATCC 700926<biblio>1.Blattner-Science-1997 pmid=9278503</biblio>∙More accurate sequence correcting 243 errors in the original sequencing Horiuchi2006. New Genbank accession number U00096.2 48.OmniMAX2From Invitrogen: "This strain overexpresses the Lac repressor (lacIq gene). For blue/white screening, you will need to add IPTG to induce expression from the lac promoter. Strain is resistant to T1 bacteriophage."F′ {proAB+ lacIq lacZΔM15 Tn10(TetR) Δ(ccdAB)} mcrAΔ(mrr-hsdRMS-mcrBC) φ80(lacZ)ΔM15 Δ(lacZYA-argF) U169 endA1 recA1 supE44 thi-1 gyrA96 relA1 tonA panD49.OverExpress(tm)C41(DE3) (Lucigen)F– ompT gal dcm hsdSB (rB- mB-)(DE3)∙The OverExpress strains contain genetic mutations phenotypically selected for conferring tolerance to toxic proteins. The strain C41(DE3) was derived from BL21(DE3). This strain has at least one uncharacterized mutation, which prevents cell death associated with expression of many recombinant toxic proteins. The strain C43(DE3) was derived from C41(DE3) by selecting for resistance toa different toxic protein and can express a different set of toxicproteins to C41(DE3).∙an E. coli B strain with DE3, a λ prophage carrying the T7 RNA polymerase gene and lacI q∙Transformed plasmids containing T7 promoter driven expression are repressed until IPTG induction of T7 RNA polymerase from a lacpromoter.50.OverExpress(tm)C41(DE3)pLysS (Lucigen)F– ompT gal dcm hsdSB (rB- mB-)(DE3)pLysS (Cm r)∙The OverExpress strains contain genetic mutations phenotypically selected for conferring tolerance to toxic proteins. The strain C41(DE3) was derived from BL21(DE3). This strain has at least one uncharacterized mutation, which prevents cell death associatedwith expression of many recombinant toxic proteins. The strainC43(DE3) was derived from C41(DE3) by selecting for resistance toa different toxic protein and can express a different set of toxicproteins to C41(DE3).∙an E. coli B strain with DE3, a λ prophage carrying the T7 RNA polymerase gene and lacI q∙Transformed plasmids containing T7 promoter driven expression are repressed until IPTG induction of T7 RNA polymerase from a lacpromoter.∙The pLysS plasmid encodes T7 phage lysozyme, an inhibitor for T7 polymerase which reduces and almost eliminates expression fromtransformed T7 promoter containing plasmids when not induced. 51.OverExpress(tm)C43(DE3) (Lucigen)F– ompT gal dcm hsdSB (rB- mB-)(DE3)∙The OverExpress strains contain genetic mutations phenotypically selected for conferring tolerance to toxic proteins. The strain C41(DE3) was derived from BL21(DE3). This strain has at least one uncharacterized mutation, which prevents cell death associated with expression of many recombinant toxic proteins. The strain C43(DE3) was derived from C41(DE3) by selecting for resistance toa different toxic protein and can express a different set of toxicproteins to C41(DE3).∙an E. coli B strain with DE3, a λ prophage carrying the T7 RNA polymerase gene and lacI q∙Transformed plasmids containing T7 promoter driven expression are repressed until IPTG induction of T7 RNA polymerase from a lacpromoter.52.OverExpress(tm)C43(DE3)pLysS (Lucigen)F– ompT gal dcm hsdSB (rB- mB-)(DE3)pLysS (Cm r)∙The OverExpress strains contain genetic mutations phenotypically selected for conferring tolerance to toxic proteins. The strain C41(DE3) was derived from BL21(DE3). This strain has at least one uncharacterized mutation, which prevents cell death associatedwith expression of many recombinant toxic proteins. The strainC43(DE3) was derived from C41(DE3) by selecting for resistance toa different toxic protein and can express a different set of toxicproteins to C41(DE3).∙an E. coli B strain with DE3, a λ prophage carrying the T7 RNA polymerase gene and lacI q∙Transformed plasmids containing T7 promoter driven expression are repressed until IPTG induction of T7 RNA polymerase from a lacpromoter.∙The pLysS plasmid encodes T7 phage lysozyme, an inhibitor for T7 polymerase which reduces and almost eliminates expression fromtransformed T7 promoter containing plasmids when not induced. 53.Rosetta(DE3)pLysSF-ompT hsdSB (RB-mB-) gal dcm λ(DE3 [lacI lacUV5-T7 gene 1 ind1 sam7 nin5])pLysSRARE (Cam R)∙an E. coli B strain with DE3, a λ prophage carrying the T7 RNA polymerase gene and lacI q∙Transformed plasmids containing T7 promoter driven expression are repressed until IPTG induction of T7 RNA polymerase from a lacpromoter.∙Chloramphenicol resistant∙pLysSRARE contains tRNA genes argU, argW, ileX, glyT, leuW, proL, metT, thrT, tyrU, and thrU. The rare codons AGG, AGA, AUA, CUA, CCC, and GGA are supplemented.∙The pLysS plasmid encodes T7 phage lysozyme, an inhibitor for T7 polymerase which reduces and almost eliminates expression fromtransformed T7 promoter containing plasmids when not induced.∙see Moffatt87 for details of pLysS and pLysE plasmids∙Novagen strain manual54.Rosetta-gami(DE3)pLysSΔ(ara-leu)7697 ΔlacX74 ΔphoA PvuII phoR araD139 ahpC galE galK rpsL (DE3) F'[lac+ lacI q pro] gor522::Tn10 trxB pLysSRARE (Cam R, Str R, Tet R)∙an E. coli K-12 strain with DE3, a λ prophage carrying the T7 RNA polymerase gene and lacI q∙Transformed plasmids containing T7 promoter driven expression are repressed until IPTG induction of T7 RNA polymerase from a lacpromoter.∙ahpC mutation allows trxB/gor double mutants to grow in the absence of reducing medium∙pLysSRARE contains tRNA genes argU, argW, ileX, glyT, leuW, proL, metT, thrT, tyrU, and thrU. The rare codons AGG, AGA, AUA, CUA, CCC, and GGA are supplemented.∙The pLysS plasmid encodes T7 phage lysozyme, an inhibitor for T7 polymerase which reduces and almost eliminates expression fromtransformed T7 promoter containing plasmids when not induced.∙see Moffatt87 for details of pLysS and pLysE plasmids∙Chloramphenicol resistant∙Kanamycin resistant∙Tetracycline resistant∙Streptomycin resistant∙Novagen strain manual55.RR1HB101 recA+56.RV308lacIq-, su-, ΔlacX74, gal IS II::OP308, strA K12 derivative used for industrial protein production. ATCC strain 31608, deposited by Genentech.57.SOLR (Stratagene)e14-(McrA-) Δ(mcrCB-hsdSMR-mrr)171 sbcC recB recJ uvrC umuC::Tn5 (Kan r) lac gyrA96 relA1 thi-1 endA1 λR [F’ proAB lacI q Z ΔM15]C Su-∙Used in phagemid recovery (LambdaZap)∙Kanamycin resistant∙Stratagene E. coli Genotype Strains58.SS320 (Lucigen)F'[proAB+lacIqlacZΔM15 Tn10 (tet r)]hsdR mcrB araD139Δ(araABC-leu)7679 Δlac X74 galUgalK rpsL thi∙Useful for phage display.∙Sidhu, S.S., Weiss, G.A., and Wells, J.A. (2000) J. Mol. Biol. 296, 487-495.59.STBL2 (Invitrogen)F- endA1 glnV44 thi-1 recA1 gyrA96 relA1 Δ(lac-proAB) mcrAΔ(mcrBC-hsdRMS-mrr) λ-∙host for unstable sequences such as retroviral sequences and direct repeats∙nalidixic acid resistant∙References: Trinh, T., Jessee, J., Bloom, F.R., and Hirsch, V. (1994) FOCUS 16, 78.60.STBL3 (Invitrogen)F- glnV44 recA13 mcrB mrr hsdS20(rB-, mB-) ara-14 galK2 lacY1 proA2 rpsL20 xyl-5 leu mtl-1∙Streptomycin resistant∙endA+, use care in preparing DNA from this strain61.STBL4endA1 glnV44 thi-1 recA1 gyrA96 relA1 Δ(lac-proAB) mcrAΔ(mcrBC-hsdRMS-mrr) λ- gal F'[ proAB+ lacI q lacZΔM15 Tn10]∙Tetracycline resistant (from Tn10 insertion)∙STBL2 + blue/white selection62.SURE (Stratagene)。
