pGL3-Promoter哺乳动物表达载体说明
pFR-luc使用说明

pFR-luc编号载体名称北京华越洋VECT76052pFR-lucpFR-luc载体相关的哺乳动物表达载体:SuperCos I pDsRed2-Bid pNFκB-MetLuc2-ReporterpYr-adshuttle-3pAcGFP1-N1pEF1α-IRES-DsRed-Express2pVitro2-neo-mcs pSecTag2A pCMV-DsRed-Express2pUB6/V5-His/LacZ pGL4.27pcDNA3.1/NT-GFP-TOPOpUB6/V5-His C pGL4.26pEF1α-IRES-ZsGreen1pUB6/V5-His B pACT pCMV-Tag2ApUB6/V5-His A pBIND-Id Control pCMV-Tag5BpTracer-CMV2pTRE2pAcGFP1-C In-Fusion ReadypSV-β-Galactosidase pRevTRE p3XFLAG-CMV-14pSI pTK-hyg p3XFLAG-CMV-8pSG5pTRE3G-Luc pFLAG-CMV-2pSFV1pSwitch pcDNA3.3-TOPOpSecTag2/Hygro A pcDNA4/His C pcDNA6.2/cLumio-DESTpSecTag B c-Flag pcDNA3pCMV-tdTomatopRluc-N2pcDNA4/TO/Myc-His A pAcGFP1-MitopPICZalpha D pcDNA6/myc-His B pAcGFP1-N In-Fusion Ready pORF-lacZ pcDNA6/V5-His B pDsRed-Monomer-N In-Fusion Ready pORF-HSV1tk pcDNA6.2/nTC-Tag-DEST pcDNA4/TO/Myc-His BpOG44pOptiVEC-TOPO pIRES2-EGFPpNTAP-B pcDNA5/FRT pcDNA3.1/His CpMEP4pGL4.30pcDNA3.1/CT-GFP-TOPOpLVX-ZsGreen-miRNA-Puro pGL4.19pEF1α-IRES-AcGFP1pLVX-IRES-Puro-3xFlag pACT-MyoD pcDNA3.2/V5/GW/D-TOPO pLPCX pCMV-BD pcDNA4/TO/Myc-His/LacZ pLEGFP-N1pCMV-Tet3G pcDNA4/HisMax-TOPOpKH3pTet on advanced p3XFLAG-CMV-13pIRES-puro2pTRE-Tight p3xFLAG-CMV-10phRL-TK pIND pFLAG-CMV-3pG5lac pGene/V5-His B pcDNA4/TO/Myc-His CpFR-luc pOPRSVI pcDNA6.2/C-YFP-DESTpEF6/myc-His C pcDNA4/HisMax A pcDNA6.2/cTC-Tag-DESTpEF4/V5-His A pIRESpuro3pcDNA6.2/nGeneBLAzer-DESTpEF1/myc-His lacZ pIRESneo3pCRE-MetLuc2-ReporterpEF1/myc-His C pIRESneo2pEF1α-DsRed-Express2pEF1/myc-His B pcDNA4/myc-His A pDsRed-Express-C1pEF1/myc-His A pCMV-PKA pEF1α-DsRed-Monomer-N1 pECFP-Mito pAcGFP1-N3pDD-AmCyan1ReporterpECFP-ER pcDNA5/FRT/TO pCRE-DD-AmCyan1pDP8rs pBApo-CMV-Pur pIRES2-DsRed-Express2pDP5rs pBApo-EF1α-pur pDsRed-Express-N1pDP4rs ptdTomato-N1pcDNA6.2/nLumio-DESTpDP3rs pAcGFP1-Golgi pcDNA6/myc-His ApDP2rs pAcGFP1-p53pcDNA6/V5-His ApDP1rs pAcGFP1-Actin pcDNA5/FRT/TO-TOPOpCMV-Myc-C pBI-CMV2pcDNA6.2/V5/GW/D-TOPOpCMV-HA-N pEF1α-tdTomato pcDNA6.2/cGeneBLAzer-DEST pCMV-HA-C pEF1α-tdTomato pcDNA6.2/nGeneBLAzer-GW/D-TOPO pCMV6-XL4pEBVHis A pcDNA5/TOpCMV6-AC-GFP pGL4.10pBApo-CMV-neopCMV5pGL4.29pDsRed-MonomerpCI pGL4.13pIRESpCGN pG5luciferase pIRES-hrGFP-1apcDNA6.2/EmGFP-Bsd/V5-DEST pCMV-AD pDsRed-Express2-N1pcDNA4/V5-His A pRevTet-Off pCMV-Tag3BpcDNA3-mRFP pTet-Off pCRE-hrGFPpcDNA3-CFP pTRE2-hygro pDsRED2-MitopcDNA3.1(+)/myc-His C pVgRxR pAcGFP1-FpcDNA3.1(+)/myc-His A pOPI3CAT pAcGFP1-C1pcDNA3.1(+)/CAT pBK-RSV pAsRed2-C1pcDNA3.1(-)/myc-His C pIRES2-DsRed2pAsRed2-N1pcDNA3.1/Zeo(+)pCMV-Myc pAcGFP1-LampcDNA3.1/Zeo(-)pCMV-Tag2C pAcGFP1-CpcDNA3.1/V5-His C pCMV-Tag5A pSEAP2-BasicpcDNA3.1/Hygro(+)pCMV-Tag3C pBI-CMV3pcDNA3.1/Hygro(-)p3XFLAG-CMV-7pNFkB-DD-tdTomato pcDNA3.1/GS p3XFLAG-CMV-9pcDNA3.1/His A pBS185CMV-Cre pFLAG-CMV-4pEBVHis B pBIND-GFP pBI-CMV4pGL4.75pBiFC-VN155(I152L)pcDNA4/His A pGL4.20pBiFC-CC155pcDNA4/myc-His B pCMV-SPORT6 pBiFC-CrN173pcDNA4/HisMax C pCMV-SPORT6 pBiFC-bJunVN155(I152L)pCMV-Tag4A pBINDpBD-p53pcDNA6/myc-His C pBD-NF-κBpBD-NF-kB pCMV-Tag2B pRevTet-OnpBC1pGRN145pTet-OnpAD-TRAF pCMV-MEK1pTRE3GpAD-SV40T pCMV-Tag3A pcDNA4/TO pAAV-ZsGreen-miRNA pCMVLacI pcDNA4/His B pAAV-tTS-shRNA pBI-CMV1pcDNA4/HisMax B pIREShyg3pEF1α-AcGFP1-N1pcDNA4/myc-His C pcDNA6/TR pCMV-LacZ pcDNA3.1/His B pDsRed-Express2-C1pCMV-Tag4B pcDNA6/V5-His C pBI-CMV5pCMV-Tag5C pCHO1.0 pAmCyan1-C1plRES2-ZsGreen1pGL3-Promoter pAcGFP1-Mem p3XFLAG-CMV-7.1pCMV-MEKK1 pAcGFP1-C3pFLAG-CMV-5a pFLAG-CMV2pTT5pBudCE4.1pAcGFP1-C2 pSEAP2-Control pREP4ptdTomato-C1 pIRES2-AcGFP1pBApo-CMV pCRE-DD-tdTomato pAcGFP1-Hyg-C1。
报告基因和荧光淬灭
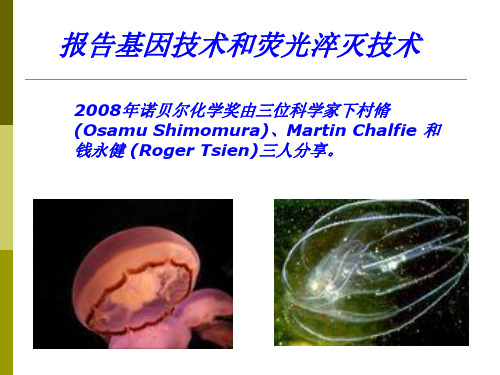
利用某种物质对某一种荧光物质的荧光猝灭作用而建立的对该猝灭剂 的荧光测定方法,即为荧光猝灭法
荧光猝灭法比直接荧光测定法更为灵敏,具有更高的选择性
肿瘤转移过程
应用举例:
MMP-2荧光分析药物筛选原理图
加 入 底 物 Mca-Pro-Leu-Gly-Leu-Dpa-Ala-Arg-NH2 , 在 37C温育过程中,MMP-2水解底物,释放出Mca发光基团和 Dpa淬灭基团。此时可以检测到荧光,随着时间延长,两种基团 浓度增加,基团之间相互碰撞机率增大,导致发光基团荧光逐渐 被淬灭。
荧光显微镜
多管水母科:Aequorleidae 27-30kD, 395nM
激光共聚焦显微镜
海紫罗兰科:Renillidae 54 kD, 498nM
荧光激活的细胞分拣器(FACS)
共定位
FRET,荧光能量共振转移
FRET,即荧光能量共振转移(fluorescence resonance energy transfer,) 随着绿色荧光蛋白应用技术的发展,成为检测活体中生物大分子纳米级距离和 纳米级距离变化的有力工具,在生物大分子相互作用分析、细胞生理研究、免 疫分析等方面有着广泛的应用。 原理:荧光能量共振转移是距离很近的两个荧光分子间产生的 一种能量转移现 象。当供体荧光分子的发射光谱与受体荧光分子的吸收光谱重叠,并且两个分 子的距离在10nm范围以内时,就会发生一种非放射性的能量转移,即FRET现象, 使得供体的荧光强度比它单独存在时要低的多(荧光猝灭),而受体发射的荧 光却大大增强(敏化荧光)。
发光机理:
BL 以脂肪醛(RCHO)为底物,在还原型黄素单核苷酸(FMNH2)及氧 脂肪醛氧化成脂肪酸+光子(490n荧光)
pDsRed2-Bid哺乳动物表达载体说明

pDsRed2-Bid编号 载体名称北京华越洋生物VECT6108 pDsRed2-‐BidpDsRed2-‐Bid载体基本信息载体名称: pDsRed2-Bid质粒类型: 哺乳动物细胞表达载体;信号通路报告载体高拷贝/低拷贝: 高拷贝克隆方法: 限制性内切酶,多克隆位点启动子: CMV IE载体大小: 5.3 kb5' 测序引物及序列: --3' 测序引物及序列: --载体标签: --载体抗性: 卡那霉素筛选标记: 新霉素(Neomycin)克隆菌株: DH5α, HB101宿主细胞(系): 常规细胞系,293、CV-1、CHO等备注: pDsRed2-Bid载体是红色荧光报告载体,用于研究依赖Bid的细胞凋亡信号通路。
稳定性: 瞬表达或稳表达组成型/诱导型: 组成型病毒/非病毒: 非病毒pDsRed2-‐Bid载体质粒图谱和多克隆位点信息pDsRed2-Bid载体描述pDsRed2-Bid is a mammalian expression vector that encodes a fusion of Discosoma sp. red fluorescent protein (DsRed2; 1, 2) and Bid, a member of the Bcl-2 “pro-apoptosis” family (3). Developed for our ApoAlert line, pDsRed2-Bid is designed to help researchers study Bid-dependent apoptosis pathways. Because of its fluorescent label, the Bid-DsRed2 fusion is easily detected by microscopy, allowing researchers to track its movements in response to certain apoptosis inducing agents (e.g., TNF-α).Bid’s activity is closely tied to its location in the cell. In healthy, non-apoptotic cells, Bid normally resides in the cytosol. But soon after the induction of apoptosis, it translocates to mitochondria, where it stimulates the release of cytochrome c—a key amplification step in the apoptotic cascade (4–7). The translocation is triggered by caspase-8, which, when activated by a death signal, cleaves Bid to produce a 15-kDa C-terminal fragment. The fragment, often referred to as truncated Bid or tBid, transmits the death signal further by translocating to the mitochondria. You can monitor this event visually by expressing Bid as a DsRed2 fusion. Bid-DsRed2, thefusion expressed by this vector, for example, emits red fluorescence—even after truncation—so it can be followed as it moves from the cytosol to the mitochondria. To drive expression of Bid-DsRed2, this vector contains the immediate early promoter of cytomegalovirus (PCMV IE), positioned just upstream of the Bid sequence. A short linker joins the Bid coding sequence to the 5'-end of DsRed2. Farther downstream, the vector contains a pair of SV40 polyadenylation signals, which direct proper processingof the 3'-end of the Bid-DsRed2 mRNA. The vector also contains an SV40 origin for replication in mammalian cells expressing the SV40 T antigen, a pUC origin of replication for propagation in E. coli, and an f1 origin for single-stranded DNA production. A neomycin-resistance cassette (Neor), consisting of the SV40 early promoter, the neomycin/kanamycin resistance gene of Tn5, and polyadenylation signals from the Herpes simplex virus thymidine kinase (HSV TK) gene, allows stably transfected eukaryotic cells to be selected using G418 (8). A bacterial promoter upstream of the cassette confers kanamycin resistance (Kanr) to E. coli.Cells transfected with pDsRed2-Bid constitutively express Bid as an N-terminal fusionto DsRed2. pDsRed2-Bid can be introduced into a variety of mammalian cell lines using any standard transfection method. If required, stable transfectants can be selected using G418 (8).Propagation in E. coliSuitable host strains: DH5α, HB101 and other general purpose strains. Single-stranded DNA production requiresa host containing an F plasmid such as JM109 or XL1-Blue.Selectable marker: plasmid confers resistance to kanamycin (50 μg/ml) to E. coli hosts.E. coli replication origin: pUCCopy number: ~500Plasmid incompatibility group: pMB1/ColE1Red Fluorescent Protein (DsRed2)Excitation/Emission Maxima: 558 nm / 583 nm其他哺乳动物表达载体:pCHO1.0 pBApo-CMV-Pur pOPRSVIpcDNA3.1/His C pcDNA5/FRT/V5-His-TOPO pREP4pcDNA3.1/His B pcDNA5/FRT/TO-TOPO pDual-GCpcDNA3.1/His A pcDNA5/TO pBK-RSVpIRESpuro3 pcDNA5/FRT/TO pBK-CMVpIRES2-EGFP pcDNA5/FRT pBI-CMV4pTT5 pFLAG-CMV2 pcDNA4/TO/Myc-His/LacZ pNFkB-DD-tdTomato pcDNA3.1/CT-GFP-TOPO pOPI3CATpBI-CMV5 pcDNA3.1/NT-GFP-TOPO pGene/V5-His B pSEAP2-Basic pOptiVEC-TOPO pSwitchpSEAP2-Control pCMV-MEKK1 pCMVLacIpBI-CMV3 pCMV-MEK1 pVgRxRpBI-CMV2 pCMV-PKA pINDpBI-CMV1 pcDNA6.2/nTC-Tag-DEST pTRE3G-Luc pNFκB-MetLuc2-Reporter pcDNA6.2/cTC-Tag-DEST pTRE3GpCRE-MetLuc2-Reporter pcDNA3.2/V5/GW/D-TOPO pTRE2-hygro pAcGFP1-Actin pcDNA6.2/V5/GW/D-TOPO pTRE-Tight pAcGFP1-N In-Fusion Ready pcDNA6.2/nGeneBLAzer-GW/D-TOPO pTK-hyg pAcGFP1-C3 pcDNA6.2/C-YFP-DEST pTet-On pAcGFP1-C pcDNA6.2/cGeneBLAzer-DEST pTet-Off pAcGFP1-p53 pcDNA6/V5-His A pTet on advanced pAcGFP1-Mito pcDNA6/V5-His B pRevTRE pAcGFP1-Mem pcDNA6/V5-His C pRevTet-On pAcGFP1-Lam pcDNA6/myc-His C pRevTet-Off pAcGFP1-Golgi pcDNA6/myc-His A pCMV-Tet3G pAcGFP1-F pcDNA6/myc-His B pTRE2 pAcGFP1-Hyg-C1 pcDNA6.2/nGeneBLAzer-DEST pBD-NF-κB pAsRed2-N1 pcDNA4/HisMax-TOPO pCMV-AD ptdTomato-N1 pcDNA6.2/nLumio-DEST pCMV-BD pCMV-tdTomato pcDNA6.2/cLumio-DEST pBIND-Id Control pCRE-DD-tdTomato pcDNA4/myc-His C pBINDpCMV-DsRed-Express2 pcDNA4/HisMax C pG5 luciferase pEF1α-tdTomato pcDNA4/HisMax A pACT-MyoD pCRE-hrGFP c-Flag pcDNA3 pACT ptdTomato-C1 pcDNA4/HisMax B pCMV-SPORT6 pAsRed2-C1 pcDNA4/myc-His B pGL4.13pGL3-Promoter pcDNA4/myc-His A pGL4.19pGL3 basic pcDNA4/His C pGL4.26 pAcGFP1-C2 pcDNA4/His B pGL4.20 pAcGFP1-C1 pcDNA4/His A pGL4.29 pAcGFP1-N3 pcDNA6/TR pGL4.30 pAcGFP1-N2 pcDNA4/TO/Myc-His A pGL4.27 pAcGFP1-N1 pcDNA4/TO pGL4.75 pAcGFP1-C In-Fusion Ready pcDNA4/TO/Myc-His B pGL4.10pCRE-DD-AmCyan1 pcDNA4/TO/Myc-His C pGRN145 pNFkB-DD-AmCyan1 pcDNA3.3-TOPO pSecTag2 A pDsRed2-Bid pBudCE4.1 pEBVHis B pDsRED2-Mito pFLAG-CMV-4 pEBVHis ApDD-AmCyan1 Reporter pFLAG-CMV-3 pCMV-Tag 3C pAmCyan1-N1 pFLAG-CMV-2 pCMV-Tag 3A pAmCyan1-C1 pFLAG-CMV-5a pCMV-Tag 3B pEF1α-IRES-DsRed-Express2 p3XFLAG-CMV-9 pCMV-Tag 5C pEF1α-DsRed-Monomer-N1 p3xFLAG-CMV-10 pCMV-Tag 5A pDsRED-Monomer-N1 p3XFLAG-CMV-8 pCMV-Tag 4ApDsRed-Express-N1 p3XFLAG-CMV-7.1 pCMV-Tag 5Bp3XFLAG-CMV-7 pDsRed-Monomer-N In-Fusion Ready pCMV-Tag 4B pDsRed-Express-C1 p3XFLAG-CMV-13 pCMV-Tag 2C pIRES2-ZsGreen1 p3XFLAG-CMV-14 pCMV-Tag 2B pDsRed-Express2-C1 plRES2-ZsGreen1 pCMV-Tag 2A pDsRed-Express2-N1 pBApo-EF1α-pur pCMV-LacZpEF1α-DsRed-Express2 pBApo-EF1α-neo pCMV-MycpIRES2-DsRed-Express pBApo-CMV pEF1α-IRES-AcGFP1 pIRES2-DsRed-Express2 pBApo-CMV-neo pEF1α-IRES-ZsGreen1 pIRES-hrGFP-1a pIRES-EGFP pEF1α-AcGFP1-N1 pIRESneo2 pIRESneo3 pIRES2-DsRed2 pIRESneo pDsRed-Monomer pIRES2-AcGFP1 pIREShyg3 pIRES。
pGL3—EPO—α—MHC启动子荧光素酶报告基因质粒的构建及缺氧调控功能考察

pGL3—EPO—α—MHC启动子荧光素酶报告基因质粒的构建及缺氧调控功能考察目的考察EPO-α-MHC启动子在缺氧条件下的调控表达作用。
方法构建pGL3-EPO-α-MHC启动子编码荧光素酶报告基因的质粒,以含pGL3-EPO-α-MHC和pGL3-CMV启动子的质粒分别转染缺氧和常氧的心肌细胞(H9C2),检测荧光素酶的表达。
结果pGL3-CMV启动子质粒在缺氧条件下的报告基因表达与常氧条件相比显著下降(P < 0.05),而pGL3-EPO-α-MHC启动子在缺氧条件下能够显著上調报告基因的表达 4.3倍(P < 0.01)。
结论EPO-α-MHC启动子具有缺氧调控功能。
标签:pGL3-EPO-α-MHC启动子;荧光素酶报告基因;质粒构建;缺氧调控α-MHC promote是研究报道中常用的一种心肌特异性启动子,用于调控治疗基因在心肌组织的特异性表达[1]。
为了增加治疗基因在心肌缺血缺氧区域的表达,本研究选用促红细胞生成素(erythropoietin,EPO)作为心肌特异性启动子的缺氧反应调控元件。
构建pGL3-EPO-α-MHC启动子编码荧光素酶报告基因的质粒(pGL3-EPO-α-MHC-LUC),考察该质粒在缺氧条件下的表达,评价EPO-α-MHC启动子的缺氧调控功能。
1 材料与方法1.1 仪器与试剂荧光化学发光酶标仪(美国Thermo公司);紫外分光光度计(美国Thermo 公司)。
荧光素酶检测试剂盒、BCA检测试剂盒、LDH检测试剂盒(美国Promega 公司);Lipofectamine 2000(美国Invitrogen 公司);DMEM培养液(美国GIBCO 公司);H9C2细胞(中科院上海细胞库);pGL3- CMV-LUC质粒(美国Promega 公司);质粒抽提试剂盒(美国Axygen公司);其他试剂均为国产分析纯。
1.2 pGL3-EPO-α-MHC-LUC质粒构建pGL3-EPO-α-MHC-LUC质粒构建[2-3],委托上海生工合成EPO-α-MHC基因序列,限制性内切酶NheⅠ和XhoⅠ对pGL3- CMV-LUC质粒进行双酶切,与EPO-α-MHC启动子基因片段于16℃酶连过夜。
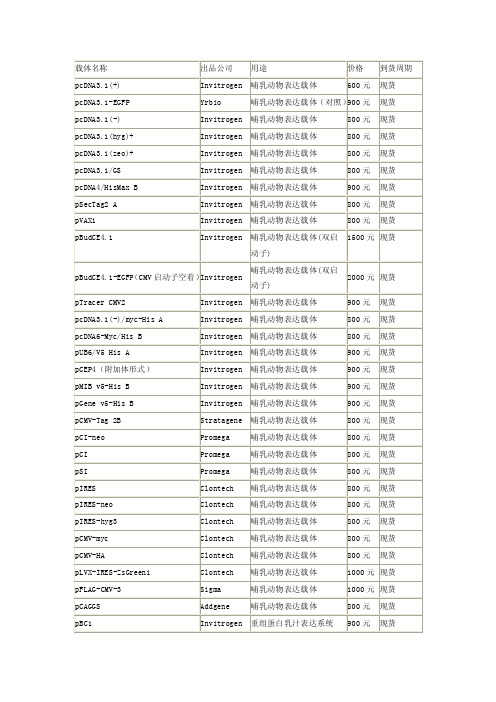
载体——精选推荐
NEB
原核表达 载体
NEB
配套大肠 杆菌
Amersham 原核表达载体
Amersham 原核表达载体
Amersham 原核表达载体
Invitrogen 原核表达载体
Lucigen
原核表达 菌株
Stratagene 原核表达菌株
Qiagen
原核表达 载体
Qiagen
原核表达 载体
Invitrogen 基因载体
700 元 现货 800 元 现货
900 元 现货
800 元 现货 800 元 现货 800 元 现货 800 元 现货 800 元 现货 700 元 现货 800 元 现货 800 元 现货 800 元 现货 800 元 现货 900 元 现货 1000 元 现货 1000 元 现货 1000 元 现货 900 元 现货 1000 元 现货 1500 元 现货 600 元 现货 300 元 现货 300 元 现货 600 元 现货 600 元 现货 800 元 现货 900 元 现货 900 元 现货 900 元 现货
BL21(DE3) BL21(DE3)plysS Rosetta(DE3) Rosetta(DE3)plysS Rosetta-gami(DE3) Rosetta-gami B(DE3)pLysS Orgami(DE3) OrgamiB(DE3) BLR(DE3) pTWIN1 pTYB11 pMXB10 pTXB1 ER2566 pGEX-6P-1 pGEX-4T-2 pGEX-4T-3 pRset-a C41(DE3) BL21 codonplus(DE3)RIPL TAGZyme pQE-2 pQE-Trisystem pOG44 pORF-lacZ pORF-HSV1tk pSP73 pUC18 pUC19 pBR322 pSP64 pBluescript II SK(+) SuperCos 1 pGAPZα A pPICZα A
pTT5哺乳动物表达载体说明

pTT5编号 载体名称北京华越洋生物VECT6098 pTT5pTT5载体基本信息载体名称: pTT5质粒类型: 哺乳动物细胞载体高拷贝/低拷贝: 高拷贝克隆方法: 限制性内切酶,多克隆位点启动子: CMV载体大小: 4401 bp5' 测序引物及序列: CMV-F:CGCAAATGGGCGGTAGGCGTG 3' 测序引物及序列: --载体标签: 无载体抗性: 氨苄青霉素筛选标记: --克隆菌株: DH5α等宿主细胞(系): 常规细胞系(293、CV-1、CHO等)备注: 利用该质粒构建的基因表达是非融合表达,在构建质粒时需要在基因前面添加Kozak序列来增加蛋白表达效率。
稳定性: 瞬时表达组成型/诱导型: 诱导性病毒/非病毒: 非病毒pTT5载体质粒图谱和多克隆位点信息pTT5载体简介pTT5载体是很好的哺乳细胞瞬时蛋白表达载体,经常用于哺乳细胞的抗体轻链或者重链的表达。
pTT5载体序列ORIGIN1 GTACATTTAT ATTGGCTCAT GTCCAATATG ACCGCCATGT TGACATTGAT TATTGACTAG 61 TTATTAATAG TAATCAATTA CGGGGTCATT AGTTCATAGC CCATATATGG AGTTCCGCGT 121 TACATAACTT ACGGTAAATG GCCCGCCTGG CTGACCGCCC AACGACCCCC GCCCATTGAC 181 GTCAATAATG ACGTATGTTC CCATAGTAAC GCCAATAGGG ACTTTCCATT GACGTCAATG 241 GGTGGAGTAT TTACGGTAAA CTGCCCACTT GGCAGTACAT CAAGTGTATC ATATGCCAAG 301 TCCGCCCCCT ATTGACGTCA ATGACGGTAA ATGGCCCGCC TGGCATTATG CCCAGTACAT 361 GACCTTACGG GACTTTCCTA CTTGGCAGTA CATCTACGTA TTAGTCATCG CTATTACCAT 421 GGTGATGCGG TTTTGGCAGT ACACCAATGG GCGTGGATAG CGGTTTGACT CACGGGGATT 481 TCCAAGTCTC CACCCCATTG ACGTCAATGG GAGTTTGTTT TGGCACCAAA ATCAACGGGA 541 CTTTCCAAAA TGTCGTAATA ACCCCGCCCC GTTGACGCAA ATGGGCGGTA GGCGTGTACG 601 GTGGGAGGTC TATATAAGCA GAGCTCGTTT AGTGAACCGT CAGATCCTCA CTCTCTTCCG 661 CATCGCTGTC TGCGAGGGCC AGCTGTTGGG CTCGCGGTTG AGGACAAACT CTTCGCGGTC721 TTTCCAGTAC TCTTGGATCG GAAACCCGTC GGCCTCCGAA CGGTACTCCG CCACCGAGGG 781 ACCTGAGCGA GTCCGCATCG ACCGGATCGG AAAACCTCTC GAGAAAGGCG TCTAACCAGT 841 CACAGTCGCA AGGTAGGCTG AGCACCGTGG CGGGCGGCAG CGGGTGGCGG TCGGGGTTGT 901 TTCTGGCGGA GGTGCTGCTG ATGATGTAAT TAAAGTAGGC GGTCTTGAGA CGGCGGATGG 961 TCGAGGTGAG GTGTGGCAGG CTTGAGATCC AGCTGTTGGG GTGAGTACTC CCTCTCAAAA 1021 GCGGGCATTA CTTCTGCGCT AAGATTGTCA GTTTCCAAAA ACGAGGAGGA TTTGATATTC 1081 ACCTGGCCCG ATCTGGCCAT ACACTTGAGT GACAATGACA TCCACTTTGC CTTTCTCTCC 1141 ACAGGTGTCC ACTCCCAGGT CCAAGTTTAA ACGGATCTCT AGCGAATTCC CTCTAGAGGG 1201 CCCGTTTCTG CTAGCAAGCT TGCTAGCGGC CGCTCGAGGC CGGCAAGGCC GGATCCCCCG 1261 ACCTCGACCT CTGGCTAATA AAGGAAATTT ATTTTCATTG CAATAGTGTG TTGGAATTTT 1321 TTGTGTCTCT CACTCGGAAG GACATATGGG AGGGCAAATC ATTTGGTCGA GATCCCTCGG 1381 AGATCTCTAG CTAGAGGATC GATCCCCGCC CCGGACGAAC TAAACCTGAC TACGACATCT 1441 CTGCCCCTTC TTCGCGGGGC AGTGCATGTA ATCCCTTCAG TTGGTTGGTA CAACTTGCCA 1501 ACTGAACCCT AAACGGGTAG CATATGCTTC CCGGGTAGTA GTATATACTA TCCAGACTAA 1561 CCCTAATTCA ATAGCATATG TTACCCAACG GGAAGCATAT GCTATCGAAT TAGGGTTAGT 1621 AAAAGGGTCC TAAGGAACAG CGATGTAGGT GGGCGGGCCA AGATAGGGGC GCGATTGCTG 1681 CGATCTGGAG GACAAATTAC ACACACTTGC GCCTGAGCGC CAAGCACAGG GTTGTTGGTC 1741 CTCATATTCA CGAGGTCGCT GAGAGCACGG TGGGCTAATG TTGCCATGGG TAGCATATAC 1801 TACCCAAATA TCTGGATAGC ATATGCTATC CTAATCTATA TCTGGGTAGC ATAGGCTATC 1861 CTAATCTATA TCTGGGTAGC ATATGCTATC CTAATCTATA TCTGGGTAGT ATATGCTATC 1921 CTAATTTATA TCTGGGTAGC ATAGGCTATC CTAATCTATA TCTGGGTAGC ATATGCTATC 1981 CTAATCTATA TCTGGGTAGT ATATGCTATC CTAATCTGTA TCCGGGTAGC ATATGCTATC 2041 CTAATAGAGA TTAGGGTAGT ATATGCTATC CTAATTTATA TCTGGGTAGC ATATACTACC 2101 CAAATATCTG GATAGCATAT GCTATCCTAA TCTATATCTG GGTAGCATAT GCTATCCTAA 2161 TCTATATCTG GGTAGCATAG GCTATCCTAA TCTATATCTG GGTAGCATAT GCTATCCTAA 2221 TCTATATCTG GGTAGTATAT GCTATCCTAA TTTATATCTG GGTAGCATAG GCTATCCTAA 2281 TCTATATCTG GGTAGCATAT GCTATCCTAA TCTATATCTG GGTAGTATAT GCTATCCTAA 2341 TCTGTATCCG GGTAGCATAT GCTATCCTCA TGATAAGCTG TCAAACATGA GAATTAATTC 2401 TTGAAGACGA AAGGGCCTCG TGATACGCCT ATTTTTATAG GTTAATGTCA TGATAATAAT 2461 GGTTTCTTAG ACGTCAGGTG GCACTTTTCG GGGAAATGTG CGCGGAACCC CTATTTGTTT 2521 ATTTTTCTAA ATACATTCAA ATATGTATCC GCTCATGAGA CAATAACCCT GATAAATGCT 2581 TCAATAATAT TGAAAAAGGA AGAGTATGAG TATTCAACAT TTCCGTGTCG CCCTTATTCC 2641 CTTTTTTGCG GCATTTTGCC TTCCTGTTTT TGCTCACCCA GAAACGCTGG TGAAAGTAAA 2701 AGATGCTGAA GATCAGTTGG GTGCACGAGT GGGTTACATC GAACTGGATC TCAACAGCGG 2761 TAAGATCCTT GAGAGTTTTC GCCCCGAAGA ACGTTTTCCA ATGATGAGCA CTTTTAAAGT 2821 TCTGCTATGT GGCGCGGTAT TATCCCGTGT TGACGCCGGG CAAGAGCAAC TCGGTCGCCG 2881 CATACACTAT TCTCAGAATG ACTTGGTTGA GTACTCACCA GTCACAGAAA AGCATCTTAC 2941 GGATGGCATG ACAGTAAGAG AATTATGCAG TGCTGCCATA ACCATGAGTG ATAACACTGC 3001 GGCCAACTTA CTTCTGACAA CGATCGGAGG ACCGAAGGAG CTAACCGCTT TTTTGCACAA 3061 CATGGGGGAT CATGTAACTC GCCTTGATCG TTGGGAACCG GAGCTGAATG AAGCCATACC 3121 AAACGACGAG CGTGACACCA CGATGCCTGC AGCAATGGCA ACAACGTTGC GCAAACTATT 3181 AACTGGCGAA CTACTTACTC TAGCTTCCCG GCAACAATTA ATAGACTGGA TGGAGGCGGA 3241 TAAAGTTGCA GGACCACTTC TGCGCTCGGC CCTTCCGGCT GGCTGGTTTA TTGCTGATAA 3301 ATCTGGAGCC GGTGAGCGTG GGTCTCGCGG TATCATTGCA GCACTGGGGC CAGATGGTAA3361 GCCCTCCCGT ATCGTAGTTA TCTACACGAC GGGGAGTCAG GCAACTATGG ATGAACGAAA3421 TAGACAGATC GCTGAGATAG GTGCCTCACT GATTAAGCAT TGGTAACTGT CAGACCAAGT3481 TTACTCATAT ATACTTTAGA TTGATTTAAA ACTTCATTTT TAATTTAAAA GGATCTAGGT3541 GAAGATCCTT TTTGATAATC TCATGACCAA AATCCCTTAA CGTGAGTTTT CGTTCCACTG3601 AGCGTCAGAC CCCGTAGAAA AGATCAAAGG ATCTTCTTGA GATCCTTTTT TTCTGCGCGT3661 AATCTGCTGC TTGCAAACAA AAAAACCACC GCTACCAGCG GTGGTTTGTT TGCCGGATCA3721 AGAGCTACCA ACTCTTTTTC CGAAGGTAAC TGGCTTCAGC AGAGCGCAGA TACCAAATAC3781 TGTTCTTCTA GTGTAGCCGT AGTTAGGCCA CCACTTCAAG AACTCTGTAG CACCGCCTAC3841 ATACCTCGCT CTGCTAATCC TGTTACCAGT GGCTGCTGCC AGTGGCGATA AGTCGTGTCT3901 TACCGGGTTG GACTCAAGAC GATAGTTACC GGATAAGGCG CAGCGGTCGG GCTGAACGGG3961 GGGTTCGTGC ACACAGCCCA GCTTGGAGCG AACGACCTAC ACCGAACTGA GATACCTACA4021 GCGTGAGCTA TGAGAAAGCG CCACGCTTCC CGAAGGGAGA AAGGCGGACA GGTATCCGGT4081 AAGCGGCAGG GTCGGAACAG GAGAGCGCAC GAGGGAGCTT CCAGGGGGAA ACGCCTGGTA4141 TCTTTATAGT CCTGTCGGGT TTCGCCACCT CTGACTTGAG CGTCGATTTT TGTGATGCTC4201 GTCAGGGGGG CGGAGCCTAT GGAAAAACGC CAGCAACGCG GCCTTTTTAC GGTTCCTGGC4261 CTTTTGCTGG CCTTTTGCTC ACATGTTCTT TCCTGCGTTA TCCCCTGATT CTGTGGATAA4321 CCGTATTACC GCCTTTGAGT GAGCTGATAC CGCTCGCCGC AGCCGAACGA CCGAGCGCAG4381 CGAGTCAGTG AGCGAGGAAG C//其他哺乳动物表达载体:pCHO1.0 pBApo-CMV-Pur pOPRSVIpcDNA3.1/His C pcDNA5/FRT/V5-His-TOPO pREP4pcDNA3.1/His B pcDNA5/FRT/TO-TOPO pDual-GCpcDNA3.1/His A pcDNA5/TO pBK-RSVpIRESpuro3 pcDNA5/FRT/TO pBK-CMVpIRES2-EGFP pcDNA5/FRT pBI-CMV4pTT5 pFLAG-CMV2 pcDNA4/TO/Myc-His/LacZ pNFkB-DD-tdTomato pcDNA3.1/CT-GFP-TOPO pOPI3CATpBI-CMV5 pcDNA3.1/NT-GFP-TOPO pGene/V5-His B pSEAP2-Basic pOptiVEC-TOPO pSwitchpSEAP2-Control pCMV-MEKK1 pCMVLacIpBI-CMV3 pCMV-MEK1 pVgRxRpBI-CMV2 pCMV-PKA pINDpBI-CMV1 pcDNA6.2/nTC-Tag-DEST pTRE3G-LucpNFκB-MetLuc2-Reporter pcDNA6.2/cTC-Tag-DEST pTRE3GpCRE-MetLuc2-Reporter pcDNA3.2/V5/GW/D-TOPO pTRE2-hygropAcGFP1-Actin pcDNA6.2/V5/GW/D-TOPO pTRE-TightpAcGFP1-N In-Fusion Ready pcDNA6.2/nGeneBLAzer-GW/D-TOPO pTK-hygpAcGFP1-C3 pcDNA6.2/C-YFP-DEST pTet-OnpAcGFP1-C pcDNA6.2/cGeneBLAzer-DEST pTet-OffpAcGFP1-p53 pcDNA6/V5-His A pTet on advanced pAcGFP1-Mito pcDNA6/V5-His B pRevTREpAcGFP1-Mem pcDNA6/V5-His C pRevTet-OnpAcGFP1-Lam pcDNA6/myc-His C pRevTet-Off pAcGFP1-Golgi pcDNA6/myc-His A pCMV-Tet3G pAcGFP1-F pcDNA6/myc-His B pTRE2pAcGFP1-Hyg-C1 pcDNA6.2/nGeneBLAzer-DEST pBD-NF-κB pAsRed2-N1 pcDNA4/HisMax-TOPO pCMV-AD ptdTomato-N1 pcDNA6.2/nLumio-DEST pCMV-BDpCMV-tdTomato pcDNA6.2/cLumio-DEST pBIND-Id Control pCRE-DD-tdTomato pcDNA4/myc-His C pBINDpCMV-DsRed-Express2 pcDNA4/HisMax C pG5 luciferasepEF1α-tdTomato pcDNA4/HisMax A pACT-MyoDpCRE-hrGFP c-Flag pcDNA3 pACTptdTomato-C1 pcDNA4/HisMax B pCMV-SPORT6 pAsRed2-C1 pcDNA4/myc-His B pGL4.13pGL3-Promoter pcDNA4/myc-His A pGL4.19pGL3 basic pcDNA4/His C pGL4.26pAcGFP1-C2 pcDNA4/His B pGL4.20pAcGFP1-C1 pcDNA4/His A pGL4.29pAcGFP1-N3 pcDNA6/TR pGL4.30pAcGFP1-N2 pcDNA4/TO/Myc-His A pGL4.27pAcGFP1-N1 pcDNA4/TO pGL4.75pAcGFP1-C In-Fusion Ready pcDNA4/TO/Myc-His B pGL4.10pCRE-DD-AmCyan1 pcDNA4/TO/Myc-His C pGRN145pNFkB-DD-AmCyan1 pcDNA3.3-TOPO pSecTag2 A pDsRed2-Bid pBudCE4.1 pEBVHis B pDsRED2-Mito pFLAG-CMV-4 pEBVHis ApDD-AmCyan1 Reporter pFLAG-CMV-3 pCMV-Tag 3C pAmCyan1-N1 pFLAG-CMV-2 pCMV-Tag 3A pAmCyan1-C1 pFLAG-CMV-5a pCMV-Tag 3BpEF1α-IRES-DsRed-Express2 p3XFLAG-CMV-9 pCMV-Tag 5CpEF1α-DsRed-Monomer-N1 p3xFLAG-CMV-10 pCMV-Tag 5A pDsRED-Monomer-N1 p3XFLAG-CMV-8 pCMV-Tag 4A pDsRed-Express-N1 p3XFLAG-CMV-7.1 pCMV-Tag 5Bp3XFLAG-CMV-7 pDsRed-Monomer-N In-Fusion Ready pCMV-Tag 4B pDsRed-Express-C1 p3XFLAG-CMV-13 pCMV-Tag 2C pIRES2-ZsGreen1 p3XFLAG-CMV-14 pCMV-Tag 2B pDsRed-Express2-C1 plRES2-ZsGreen1 pCMV-Tag 2A pDsRed-Express2-N1 pBApo-EF1α-pur pCMV-LacZpEF1α-DsRed-Express2 pBApo-EF1α-neo pCMV-MycpIRES2-DsRed-Express pBApo-CMV pEF1α-IRES-AcGFP1 pIRES2-DsRed-Express2 pBApo-CMV-neo pEF1α-IRES-ZsGreen1 pIRES-hrGFP-1a pIRES-EGFP pEF1α-AcGFP1-N1 pIRESneo2 pIRESneo3 pIRES2-DsRed2 pIRESneo pDsRed-Monomer pIRES2-AcGFP1 pIREShyg3 pIRES。
第十六次全国动物遗传育种学术讨论会系列学术报告会

脂肪型猪与瘦肉型猪不同发育时期骨骼肌的转录谱比较研究(莫德林中山大学)
利用高密度SNP芯片进行中国荷斯坦牛体型性状全基因组关联分析(吴晓平中国农业大学)
Transcriptomic comparison of duroc-erhualian F2pigs with extreme immune responses to Poly I:C(刘向东华中农业大学)
(分会场部分:细胞工程与转基因动物)
时间
报告人及报告名称
5月15日14:00-18:15
Foxo1参与卵巢卵泡颗粒细胞凋亡(刘红林南京农业大学)
猪胎盘发生的分子调控机制研究(余梅华中农业大学)
山羊精子介导基因转移(SMGT)机理研究(赵永聚西南大学)
奶山羊生殖细胞发育分化的研究(华进联西北农林科技大学)
第十六次全国动物遗传育种学术讨论会系列学术报告会(一)
(特邀报告及大会报告部分)
时间
报告名称及报告人
5月14日
上午10:00-12:00
下午15:50-17:50
Post-sequencing advances in cattle genomics
(Prof. James E Womack美国Texas A & M University)
Snat-a bovine SNP annotation tool for post-GWAS functional studies(蒋纪才中国农业大学)
5月16日08:00-12:15
肉牛基因组选择的初步探讨(李俊雅中国农业科学院北京畜牧兽医研究所)
PU.1 regulates adipogenesis(庞卫军西北农林科技大学动物科技学院)
常见的真核和原核表达系统的启动子(promoters)

常规表达用
mRNA
猿猴空泡病毒40来源的哺乳动物表达启动子
组成型
可能包含一个增强子
PGK1 (人/小鼠)
常规表达用
mRNA
磷酸甘油酸酯激酶基因来源的哺乳动物启动子
组成型
广泛表达,但可能因细胞类型而异。由于甲基化或脱乙酰作用,倾向于抵抗启动子下调。
Ubc
常规表达用
mRNA
人类泛素C基因来源的哺乳动物启动子
CaMV35S
常规表达用
mRNA
花椰菜花叶病毒的强植物启动子
组成型
在双子叶植物激活,在单子叶植物中稍微弱一些,在一些动物细胞中有活性。
Ubi
常规表达用
mRNA
玉米泛素基因的植物启动子
组成型
在植物中促进高表达
H1
小RNA表达
shRNA
来源于人聚合酶III RNA启动子
组成型
可能比U6启动子弱一点,在神经元细胞中表现更佳
U6
小RNA表达
shRNA
来源于人U6小核启动子
组成型
小鼠U6也使用,但效率略差。
常用的原核表达系统启动子
启动子
主要用途
描述
表达
备注
T7
体外转录/常规表达
T7噬菌体来源的启动子
组成型,需要T7 RNA聚合酶
当用于体外转录的时候,外转方向有可能是正向的也可能是反向的,取决于启动子相对于目的基因的方向
T7lac
araBAD
常规表达用
阿拉伯糖代谢操纵子的启动子
阿拉伯糖诱导
弱,常用与pBAD载体。适合于快速调控和低的本底表达
trp
高水平基因表达
色氨酸操纵子来源的启动子
双荧光报告实验步骤

双荧光报告实验步骤:荧光步骤实验报告荧光酶报告实验植物写物理实验报告的步骤实验报告模板篇一:双荧光报告系统报告基因Promega中文通讯第2期2002荧光素酶双荧光素酶报告基因测试∶结合萤火虫和海洋腔肠荧光素酶先进的共报告基因测试技术在用萤火虫荧光素酶定量基因表达时,通常采用第二个报告基因来减少实验的变化因素。
但传统的共报告基因(比如CAT,β-Gal,GUS)不够便利,因为各自的测试化学,处理要求,检测特点存在差异。
Promega提供一种先进的双报告基因技术,结合了萤火虫荧光素酶测试和海洋腔肠荧光素酶测试。
双荧光素酶报告基因测试系统,结合pRL载体系统,表达第二个报告基因海洋腔肠荧光素酶,在单管中进行双荧光素酶报告基因测试,快速,灵敏,简便。
系统还提供PLB裂解液,用来裂解在多孔板中培养的哺乳细胞,不需操作单个样品。
对于正在使用萤火虫荧光素酶报告基因载体的研究人员。
双荧光素酶报告基因测试系统将使他们立即体会到该系统的便利。
介绍双报告基因用于实验系统中作相关的或成比例的检测, 通常一个报告基因作为内对照, 使另一个报告基因的检测均一化。
检测基因表达时双报告基因通常用来瞬时转染培养细胞,带有实验报告基因的载体共转染带有不同的报告基因作为对照的第二个载体。
通常实验报告基因偶联到调控的启动子, 研究调控基因的结构和生理基础。
报告基因表达活力的相对改变与偶联调控启动子转录活力的改变相关,偶联到组成型启动子的第二个报告基因,提供转录活力的内对照, 使测试不被实验条件变化所干扰。
通过这种方法, 可减少内在的变化因素所削弱的实验准确性, 比如, 培养细胞的数目和活力的差别, 细胞转染和裂解的效率。
使用萤火虫荧光素酶,结合氯霉素乙酰转移酶(CAT), β-半乳糖苷酶(β-Gal), 或葡萄醛酸糖苷酶(GUS)的双报告基因,近几年已普遍使用。
但这些双报告基因组合削弱了荧光素酶操作的优势, 比如荧光素酶测试和定量可在几秒钟内进行, 但CAT, β-Gal和GUS测试法, 则在定量前需要长时间的保温。
真核细胞常见表达载体

真核细胞常见表达载体真核细胞, 表达载体1、pCMVp-NEO-BAN载体特点:该真核细胞表达载体分子量为6600碱基对,主要由CMVp启动子、兔β-球蛋白基因内含子、聚腺嘌呤、氨青霉素抗性基因和抗neo基因以及pBR322骨架构成,在大多数真核细胞内都能高水平稳定地表达外源目的基因。
更重要的是,由于该真核细胞表达载体中抗neo基因存在,转染细胞后,用G418筛选,可建立稳定的、高表达目的基因的细胞株。
插入外源基因的克隆位点包括Sal1、BamH1和EcoR1位点。
注意在此载体中有二个EcoR1位点存在。
2、pEGFP,增强型绦色荧光蛋白表达载体(Enhanced Fluorecent Protein Vector)特点:pEGFP表达载体中含有绿色荧光蛋白,在PCMV启动子驱动下,在真核细胞中高水平表达。
载体骨架中的SV40origin使该载体在任何表达SV40 T抗原的真核细胞内进行复制。
Neo抗性盒由SV40早期启动子、Tn5的neomycin/kanamycin抗性基因以及HSV—TK基因的聚腺嘌呤信号组成,能应用G418筛选稳定转染的真核细胞株。
此外,载体中的pUC origin 能保证该载体在大肠杆菌中的复制,而位于此表达盒上游的细菌启动子能驱动kanamycin抗性基因在大肠杆菌中的表达.用途: 该表达载体EGFP上游有Nde1、Eco47111和Age1克隆位点,将外源基因扦入这些位点,将合成外源基因和EGFP的融合基因。
借此可确定外源基因在细胞内的表达和/或组织中的定位.亦可用于检测克隆的启动子活性(取代CMV启动子,Acet1-Nhe1)。
3、pEGFT-Actin,增强型绿色荧光蛋白/人肌动蛋白表达载体特点:pEGFP—Actin表达载体中含有绿色荧光蛋白和人胞浆β-肌动蛋白基因,在PCMV启动子驱动下,在真核细胞中高水平表达.载体骨架中的SV40origin使该载体在任何表达SV40 T抗原的真核细胞内进行复制。
(完整版)所有质粒载体汇总,推荐文档

酿酒酵母表达载体pYES2,pYES2/NT,pYES2/CT,pYES3,pYES6, pYCplac22-GFP,酵母载体pAUR123,pRS303TEF,pRS304, pRS305,pRS306,pY13TEF,pY14TEF,pY15TEF,pY16TEF,酵母基因重组表达载体pUG6, pSH47,酵母单杂载体pHISi,pLacZi,pHIS2, pGAD424, 酵母双杂交系统:酿酒酵母Y187, 酿酒酵母AH109;质粒pGADT7,pGBKT7;对照质粒pGBKT7-53,pGBKT7-lam,pGADT7-T,PCL1,酿酒酵母菌株INVSc1,YM4271, AH109,Y187,Y190,毕赤酵母表达载体pPIC9K,pPIC9K-His,pPIC3.5K,pPICZalphaA,B,C,pPICZA,B,C,pGAPZαA,pAO815,pPIC9k-His,pHIL-S1,pPink hc,配套毕赤酵母Pichiapink,毕赤酵母宿主X33,KM71,KM71H,GS115,原核表达载体pQE30,31,32,40,60,61,62,等原核表达载体,包括pET系列,pET-GST,pGEX系列(含GST标签),pMAL系列pMAL-c2x,-c4x,-c4e,-c5x,-p5x,pBAD,pBADHis,pBADmycHis系列,pQE系列,pTrc99a,pTrcHis系列,pBV220,221,222,pTXB系列,pLLP-ompA,pIN-III-ompA (分泌型表达系列),pQBI63(原核表达带荧光)pET3a, pET 3d, pET 11a, pET 12a, pET 14b, pET 15b, pET 16b, pET 17b, pET 19b, pET 20b, pET 21a,b,d, pET 22b, pET 23a, pET 23b, pET 24a,b, pET 25b, pET 26b, pET 27b, pET 28a,b, pET 29a, pET 30a, pET 31b, pET 32a, pET 35b, pET 38b, pET 39b, pET 40b, pET 41a,b pET 42a, pET 43.1a,b pET 44a, pET 49b pET302,303 pET His,pET Dsb,pET GST,pET Trx pQE2, pQE9 pQE30,31,32, pQE 40 pQE70 pQE80L pQETirs system pRSET-A pRSET-B pRSET-C pGEX4T-1,-2,-3,5x-1,6p-1,6p-2,2tk,3c pBV220,221,222 pTrcHisA,B,C pBAD24,34,43 pBAD HisA,B,C pPinPoint-Xa1,Xa2,Xa3 pMALc2x, p2x pBV220 pGEM Ex1, pGEM7ZF(+), pTrc99A, pTwin1, pEZZ18 pkk232-8,pkk233-3,pACYC184,pBR322,pUC119 pTYB1,pTYB2,pTYB4,pTYB11 pBlueScript SK (+),pBlueScript SK(-)pLLP ompA, pINIIIompA, pMBP-P ,pMBP-C, 大肠杆菌冷激质粒: pColdI pColdII pColdIII pColdTF 原核共表达质粒:pACYCduet-1,pETduet-1,pCDFduet-1,pRSFduet-1 Takara公司大肠杆菌分子伴侣: pG-KJE8 pGro7 pKJE7 pGTf2 pTf16 大肠杆菌宿主细胞: DH5a JM101 JM103JM105 JM107 JM109 JM110 Top10 Top10F BL21(DE3)HB101 ER2529 E2566 C2566 MG1655 XL-10gold XL blue M15 JF1125 K802 SG1117 BL21(AI)BL21(DE3)plysS TG1 TB1 DH5a(pir)Tuner(DE3)Bl21 codonplusRIPL Novablue (DE3)Rosetta Rosetta(DE3)Rosetta(DE3)plys Rosetta-gami(DE3)Rosetta-gamiB(DE3), Rosetta-gamiB(DE3)plysS Orgami(DE3)OrgamiB(DE3)HMS174(DE3)植物表达/RNAi载体农杆菌pBI121,pBI121-GFP,pBI101,pBI221,pSN1301,pUN1301,pRTL2 , pRTL2-GFP , pRTL2-CFP, pRTL2-RFP , pRTL2-YFP,pCAMBIA 1300, 1301, 1302,1303,1304,1305, 1381Z,1391Z,2300, 2301,3300,3301,pCAMBIA super1300,pCAMBIAsuper1300-GFP,pPZP212,pPZP2121,pPZP212-GFP,pGDG,RNAi载体pART27,pHANNIBAL,pKANNIBAL, pFGC5941,pTCK303, pTRV1,pTRV2,T-DNA插入载体(随机突变体库)pSKI015,pSKI074,真菌ATMT载体pBIG2RHPH2-GUS-GFP,pBHt1枯草芽孢杆菌表达载体pWB980,pHT43,pHP13,pHP43,pBE2,pMUTIN4,pUB110,pE194,pMA5, pMK3,pMK4,pHT304,pHY300PLK,pBest502,pDG1363,pSG1154,pAX01, pSAS144,pDL,pDG148-stu,pDG641,pAL12,pUCX05-bgaB,pHT01,配套菌株BS 168,WB600,WB800,WB700,WB800N,1012,FZB42,1A747,广宿主质粒pVLT33RNAi基因沉默干扰敲除载体pSilencer1.0,pSilencer 2.1-U6 hygro, pSilencer 3.1-H1 hygro,pSilencer 3.1-H1 neo, pSilencer 4.1-CMV neo, pSilencer 4.1-CMV puro pMIR-REPORT Luciferase RNAi载体(oligoengine)pSuper-puro RNAi逆转录病毒载体(clontech): RNAi-Ready pSIREN-Retro Q, RNAi-ReadypSIREN-RetroQ-ZsGreen(Luciferase shRNA Annealed Oligonucleotide)RNAi慢病毒载体(addgene): pLKO.1哺乳动物表达载体pcDNA3.1+/-,pcDNA4/HisMax B,pSecTag2 A,pVAX1,pBudCE4.1,pTracer CMV2,pcDNA3.1(-)/myc-His A ,pcDNA6-Myc/His B,pCEP4, pIRES,pIRESneo,pIRES hyg3,pCMV-myc,pCMV-HA,pIRES-puro3,pIRES-neo3,pCAGGS哺乳动物双杂交系统pACT,pBIND,pACT-MyoD,pBIND-Id,pG5luc,pCMV-BD, pCMV-AD, pBD-p53, pFR-luc,Cytotrap Two-Hybrid System:pSos, pSos MAFB, pMyr蜕皮激素诱导系统pIND, pVgRxR,LacSwith II哺乳动物诱导表达系统:pOPRSVI ,pOPI3CAT,pCMVLacI,GeneSwitch System:pSwitch哺乳动物表面展示系统:pDisplay, 四环素调控系统(Invitrogen):pcDNA4/TO/Myc-His A,pcDNA4/TO/Myc-His B,pcDNA4/TO/Myc-His C,pcDNA4/TO/Myc-His/LacZ,pcDNA6/TR四环素调控系统(Clontech):pTet-On,pTet-Off,pTRE2,pRevTRE,pRevTet-On,pRevTet-off信号通路报告载体:pGAS-TA-Luc,pSTAT3-TA-Luc, pISRE-TA-Luc, pTA-Luc,pIκB-EGFP,pNFAT-TA-Luc,pCaspase3-sensor,pAP1(PMA)-Luc;pGL4.26[luc2P/minP/Hygro],pGL4.29[luc2P/CRE/Hygro],pGL4.30[luc2P/NFA T-RE/Hygro],pGL4.75;p53-Luc,pAP-1-Luc, pNF-κB-Luc,pSRE-Luc,pFA2-Elk1,pFC-MEKK,pFR-luc,Gateway系统(invitrogen)pcDNA6.2-GWEmGFP-miR negative, pLenti 6/TR,pcDNA 6.2-GW EmGFP-miR,乳酸菌表达载体及各种乳酸菌乳酸杆菌菌株,pNZ8148,pLEISS,pMG36e,pBBR1MCS-5,pBBR1MCS-6,pRV610,pLEM415,pHY3 00PLK,分泌型乳酸菌表达载体pVE5523,pPG611.1,pPG612.1等和乳酸杆菌菌株宿主菌NZ9000,MG1363,Lactobacillus casei 1.539,Lactobacillus casei,acidophilus NCFM,1.2,Lactobacillus sakei 23K,L.plantarum,L.rhamnosusGG,B.coagulans,Bifidobacterium bifidum,Bifidobacterium infantis,Lactococcus lactis M17,1663,Lactobacillus reuterii广宿主表达载体链球菌表达敲除载体假单胞菌表达载体pVLT33,pBBR1MCS-2,3,4,5,6, pJRD215,pJN105,pME6032,Cos载体pLAFR3,pMP2444(GFP), pHY300PLK,pRT102,pRL1063a, 转座子载体pUT-miniTn5,pMGS100, pWHM10,pKC1139,pSET152,pOJ260,pPG611.1,pPG612.1,腺病毒载体/慢病毒,逆转录病毒表达载体及包装包膜质粒,腺病毒系统(Stratagene): pAdEasy-1,pShuttle-CMV,pShuttle,pAdTrack, pAdTrack-CMV, pShuttle-IRES-hrGFP-1、pShuttle-IRES-hrGFP-2、pShuttle-CMV-lacZ,pShuttle-CMV-EGFP-C,pXC1, pBHGE3, 配套大肠杆菌BJ5183,293,293T cellline 腺相关病毒系统(Stratagene): pAAV-MCS,pAAV-RC,pHelper,pAAV-LacZ,pAAV-IRES-hrGFP,pCMV-MCS,慢病毒载体:pLVX-DsRed-Monomer-N1,pLVX-IRES-ZsGreen1,pLVX-AcGFP1-N1,Lenti6/v 5-EDST-EGFP,pWPXL, FUGW,pLentilox 3.7,RNAi-Ready pSIREN-Retro Q,RNAi-Ready pSIREN-Retro Q-ZsGreen,pSUPER.Retro-GFP/Neo,pSUPER-Retro-Neo, pSUPER.Retro-puro,PLNCX PLNCX2 pMSCV-HYG pMSCV-neo pMSCV-puro pLEGFP-C1 pLOX-CW-CRE pLOX-GFP-IRES-TK pRetroX-IRES-DsRedExpress, pLVX-IRES-mCherry质粒载体。
在CoCl2模拟低氧条件下HIF-1α直接调控PPARγ2的表达

在CoCl2模拟低氧条件下HIF-1α直接调控PPARγ2的表达刘晓玲;包韩乌云;赵华路;张俊武【摘要】目的探讨在CoCl2模拟低氧条件下,低氧诱导因子1 α(HIF-1α)对于过氧化物酶体增殖物激活受体γ2(PPARγ2)是否具有调控作用.方法用real time PCR 和Western blot检测CoCl2模拟低氧条件下PPARγ2和低氧诱导因子1α(HIF-1α) mRNA及蛋白表达.用双荧光报告系统检测HIF-1α对于PPARγ调控的剂量依赖性,并通过染色质免疫共沉淀技术进一步验证.结果 CoCl2模拟低氧条件下PPARγ2和HIF-1α的mRNA及蛋白表达水平均随着诱导时间的增加而增加.过表达HIF-1α导致PPARγ2表达上调(P<0.01),而抑制HIF-1α则导致PPARγ2表达下调(P<0.05).HIF-1α对PPARγ2基因的表达具有正调控作用,通过结合于PPARγ2上游调控区特定的低氧应答元件(HRE)而调控其表达.结论PPARγ2通过HIF-1α依赖的途径在CoCl2模拟的低氧条件下发挥低氧适应作用.【期刊名称】《基础医学与临床》【年(卷),期】2015(035)005【总页数】5页(P585-589)【关键词】低氧;CoCl2;PPARγ2;HIF-1α;HRE【作者】刘晓玲;包韩乌云;赵华路;张俊武【作者单位】中国医学科学院基础医学研究所医学分子生物学国家重点实验室,北京100005;中国医学科学院基础医学研究所医学分子生物学国家重点实验室,北京100005;中国医学科学院基础医学研究所医学分子生物学国家重点实验室,北京100005;中国医学科学院基础医学研究所医学分子生物学国家重点实验室,北京100005【正文语种】中文【中图分类】R34过氧化物酶体增殖物激活受体γ(peroxisome proliferator-activated receptor γ,PPARγ)属于核受体超家族,具有4种不同的剪接体PPARγ1- 4,产生两种不同的蛋白。
鸡卵清蛋白启动子功能分析及验证

鸡卵清蛋白启动子功能分析及验证摘要转基因鸡输卵管生物反应器具有高产蛋能力,转基因鸡群快速扩繁能力和与人类相似的蛋白糖基化形式,在药用蛋白生产方面具有不可替代的作用。
到目前为止,相继建立了鸡蛋卵白(eggwhite)中表达干扰素、溶菌酶、抗体、促红细胞生成素、表皮生长因子等重组蛋白的转基因鸡。
但是对于不同长度卵清蛋白启动子(OVP)活性,以及雌激素应答元件(ERE)和卵清蛋白上游启动子转录因子(COUP-TF)调控作用缺乏系统研究;其次,作为外源基因转移的有效载体,鸡原始生殖细胞(PGCs)分离培养方法也待探讨。
本研究的目的是确定鸡OVP核心区域,筛选高转录活性的OVP; 探讨ERE和COUP-TF调控作用;摸索鸡PGCs适宜培养方法;通过体外和体内验证试验,确定可望用于制备输卵管生物反应器的OVP。
1. CRISPR-dCas9-VP64介导的鸡卵清蛋白启动子转录活性分析。
将启动子cERE-cOVP-2785、cOVP-2785分别亚克隆至pGL3-Basic载体,构建了荧光素酶表达载体pGL3-cERE-cOVP-2785、pGL3 -cOVP-2785。
利用Lipofectamine™ 2000将上述2个质粒以及pGL3cOVP-1548分别与pRL-TK质粒共转染293T细胞,检测荧光素酶活性,评估三个启动子的转录活性。
利用在线软件Optimized CRISPR Design,依据鸡卵清蛋白(OV A)基因第一内含子、转录起始位点上游区域DNA序列,共设计11对sgRNA;依据鸡ERE序列设计了2对sgRNA, 分别构建了11个pGL3-U6-OVPsgRNA 和2个pGL3-U6-EREsgRNA重组质粒;将pGL3-cERE-cOVP-2785、pcDNA-dCas9或pcDNA-dCas9-VP64质粒分别与单个或多个sgRNA表达质粒共转染293T细胞,检测荧光素酶活性,评估启动子和ERE对荧光素酶表达的影响。
5、转录调控

分子机制研究套路(五)转录调控课题:转录因子A对B基因的转录调控1.概念介绍:转录水平的调控是真核生物基因表达调控中重要环节。
真核细胞RNA 聚合酶自身对启动子并无特殊亲和力,单独不能进行转录,也就是说基因是无活性的。
因此,转录需要众多的转录因子和辅助转录因子形成复杂的转录装置。
在基因转录起始阶段,通用转录因子协助RNA 聚合酶与启动子结合,但其作用很弱,不能高效率地启动转录。
只有在反式作用因子(基因特异性转录因子)的协助下,RNA 聚合酶Ⅱ和TFⅡ才能有效地形成转录起始复合物。
反式作用因子(trans acting factor)在转录调节中具有特殊的重要性。
它是能直接或间接地识别或结合在顺式作用元件8~12bp 核心序列上,参与调控靶基因转录效率的一组蛋白质。
这类DNA 结合蛋白有多种,能特异性识别这类蛋白的序列也有多种,正是不同的DNA 结合蛋白与不同的识别序列之间的空间结构上的相互作用,以及蛋白质与蛋白质之间的相互作用构成了复杂的基因转录调控机制的基础。
在真核生物中转录因子的调控是最重要,也是研究得最多的。
蛋白质相互作用在转录因子活性的调控方面具有重要的意义。
细胞内的反式作用因子都是处于有活性和无活性两种状态,这两种状态是可以转换的。
反式作用因子处于无活性状态时,与之相应的基因就不能表达;反式作用因子处于有活性状态、并与相应的顺式作用元件结合时,就可以促进RNA 聚合酶和通用转录因子与相应的启动子结合,形成转录起始复合物。
所以,真核基因的表达调控主要是调节反式作用因子的活性,随后反式作用因子调控基因的转录起始。
转录因子被激活后,即可识别并结合上游启动子元件和增强子,对基因转录发挥调控作用。
大部分转录因子在激活以后与顺式作用元件结合,但也可能有一些转录因子是先结合DNA,被激活后才发挥调节功能。
增强子和上游启动子元件可以结合一些相同的蛋白质,在不同的基因中,存在着同样的顺式作用元件,这表明一些数量有限的基因调控蛋白控制着真核细胞的基因表达。
报告基因和荧光淬灭

Co of mRNA copy
3’end Of processed mRNA copy
Startpoint initiator codon terminator codon termination site For for protein for protein synthesis for transcription Transcription synthesis (UGA,UAA, or UAG) signal for site of 3’end trimming and poly (A) tail addition
1974年,他们得到了这个蛋白,当时称绿色蛋白、以后称绿色荧光蛋白(GFP)。GFP在水母中之所以能发光,是因为水母素和GFP之间发生了能量转移。水母素在钙刺激下发光,其能量可转移到GFP,刺激GFP发光。这是物理化学中已知的荧光共振能量转移(FRET)在生物中的发现。
直到1992年,道格拉斯•普瑞舍克隆并测序了野生型的GFP,文章发表在《Gene》杂志上。但具有讽刺意味的是,基金评审委员会认为普瑞舍的工作没有意义,不愿提供经费。普瑞舍一气之下,离开了科学界,将GFP的cDNA送给了几个实验室。
紫外灯
单击此处添加正文,文字是您思想的提炼,为了演示发布的良好效果,请言简意赅地阐述您的观点。您的内容已经简明扼要,字字珠玑,但信息却千丝万缕、错综复杂,需要用更多的文字来表述;但请您尽可能提炼思想的精髓,否则容易造成观者的阅读压力,适得其反。正如我们都希望改变世界,希望给别人带去光明,但更多时候我们只需要播下一颗种子,自然有微风吹拂,雨露滋养。恰如其分地表达观点,往往事半功倍。当您的内容到达这个限度时,或许已经不纯粹作用于演示,极大可能运用于阅读领域;无论是传播观点、知识分享还是汇报工作,内容的详尽固然重要,但请一定注意信息框架的清晰,这样才能使内容层次分明,页面简洁易读。如果您的内容确实非常重要又难以精简,也请使用分段处理,对内容进行简单的梳理和提炼,这样会使逻辑框架相对清晰。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
pGL3-Promoter编号 载体名称北京华越洋生物VECT6010 pGL3-‐PromoterpGL3-‐Promoter载体基本信息载体名称: pGL3-promoter, pGL3promoter质粒类型: 荧光素酶报告系统载体高拷贝/低拷贝: 高拷贝启动子: SV40克隆方法: 多克隆位点,限制性内切酶载体大小: 5010bp5' 测序引物及序列: RV primer3:CTAGCAAAATAGGCTGTCCC3' 测序引物及序列: GLprimer2: CTTTATGTTTTTGGCGTCTTCCA 载体标签: --载体抗性: 氨苄筛选标记: --备注: 用于快速定量评估影响哺乳动物细胞特定基因表达的因子及其影响能力。
稳定性: 稳定组成型: 非组成型病毒/非病毒: 非病毒pGL3-‐Promoter载体质粒图谱和多克隆位点信息pGL3-‐Promoter载体序列ORIGIN1 GGTACCGAGC TCTTACGCGT GCTAGCCCGG GCTCGAGATC TGCGATCTGC ATCTCAATTA 61 GTCAGCAACC ATAGTCCCGC CCCTAACTCC GCCCATCCCG CCCCTAACTC CGCCCAGTTC 121 CGCCCATTCT CCGCCCCATC GCTGACTAAT TTTTTTTATT TATGCAGAGG CCGAGGCCGC 181 CTCGGCCTCT GAGCTATTCC AGAAGTAGTG AGGAGGCTTT TTTGGAGGCC TAGGCTTTTG241 CAAAAAGCTT GGCATTCCGG TACTGTTGGT AAAGCCACCA TGGAAGACGC CAAAAACATA 301 AAGAAAGGCC CGGCGCCATT CTATCCGCTG GAAGATGGAA CCGCTGGAGA GCAACTGCAT 361 AAGGCTATGA AGAGATACGC CCTGGTTCCT GGAACAATTG CTTTTACAGA TGCACATATC 421 GAGGTGGACA TCACTTACGC TGAGTACTTC GAAATGTCCG TTCGGTTGGC AGAAGCTATG 481 AAACGATATG GGCTGAATAC AAATCACAGA ATCGTCGTAT GCAGTGAAAA CTCTCTTCAA 541 TTCTTTATGC CGGTGTTGGG CGCGTTATTT ATCGGAGTTG CAGTTGCGCC CGCGAACGAC 601 ATTTATAATG AACGTGAATT GCTCAACAGT ATGGGCATTT CGCAGCCTAC CGTGGTGTTC 661 GTTTCCAAAA AGGGGTTGCA AAAAATTTTG AACGTGCAAA AAAAGCTCCC AATCATCCAA 721 AAAATTATTA TCATGGATTC TAAAACGGAT TACCAGGGAT TTCAGTCGAT GTACACGTTC 781 GTCACATCTC ATCTACCTCC CGGTTTTAAT GAATACGATT TTGTGCCAGA GTCCTTCGAT 841 AGGGACAAGA CAATTGCACT GATCATGAAC TCCTCTGGAT CTACTGGTCT GCCTAAAGGT 901 GTCGCTCTGC CTCATAGAAC TGCCTGCGTG AGATTCTCGC ATGCCAGAGA TCCTATTTTT 961 GGCAATCAAA TCATTCCGGA TACTGCGATT TTAAGTGTTG TTCCATTCCA TCACGGTTTT 1021 GGAATGTTTA CTACACTCGG ATATTTGATA TGTGGATTTC GAGTCGTCTT AATGTATAGA 1081 TTTGAAGAAG AGCTGTTTCT GAGGAGCCTT CAGGATTACA AGATTCAAAG TGCGCTGCTG 1141 GTGCCAACCC TATTCTCCTT CTTCGCCAAA AGCACTCTGA TTGACAAATA CGATTTATCT 1201 AATTTACACG AAATTGCTTC TGGTGGCGCT CCCCTCTCTA AGGAAGTCGG GGAAGCGGTT 1261 GCCAAGAGGT TCCATCTGCC AGGTATCAGG CAAGGATATG GGCTCACTGA GACTACATCA 1321 GCTATTCTGA TTACACCCGA GGGGGATGAT AAACCGGGCG CGGTCGGTAA AGTTGTTCCA 1381 TTTTTTGAAG CGAAGGTTGT GGATCTGGAT ACCGGGAAAA CGCTGGGCGT TAATCAAAGA 1441 GGCGAACTGT GTGTGAGAGG TCCTATGATT ATGTCCGGTT ATGTAAACAA TCCGGAAGCG 1501 ACCAACGCCT TGATTGACAA GGATGGATGG CTACATTCTG GAGACATAGC TTACTGGGAC 1561 GAAGACGAAC ACTTCTTCAT CGTTGACCGC CTGAAGTCTC TGATTAAGTA CAAAGGCTAT 1621 CAGGTGGCTC CCGCTGAATT GGAATCCATC TTGCTCCAAC ACCCCAACAT CTTCGACGCA 1681 GGTGTCGCAG GTCTTCCCGA CGATGACGCC GGTGAACTTC CCGCCGCCGT TGTTGTTTTG 1741 GAGCACGGAA AGACGATGAC GGAAAAAGAG ATCGTGGATT ACGTCGCCAG TCAAGTAACA 1801 ACCGCGAAAA AGTTGCGCGG AGGAGTTGTG TTTGTGGACG AAGTACCGAA AGGTCTTACC 1861 GGAAAACTCG ACGCAAGAAA AATCAGAGAG ATCCTCATAA AGGCCAAGAA GGGCGGAAAG 1921 ATCGCCGTGT AATTCTAGAG TCGGGGCGGC CGGCCGCTTC GAGCAGACAT GATAAGATAC 1981 ATTGATGAGT TTGGACAAAC CACAACTAGA ATGCAGTGAA AAAAATGCTT TATTTGTGAA 2041 ATTTGTGATG CTATTGCTTT ATTTGTAACC ATTATAAGCT GCAATAAACA AGTTAACAAC 2101 AACAATTGCA TTCATTTTAT GTTTCAGGTT CAGGGGGAGG TGTGGGAGGT TTTTTAAAGC 2161 AAGTAAAACC TCTACAAATG TGGTAAAATC GATAAGGATC CGTCGACCGA TGCCCTTGAG 2221 AGCCTTCAAC CCAGTCAGCT CCTTCCGGTG GGCGCGGGGC ATGACTATCG TCGCCGCACT 2281 TATGACTGTC TTCTTTATCA TGCAACTCGT AGGACAGGTG CCGGCAGCGC TCTTCCGCTT 2341 CCTCGCTCAC TGACTCGCTG CGCTCGGTCG TTCGGCTGCG GCGAGCGGTA TCAGCTCACT 2401 CAAAGGCGGT AATACGGTTA TCCACAGAAT CAGGGGATAA CGCAGGAAAG AACATGTGAG 2461 CAAAAGGCCA GCAAAAGGCC AGGAACCGTA AAAAGGCCGC GTTGCTGGCG TTTTTCCATA 2521 GGCTCCGCCC CCCTGACGAG CATCACAAAA ATCGACGCTC AAGTCAGAGG TGGCGAAACC 2581 CGACAGGACT ATAAAGATAC CAGGCGTTTC CCCCTGGAAG CTCCCTCGTG CGCTCTCCTG 2641 TTCCGACCCT GCCGCTTACC GGATACCTGT CCGCCTTTCT CCCTTCGGGA AGCGTGGCGC 2701 TTTCTCATAG CTCACGCTGT AGGTATCTCA GTTCGGTGTA GGTCGTTCGC TCCAAGCTGG 2761 GCTGTGTGCA CGAACCCCCC GTTCAGCCCG ACCGCTGCGC CTTATCCGGT AACTATCGTC 2821 TTGAGTCCAA CCCGGTAAGA CACGACTTAT CGCCACTGGC AGCAGCCACT GGTAACAGGA2881 TTAGCAGAGC GAGGTATGTA GGCGGTGCTA CAGAGTTCTT GAAGTGGTGG CCTAACTACG 2941 GCTACACTAG AAGAACAGTA TTTGGTATCT GCGCTCTGCT GAAGCCAGTT ACCTTCGGAA 3001 AAAGAGTTGG TAGCTCTTGA TCCGGCAAAC AAACCACCGC TGGTAGCGGT GGTTTTTTTG 3061 TTTGCAAGCA GCAGATTACG CGCAGAAAAA AAGGATCTCA AGAAGATCCT TTGATCTTTT 3121 CTACGGGGTC TGACGCTCAG TGGAACGAAA ACTCACGTTA AGGGATTTTG GTCATGAGAT 3181 TATCAAAAAG GATCTTCACC TAGATCCTTT TAAATTAAAA ATGAAGTTTT AAATCAATCT 3241 AAAGTATATA TGAGTAAACT TGGTCTGACA GTTACCAATG CTTAATCAGT GAGGCACCTA 3301 TCTCAGCGAT CTGTCTATTT CGTTCATCCA TAGTTGCCTG ACTCCCCGTC GTGTAGATAA 3361 CTACGATACG GGAGGGCTTA CCATCTGGCC CCAGTGCTGC AATGATACCG CGAGACCCAC 3421 GCTCACCGGC TCCAGATTTA TCAGCAATAA ACCAGCCAGC CGGAAGGGCC GAGCGCAGAA 3481 GTGGTCCTGC AACTTTATCC GCCTCCATCC AGTCTATTAA TTGTTGCCGG GAAGCTAGAG 3541 TAAGTAGTTC GCCAGTTAAT AGTTTGCGCA ACGTTGTTGC CATTGCTACA GGCATCGTGG 3601 TGTCACGCTC GTCGTTTGGT ATGGCTTCAT TCAGCTCCGG TTCCCAACGA TCAAGGCGAG 3661 TTACATGATC CCCCATGTTG TGCAAAAAAG CGGTTAGCTC CTTCGGTCCT CCGATCGTTG 3721 TCAGAAGTAA GTTGGCCGCA GTGTTATCAC TCATGGTTAT GGCAGCACTG CATAATTCTC 3781 TTACTGTCAT GCCATCCGTA AGATGCTTTT CTGTGACTGG TGAGTACTCA ACCAAGTCAT 3841 TCTGAGAATA GTGTATGCGG CGACCGAGTT GCTCTTGCCC GGCGTCAATA CGGGATAATA 3901 CCGCGCCACA TAGCAGAACT TTAAAAGTGC TCATCATTGG AAAACGTTCT TCGGGGCGAA 3961 AACTCTCAAG GATCTTACCG CTGTTGAGAT CCAGTTCGAT GTAACCCACT CGTGCACCCA 4021 ACTGATCTTC AGCATCTTTT ACTTTCACCA GCGTTTCTGG GTGAGCAAAA ACAGGAAGGC 4081 AAAATGCCGC AAAAAAGGGA ATAAGGGCGA CACGGAAATG TTGAATACTC ATACTCTTCC 4141 TTTTTCAATA TTATTGAAGC ATTTATCAGG GTTATTGTCT CATGAGCGGA TACATATTTG 4201 AATGTATTTA GAAAAATAAA CAAATAGGGG TTCCGCGCAC ATTTCCCCGA AAAGTGCCAC 4261 CTGACGCGCC CTGTAGCGGC GCATTAAGCG CGGCGGGTGT GGTGGTTACG CGCAGCGTGA 4321 CCGCTACACT TGCCAGCGCC CTAGCGCCCG CTCCTTTCGC TTTCTTCCCT TCCTTTCTCG 4381 CCACGTTCGC CGGCTTTCCC CGTCAAGCTC TAAATCGGGG GCTCCCTTTA GGGTTCCGAT 4441 TTAGTGCTTT ACGGCACCTC GACCCCAAAA AACTTGATTA GGGTGATGGT TCACGTAGTG 4501 GGCCATCGCC CTGATAGACG GTTTTTCGCC CTTTGACGTT GGAGTCCACG TTCTTTAATA 4561 GTGGACTCTT GTTCCAAACT GGAACAACAC TCAACCCTAT CTCGGTCTAT TCTTTTGATT 4621 TATAAGGGAT TTTGCCGATT TCGGCCTATT GGTTAAAAAA TGAGCTGATT TAACAAAAAT 4681 TTAACGCGAA TTTTAACAAA ATATTAACGC TTACAATTTG CCATTCGCCA TTCAGGCTGC 4741 GCAACTGTTG GGAAGGGCGA TCGGTGCGGG CCTCTTCGCT ATTACGCCAG CCCAAGCTAC 4801 CATGATAAGT AAGTAATATT AAGGTACGGG AGGTACTTGG AGCGGCCGCA ATAAAATATC 4861 TTTATTTTCA TTACATCTGT GTGTTGGTTT TTTGTGTGAA TCGATAGTAC TAACATACGC 4921 TCTCCATCAA AACAAAACGA AACAAAACAA ACTAGCAAAA TAGGCTGTCC CCAGTGCAAG 4981 TGCAGGTGCC AGAACATTTC TCTATCGATA//其他哺乳动物表达载体:pCHO1.0 pBApo-CMV-Pur pOPRSVIpcDNA3.1/His C pcDNA5/FRT/V5-His-TOPO pREP4pcDNA3.1/His B pcDNA5/FRT/TO-TOPO pDual-GCpcDNA3.1/His A pcDNA5/TO pBK-RSV pIRESpuro3 pcDNA5/FRT/TO pBK-CMVpIRES2-EGFP pcDNA5/FRT pBI-CMV4pTT5 pFLAG-CMV2 pcDNA4/TO/Myc-His/LacZ pNFkB-DD-tdTomato pcDNA3.1/CT-GFP-TOPO pOPI3CATpBI-CMV5 pcDNA3.1/NT-GFP-TOPO pGene/V5-His B pSEAP2-Basic pOptiVEC-TOPO pSwitchpSEAP2-Control pCMV-MEKK1 pCMVLacIpBI-CMV3 pCMV-MEK1 pVgRxRpBI-CMV2 pCMV-PKA pINDpBI-CMV1 pcDNA6.2/nTC-Tag-DEST pTRE3G-LucpNFκB-MetLuc2-Reporter pcDNA6.2/cTC-Tag-DEST pTRE3GpCRE-MetLuc2-Reporter pcDNA3.2/V5/GW/D-TOPO pTRE2-hygropAcGFP1-Actin pcDNA6.2/V5/GW/D-TOPO pTRE-TightpAcGFP1-N In-Fusion Ready pcDNA6.2/nGeneBLAzer-GW/D-TOPO pTK-hygpAcGFP1-C3 pcDNA6.2/C-YFP-DEST pTet-OnpAcGFP1-C pcDNA6.2/cGeneBLAzer-DEST pTet-OffpAcGFP1-p53 pcDNA6/V5-His A pTet on advanced pAcGFP1-Mito pcDNA6/V5-His B pRevTREpAcGFP1-Mem pcDNA6/V5-His C pRevTet-OnpAcGFP1-Lam pcDNA6/myc-His C pRevTet-OffpAcGFP1-Golgi pcDNA6/myc-His A pCMV-Tet3GpAcGFP1-F pcDNA6/myc-His B pTRE2pAcGFP1-Hyg-C1 pcDNA6.2/nGeneBLAzer-DEST pBD-NF-κBpAsRed2-N1 pcDNA4/HisMax-TOPO pCMV-ADptdTomato-N1 pcDNA6.2/nLumio-DEST pCMV-BDpCMV-tdTomato pcDNA6.2/cLumio-DEST pBIND-Id ControlpCRE-DD-tdTomato pcDNA4/myc-His C pBINDpCMV-DsRed-Express2 pcDNA4/HisMax C pG5 luciferasepEF1α-tdTomato pcDNA4/HisMax A pACT-MyoDpCRE-hrGFP c-Flag pcDNA3 pACTptdTomato-C1 pcDNA4/HisMax B pCMV-SPORT6 pAsRed2-C1 pcDNA4/myc-His B pGL4.13pGL3-Promoter pcDNA4/myc-His A pGL4.19pGL3 basic pcDNA4/His C pGL4.26pAcGFP1-C2 pcDNA4/His B pGL4.20pAcGFP1-C1 pcDNA4/His A pGL4.29pAcGFP1-N3 pcDNA6/TR pGL4.30pAcGFP1-N2 pcDNA4/TO/Myc-His A pGL4.27pAcGFP1-N1 pcDNA4/TO pGL4.75pAcGFP1-C In-Fusion Ready pcDNA4/TO/Myc-His B pGL4.10pCRE-DD-AmCyan1 pcDNA4/TO/Myc-His C pGRN145pNFkB-DD-AmCyan1 pcDNA3.3-TOPO pSecTag2 ApDsRed2-Bid pBudCE4.1 pEBVHis BpDsRED2-Mito pFLAG-CMV-4 pEBVHis ApDD-AmCyan1 Reporter pFLAG-CMV-3 pCMV-Tag 3CpAmCyan1-N1 pFLAG-CMV-2 pCMV-Tag 3A pAmCyan1-C1 pFLAG-CMV-5a pCMV-Tag 3BpEF1α-IRES-DsRed-Express2 p3XFLAG-CMV-9 pCMV-Tag 5CpEF1α-DsRed-Monomer-N1 p3xFLAG-CMV-10 pCMV-Tag 5A pDsRED-Monomer-N1 p3XFLAG-CMV-8 pCMV-Tag 4A pDsRed-Express-N1 p3XFLAG-CMV-7.1 pCMV-Tag 5Bp3XFLAG-CMV-7 pDsRed-Monomer-N In-Fusion Ready pCMV-Tag 4B pDsRed-Express-C1 p3XFLAG-CMV-13 pCMV-Tag 2C pIRES2-ZsGreen1 p3XFLAG-CMV-14 pCMV-Tag 2B pDsRed-Express2-C1 plRES2-ZsGreen1 pCMV-Tag 2A pDsRed-Express2-N1 pBApo-EF1α-pur pCMV-LacZpEF1α-DsRed-Express2 pBApo-EF1α-neo pCMV-MycpIRES2-DsRed-Express pBApo-CMV pEF1α-IRES-AcGFP1 pIRES2-DsRed-Express2 pBApo-CMV-neo pEF1α-IRES-ZsGreen1 pIRES-hrGFP-1a pIRES-EGFP pEF1α-AcGFP1-N1 pIRESneo2 pIRESneo3 pIRES2-DsRed2 pIRESneo pDsRed-Monomer pIRES2-AcGFP1 pIREShyg3 pIRES。
