生物信息学作业
生物信息学作业(一)

生物信息学实验作业一1、了解NCBI、DDBJ、EMBL上网的方法自学各网站相关介绍。
答:(1)、NCBI: (National Center of Biotechnology Information,简称NCBI)美国国立生物技术信息中心。
其主页为:。
NCBI 是在NIH的国立医学图书馆(NLM)的一个分支。
NLM是因为它在创立和维护生物信息学数据库方面的经验被选择的,而且这可以建立一个内部的关于计算分子生物学的研究计划。
NCBI的任务是发展新的信息学技术来帮助对那些控制健康和疾病的基本分子和遗传过程的理解。
NCBI有一个多学科的研究小组包括计算机科学家,分子生物学家,数学家,生物化学家,实验物理学家,和结构生物学家,集中于计算分子生物学的基本的和应用的研究。
他们一起用数学和计算的方法研究在分子水平上的基本的生物医学问题。
这些问题包括基因的组织,序列的分析,和结构的预测。
在1992年10月,NCBI承担起对GenBank DNA序列数据库的责任。
NCBI 受过分子生物学高级训练的工作人员通过来自各个实验室递交的序列和同国际核酸序列数据库(EMBL和DDBJ)交换数据建立起数据库。
同美国专利和商标局的安排使得专利的序列信息也被整合。
BLAST是一个NCBI开发的序列相似搜索程序,还可作为鉴别基因和遗传特点的手段。
BLAST能够在小于15秒的时间内对整个DNA数据库执行序列搜索。
NCBI提供的附加的软件工具有:开放阅读框寻觅器(ORF Finder),电子PCR,和序列提交工具,Sequin和BankIt。
所有的NCBI数据库和软件工具可以从WWW 或FTP来获得。
NCBI还有E-mail服务器,提供用文本搜索或序列相似搜索访问数据库一种可选方法。
主要任务:(1)建立关于分子生物学,生物化学,和遗传学知识的存储和分析的自动系统(2)实行关于用于分析生物学重要分子和复合物的结构和功能的基于计算机的信息处理的,先进方法的研究(3)加速生物技术研究者和医药治疗人员对数据库和软件的使用。
生物信息学习题

第六章 分子系统发生分析(问题与练习)
1、构建系统发生树,应使用
A、BLAST
B、FASTA
C、UPGMA
D、Entrez
2、构建系统树的主要方法有
、
、
等。
3、根据生物分子数据进行系统发生分析有哪些优点?
4、在 5 个分类单元所形成的所有可能的有根系统发生树中,随机抽取一棵树是反映真实关
系的树的可能性是多少?从这些分类单元所有可能的无根系统发生树中,随机选择一棵
库
8、TreeBASE 系统主要用于
A、发现新基因 B、系统生物学研究 C、类群间系统发育关系研究 D、序列比对
二、 问答题
1、 为什么说 SWISS-PROT 是最重要的蛋白质一级数据库?
2、 构建蛋白质二级数据库的基本原则是什么?
3、 构建蛋白质二级数据库的主要方法有哪些?
4、 叙述 SCOP 数据库对蛋白质分类的主要依据
第八章 后基因组时代的生物信息学(问题与练习)
1、 比较生物还原论与生物综合论的异同 2、 简述“后基因组生物信息学”的基本研究思路 3、 后基因组生物信息学的主要挑战是什么? 4、 功能基因组系统学的基本特征是什么? 5、 说明后基因组生物信息学对信息流动的最新理解 6、 列举几种预测蛋白质-蛋白质相互作用的理论方法 7、 解释从基因表达水平关联预测蛋白质-蛋白质相互作用的理论方法 8、 解释基因保守近邻法预测蛋白质-蛋白质相互作用的理论方法 9、 解释基因融合法预测蛋白质-蛋白质相互作用的理论方法 10、解释种系轮廓发生法预测蛋白质-蛋白质相互作用的理论方法
1、蛋白质得分矩阵类型有 、
、、
和
等。
2、对位排列主要有局部比对和 三、运算题 1、画出下面两条序列的简单点阵图。将第一条序列放在 x 坐标轴上,将第二条序列放在 y
生物信息学作业1.doc

生物信息学实验作业试验一一.找到编码拟南芥(arabidopsis)phyA(光敏色素A)基因的核酸序列编号, 并记录查找过程。
GI:224576211步骤1.进入NCBI主页2.搜索arabidopsis phyA3.Arabidopsis thaliana phytochrome A (PHYA) gene, partial cds4.VERSION:GI:224576211二.以phyA为检索词,在pubmed数据库中分别检索在题目和关键词字段中含有该检索词的文献,记录检索出的条目数目。
Results: 614三.仔细阅读所查询核酸序列在NCBI和EMBL数据库中格式的解释,理解各字段的含义,并比较NCBI 与EMBL中序列格式的异同。
实验二一.分析你感兴趣核酸序列的分子质量、碱基组成。
Composition 35 A; 25 C; 35 G; 15 T; 0 OTHERPercentage: 32% A; 23% C; 32% G; 14% T; 0%OTHERMolecular Weight (kDa): ssDNA: 34.26 dsDNA: 67.8二.列出你所分析核酸序列(或部分序列)的互补序列、反向序列、反向互补序列、DNA双链序列和RNA 序列。
R S1 ACTACTCGAG AAGCAGCGAC AGAGGCGTTA GCCCGCTCAG CAGACTGGCA GTTCTCTACC61 GACAAAAAAG AGGTAGGAGG CACAGTAATG ATACAGGCGT AGCAGGAGGGC S1 CCCTCCTGCT ACGCCTGTAT CATTACTGTG CCTCCTACCT CTTTTTTGTC GGTAGAGAAC61 TGCCAGTCTG CTGAGCGGGC TAACGCCTCT GTCGCTGCTT CTCGAGTAGTR C S1 TGATGAGCTC TTCGTCGCTG TCTCCGCAAT CGGGCGAGTC GTCTGACCGT CAAGAGATGG61 CTGTTTTTTC TCCATCCTCC GTGTCATTAC TATGTCCGCA TCGTCCTCCCD DNA S1 GGGAGGACGA TGCGGACATA GTAATGACAC GGAGGATGGA GAAAAAACAG CCATCTCTTGCCCTCCTGCT ACGCCTGTAT CATTACTGTG CCTCCTACCT CTTTTTTGTC GGTAGAGAAC61 ACGGTCAGAC GACTCGCCCG ATTGCGGAGA CAGCGACGAA GAGCTCATCATGCCAGTCTG CTGAGCGGGC TAACGCCTCT GTCGCTGCTT CTCGAGTAGTRNA S1 GGGAGGACGA UGCGGACAUA GUAAUGACAC GGAGGAUGGA GAAAAAACAG CCAUCUCUUG61 ACGGUCAGAC GACUCGCCCG AUUGCGGAGA CAGCGACGAA GAGCUCAUCA三.列出核酸序列的限制性酶切位点分析结果(酶及识别位点)。
生物信息学作业

结论一:这是什么基因1.该基因为人的CD226 抗原分子(CD226),染色体定位18号染色体67624232 -67530192基因标识符:NM_006566.22.功能:细胞粘附功能,整合素结合,蛋白结合,蛋白激酶结合;参与细胞粘合,细胞识别,细胞因子产生,正向调控Fc受体介导的刺激性信号通路,正向调控免疫球蛋白介导的免疫反应,正向调控肥大细胞的活化正向调控NK细胞介导的细胞毒性,正向调控NK细胞介导的针对肿瘤细胞靶标的细胞毒活性,调节免疫反应,信号转导等途径。
结论二:编码的蛋白质序列是怎样的蛋白标识符:"NP_006557.2" 336 aa蛋白序列为:MDYPTLLLAL LHVYRALCEE VLWHTSVPFA ENMSLECVYP SMGILTQVEWFKIGTQQDSI AIFSPTHGMV IRKPYAERVY FLNSTMASNN MTLFFRNASE DDVGYYSCSL YTYPQGTWQK VIQVVQSDSF EAAVPSNSHI VSEPGKNVTL TCQPQMTWPV QAVRWEKIQP RQIDLLTYCN LVHGRNFTSK FPRQIVSNCS HGRWSVIVIP DVTVSDSGLY RCYLQASAGE NETFVMRLTV AEGKTDNQYT LFVAGGTVLL LLFVISITTI IVIFLNRRRR RERRDLFTES WDTQKAPNNY RSPISTSQPT NQSMDDTRED IYVNYPTFSR RPKTRV结论三:有没有功能保守的结构序列?该蛋白有Ig的保守结构序列结论四;:它的功能是?功能:细胞黏附相关受体,淋巴细胞信号转导,CTL和NK介导的细胞毒性和淋巴因子分泌亚单元结构:与PVR和PVRL2相互作用亚细胞定位:细胞膜,Ⅰ类信号传播膜蛋白组织特异性:外周血T细胞表达序列:包含2个Ig-like C2型(免疫球蛋白样)结构域结论五:在真核生物中保守吗?在酵母中不存在其同源物,在一些灵长类动物存在一些同源性较高的序列,在其他的哺乳动物如:褐家鼠,野猪等中也存在一些同源性较高的序列。
生物信息学作业10

调和序列
1 Ⅰ Ⅱ Ⅲ Ⅳ Ⅴ Y Y F F Y y 2 D D E D E d 3 G G G G G 4 G G G G G G 5 A I I A A/I 6 V L L V 7 V V V 8 E E E Q Q 9 A A A A A 1 L L L V L
多序列比对的方法
同源性分析中常常要通过多序列比对来找出序列之间的相 互关系,和blast的局部匹配搜索不同,多序列比对大多 都是采用全局比对的算法。这样对于采用计算机程序的自 动多序列比对是一个非常复杂且耗时的过程,特别是序列 数目多,且序列长的情况下。
Clustal程序有许多版本。 Clustal是免费软件,很容易从互联网上下载,和其它软 件一起,广泛用于序列分析。Clustal所支持的数据格式包 括EMBL/SWISSPROT、PIR、Pearson/FastA以及 Clustal本身定义的格式。它的输出格式是Clustal格式。
FASTA格式
以上这个FASTA文件中包含了gi号码、GenBank检索号码、LOCUS名称、 以及GenBank记录中的DEFINATION字段。 第一行( 〉)表示一个新的序列文件的开始,为标记符。后面可以加上文 字说明, gi号码、GenBank检索号码、LOCUS名称等信息。 第二行 序列本身,为DNA或蛋白质的标准符号。通常核苷酸符号大小写 均可,而氨基酸一般用大写字母。
正因为存在这样的关系,很多时候对序列的相似性和 同源性就没有做很明显的区分,造成经常等价混用两个 名词。所以有出现A序列和B序列的同源性为80%一说。 不能把相似性和同源性混为一谈。所谓“具有50%同 源性”,或“这些序列高度同源”等说法,都是不确切 的,应该避免使用。
序列相似性比较和序列同源性分析
生物信息学作业

生物信息学作业生物信息学试题1、构建分子系统树的主要方法有哪些?并简要说明构建分子进化树的一般步骤。
(20分)答:(1)构建进化树的方法包括两种:一类是序列类似性比较,主要是基于氨基酸相对突变率矩阵(常用PAM250)计算不同序列差异性积分作为它们的差异性量度(序列进化树);另一类在难以通过序列比较构建序列进化树的情况下,通过蛋白质结构比较包括刚体结构叠合和多结构特征比较等方法建立结构进化树(2)序列比对——选取所需序列——软件绘制具体如下:a测序获取序列或者在NCBI上搜索所需的目的序列b在NCBI上做blast:比对相似度较高的基因,并以fast格式下载,整合在*txt文档中。
c比对序列,比对序列转化成*meg格式d打开保存的*meg格式文件,构建系统进化树2、氨基酸序列打分矩阵PAM和BLOSUM中序号有什么意义?它们各自的规律是什么?(10分)(1)PAM矩阵:基于进化的点突变模型,如果两种氨基酸替换频繁,说明自然界接受这种替换,那么这对氨基酸替换得分就高。
一个PAM就是一个进化的变异单位, 即1%的氨基酸改变。
BLOSUM矩阵:首先寻找氨基酸模式,即有意义的一段氨基酸片断,分别比较相同的氨基酸模式之间氨基酸的保守性(某种氨基酸对另一种氨基酸的取代数据),然后,以所有60%保守性的氨基酸模式之间的比较数据为根据,产生BLOSUM60;以所有80%保守性的氨基酸模式之间的比较数据为根据,产生BLOSUM80。
(2)PAM用于家族内成员相比,然后把所有家族中对某种氨基酸的比较结果加和在一起,产生“取代”数据(PAM-1 );PAM-1自乘n次,得PAM-n。
PAM-n中,n 越小,表示氨基酸变异的可能性越小;相似的序列之间比较应该选用n值小的矩阵,不太相似的序列之间比较应该选用n 值大的矩阵。
PAM-250用于约 20%相同序列之间的比较。
BLOSUM-n中,n越小,表示氨基酸相似的可能性越小;相似的序列之间比较应该选用n 值大的矩阵,不太相似的序列之间比较应该选用n值小的矩阵。
生物信息学作业

B7家族成员生物信息学分析作业
T细胞最适活化除了需要TCR传导的第一信号外,还需共刺激分子传导的第二信号。
其中研究最多、最清楚、认为最有意义的共刺激分子是B7-1和B7-2分子,它们与受体CD28分子或CTLA-4分子相互作用,在T细胞生长、分化和死亡中起重要作用。
在寻找B7-1和B7-2的同源分子过程中,最近发现了B7家族的新成员B7H1(B7 homolog 1)、B7H2(B7 homolog 2)、B7H3(B7 homolog 3)和B7H4(B7 homolog 4)分子。
作业要求对B7家族的这几个成员进行了生物信息学分析(主要包括:人源、鼠源B7家族成员基因cDNA序列之间、氨基酸序列之间的同源性比较及发生树分析、蛋白质结构分析、三级结构预测等),并对扩增和检测B7H4基因进行了引物和探针设计。
B7家族人源、鼠源等自己从相关数据库获得的、相关生物信息学分析越多加分。
作业提供了B7家族人源、鼠源的相关序列文件。
数据获得、利用软件分析、思路等需要文字说明和截图说明,不详细的扣分。
作业在开学第一周交,即2月28日之前。
发到jiabinxj@。
《生物信息学》题集

《生物信息学》题集一、选择题(每题3分,共30分)1.生物信息学的主要研究对象是什么?A. 蛋白质结构B. 基因序列C. 生态系统D. 细胞代谢2.下列哪项技术不是生物信息学中常用的数据库技术?A. BLASTB. GenBankC. PubMedD. SWISS-PROT3.在生物信息学中,进行多序列比对时常用的软件是什么?A. MATLABB. ClustalWC. ExcelD. PowerPoint4.哪种算法常用于基因表达数据的聚类分析?A. K-meansB. DijkstraC. A*D. Floyd5.生物信息学中,下列哪项不是常用的序列分析技术?A. PCRB. 测序C. 质谱分析D. 芯片技术6.下列哪项不是生物信息学在医学领域的应用?A. 疾病诊断B. 药物设计C. 天气预报D. 个性化医疗7.下列哪项技术常用于生物大分子的结构预测?A. NMRB. X射线衍射C. 同源建模D. 质谱分析8.在生物信息学中,下列哪项不是基因注释的内容?A. 基因功能B. 基因表达水平C. 基因在染色体上的位置D. 基因的长度9.下列哪项技术不是高通量测序技术?A. Sanger测序B. Illumina测序C. 454测序D. SOLiD测序10.下列哪项不是生物信息学在农业领域的应用?A. 作物育种B. 病虫害防治C. 土壤成分分析D. 农产品品质改良二、填空题(每题2分,共20分)1.生物信息学是一门交叉学科,它主要涉及______、计算机科学和数学等领域。
2.在生物信息学中,______技术常用于基因序列的相似性搜索。
3.生物信息学在药物研发中的主要应用包括______和药物靶点的预测。
4.在基因表达数据分析中,______是一种常用的数据标准化方法。
5.生物信息学中,______技术常用于蛋白质结构的预测和分析。
6.在生物信息学数据库中,GenBank主要存储的是______数据。
生物信息学作业2

生物信息学实验三1.了解什么是BLAST,它有哪些应用。
BLAST (Basic Local Alignment Search Tool)是一套在蛋白质数据库或DNA数据库中进行相似性比较的分析工具。
BLAST程序能迅速与公开数据库进行相似性序列比较。
BLAST结果中的得分是对一种对相似性的统计说明。
BLAST对一条或多条序列(可以是任何形式的序列)在一个或多个核酸或蛋白序列库中进行比对。
BLAST还能发现具有缺口的能比对上的序列。
2.请在NCBI中查找你感兴趣的某一基因或蛋白,通过BLAST工具检索与其高度相似的序列,并将你查到的这一基因或蛋白与你检索到的与其相似的序列(其中一条)的比对结果列出来,简单说明序列比对评分和检索过程。
Homo sapiens coagulation factor VIII, procoagulant component(F8), transcript variant 1, mRNA Length=9048Score = 398 bits (212), Expect = 1e-108Identities = 214/215 (99%), Gaps = 0/215 (0%)Strand=Plus/PlusQuery 15 GGAGCTGAATATGATGATCAGACCAGTCAAAGGGAGAAAGAAGATGATAAAGTCTTCCCT 74||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||Sbjct 559 GGAGCTGAATATGATGATCAGACCAGTCAAAGGGAGAAAGAAGATGATAAAGTCTTCCCT 618Query 75 GGTGGAAGCCATACATATGTCAGGCAGGTCCTGAAAGAGAATGGTCCAATGGCCTCTGAC 134||||||||||||||||||||| ||||||||||||||||||||||||||||||||||||||Sbjct 619 GGTGGAAGCCATACATATGTCTGGCAGGTCCTGAAAGAGAATGGTCCAATGGCCTCTGAC 678Query 135 CCACTGTGCCTTACCTACTCATATCTTTCTCATGTGGACCTGGTAAAAGACTTGAATTCA 194||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||Sbjct 679 CCACTGTGCCTTACCTACTCATATCTTTCTCATGTGGACCTGGTAAAAGACTTGAATTCA 738Query 195 GGCCTCATTGGAGCCCTACTAGTATGTAGAGAAGG 229|||||||||||||||||||||||||||||||||||Sbjct 739 GGCCTCATTGGAGCCCTACTAGTATGTAGAGAAGG 773Homo sapiens chromosome X genomic contig, GRCh37.p5 Primary AssemblyLength=6178498Score = 451 bits (240), Expect = 2e-124Identities = 242/243 (99%), Gaps = 0/243 (0%)Strand=Plus/MinusQuery 1 TTCTTCCTGCTATAGGAGCTGAATATGATGATCAGACCAGTCAAAGGGAGAAAGAAGATG 60||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||Sbjct 5139376 TTCTTCCTGCTATAGGAGCTGAATATGATGATCAGACCAGTCAAAGGGAGAAAGAAGATG 5139317Query 61 ATAAAGTCTTCCCTGGTGGAAGCCATACATATGTCAGGCAGGTCCTGAAAGAGAATGGTC 120||||||||||||||||||||||||||||||||||| ||||||||||||||||||||||||Sbjct 5139316 ATAAAGTCTTCCCTGGTGGAAGCCATACATATGTCTGGCAGGTCCTGAAAGAGAATGGTC 5139257Query 121 CAATGGCCTCTGACCCACTGTGCCTTACCTACTCATATCTTTCTCATGTGGACCTGGTAA 180||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||Sbjct 5139256 CAATGGCCTCTGACCCACTGTGCCTTACCTACTCATATCTTTCTCATGTGGACCTGGTAA 5139197Query 181 AAGACTTGAATTCAGGCCTCATTGGAGCCCTACTAGTATGTAGAGAAGGTAAGTGTATGA 240||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||Sbjct 5139196 AAGACTTGAATTCAGGCCTCATTGGAGCCCTACTAGTATGTAGAGAAGGTAAGTGTATGA 5139137Query 241 AAG 243|||Sbjct 5139136 AAG 5139134Homo sapiens chromosome X genomic contig, alternate assemblyHuRef SCAF_1103279188170, whole genome shotgun sequence Length=869535Score = 451 bits (240), Expect = 2e-124Identities = 242/243 (99%), Gaps = 0/243 (0%)Strand=Plus/MinusQuery 1 TTCTTCCTGCTATAGGAGCTGAATATGATGATCAGACCAGTCAAAGGGAGAAAGAAGATG 60||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||Sbjct 406733 TTCTTCCTGCTATAGGAGCTGAATATGATGATCAGACCAGTCAAAGGGAGAAAGAAGATG 406674Query 61 ATAAAGTCTTCCCTGGTGGAAGCCATACATATGTCAGGCAGGTCCTGAAAGAGAATGGTC 120||||||||||||||||||||||||||||||||||| ||||||||||||||||||||||||Sbjct 406673 ATAAAGTCTTCCCTGGTGGAAGCCATACATATGTCTGGCAGGTCCTGAAAGAGAATGGTC 406614Query 121 CAATGGCCTCTGACCCACTGTGCCTTACCTACTCATATCTTTCTCATGTGGACCTGGTAA 180||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||Sbjct 406613 CAATGGCCTCTGACCCACTGTGCCTTACCTACTCATATCTTTCTCATGTGGACCTGGTAA 406554Query 181 AAGACTTGAATTCAGGCCTCATTGGAGCCCTACTAGTATGTAGAGAAGGTAAGTGTATGA 240||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||Sbjct 406553 AAGACTTGAATTCAGGCCTCATTGGAGCCCTACTAGTATGTAGAGAAGGTAAGTGTATGA 406494Query 241 AAG 243|||Sbjct 406493 AAG 4064911)于NCBI下载一条序列(FASTA格式)2)在BLAST中载入该序列文件3)调节各参数4)点击BLAST进行比对3.理解BLAST不同参数的含义,以及如何调整和适用情况。
生物信息学作业

作业个人感觉entrez系统查询序列比SRS更精准,推荐用entrez完成作业。
1.Do a search for the 16S ribosomal RNA gene from Aeromonas hydrophila strainAE7.a.Give the search details that you used to find this sequence.b.What is the accession number?c.How many base pairs are in this sequence?d.When was the entry last modified?e.Is there another organism that produces the same gene? If so, name theorganism and show your evidence.答案:a.进入网址/Entrez,在搜索栏中输入“16Sribosomal RNA gene from Aeromonas hydrophila strain AE7”点击GO,在Nucleotide就能找到结果。
b.DQ855289c. 992 bpd. 21-AUG-2006e. there are a few organisms like Drosophila melanogaster and it was published onScience 316 (5831), 1625-1628 (2007)2.Search for the nucleotide sequence with accession number NM_013161.a.What organism is this sequence from?b.What is the accession number of the protein linked to this sequence?c.What is the function of this protein?d.Find a reference by Hjorth, et al, related to this protein. What is the PubMedID for this article?e.In your own words, briefly describe what the researchers reported in thearticle.答案:a.Rattus norvegicus (Norway rat)b. NP_037293.1c.The protein named Pancreatic triglyceride lipase (胰甘油三酯脂酶, PTL), is an enzyme ofdigestive system, which plays very important roles in the digestion and absorption of lipids. And there has new data suggested that PTL may be involved in the pathophysiology of TBI (脑外伤) and that PTL may be implicated in the proliferation (增殖) of astrocytes (星形胶质细胞) and the recovery of neurological outcomes.d. 8490016 Pancreatic lipase structure-function relationships by domain exchange (通过改变结构域来研究胰脂酶三级结构与功能的关系).e.试验通过交换古典人类胰脂肪酶(HPL)和几内亚猪胰脂肪酶相关蛋白2(GPLRP2)帽子区域设计嵌合突变体,测定两种嵌合体C-端磷脂酶与脂肪酶活性发现脂肪分解酵素在界面锚定和稳定的效应,这个嵌合体的动力学特性首次揭示了胰脂肪酶的界面稳定性取决于它的C端结构域的结构。
生物信息技术第二次作业

生物信息技术第二次作业第一篇:生物信息技术第二次作业实用生物信息技术课程第2次作业人、小鼠、大鼠血红蛋白及其编码基因序列分析1.认真阅读NCBI书架(Bookshelf)、PDB分子月报(Molecule of the Month)、UniProt蛋白质精选(Protein Spotlight)以及维基百科(Wikipedia)等网站中有关血红蛋白的介绍,了解血红蛋白的生理功能、空间结构、亚基组成等基本知识。
血红蛋白是高等生物体内负责运载氧的一种蛋白质,具有四聚体蛋白质四级空间结构。
人体内的血红蛋白由四个亚基构成,分别为两个α亚基和两个β亚基,每个亚基由一条肽链和一个血红素分子构成。
血红蛋白的每个亚基由一条肽链和一个血红素分子构成,肽链在生理条件下会盘绕折叠成球形,把血红素分子抱在里面,这条肽链盘绕成的球形结构又被称为珠蛋白。
血红素分子是一个具有卟啉结构的小分子,在卟啉分子中心,由卟啉中四个吡咯环上的氮原子与一个亚铁离子配位结合,珠蛋白肽链中第8位的一个组氨酸残基中的吲哚侧链上的氮原子从卟啉分子平面的上方与亚铁离子配位结合,当血红蛋白不与氧结合的时候,有一个水分子从卟啉环下方与亚铁离子配位结合,而当血红蛋白载氧的时候,就由氧分子顶替水的位置。
2.查阅ENSEMBL基因组数据库中已经或正在进行基因组测序的物种树,了解人、小鼠、大鼠三个物种之间演化关系;检索物种分歧时间数据库TimeTree,了解人和小鼠、小鼠和大鼠之间的分歧时间。
3.从UniProt数据库中检索并提取人、小鼠、大鼠血红蛋白alpha 亚基蛋白质序列,利用WebLab或JEMBOSS软件包中的序列比对程序Needle,选择默认计分矩阵BLOSUM62和默认空位罚分进行序列比对,将比对结果填入表1。
分析比对结果,说明得到上述结果的原因和进一步分析思路。
表1 人、小鼠、大鼠血红蛋白alpha亚基蛋白质序列比对结果物种Species 人/小鼠人/大鼠登录号AccessionNP_000549/NP_032244 NP_000549/NP_037228得分 Score 653 587相同氨基酸 Identity 123/142(86.6%)111/142(78.2%)相同和相似氨基酸Similarity 131/142(92.3%)120/142(84.5%)空位 Gaps 0 0实用生物信息技术课程第2次作业小鼠/大鼠 NP_032244/NP_037228632 120/142(84.5%)127/142(89.4%)0 4.检索RefSeq数据库中人、小鼠、大鼠血红蛋白alpha亚基编码基因的mRNA序列,下载其编码区序列和所编码的氨基酸序列,进行序列比对,将结果填入下面表2中。
生物信息学课后题及答案

生物信息学课后习题及答案(由10级生技一、二班课代表整理)一、绪论1.你认为,什么是生物信息学?采用信息科学技术,借助数学、生物学的理论、方法,对各种生物信息(包括核酸、蛋白质等)的收集、加工、储存、分析、解释的一门学科。
2.你认为生物信息学有什么用?对你的生活、研究有影响吗?(1)主要用于:在基因组分析方面:生物序列相似性比较及其数据库搜索、基因预测、基因组进化和分子进化、蛋白质结构预测等在医药方面:新药物设计、基因芯片疾病快速诊断、流行病学研究:SARS、人类基因组计划、基因组计划:基因芯片。
(2)指导研究和实验方案,减少操作性实验的量;验证实验结果;为实验结果提供更多的支持数据等材料。
3.人类基因组计划与生物信息学有什么关系?人类基因组计划的实施,促进了测序技术的迅猛发展,从而使实验数据和可利用信息急剧增加,信息的管理和分析成为基因组计划的一项重要的工作。
而这些数据信息的管理、分析、解释和使用促使了生物信息学的产生和迅速发展。
4简述人类基因组研究计划的历程。
通过国际合作,用15年时间(1990-2005)至少投入30亿美元,构建详细的人类基因组遗传图和物理图,确定人类DNA的全部核苷酸序列,定位约10万基因,并对其他生物进行类似研究。
1990,人类基因组计划正式启动。
1996,完成人类基因组计划的遗传作图,启动模式生物基因组计划。
1998完成人类基因组计划的物理作图,开始人类基因组的大规模测序。
Celera公司加入,与公共领域竞争启动水稻基因组计划。
1999,第五届国际公共领域人类基因组测序会议,加快测序速度。
2000,Celera公司宣布完成果蝇基因组测序,国际公共领域宣布完成第一个植物基因组——拟南芥全基因组的测序工作。
2001,人类基因组“中国卷”的绘制工作宣告完成。
2003,中、美、日、德、法、英等6国科学家宣布人类基因组序列图绘制成功,人类基因组计划的.目标全部实现。
2004,人类基因组完成图公布。
生物信息学作业题

生物信息学作业题生物信息学作业题绪论1.什么是生物信息学?2.生物信息学有哪些主要研究领域?第一章生物信息学的分子生物学基础1.DNA的双螺旋结构要点是什么?2.什么是基因组和蛋白质组?对它们的研究有何意义?第二章生物信息学的计算机基础1.简述网络操作系统的类型。
第三章核酸序列分析1.什么是全局比对?2.什么是局部比对?有哪些优点?第四章分子进化分析1.分子进化分析具有哪些优点?2. 简述分子进化的中性学说。
第五章基因组分析1. 什么是基因组学?其主要研究内容是什么?2.简述基因预测分析的一般步骤。
第六章蛋白质组分析1. 蛋白质组学的概念和主要研究的大致方向是什么?2. 蛋白质组功能预测的程序是怎样的?第七章生物芯片数据分析1. 什么是生物芯片?2. 生物芯片有哪些方面的应用?第八章核酸与蛋白质结构预测1. RNA二级结构典型的预测方法有哪些?2. 基于统计学的预测蛋白质二级结构的方法有哪些?第九章生物信息学平台与工具软件1. 请利用Clustal X软件对下列6条蛋白质序列进行多重比对(比对结果用BioEdit软件打开,用“截图”方式显示比对结果)。
>1mqngkvkwfn sekgfgfiev eggedvfvhf saiqgegfkt leegqevtfe veqgnrgpqatnvnkk>2mqgkvkwfnn ekgfgfieie gaddvfvhfs aiqgegykal eegqevsfdi tegnrgpqaanvvkl>3mqngkvkwfn sekgfgfiev eggedvfvhf saiqgegfkt leegqevtfe veqgnrgpqatnvnkk>4mqgkvkwfnn ekgfgfieie gaddvfvhfs aiqgegykal eegqevsfdi tegnrgpqaanvvkl>5mqngkvkwfn sekgfgfiev eggedvfvhf saiqgegfkt leegqevtfe veqgnrgpqatnvnkk>6mqgkvkwfnn ekgfgfieie gaddvfvhfs aiqgegykal eegqevsfdi tegnrgpqaanvvkl2. 现有一ZmPti1b蛋白质序列,请用DNAMAN软件分析其二级结构,给出分析结果。
生物信息上机作业

生物信息学上机作业上机一生物信息数据库信息检索上机内容:1、了解NCBI、DDBJ、EMBL上网的方法自学各网站相关介绍。
2、了解北大生物信息学中心等几大中文生物信息学网站。
3、了解一些生物论坛中有关生物信息学的部分。
如:Biooo和Bioon。
4、利用NCBI的Entrenz查询系统和EBI的SRS检索文献和核酸或蛋白质序列。
(phyA)并对照所学复习各字段的含义。
5、将所得记录的ID或Accession记录下来备用。
作业:1、记录相关网站及论坛网址(或如何查询到该网址的方法)。
(1)NCBI :/(2)DDBJ :http://www.ddbj.nig.ac.jp/(3)EMBL :/(4)北大生物信息学中心 /chinese/(5)中科院计算所智能信息处理重点上机室生物信息学:/index.php(6)北大生物信息中心:/chinese/documents/bioinfor/overview/web1/1.html (7)生物谷生物信息学:/bioinfo.htm(8)中国生物论坛:/(9)中国生物谷论坛:/(10)生物谷:/2、找到编码拟南芥(arabidopsis)phyA(光敏色素A)基因的核酸序列编号。
并记录查找过程。
上机二核酸及蛋白质序列的比对一、上机内容利用检索出的蛋白质和核酸序列进行序列比对并进行分子进化树分析。
二、作业1、绘制分子进化树,并标明各个物种phyA蛋白之间的序列相似性。
2、根据你所学生物分类的知识,试解释该分子进化树的合理性。
3、找出一条可能的保守序列(多条蛋白共同的氨基酸序列)。
上机三核酸序列分析(一)一、上机内容1、使用DNAstar进行核酸基本信息分析2、ORF分析二、作业1、记录拟南芥phyA NM_100828序列的序列组成2、记录拟南芥phyA NM_100828序列最长的ORF的起止区间。
上机四核酸序列分析(二)一、上机内容1、PCR引物设计2、核酸序列的电子基因定位二、作业1、记录拟南芥phyA NM_100828序列最长的ORF的起止区间。
研究生《生物信息学》作业模板

研究生《医学生物信息学》作业班级:专业:姓名:一、实验目的:(1)掌握中文文献全文的检索和获得方法。
(2)掌握Pubmed数据库文献的检索和交大图书馆英文数据库全文的获得方法。
(3)掌握核酸序列搜索的方法。
(4)掌握核酸序列相似性分析的方法。
(5)掌握PCR引物设计软件的原理、使用及特点。
(6)掌握蛋白质序列搜索的方法。
(7)掌握蛋白质序列分析常用软件的使用方法。
二、研究背景:AIB1基因为近年来发现的p160类固醇受体转录共激活因子SRC-1家族成员,是新定义的一个原癌基因[1]。
该基因表达的蛋白在许多生物学过程中发挥重要作用,如细胞生长,增殖,分化,性成熟,女性生殖功能等[2]。
近年发现,该基因的表达异常与多种肿瘤的发生发展有关,以在乳腺癌中研究最多。
AIB1基因的高表达与乳腺癌的发生和发展有关[3]。
AIB1蛋白通过与雌激素受体相互作用,能强烈地增强雌激素受体的促进靶基因转录的效应,进而引起细胞增殖和肿瘤形成,此外,AIB1蛋白还在多条信号传导通路中发挥作用[4]。
AIB1基因(amplified in breast cancer1)又称为ACTR,TRAM1,RAC3,SRC3,NCoA3,P/CIP等。
本人选择其为研究对象。
三、实验方法、步骤及结果:1.在中国知网(CNKI)中查找中文文献:2.在PubMed中查找英文文献:登陆NCBI主页,网址:/guide/,选择gene数据库4. 使用NCBI网站中的BLAST工具进行序列比对登陆/,选择核酸序列比对nucleotide BLAST,界面显示如下,输入登录号,AF012108,点击“BLAST”。
结果如下:共有2条核苷酸序列和2条基因组序列和其匹配:第一条核苷酸序列为“Homo sapiens nuclear receptor coactivator 3 (NCOA3), transcript variant 2, mRNA”,登录号:NM_006534。
生物信息学作业
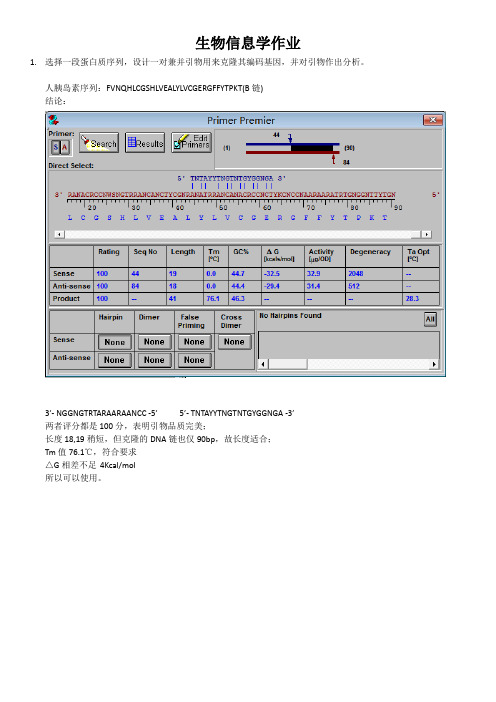
生物信息学作业1.选择一段蛋白质序列,设计一对兼并引物用来克隆其编码基因,并对引物作出分析。
人胰岛素序列:FVNQHLCGSHLVEALYLVCGERGFFYTPKT(B链)结论:3’- NGGNGTRTARAARAANCC -5’ 5’- TNTAYYTNGTNTGYGGNGA -3’两者评分都是100分,表明引物品质完美;长度18,19稍短,但克隆的DNA链也仅90bp,故长度适合;Tm值76.1℃,符合要求△G相差不足4Kcal/mol所以可以使用。
Step1:打开primer premier 5.0 输入蛋白质链,转化为DNA链。
获得DNA链。
2.选择一段基因,预测期编码RNA的二级结构,并分析功能。
取一段基因:ACGCG GGCGG GCATG TGGGC AGCTT TACCC AGTGC TACTG TGCTG GCCAGCACTG AAACA GGGGC ACTGG TTTGG GGTGG ATGAA GGGTA GAAGT GCAAGTTCCA TTGCC TGTGC AATCC CTGCC TTGCT CAGAC CCTGC TCACT CCTCAGGCCC CATCA GCCCC TCAAC TCTGC TAACC ATGGT GGTAG AAATC AGCTACAATA AACCC TGGAG CCAGT AAAAA AAAAA AAAAA AAAAA AAAAA AAAGT点击Fold as RNA点击START点击Draw Stuclture得到RNA二级结构RNA功能预测打开网址http://sidirect2.rnai.jp/输入DNA序列得出结论:。
《生物信息学》上机作业

《生物信息学》上机作业题目:对人血红蛋白(HBA1)编码基因序列的生物信息分析目录引言 .............................................................................................................................................. - 1 -1 正文......................................................................................................................................... - 2 -1.1 NCBI上对相关核苷酸序列的查找............................................................................ - 2 -1.2 BLAST运行及其结果.................................................................................................. - 2 -1.3 BLASTX运行及其结果................................................................................................ - 6 -2 其他软件的运行及其结果..................................................................................................... - 8 -2.1 Clustal W运行及其结果 ............................................................................................. - 9 -2.2 MEGA4.0运行及其结果............................................................................................. - 10 -结论 ............................................................................................................................................ - 10 -引言血红蛋白又称血色素,是红细胞的主要组成部分,能与氧结合,运输氧和二氧化碳。
生物信息学试题及答案

生物信息学试题及答案一、单项选择题(每题2分,共20分)1. 生物信息学的主要研究对象是()。
A. 生物数据B. 生物实验C. 生物模型D. 生物技术答案:A2. 下列哪项不是生物信息学中的常用数据库()。
A. GenBankB. Swiss-ProtC. PubMedD. Google Scholar答案:D3. 蛋白质序列比对的主要目的是()。
A. 确定蛋白质的三维结构B. 预测蛋白质的功能C. 比较蛋白质的氨基酸序列D. 计算蛋白质的分子量答案:B4. 在生物信息学中,以下哪种算法不是用于序列比对的()。
A. BLASTB. FASTAC. Smith-WatermanD. Hidden Markov Model答案:D5. 下列哪种生物信息学工具主要用于基因表达分析()。
A. ClustalWB. Primer3C. R语言D. PDB答案:C6. 以下哪种技术不是用于蛋白质结构预测的()。
A. 同源建模B. 从头预测C. 序列比对D. 折叠识别答案:C7. 以下哪种生物信息学工具主要用于基因组注释()。
A. BLASTC. GATKD. Primer3答案:B8. 在生物信息学中,以下哪种方法不用于基因表达数据的聚类分析()。
A. K-meansB. Hierarchical clusteringC. Principal component analysisD. Multiple sequence alignment答案:D9. 下列哪种生物信息学工具主要用于蛋白质-蛋白质相互作用网络分析()。
A. STRINGB. BLASTD. Primer3答案:A10. 在生物信息学中,以下哪种数据库不包含蛋白质结构信息()。
A. PDBB. UniProtC. RCSBD. GenBank答案:D二、多项选择题(每题3分,共15分)11. 生物信息学中常用的序列比对工具包括()。
A. BLASTB. FASTAC. ClustalWD. Pfam答案:ABC12. 以下哪些是生物信息学中常用的基因表达分析软件()。
《生物信息学》练习题及答案

《生物信息学》练习题及答案1、在Genbank中查找以下6个植物蛋白序列:protein1:NP_974673.2;protein2:NP_187969.1;protein3: NP_190855.1;protein4:NP_565618.1;protein5: NP_200511.1;protein6:NP_191407.1(以FASTA格式)。
(1)用EBI上的ClustalW2工具对其进行多序列比对,分析各蛋白序列之间的同源性。
序列比对结果比对结果表明:protein1:NP_974673.2和protein4: NP_565618.1的亲缘关系最近。
(2)利用Phylip软件,选择距离法构建其进化树(要求写出具体的建树步骤)。
1.将蛋白序列保存为FASTA格式,存于txt文档;2.用Clustalx打开txt文本,保存为*.phy文件;3.用seqboot程序打开phy文件,输出结果文件*_seqboot4.用protdist程序打开*_seqboot文件,输出为*_protdist文件5.用neighbor程序打开*_protdist文件,输出为*_neighbor 文件6.用consense程序打开*_neighbor文件,输出为*_consense 文件7.用dratree程序打开*_consense文件得到进化树。
(注:由于seqboot软见无法正常运行,因此进化树无法显示)(3)任意选取其中的一个蛋白进行蛋白质一级序列分析、二级结构预测及三维结构的模拟。
选择protein3:NP_190855.1一级结构网址:/doc/479b86d06edb6f1afe001f6e.html /tools/protparam.htmlNumber of amino acids:456氨基酸数目Molecular weight:51154.5相对分子质量Theoretical pI:8.69理论pI值Amino acid composition氨基酸组成Ala(A)306.6%Arg(R)286.1%Asn(N)153.3%Asp(D)275.9%Cys(C)51.1%Gln(Q)183.9%Glu(E)286.1%Gly(G)378.1%His(H)163.5%Ile(I)163.5%Leu(L)429.2%Lys(K)327.0%Met(M)51.1%Phe(F)173.7%Pro(P)163.5%Ser(S)4610.1%Thr(T)214.6%Trp(W)81.8%Tyr(Y)194.2%Val(V)306.6%Pyl(O)00.0%Sec(U)00.0%(B)00.0%(Z)00.0%(X)00.0%正/负电荷残基数Total number of negatively charged residues(Asp+Glu): 55Total number of positively charged residues(Arg+Lys): 60Atomic composition:原子组成Carbon C2270Hydrogen H3531Nitrogen N645Oxygen O686Sulfur S10Formula:C2270H3531N645O686S10分子式Total number of atoms:7142总原子数Extinction coefficients:消光系数Extinction coefficients are in units of M-1cm-1,at280 nm measured in water.Ext.coefficient72560Abs0.1%(=1g/l)1.418,assuming all pairs of Cys residues form cystines Ext.coefficient72310Abs0.1%(=1g/l) 1.414,assuming all Cys residues are reducedEstimated half-life:半衰期The N-terminal of the sequence considered is M(Met). The estimated half-life is:30hours(mammalian reticulocytes,in vitro).>20hours(yeast,in vivo).>10hours(Escherichia coli,in vivo).Instability index:不稳定系数The instability index(II)is computed to be48.99This classifies the protein as unstable.Aliphatic index:75.26脂肪系数Grand average of hydropathicity(GRAVY):-0.554总平均亲水性蛋白质亲疏水性分析所用氨基酸标度信息Ala:1.800Arg:-4.500Asn:-3.500Asp:-3.500Cys:2.500 Gln:-3.500Glu:-3.500Gly:-0.400His:-3.200Ile:4.500 Leu:3.800Lys:-3.900Met:1.900Phe:2.800Pro:-1.600 Ser:-0.800Thr:-0.700Trp:-0.900Tyr:-1.300Val: 4.200:-3.500:-3.500:-0.490分析所用参数信息Weights for window positions1,..,9,using linear weight variation model:1234567891.001.001.001.001.001.001.001.001.00edge center edge跨膜结构预测结果(没有跨膜结构)信号肽分析:二级结构预测三级结构预测网站/doc/479b86d06edb6f1afe001f6e.html/~phyre2、在拟南芥基因组数据库中(/doc/479b86d06edb6f1afe001f6e.ht ml/)查找编号分别为At4G33050,At3G13600,At3G52870或At2G26190基因,针对所查找的基因进行初步的生物信息学分析(每人任选其中一个基因)。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
乳腺癌易感基因BRCA1的研究
班级:5061专业:药剂学姓名:孙建梅
一、实验目的:
(1)掌握中文文献全文的检索和获得方法。
(2)掌握Pubmed数据库文献的检索和交大图书馆英文数据库全文的获得方法。
(3)掌握核酸序列搜索的方法。
(4)掌握核酸序列相似性分析的方法。
(5)掌握PCR引物设计软件的原理、使用及特点。
(6)掌握蛋白质序列搜索的方法。
(7)掌握蛋白质序列分析常用软件的使用方法。
二、研究背景:
乳腺癌易感基因(BRCA1)的突变率与35%~40%的家族性乳腺癌和卵巢癌有关。
该基因常以染色体显性方式遗传,并有很高的外显率。
外显率在乳腺癌为60%~80%,卵巢癌也可达15%~40%。
该基因作为一种抑癌基因, 不仅能抑制细胞生长, 还参与细胞周期调控、基因转录调节、DNA 损伤修复及其凋亡等重要细胞活动, 在维持基因稳定性中起重要作用。
BRCA1是目前所发现的最重要的乳腺癌易感基因之一,本人选择其为研究对象。
三、实验方法、步骤及结果:
1.在中国知网(CNKI)中查找中文文献:
2.在PubMed中查找英文文献:
3 在Genbank中查找BRCA1基因及其序列:
登陆NCBI主页,网址:/guide/,选择gene数据库
4. 使用NCBI网站中的BLAST工具进行序列比对
登陆/,选择核酸序列比对nucleotide BLAST,界面显示如下,
输入登录号,NM-007294.3,点击“BLAST”。
结果如下:
与其匹配的核苷酸序列和基因组序列如下:
一条核苷酸序列为“Homo sapiens breast cancer 1(BRCA1), transcript variant 1, mRNA”,登录号:NM_007294.3。
另一条核苷酸序列为“Homo sapiens breast cancer 1(BRCA1), transcript variant 2, mRNA”,登录号:NM_007300.3。
5.蛋白质序列的比对检索页面:
结果输出:
6. 根据序列,设计PCR引物:
(1)利用peimer3进行引物设计
登陆引物设计软件primer3网址/primer3/。
输入FASTA格式的核苷酸序列,运算得到:
上游引物:5’caccctctgctctgggtaaa 3’
下游引物:5’aagctcattcttggggtcct 3’
产物:5680bp。
引物与模板结合的位点显示如下:
7.蛋白质预测
BRCA1蛋白在NCBI protein数据库中的登录号为NP-009225.3,共1863个氨基酸残基组成。
(1)使用protparam在线软件分析蛋白质基本理化性质。
结果如下,分子量:207720.8,理论等电点:5.29,为近似不稳定蛋白,亲水。
(2)跨膜区序列和方向预测:
/software/TMPRED_form.html,结果如下,分析显示该蛋白没有跨膜区。
(3)使用SignalP(http://www.cbs.dtu.dk/services/SignalP/)预测信号肽切割位点。
神经网络及隐氏马尔可夫模型均认为该蛋白为非分泌性蛋白,无信号肽位点。
(4)使用sopma软件预测蛋白质的二级结构:
该蛋白二级结构以为a螺旋为主,还包括随意卷曲和β折叠。
(5)使用PDB /pdb/search/advSearch.do?st=SequenceQuery,预测蛋白质三级结构及功能,没有找到匹配结构。
四、分析与讨论:
近年来,BRCA1的研究越来越广泛。
BRCA1基因覆盖大约100 k b的基因组DNA, 含有24个外显子, 其中22个为编码的外显子。
BRCA1基因编码1863个氨基酸, 与已知蛋白的同源性较低。
在Genbank中查找BRCA1基因,得到对该基因的概述:BRCA1基因能够编码一种在维持基因组稳定性中起作用的核蛋,它也被视为一种肿瘤抑制基因。
其编码的蛋白质结合其他肿瘤抑制基因,DNA损伤传感器及信号转导形成一个
大的多亚基蛋白复合物称为相关基因组监控复杂的BRCA1(BASC),该基因产物与RNA聚合酶II结合,并通过C端结构域,与组蛋白去乙酰化酶复合物相互作用。
这种蛋白在转录、核酸修复、双链断裂和重组中发挥了作用。
该基因突变约40%导致遗传性乳腺癌,而高达80%以上则会导致遗传性乳腺癌和卵巢癌。
选择性剪接在调节该基因的亚细胞定位和生理功能中起着调节作用。
许多剪接体的变体并未被描述,但是其中一些与基因突变导致的疾病相关的变体的全长序列已被描述。
位于17号染色体上相关假基因也已被确定。
BRCA1基因mRNA在NCBI数据库中的登录号为NM-007294.3,BRCA1蛋白在NCBI protein数据库中的登录号为NP-009225.3,共1863个氨基酸残基组成。
在进行核酸及蛋白质序列比对过程中,发现了若干条核苷酸序列和基因组序列和其匹配。
根据序列,设计PCR引物,可得上游引物:5’caccctctgctctgggtaaa 3’,下游引物:5’aagctcattcttggggtcct 3’,产物:5680bp以及上、下游引物与模板结合的位点。
蛋白质预测中,可得到以下结论:
1、BRCA1蛋白在NCBI protein数据库中的登录号为NP-009225.3,1863个氨基酸残基组成。
使用protparam在线软件分析蛋白质基本理化性质。
结果如下,分子量:207720.8,理论等电点:5.29,为近似不稳定蛋白,亲水。
2、跨膜区序列和方向预测:结果分析显示该蛋白没有跨膜区。
3、预测信号肽切割位点,结果显示神经网络及隐氏马尔可夫模型均认为该蛋白为非分泌性蛋白,无信号肽位点。
4、蛋白质二级结构预测认为该蛋白二级结构以a螺旋为主,还包括随意卷曲和β折叠。
5、预测蛋白质三级结构及功能,没有找到匹配结构。
总之,关于BRCA1 的研究已经取得了较大进展, 如BRCA1 在转录调节及DNA 修复中的作用已经被肯定。
BRCA1 基因异常还在人类许多癌症中被证实, 包括乳腺癌、卵巢癌、前列腺癌、直肠癌、子宫内膜癌等。
但是有关BRCA1 的抑癌机制, 如何提高基因的检测率以及怎样对基因突变携带者进行有效的预防等问题, 仍需更加深入和系统地进行研究。
五、参考文献
[1]严景华,叶棋浓,黄翠芬. 乳腺癌易感基因BRCA1研究进展[J]. 遗传,2004,03:367-372.
[2]崔珅,韩世愈,林英姬. 乳腺癌易感基因(BRCA1)研究进展[J]. 中国肿瘤,2008,04:290-292.
[3]王曦,杨名添,曾益新. 乳腺癌易感基因BRCA1和BRCA2的突变检测及其临床意义[J]. 国外医学(肿瘤学分册),2001,01:63-65.
[4]Daley, J.M.; Sung, P. 53BP1, BRCA1, and the choice between recombination and end joining at DNA double-strand breaks. Mol.Cell Biol., 2014, 34(8), 1380-1388.
[5] Zhu, Q.; Pao, G.M.; Huynh, A.M.; Suh, H.; Tonnu, N.; Nederlof,P.M.; Gage, F.H.;
Verma, I.M. BRCA1 tumour suppression occurs via heterochromatin-mediated silencing. Nature, 2011, 477(7363),179-184.
[6] Kurian, A.W. BRCA1 and BRCA2 mutations across race and ethnicity:
distribution and clinical implications. Curr. Opin. Obstet.Gynecol., 2010, 22(1), 72-78.。
