用探针法进行基因分型的方法
荧光pcr法和电泳

荧光pcr法和电泳荧光PCR法和电泳引言PCR技术是一种重要的基因分析技术,它能够扩增DNA,从而使得检测更加敏感。
荧光PCR法和电泳是PCR技术中常用的两种方法。
本文将详细介绍荧光PCR法和电泳的原理、应用以及优缺点。
一、荧光PCR法1.1 原理荧光PCR法是一种利用荧光探针检测扩增产物的方法。
在扩增过程中,引物结合到模板DNA上,并且在DNA聚合酶的作用下进行扩增。
同时,荧光探针也会结合到目标区域上,并且与DNA结合后发生融合,导致发射出特定波长的荧光信号。
这个信号可以被检测器捕获并且分析。
1.2 应用荧光PCR法被广泛应用于病毒、细菌等微生物检测以及基因突变、SNP等基因分型等领域。
例如,在乙型肝炎病毒检测中,可以利用荧光探针来检测病毒载量;在基因突变分析中,可以利用荧光探针来检测SNP等基因多态性。
1.3 优缺点荧光PCR法具有高灵敏度、高特异性、高通量等优点。
同时,荧光PCR法可以进行实时检测,从而减少了实验时间和成本。
但是,荧光PCR法的探针设计比较困难,而且需要使用昂贵的仪器。
二、电泳2.1 原理电泳是一种利用电场对DNA分子进行分离的方法。
在电泳过程中,DNA分子会在电场作用下向阳极或阴极移动,移动速度与DNA分子大小成反比。
因此,在一定时间内,不同大小的DNA分子会被分离开来,并且可以通过染色剂进行可视化。
2.2 应用电泳被广泛应用于DNA测序、基因型鉴定、病原体检测等领域。
例如,在DNA测序中,可以利用电泳将不同长度的DNA片段分离出来,并且进行序列测定;在基因型鉴定中,可以利用电泳将不同基因型的PCR产物分离开来,并且通过染色剂进行可视化。
2.3 优缺点电泳具有简单、易操作、成本低等优点。
同时,电泳可以进行大规模样品分析,从而适用于高通量分析。
但是,电泳需要使用大量的试剂和设备,并且对于低浓度的DNA样品灵敏度较低。
结论荧光PCR法和电泳是PCR技术中常用的两种方法。
荧光PCR法具有高灵敏度、高特异性、高通量等优点,适用于微生物检测和基因分型等领域;而电泳具有简单、易操作、成本低等优点,适用于DNA测序和基因型鉴定等领域。
taqman法SNP基因分型引物设计及Primer expressss教程

①Байду номын сангаас
②
3. 之后,上图中黑色区域会变为白色活动工作页面;将在NCBI中复制的FASTA格 式的序列全部粘贴进去。 ③因无法识别Y,该图标为灰色状态;在下一步
骤会激活
②
②注意,Y也会被粘贴进去,但是软件不会识别这种复制粘贴进去的Y,需跟随下 一个步骤,人工指定。
4. 人工指定SNP。选中上图中的Y,将其改为C①;再选中改好的C,此时,菜单栏 中的图标②会亮起,点击图标后,软件会要求使用者根据多态性选择另一个碱基 ③,本例中其多态性为T;点击T后,选中的字母C会再次变回Y。此时,完成了指 定SNP位点。
向3’端,但不可以放在最后两个碱基的位置上。
SNP位置 SNP位置必要时可以移向3’端
SNP位置最好在探针1/3-2/3间
但不可以在最后两个碱基
primer express软件设计SNP引物教程以rs2243106为例
• 1.SNP序列的获得 (1)在NCBI的SNP数据库(https:///snp/)中输入SNP 号,可以很方便的获得位点信息,但显示的序列较短,无法用于设计引物。 即:ACTATTGCTACACTCAGTTTCCCAA[C/T]TGCTCCTGTATAATCTTGTCTTCTG。如 下图所示:
①
5. 完成指定SNP位点后,会变回字母Y(红底),也就是指定SNP位点成功了。此 时,就可以点击“绿色播放”按钮①,软件就会自动设计上下游引物和探针。
6. 软件完成设计后,会生成下图结果报告。在candidate primers&probes中,可以 方便的选择引物对,选定之后,可以点击sequence标签,查看所在的位置,或者 点击order标签内底部的export,可以方便的输出,以便复制粘贴,用于合成引物。
taqman基因分型原理

taqman基因分型原理Taqman基因分型原理引言:Taqman基因分型是一种常用的分子生物学技术,用于检测和分析DNA序列中的遗传变异,尤其是单核苷酸多态性(SNP)。
该技术利用了Taqman探针的特性,通过荧光信号的定量测量来确定基因型。
本文将介绍Taqman基因分型的原理、步骤及其应用。
一、Taqman探针的构成和特性:Taqman探针由三个部分组成:荧光染料(如FAM、VIC等)标记的探针序列、与目标序列互补的引物以及含有非荧光的猝灭探针序列。
在无目标序列存在时,猝灭探针与引物结合,导致荧光信号被猝灭;而在目标序列存在时,引物与目标序列结合,猝灭探针无法结合,荧光信号得以释放。
这种荧光信号的变化可以通过PCR扩增过程中的实时荧光定量测量。
二、Taqman基因分型的步骤:1. DNA提取:从样本中提取DNA,并对其进行纯化处理,以消除潜在的污染和抑制因素。
2. PCR反应:将待分型的DNA样本与引物和Taqman探针一起加入PCR反应体系中。
PCR反应过程中,引物与目标序列特异性结合,Taq DNA聚合酶将引物向前延伸,同时Taqman探针也结合在目标序列上。
3. 荧光信号检测:在PCR反应过程中,荧光信号会随着PCR产物的增加而增加。
通过荧光定量PCR仪器实时监测PCR反应过程中的荧光信号,可以获得实时的PCR产物数量。
4. 数据分析:根据荧光信号的变化情况,可以确定每个样本的基因型。
通过设定阈值,可以将样本分为阳性和阴性。
同时,根据荧光信号的相对强度,可以判断所检测的SNP位点的基因型。
三、Taqman基因分型的应用:Taqman基因分型技术广泛应用于遗传病的研究、药物反应性研究、个体差异研究等领域。
例如,在遗传病研究中,Taqman基因分型可用于检测与疾病相关的SNP位点,从而确定个体携带的风险基因型。
在药物反应性研究中,Taqman基因分型可以帮助确定个体对特定药物的代谢能力,从而指导个体化用药。
荧光str法

荧光str法荧光str法是一种高通量的DNA分型技术,用于分离、鉴定和定量DNA。
该技术广泛用于基因组学研究、遗传学研究、疾病诊断以及种系追溯等领域。
荧光STR法的原理是基于PCR扩增后的DNA片段长度差异,通过荧光标记区别不同的片段,再利用荧光探针进行荧光标记,用荧光检测仪对不同位点的荧光信号进行检测,从而分离鉴定DNA序列。
该技术主要应用于分离指纹基因和DNA遗传标记,可以成功地确定DNA提取物的来源和个体之间的关系。
该技术需要使用多个PCR反应,每个反应都包含一个引物对和一个荧光探针对,每个探针标记一个不同的颜色。
PCR扩增后的产物经过电泳分离,荧光探针发出荧光信号进行检测。
每个PCR反应检测的结果就是一个STR位点的多样性,可以根据这些位点的不同组合来鉴定个体或样品的DNA特征。
荧光STR法与传统的STR方法相比,具有以下优点:1.高通量性:荧光STR法能够同时检测多个位点,因此可以对大量的样品进行快速的鉴定和分析。
2.高灵敏度:荧光探针的标记能够增强检测灵敏度,使得荧光STR法在样品低浓度下也能够进行准确的分析。
3.高准确性:荧光STR法具有高度的鉴定准确性,在进行人类遗传学分析和生物学样品分析时具有重要的应用价值。
4.自动化程度高:荧光STR法可以进行高度自动化操作,并且可以快速处理大量的样品,减少操作时间和劳动力成本。
荧光STR法的应用包括以下领域:1.人类遗传学分析:荧光STR法可以用于人类个体DNA的鉴定和分析,包括亲子关系鉴定、婚姻和家族关系鉴定、犯罪嫌疑人DNA鉴定等。
2.生物学分析:荧光STR法可以用于分析动物和植物的DNA序列,以确定基因型和个体之间的遗传关系。
3.医学研究:荧光STR法可以用于疾病诊断和治疗,包括肿瘤DNA诊断、遗传疾病诊断等。
4.食品安全与环境监测:荧光STR法可以用于检测食品和环境中的微生物、细菌等,以确定其种类和数量。
总之,荧光STR法是一种高度自动化、高通量、高灵敏度和高准确性的DNA分型技术,广泛应用于生物学、医学、食品安全和环境监测等领域。
基于TaqMan-TAMRA探针技术的ApoE基因快速分型方法研究

基于TaqMan-TAMRA探针技术的ApoE基因快速分型方法研究摘要】目的建立基于TaqMan-TAMRA探针技术的快速、准确的载脂蛋白E(ApoE)基因型检测方法,为临床诊断、治疗和预防ApoE基因相关疾病提供依据。
方法对1049例血液样本提取DNA后,利用TaqMan-TAMRA探针对ApoE基因进行检测和分型,并利用测序法进行验证。
结果 1049例样本中,共检出1048例,ApoE基因的6种基因型(ε3/ε3、ε2/ε3、ε3/ε4、ε2/ε4、ε2/ε2、ε4/ε4)所占比例分别为68.64%、13.54%、14.97%、1.72%、0.76%、0.38%。
TaqMan-TAMRA探针法分型结果与测序结果完全一致。
结论 TaqMan-TAMRA探针法操作简便,分型结果准确度高,是一种适用于临床的ApoE基因快速分型方法。
【关键词】载脂蛋白ETaqMan-TAMRA基因分型测序【中图分类号】R319 【文献标识码】A 【文章编号】2095-1752(2013)35-0132-02Development of a fast genotyping forApoEbased on TaqMan-TAMRAprobes【Abstract】Objective:To establish a method for fast and accurategenotyping ApoE with TaqMan-TAMRA probes and to support the clinical diagnosis、treatment and prevention.Methods:DNA was extracted from 1049 blood samples, then detected and genotyped with TaqMan TAMRA probes. The genotypes were compared with sequencing results. Results: DNA were detected in 1048 out of 1049 samples.Of which, six genotypes of ApoE gene (ε3/ε3, ε2/ε3, ε3/ε4, ε2/ε4, ε2/ε2, ε4/ε4) accounted for 68.64%, 13.54%, 14.97%, 1.72%, 0.76%, 0.38%.TaqMan-TAMRA probe genotypes were consistent with sequencing results. Conclusion:TaqMan-TAMRA is a fast and accurate method for genotyping ApoE.【Key words】 Apolipoprotein E TaqMan-TAMRA genotyping sequencing 载脂蛋白(ApoE)基因位于人的第19号染色体,编码由299个氨基酸组成的ApoE蛋白,该蛋白具有脂蛋白转运、代谢及修复细胞膜等功能[1,2]。
分子生物学第五章课后思考题答案【修订版】

分子生物学第五章作业1、哪些重要的科学发现和实验推动了DNA重组技术的产生及发展?答:近半个世纪来,分子生物学主要取得了三大成就:第一,20世纪40年代确定了遗传信息的携带者,即基因的分子载体是DNA而不是蛋白质,解决了遗传的物质基础问题;第二,50年代提示了DNA分子的双螺旋结构模型和半保留复制机制,解决了基因的自我复制和世代交替问题;第三,50年代末至60年代,相继提出了“中心法则”和操纵子学说,成功地破译了遗传密码,充分认识了遗传信息的流动和表达。
但事实上,DNA分子体外切割与连接技术及核苷酸序列分析技术的进步直接推动了重组DNA技术的产生和发展。
其中,限制性内切核酸酶和DNA连接酶等工具酶的发现和应用是现代生物工程技术史上最重要的事件。
DNA重组技术的产生及发展过程中比较重要的科学发现和实验如下:1957年A.Kornberg从大肠杆菌中发现了DNA聚合酶I。
1965年S. W. Holley完成了酵母丙氨酸tRNA的全序列测定;科学家证明细菌的抗药性通常由"质粒"DNA所决定。
1967年年世界上有五个实验室几乎同时宣布发现了DNA连接酶。
1970 年H.O.Smith,K.W.Wilcox和T.J.Kelley分离了第一种限制性核酸内切酶。
H.M.Temin和D.Baltimore从RNA肿瘤病毒中发现反转录酶。
1972-1973 年H.Boyer,P.Berg等人发展了DNA重组技术,于72年获得第一个重组DNA分子,73年完成第一例细菌基因克隆。
1978 年首次在大肠杆菌中生产由人工合成基因表达的人脑激素和人胰岛素。
1981 年R. D. Palmiter和R. L. Brinster获得转基因小鼠;A. C. Spradling和G. M. Rubin得到转基因果蝇。
1982 年美、英批准使用第一例基因工程药物--胰岛素;Sanger等人完成了入噬菌体48,502bp全序列测定。
hpv基因分型的原理

hpv基因分型的原理HPV(人乳头瘤病毒)是一种常见的性传播病毒,它可以通过性接触传播给他人。
目前已发现有超过200种不同的HPV型别,其中约40种可以感染人类生殖系统,引起各种疾病,包括生殖器疣和各种癌症。
对于HPV感染的分析和诊断,基因分型是非常重要的一个方面。
基因分型是指通过检测HPV基因组中特定的基因序列,来确定感染者所携带的HPV型别。
这种技术可以通过PCR(聚合酶链反应)方法,将DNA样本中的HPV基因扩增,然后使用特异性引物和荧光探针进行检测。
通过分析PCR产物的荧光信号,可以确定感染者体内存在哪些HPV型别。
HPV基因分型的原理是基于HPV的基因组结构和序列差异。
HPV的基因组由大约8个开放阅读框(ORF)组成,其中E6和E7基因是与致癌相关的关键基因。
这两个基因的序列在不同的HPV型别之间有明显的差异,因此可以作为基因分型的标志。
在进行HPV基因分型时,首先需要提取患者样本中的DNA。
可以使用不同的方法,如酚-氯仿法或商用基因提取试剂盒,从细胞中提取DNA。
提取的DNA样本经过定量和质量检测后,可以用于后续的PCR 反应。
PCR反应是基因分型的关键步骤。
通过选择特异性引物和荧光探针,可以扩增和检测特定的HPV基因序列。
引物的设计需要考虑到不同HPV型别的序列差异,以确保扩增的特异性。
荧光探针则用于检测PCR反应产物的荧光信号。
PCR反应一般分为两个步骤:扩增和检测。
扩增步骤通过一系列的温度变化,使引物与DNA结合并扩增目标序列。
检测步骤则使用荧光探针,通过荧光信号的变化来确定PCR产物中是否存在特定的HPV型别。
在进行HPV基因分型时,一般会选择多重PCR反应。
这种方法可以同时检测多种HPV型别,提高分型的准确性和效率。
多重PCR反应通常使用多对引物和多个荧光探针,以区分不同的HPV型别。
完成PCR反应后,可以通过荧光信号的分析来确定感染者所携带的HPV型别。
不同的HPV型别会产生不同的荧光信号模式,通过与已知的HPV基因型别进行比对,可以确定感染者体内存在哪些HPV型别。
KASP-基于已知SNP的高通量基因分型

KASP-基于已知SNP的⾼通量基因分型KASP——基于已知SNP的⾼通量基因分型1、什么是SNP?SNP,即单核苷酸多态性,是指在基因组⽔平上由单个核苷酸的变异⽽引起的⼀种DNA序列多态性。
SNP研究具有⼴泛意义,在农业领域中,可以进⾏性状基因的精细定位、分⼦辅助育种、种⼦资源鉴定等;在医学领域中,则主要是疾病的分⼦遗传机制研究、疾病基因定位、药物敏感或疾病易感性位点筛选等。
2、SNP研究的内容有哪些?SNP的研究主要分为SNP的发现及SNP的基因分型。
SNP的发现是应⽤的基础,需要在⼀定数量样本的全基因组范围内,经过统计学分析得到少量可能与疾病或性状关联的SNPs。
这时基因芯⽚或NGS(Next Generation Sequencing,第⼆代测序技术)技术在单个样品的⼤量SNP检测中具有优势。
SNP的基因分型是应⽤的技术⼿段。
这⼀过程需要检测⼤量样本的少量SNPs,⽽基因芯⽚或NGS技术则由于其⾼成本不太适和此阶段的研究。
KASP(Kompetitive Allele Specific PCR,竞争性等位基因特异性PCR)因其经济灵活的特点,作为国际上主流SNP检测⽅法之⼀,替代传统的⾼通量测序,在SNP分型研究中发挥重要作⽤。
3、KASP技术原理便宜⽽准确的基因SNP分型⽅法⼀直是遗传学家追求的⼿段。
KASP⽅法利⽤通⽤荧光探针,就可以通过简单的touch-down PCR的⽅法实现SNP分型。
含有SNP的单位基因-1和单位基因-2作为模板;针对等位基因SNP位点设计的两个正向引物和⼀个通⽤反向引物;每条正向引物尾部有特异性序列,可与荧光标记结合。
第⼀轮PCR,能够和模板互补的正向引物得到延伸,⽆法和模板互补的正向引物⽆法延伸;第⼆轮PCR,正向引物互补的特异性序列得以延伸,这步完成把通⽤标签序列引⼊与SNP对应的PCR产物。
随着PCR循环数增加,扩增⼦数量呈指数增长,荧光探针更多地退⽕到新合成的互补链上,发出荧光。
血型基因分型

血型基因分型1.血型基因分型技术由于绝大多数血型多态性都表现为SNP,因此检测SNP 的分子生物学技术基本上都适用于血型基因分型。
目前已报告的方法基本上都是以PCR为基础,所不同的只是在于PCR 引物序列以及检测PCR产物方法而异。
常用方法包括:①PCR-序列特异性引物(PCR-SSP)分型。
目前大部分红细胞血型基因分型,以及血小板HPA基因分型、粒细胞HNA 基因分型、白细胞HLA基因分型均可采用此技术。
②荧光标记PCR-SSP分型。
使用实时PCR扩增仪,结合使用荧光标记的SSP引物,可自动记录分析结果。
此方法主要被用于胎儿血型的产前鉴定。
③PCR-序列特异性寡核苷酸探针(PCR-SSOP)分型。
此方法通过与序列特异性寡核苷酸探针(SSOP)的杂交来鉴定相应基因。
④Luminex流式荧光技术。
在此方法中SSOP探针和微球耦联,1次PCR反应最多可同时检测100多种指标。
⑤PCR-测序分型(PCR-SBT)。
测序分型可以提供精确的DNA碱基序列信息,并有可能发现新的等位基因,使用最普遍的是Sanger双脱氧终止法。
由于PCR-SBT技术同时检测2条单体型的核酸序列,因此可能产生模棱两可分型结果。
⑥第2代测序技术。
第2代测序技术可以检测单独1条单体型核酸序列,因此用于HLA基因分型可以解决分型结果模棱两可的问题。
⑦基因克隆分型。
使用PCR扩增和分子克隆技术,将待测基因克隆到载体后再测序,每个克隆只含有1条单体型的基因片段,可用于确认新发现的等位基因。
2.血型基因分型的应用(1)疑难血型鉴定:对红细胞直接抗球蛋白试验阳性或具有多凝集作用的血液样品鉴定血型,通常都会遇到麻烦。
对于新近输血患者或多次输血患者做血型鉴定,由于外周血液中含有供者的血液,使用常规凝集试验不能正确鉴定患者血型,在这些情况下可以通过基因检测鉴定血型。
(2)无效型鉴定:使用血清学方法检测无效型个体,一般都是在患者已经产生抗体后才能被发现。
SNP分型

1、测序方法这个是最原始的,也是最简单的(操作简单,工作量不小啊!)一种方法,估计各位都明白,就不再累述了!2、Taqman探针法Taqman探针法可是一种无敌的方法,可用于基因荧光定量分析、甲基化检测、SNP分型、miRNA定量分析等等,差不多只要能用到的分子检测技术都有Taqman的身影。
Taqman探针法SNP分型技术的核心就是特异性的MGB探针技术,已证实的Taqman探针,3·段结合MGB技术,能更好的进行等位基因的区分。
MGB分子结合到DNA螺旋小沟,通过稳定MGB探针/模板联合体提高杂交的检验。
这种超强的稳定性可使短至13个碱基的探针提高错配的辨别力,并为困难多变的序列设计提供更高的灵活性。
所有的MGB探针都包括一个不发荧光的猝灭基团(NFQ),它能真正消除传统猝灭基团产生的背景荧光,提高信噪比,从而提供检测灵敏性。
Taqman探针法还有个好处据说AB公司能提供数以万计的现成探针,只要你付得起钱就可以了,什么可将illumina SNP芯片上的所有SNP位点都检测一遍。
Taqman探针法的缺点就是探针购买太贵,一般他们的试剂盒是1500人份的,价格可以咨询AB公司在国内的总代!1000份左右标本,几个位点用Taqman还是蛮核算的。
3、Beckman SNP genomestream技术Beckman技术的特点就是单碱基延伸法,设计时每个位点共设计3条引物,2条是扩增引物,1条为单碱基延伸引物(这个引物也可理解为探针)。
现在Beckman探针法可做到48重(也就是一次检测48个SNP 位点),引物及探针的设计可以直接提交给Beckman(这点同Taqman,不赚你,赚谁啊!),他们帮你有偿设计啊!Beckman的技术比较适合恰好有12个位点,48个位点检测,假如你的只有几个位点,或者恰巧不是12或48的倍数时,这个就有些不合算了!另外标本量不能太低,Beckman检测采用384板,一两百个样本那就算了吧!4、SNPshotSnaPshot技术平台是Applied Biosystems,ABI公司推出了专为检测SNP 设计的分析软件和试剂盒可对多个SNP 位点同时进行基因分型,也被称为minisequencing 。
hla分型鉴定方法

hla分型鉴定方法HLA分型鉴定方法介绍HLA(人类白细胞抗原)分型是研究人类免疫系统遗传多样性的重要手段,也是组织配型和移植匹配的关键依据。
本文将介绍几种常用的HLA分型鉴定方法。
方法一: PCR-SSP法(聚合酶链式反应-特异性引物扩增法)•基本原理:利用特异性引物扩增HLA基因区域,通过产物大小来判断HLA型别。
•优点:简单、快速、经济实惠。
•缺点:只能分析特定的HLA位点,结果可能存在假阳性或假阴性。
方法二: SBT法(序列特异性引物法)•基本原理:通过PCR扩增HLA基因区域后,应用生物信息学方法对扩增片段进行测序,进而确定HLA分型。
•优点:高分辨率、灵敏度高,可获得更详细的HLA型别信息。
•缺点:复杂度高、耗时长、成本较高。
方法三: SSOP法(特异性寡核苷酸探针法)•基本原理:利用标记的寡核苷酸探针与特定HLA位点进行杂交,通过探针的结合情况确定HLA型别。
•优点:具有较高的分辨率和特异性,无需测序。
•缺点:多个探针之间的互作用可能导致结果解读困难,技术要求较高。
方法四: NGS法(高通量测序法)•基本原理:借助新一代测序技术对HLA基因进行全基因组的测序,通过后续的生物信息学分析得到HLA分型结果。
•优点:高分辨率、高通量,可同时分析多个HLA位点。
•缺点:设备和运维成本高,数据分析复杂。
方法五: SSP法(特异性扩增PCR法)•基本原理:利用特异性引物扩增HLA基因区域,通过产物大小和特定引物的扩增结果判断HLA型别。
•优点:简单、节省成本,适用于常见位点的分型。
•缺点:分辨率较低,无法覆盖所有HLA位点。
结论每种HLA分型鉴定方法都有其特点和适用范围,在实际应用中需根据具体需求进行选择。
随着技术的不断进步,基于生物信息学的测序方法将成为未来的发展方向,为人类免疫系统遗传多样性的研究提供更准确的数据基础。
hla基因分型检测流程

hla基因分型检测流程下载温馨提示:该文档是我店铺精心编制而成,希望大家下载以后,能够帮助大家解决实际的问题。
文档下载后可定制随意修改,请根据实际需要进行相应的调整和使用,谢谢!并且,本店铺为大家提供各种各样类型的实用资料,如教育随笔、日记赏析、句子摘抄、古诗大全、经典美文、话题作文、工作总结、词语解析、文案摘录、其他资料等等,如想了解不同资料格式和写法,敬请关注!Download tips: This document is carefully compiled by theeditor. I hope that after you download them,they can help yousolve practical problems. The document can be customized andmodified after downloading,please adjust and use it according toactual needs, thank you!In addition, our shop provides you with various types ofpractical materials,such as educational essays, diaryappreciation,sentence excerpts,ancient poems,classic articles,topic composition,work summary,word parsing,copy excerpts,other materials and so on,want to know different data formats andwriting methods,please pay attention!HLA 基因分型检测是一种用于确定个体人类白细胞抗原(HLA)基因型的技术。
HLA-B5801 等位基因荧光探针PCR介绍+说明书

本试剂盒适用于 Applied Biosystems Prism 7000/7300/7500 及 Stratagene Mx3000p/3005p,整个反应约需 50 分钟。 【样本要求】 1. 血液样本采集
同时在荧光聚合酶链反应试剂中,也加入了另外一对内部对照组的核酸引物及探针,进行一常存基因——囊胞 纤维膜基因的核酸片段放大侦测,其目的在于证明聚合酶链锁反应的正常进行,当样本中的人类主要组织相容性抗 原分子核酸片段被复制放大时,内部对照组基因核酸片段将有可能因放大效率的差异而受到抑制。 【主要组成成分】 1. 分型反应 8 连管 10 排,每个检测需 3 管(MIX1,MIX2,MIX3): MIX-1,MIX-2,MIX-3 反应管中包含测定 B5801 所需的核酸引物和探针及内对照引物和探针。 注:8 连管上标记红色依次为 MIX1,MIX2,MIX3,同排空一管后依次为 MIX1,MIX2,MIX3,如下图所示:
重新测定 注2
1. 本品仅用于科学研究; 2. 需具有经验和专门培训过的人员进行操作; 3. PCR 操作各阶段应在不同的实验室中进行; 4. PCR 操作每个阶段使用专用的仪器和设备; 5. 穿工作服,戴一次性手套; 6. 必须使用一次性吸头,吸完即弃; 7. 用 10%次氯酸或 70%酒精或紫外线灯处理工作台和加样器; 8. 仅严格遵守本说明书规范操作所得到的测试结果为有效测试数据; 9. 本试剂盒所有使用的仪器设备包括荧光 PCR 仪及可调式移液器等均需依照规定进行校正; 10. 实验中使用的 Taq 核酸聚合酶(DNA polymerase) 以 Fermentas 厂牌为标准,使用其它厂牌之 Taq 核酸聚合酶可
用探针法进行基因分型的方法.doc

用非标记探针、遮蔽技术及高分辨率熔解扩增子分析对RET原癌基因进行基因分型RET原癌基因单个碱基的突变可以引发多发性内分泌腺瘤2型。
RET突变传统的基因分型方法是外显子测序。
一种闭管操作的基因分型方法已经成熟,此方法用的是一种饱和DNA 染料,非标记探针及高分辨率熔解扩增子分析。
此方法需要两个连续的聚合酶链式反应阶段,主要的和第二次实验。
主要的实验共用7个反应和8个非标记探针分析RET基因外显子10、11、13、14和16 。
主要实验基因分型了野生型外显子,外显子13普通的多态性,外显子16的一个突变及其它检测到的序列变化。
主要非标记探针数据限制检测到的RET基因序列突变的基因分型,这些突变位点的基因分型在第二次实验的2-5个反应中进行。
设计6条探针,所用方法是:用遮蔽技术和遮蔽选择的序列变化使探针下其它位置的突变的分析变得明确。
这两步RET基因分型之后,少于实验0.2%的外显子需要测序确定基因型。
对5个野生型和29个可用的RET序列突变样品(与测序结果100%一致)做盲研究。
用非标记探针和遮蔽技术进行高分辨率熔解分析是快速、精确的基因分型方法,>50的RET序列突变可以进行基因分型。
多发性内分泌腺瘤2型(MEN2)综合症,MEN2A,MEN2B及家族性甲状腺髓样癌由生殖细胞系RET基因外显子10、11、13和16的突变引发。
MEN2综合症是常染色体显性秩序失调引起的高生命危险的甲状腺癌。
RET突变的遗传学检测可以确认处于甲状腺癌危险中但是没有爆发癌症的病人,此时切除甲状腺可以增加存活的机率。
之前有许多实验方法检测或基因分型RET突变,比如说单链构象多态性,变性梯度凝胶电泳,温度梯度毛细管电泳,限制性内切酶消化PCR产物,焦磷酸测序,荧光标记杂交探针及微卫星。
但是,这些方法中的大多数需要PCR反应之后的操作来检测突变。
其中一些方法报道突变检测的敏感性为95%或少于95%,或者仅实验了RET突变样品的一小部分。
终点荧光法等位基因分型

终点荧光法等位基因分型
终点荧光法是一种用于等位基因分型的技术,它通常用于分析DNA中的单核苷酸多态性(SNP)。
在终点荧光法中,DNA样本首先经过PCR扩增,然后使用特定的荧光探针进行检测。
这些荧光探针与待检测的等位基因序列特异性结合,形成荧光信号。
通过检测荧光信号的强度和颜色,可以确定样本中的等位基因类型。
终点荧光法的优点之一是其高度特异性和灵敏度,能够准确地区分不同的等位基因。
此外,该技术具有高通量性和自动化程度高的特点,适用于大规模样本的快速分型。
在进行终点荧光法等位基因分型时,需要考虑以下几个方面:
1. 实验设计,包括PCR引物的设计、荧光探针的选择等。
2. 样本处理,DNA提取和纯化是确保实验成功的关键步骤。
3. 数据分析,对荧光信号的解读和等位基因类型的确定需要进行严格的数据分析和比对。
终点荧光法等位基因分型在遗传学研究、疾病诊断和药物反应等领域具有广泛的应用。
通过对个体的等位基因类型进行分型,可以帮助科研人员和临床医生更好地理解遗传变异与个体特征之间的关系,为个性化医疗提供重要依据。
同时,终点荧光法等位基因分型也为基因组学研究提供了高效、可靠的技术手段。
基因分型方法总结与比较
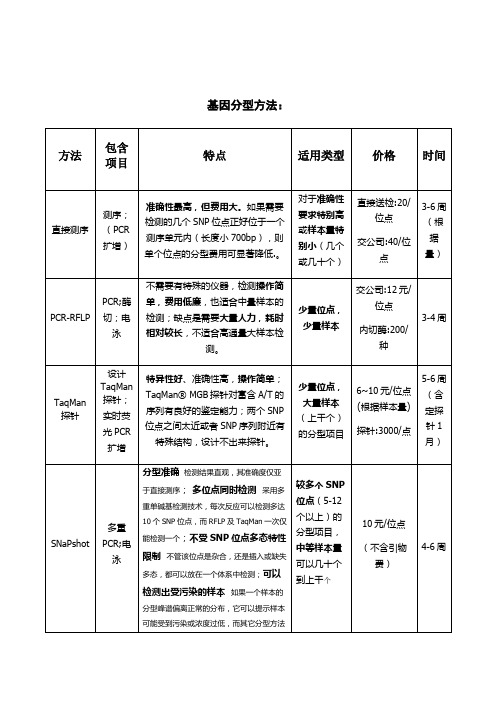
若有10多个位点可估6元/位点
4-6周
LDR(连接酶检测反应)
多重PCR;多重LDR连接反应;电泳
检测快速,由于检测条件更易控制最大优点就是实验时间短;通用性强;普遍适用,不论样本量或位点数的多少,适用于各种SNP的研究方案。而Taqman方法探针标记成本高,对样本量有一定要求;DNA芯片仅适合大量SNP位点的研究。
PCR-RFLP
PCR;酶切;电泳
不需要有特殊的仪器,检测操作简单,费用低廉,也适合中量样本的检测;缺点是需要大量人力,耗时相对较长,不适合高通量大样本检测。
少量位点,少量样本
交公司:12元/位点
内切酶:200/种
3-4周
TaqMan探针
设计TaqMan探针;实时荧光PCR扩增
特异性好、准确性高,操作简单;TaqMan® MGB探针对富含A/T的序列有良好的鉴定能力;两个SNP位点之间太近或者SNP序列附近有特殊结构,设计不出来探针。
少量位点,大量样本(上千个)的分型项目
6~10元/位点(根据样本量)
探针:3000/点
5-6周(含定探针1月)
SБайду номын сангаасaPshot
多重PCR;电泳
分型准确检测结果直观,其准确度仅亚于直接测序;多位点同时检测采用多重单碱基检测技术,每次反应可以检测多达10个SNP位点,而RFLP及TaqMan一次仅能检测一个;不受SNP位点多态特性限制不管该位点是杂合,还是插入或缺失多态,都可以放在一个体系中检测;可以检测出受污染的样本如果一个样本的分型峰谱偏离正常的分布,它可以提示样本可能受到污染或浓度过低,而其它分型方法则不能做到那样。
基因分型方法:
方法
包含项目
mbse中rflp中物理实现方法

mbse中rflp中物理实现方法MBSE(Model-Based System Engineering)是一种基于模型的系统工程方法,它通过建立系统模型来描述和分析复杂系统的行为、功能和结构。
RFLP (Restriction Fragment Length Polymorphism)是一种用于基因分型和基因诊断的分子生物学技术。
本文将介绍在MBSE中如何使用RFLP的物理实现方法。
RFLP的物理实现方法涉及以下几个主要步骤:1. DNA提取与切割:首先需要从样本中提取目标DNA,并对其进行切割。
一般采用限制性内切酶来切割DNA,酶切的位置取决于目标基因的序列。
2. 凝胶电泳:将DNA经过切割后的片段通过凝胶电泳进行分离。
凝胶电泳是一种利用电场在凝胶基质中分离DNA的技术。
较短的DNA片段会在凝胶中移动得更快,而较长的DNA片段则会移动得更慢。
3. 转移和固定:将凝胶上的DNA片段转移到一个固体基质(如纸或膜)上,以固定DNA片段在凝胶上的位置。
4. 探针杂交:在固体基质上添加一段带有特定标记的DNA探针。
探针的序列应与目标DNA的特定区域相互补充。
如果目标DNA中存在与探针互补的序列,探针将与目标DNA结合。
5. 探针检测:使用特定的检测方法来检测与探针结合的目标DNA片段。
常用的方法包括放射性同位素标记、酶标测定和荧光标记等。
在MBSE中,可以利用系统建模工具来支持RFLP的物理实现方法。
以下是在MBSE框架中应用RFLP物理实现的几个关键步骤:1. 建立系统模型:根据要分析的系统的需求和功能,创建系统模型。
模型应包括包括DNA提取与切割、凝胶电泳、转移和固定、探针杂交和探针检测等步骤的子模型。
2. 描述模型的输入和输出:为每个子模型定义输入参数和输出结果。
例如,在DNA提取与切割子模型中,输入参数可以是样本信息和限制性内切酶的切割位点,输出结果可以是切割后的DNA片段。
3. 建立模型之间的关系:通过使用系统建模工具提供的关系建立功能,将各个子模型与主模型连接起来。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
用非标记探针、遮蔽技术及高分辨率熔解扩增子分析对RET原癌基因进行基因分型RET原癌基因单个碱基的突变可以引发多发性内分泌腺瘤2型。
RET突变传统的基因分型方法是外显子测序。
一种闭管操作的基因分型方法已经成熟,此方法用的是一种饱和DNA 染料,非标记探针及高分辨率熔解扩增子分析。
此方法需要两个连续的聚合酶链式反应阶段,主要的和第二次实验。
主要的实验共用7个反应和8个非标记探针分析RET基因外显子10、11、13、14和16 。
主要实验基因分型了野生型外显子,外显子13普通的多态性,外显子16的一个突变及其它检测到的序列变化。
主要非标记探针数据限制检测到的RET基因序列突变的基因分型,这些突变位点的基因分型在第二次实验的2-5个反应中进行。
设计6条探针,所用方法是:用遮蔽技术和遮蔽选择的序列变化使探针下其它位置的突变的分析变得明确。
这两步RET基因分型之后,少于实验0.2%的外显子需要测序确定基因型。
对5个野生型和29个可用的RET序列突变样品(与测序结果100%一致)做盲研究。
用非标记探针和遮蔽技术进行高分辨率熔解分析是快速、精确的基因分型方法,>50的RET序列突变可以进行基因分型。
多发性内分泌腺瘤2型(MEN2)综合症,MEN2A,MEN2B及家族性甲状腺髓样癌由生殖细胞系RET基因外显子10、11、13和16的突变引发。
MEN2综合症是常染色体显性秩序失调引起的高生命危险的甲状腺癌。
RET突变的遗传学检测可以确认处于甲状腺癌危险中但是没有爆发癌症的病人,此时切除甲状腺可以增加存活的机率。
之前有许多实验方法检测或基因分型RET突变,比如说单链构象多态性,变性梯度凝胶电泳,温度梯度毛细管电泳,限制性内切酶消化PCR产物,焦磷酸测序,荧光标记杂交探针及微卫星。
但是,这些方法中的大多数需要PCR反应之后的操作来检测突变。
其中一些方法报道突变检测的敏感性为95%或少于95%,或者仅实验了RET突变样品的一小部分。
这些方法分析RET序列变化的限制范围,可能只以普通突变为目标而错过了稀有突变。
此外,不致病多态性的存在可以导致异常的结果。
因为这些限制,RET外显子测序保持黄金标准。
测序是一种耗时且昂贵的开管实验,需要生成PCR产物之后的额外操作。
相反,高分辨率熔解曲线的突变扫描分析是一种快速且便宜的闭管操作实验,不需要进行PCR产物之后的额外操作。
通过用一种饱和DNA染料:LC Green,高分辨率熔解曲线分析可以检测PCR扩增子之内的序列变化。
对检测PCR扩增子内杂合点突变的高分辨率熔解技术的系统研究发现,检测400bp之内的扩增子的敏感性和特异性是100%。
最近,我们证实用高分辨率熔解分析进行RET 外显子突变扫描。
虽然这种方法在盲实验中检测100%的突变,但是有些罕见的突变的熔解曲线相似,完全进行基因分型是困难的,在这种情况下,在最初的突变扫描之后需要测序。
另一种方法,添加杂交探针可以分出RET突变的绝大多数的基因型,而不需要测序。
荧光标记杂交探针分析是一种快速、闭管操作的实验,可以通过探针/等位基因的熔解温度基因分型序列变化,但是需要昂贵的寡核苷酸修饰。
非标记探针也可以通过Tm的不同进行基因分型,也是一种闭管操作的实验,需要LC Green染料。
因为用DNA饱和染料检测,非标记探针和扩增数据可以从一个熔解曲线分析。
Zhou等最近证实非标记探针基因分型分析可以补充突变扫描。
但是,在同一个外显子之内的RET所有突变不能只用一条探针进行基因分型,因为探针下特定突变的熔解温度相似或相同。
为了完成闭管基因分型实验,发明了两步RET基因分型方法,此方法比测序技术成本低,耗时少。
用于扩增熔解的第一个PCR阶段(主要实验),扫描RET外显子的序列变化,反之,非标记探针分析位于突变范围内,且分辨可能的基因型。
第二个PCR阶段(第二次实验)需要限定数目的特定突变的探针对>50的RET序列突变进行明确的基因分型。
用遮蔽技术设计的探针简化了突变分离。
当探针下有多重的可能的序列突变使分析变得复杂时,遮蔽技术简化了探针熔解的判断。
在多态位点选择含有错配的(缺失、不匹配碱基或一般碱基)可以遮蔽的序列变化。
这样的话就在遮蔽多态位点人为的创造了一个与可能的等位基因的错配。
野生型与遮蔽突变的等位基因都有一个与探针错配的碱基和相似的Tm。
然而,突变等位基因在探针下其它的位置有一个额外的错配,可以通过比仅在遮蔽位置变化的等位基因稍低的Tm值鉴定。
材料和方法样品此报道中用到的野生型RET基因的DNA基因组样品或有RET序列变化的样品在以前描述过。
RET基因序列变化改变RET蛋白的功能引发MEN2综合症的是突变,然而RET序列改变不会引发MEN2综合症是多态。
不能确定意义的稀有的或不会引起MEN2综合症的RET基因序列变化成为“序列变化”。
在这份报告中,通过密码子数列出RET序列变化,野生型编码DNA序列后面是编码序列改变,伴随着加粗突变核苷酸(例:618(TGC→T A C)),反之多态及序列变化用下划线标出。
所有测试的RET序列变化的样品均有单个核苷酸改变,并且是杂合的除非有另外的阐明。
RET突变基因型和多态性及序列变化基因型列于表1。
引物和探针用完整DNA技术合成寡核苷酸。
用Primer3设计引物。
选择引物和探针检测所有已知RET突变,不包括分析中的普通多态(表2)。
非标记探针包含有3’磷酸化或3’氨基修饰(整合DNA技术),避免PCR反应中的延伸。
设计的探针用于分析每个外显子的突变热点。
RET基因分型实验中用了4种非标记探针(表2):野生型(WT)探针,特定突变(MS)探针,只遮蔽单一多态核苷酸的遮蔽探针,遮蔽3个多态核苷酸的位置探针(一个密码子)。
比如说,外显子13遮蔽探针(遮蔽1bp 769)在多态核苷酸位置有单个碱基的缺失。
(WT13探针的“T”位置,表2)。
外显子10和11位置探针与野生探针序列相比有3个核苷酸缺失,遮蔽一个致病密码子分辨密码子突变的位置。
外显子10和11在致病密码子位置有同样的序列改变。
比如说:外显子10,MS618/620T A C探针在密码子618-620有T A C序列,如表2描述。
PCR用之前描述的不对称快速PCR扩增样品DNA,除了用表2中给出的引物浓度反应55个循环。
外显子14反应包括2个探针,0.5μmol/L的WT14A探针和0.25μmol/L的WT14B探针。
在主要实验和第二次实验一个野生型对照与每个探针一起参加反应。
外显子13, WT13主要的实验反应用2个额外的对照:杂合和纯合密码子769样品。
外显子10主要实验反应用620(TGC→A GC)突变样品作为正对照。
高分辨率熔解在高分辨率熔解仪器HR-1(爱荷华科技,盐湖城,犹他州)上进行分析,将LightCycle 毛细管加入HR-1中,0.3℃/s加热。
主要的实验中,RET外显子的扩增及非标记探针熔解数据从60℃到95℃采集。
主要实验分析了每个外显子的扩增子及探针数据,除了外显子14。
因为*序列变化是杂合的除非在文本上另作说明。
密码子DNA序列列出核苷酸改变,加粗突变且多态性或序列变化使加下划线的。
·,可用的RET序列变化样品。
†未知意义的稀有序列变化或不引发MEN2综合症;查看参考文献。
在RET致病密码子范围内密码子609或611的3个突变体与MEN2综合症没有联系。
密码子922和852的突变体以前在MTC病人上发现过,也许不会引发MEN2综合症。
‡良性多态性,0.26等位基因频率。
§806与804突变在同一等位基因时致病。
*最终引物的浓度和上下引的比例在引物序列上显示。
列出引物序列从5’到3’,上游引物在下游引物上部。
†每个探针集下是密码子的变化。
下划线密码子包括了多态或序列变化,反之其它密码子包括突变。
‡遮蔽,遮蔽1-3个核苷酸的缺失。
主要实验中的野生型探针序列用于每个外显子,除了外显子16。
如果两个野生型探针用于一个外显子,标记做A和B探针。
§探针序列从5’到3’显示,RET外显子10、13、14和16是上游探针,反之,外显子11是下游探针。
已知突变的位点加粗,可能的多态性或序列变化位点用下划线表示。
¶位置(L)探针与野生型探针序列致病密码子相比有3个核苷酸遮蔽缺失(---)。
‖用于外显子10,11突变的基因分型的MS探针的例子。
在探针内致病密码子位置的相同的特定突变序列。
这些探针有特定突变序列的名称,改变的序列加粗。
有7个可能的从TGC 野生型半胱氨酸到其他氨基酸的突变序列改变。
因此,21个MS探针中的7个特定突变探针(A GC,CGC,GGC,TA C,TCC,TTC,TGG)是用于外显子10和11突变。
用于每个突变基因分型的MS探针用图解法表示,表3和补充表格1-5。
**外显子13的主要和第二次试验用遮蔽探针。
遮蔽 1bp 769 主要探针及外显子特定突变探针在密码子769多态位置有单核苷酸缺失(缺失野生型探针序列有下划线的“T”)。
††外显子16用918(ATG-ACG)密码子特定突变探针做主要实验。
所有报道的外显子14突变都包含在非标记探针下,只分析了探针数据。
第二次实验,只分析了在60℃-89℃采集的非标记探针熔解的数据。
高分辨率熔解数据用之前描述由LabView编写的定制软件分析。
简言之,扩增子的熔解曲线数据是规范化的,温度转移之后转换成派生图或荧光差异图。
依据用于扩增分析的数据图,由于从野生型对照的视觉偏移的不同曲线形状较宽的派生熔解峰,独特形状的熔解峰(肩峰或两个峰),熔解温度的偏移,和/或荧光不同来检测序列变化。
扩增熔解曲线,派生图,荧光不同的图都可以用来检测RET序列变化。
非标记探针熔解数据直接转换成派生图。
外显子14探针数据标准化,在分析之前指数背景已消除。
在派生熔解峰的1/2出估计非标记探针数据的Tms。
杂合样品的△Tms定义为野生型等位基因Tm减去变化(突变,多态性,或序列变化等位基因)等位基因Tm的值。
注意:当变化等位基因的Tm值高于野生型的等位基因Tm(例如:和互补的特定突变探针),△Tms的值是否定的。
杂合样品的探针数据导致肩峰或平稳形状的峰,峰面积分为1/4,1/2和1/4野生型范围,变化等位基因Tms在这些区域的边界。
因为纯合子序列变化(没有野生型等位基因)或变化等位基因Tm值与野生型等位基因Tm值相同时(没有可识别的野生型等位基因)当只有单个峰是显而易见的。
之后,野生型对照样品Tm在计算△Tms时用于计算野生型等位基因Tm。
野生型样品也会导致单一的峰,所以,从野生型对照Tm减去样品的单个等位基因Tm值。
每个RET序列变化的主要探针△Tm值决定用于第二次基因分型实验的特定突变探针。
