引物设计基本方法Word版
如何设计引物 (2)

1.引物最好在模板cDNA的保守区内设计。
DNA序列的保守区是通过物种间相似序列的比较确定的。
在NCBI上搜索不同物种的同一基因,通过序列分析软件(比如DNAman)比对(Alignment),各基因相同的序列就是该基因的保守区。
2.引物长度一般在15~30碱基之间。
引物长度(primerlength)常用的是18-27bp,但不应大于38,因为过长会导致其延伸温度大于74℃,不适于TaqDNA聚合酶进行反应。
3.引物GC含量在40%~60%之间,Tm值最好接近72℃。
GC含量(composition)过高或过低都不利于引发反应。
上下游引物的GC含量不能相差太大。
另外,上下游引物的Tm值(meltingtemperature)是寡核苷酸的解链温度,即在一定盐浓度条件下,50%寡核苷酸双链解链的温度。
有效启动温度,一般高于Tm值5~10℃。
若按公式Tm=4(G+C)+2(A+T)估计引物的Tm值,则有效引物的Tm为55~80℃,其T m值最好接近72℃以使复性条件最佳。
4.引物3′端要避开密码子的第3位。
如扩增编码区域,引物3′端不要终止于密码子的第3位,因密码子的第3位易发生简并,会影响扩增的特异性与效率。
5.引物3′端不能选择A,最好选择T。
引物3′端错配时,不同碱基引发效率存在着很大的差异,当末位的碱基为A时,即使在错配的情况下,也能有引发链的合成,而当末位链为T时,错配的引发效率大大降低,G、C错配的引发效率介于A、T之间,所以3′端最好选择T。
6.碱基要随机分布。
引物序列在模板内应当没有相似性较高,尤其是3’端相似性较高的序列,否则容易导致错误引发(Falsepriming)。
降低引物与模板相似性的一种方法是,引物中四种碱基的分布最好是随机的,不要有聚嘌呤或聚嘧啶的存在。
尤其3′端不应超过3个连续的G或C,因这样会使引物在GC富集序列区错误引发。
7.引物自身及引物之间不应存在互补序列。
引物自身不应存在互补序列,否则引物自身会折叠成发夹结构(Hairpin)使引物本身复性。
【设计】NCBI引物设计方法

【关键字】设计Primer-Blast介绍Primer-BLAST,在线设计用于聚合酶链反应(PCR)的特异性寡核苷酸引物。
Primer-BLAST 可以直接从Blast主页(/)找到,或是直接用下面的链接进入:/tools/primer-blast/这个工具整合了目前流行的Primer3软件,再加上NCBI的Blast进行引物特异性的验证。
Primer-BLAST免除了用另一个站点或工具设计引物的步骤,设计好的引物程序直接用Blast进行引物特异性验证。
并且,Primer-BLAST能设计出只扩增某一特定剪接变异体基因的引物–an important feature for PCR protocols measuring tissue specific expression (注:没办法准确的翻译,只好作罢,汗!)。
Primer-BLAST有许多改进的功能,这样在选择引物方面比单个的用Primer3和NCBI BLAST更加准确。
Primer-BLAST的输入Primer-BLAST界面包括了Primer3和BLAST的功能。
提交的界面主要包括三个部分:target template(模板区), the primers(引物区), 和specificity check(特异性验证区)。
跟其它的BLAST一样,点击底部的“Advanced parameters”有更多的参数设置。
模板(Template)在“PCR Template”下面的文本框,输入目标模板的序列,FASTA格式或直接用Accession Number。
如果你在这里输入了序列,是用于引物的设计。
Primer-BLAST就会根据你输入的序列设计特异性引物,并且在目标数据库(在specificity check区选择)是唯一的。
引物(Primers)如果你已经设计好了引物,要拿来验证引物的好坏。
可以在Primer Parameters区填入你的一条或一对引物。
引物设计说明

引物设计说明(2009-05-13 09:35:28)转载标签:杂谈引物设计步骤:1.查阅自己所需要基因的研究状况。
用将各个相近或相关物种的氨基酸序列进行比对. /tools/clustalw2/index.hteml 书写格式例如:>portunus(物种名,只是一个标志)MVSRAHSRFSCQRTTLLAVVLLAVLWSSSLQQAAARVINDDCPNLMGNRDLYKKVEWICDDCANIYRSTGMASLCRKDCFFN EDFLWCVRATERSEDMMQLKQWVRILGAGRI>Cam MMSRANSRFSCQRTWLLSVVVLAALWSFGVHRAAARVINDECPNLIGNRDLYKKVEWICEDCSNIFRKTGMASLCRRNCFFNE DFVWCVHATERSEELRDLEEWVGILGAGRD>Cas MMSLAHSKFSCQRTRLLAVVLLAALWSSSLQQAAARVINDDCPNLIGNRDLYKKVEWICDDCANIYRSTGMASLCRKDCFFNE DFLWCVRATERSEDLAQLKQWVTILGAGRI>Cap MMSRTESRYSSQRTWLLSMVVLAALWSISVQRATARVINDDCPNLIGNRDLYKKVEWICEDCSNIFRNTGMATLCRKNCFFNE DFLWCVYATERTEEMSQLRQWVGILGAGRE>ChfMMSRANSRFSCQRTWLLAVVVLAAIWSSSLHQAAARVFNDDCPNLMGNRDLYKKVEWICDDCANIFRIPGMASICRKDCFFNE DFLWCVRATERTEEMMQLKQWVRILGAGRM>Mee A MYRMPMRFWLTAVVMVVVGALLLDTASASYIENTCRGVMGNRDIYKKVVRVCEDCTNIFRLPGLDGMCRDRCFNNEWFLVC LKAANRDDELDKFKVWISILNPGL>liv1 MYRLAMKTWLAIVIVVVGTSLFFDTTSASFIDGTCRGVMGNRDIYKKVVRVCEDCTNIFRLPGLDGMCRDRCFYNEWFLICLK AANREDEIEKFKVWISILNAGQ(碱基比对同理)2.根据比对结果,找出保守氨基酸即功能氨基酸的位置。
(完整word版)引物设计步骤与要点

引物设计step by step1、在NCBI上搜索到目的基因,找到该基因的mRNA,在CDS选项中,找到编码区所在位置,在下面的origin中,Copy该编码序列作为软件查询序列的候选对象。
2、用Primer Premier5搜索引物①打开Primer Premier5,点击File-New-DNA sequence, 出现输入序列窗口,Copy目的序列在输入框内(选择As),此窗口内,序列也可以直接翻译成蛋白。
点击Primer,进入引物窗口。
②此窗口可以链接到“引物搜索”、“引物编辑”以及“搜索结果”选项,点击Search按钮,进入引物搜索框,选择“PCR primers”,“Pairs”,设定搜索区域和引物长度和产物长度。
在Search Parameters里面,可以设定相应参数。
一般若无特殊需要,参数选择默认即可,但产物长度可以适当变化,因为100~200bp的产物电泳跑得较散,所以可以选择300~500bp.③点击OK,软件即开始自动搜索引物,搜索完成后,会自动跳出结果窗口,搜索结果默认按照评分(Rating)排序,点击其中任一个搜索结果,可以在“引物窗口”中,显示出该引物的综合情况,包括上游引物和下游引物的序列和位置,引物的各种信息等。
④对于引物的序列,可以简单查看一下,避免出现下列情况:3’不要出现连续的3个碱基相连的情况,比如GGG或CCC,否则容易引起错配。
此窗口中需要着重查看的包括:Tm 应该在55~70度之间,GC%应该在45%~55%间,上游引物和下游引物的Tm值最好不要相差太多,大概在2度以下较好。
该窗口的最下面列出了两条引物的二级结构信息,包括,发卡,二聚体,引物间交叉二聚体和错误引发位置。
若按钮显示为红色,表示存在该二级结构,点击该红色按钮,即可看到相应二级结构位置图示。
最理想的引物,应该都不存在这些二级结构,即这几个按钮都显示为“None”为好。
但有时很难找到各个条件都满足的引物,所以要求可以适当放宽,比如引物存在错配的话,可以就具体情况考察该错配的效率如何,是否会明显影响产物。
引物设计——精选推荐

引物设计⼀、模板序列确定注意:1.找到同源基因的蛋⽩氨基酸序列,⽐对,找到⾼度同源区域,再回到cDNA序列设计引物,核苷酸序列同源性⾼,就⽤核苷酸,否则⽤氨基酸,设计简并引物。
⼀般在mRNA序列的中间偏下游即靠3'端的部分序列设计引物(习惯问题)。
⼀般认为,如果蛋⽩的序列⼀致性为25-30%,则可认为序列同源。
2.简并引物的设计最好还是使⽤CDS序列来做⽐对,然后根据保守区设计简并引物,CDS两端的⾮翻译区在物种间变化太⼤,不适合设计引物。
把获得各个物种的CDS做成fasta格式的,序列最好处理成纯序列格式的不含其它任何字符。
然后⽤⽐对软件⽐对⼀下,如mega,DNAman或者是vectorNTI ⾥的⽐对⼯具都可以做⽐对。
在⽐对结果⾥找到⾮常保守的区域就可以设计简并引物了。
设计的简并引物可以根据经验公式⾃个算Tm值。
不⽤计较打分的问题。
因为你只能在保守区设计引物。
引物的3'端最好位于最保守的区域。
⼀般是寻找该基因的ORF框即编码区域作为该引物的保守序列。
你可以⽤软件查找ORF框,找⼀个⽐较长的位置在序列中下游的区域设计引物。
⼆、引物设计的原则⾸先引物要跟模板紧密结合,其次引物与引物之间不能有稳定的⼆聚体或发夹结构存在,再次引物不能在别的⾮⽬的位点引起DNA聚合反应(即错配)。
围绕这⼏条基本原则,设计引物需要考虑诸多因素,如引物长度(primerlength)、产物长度(product length)、序列Tm值(melting temperature)、ΔG值(internal stability)、引物⼆聚体及发夹结构(duplexformation and hairpin)、错误引发位点(false priming site)、引物及产物GC含量(composition),有时还要对引物进⾏修饰,如增加限制酶切点,引进突变等。
以使⽤Oligo 软件分析设计引物为例,笔者总结出以下的要点:1.引物的长度⼀般为15-30bp,常⽤的是18-27bp,但不能⼤于38,因为过长会导致其延伸温度⼤于74℃,即Taq 酶的最适温度。
PCR引物设计方法综述

PCR引物设计方法综述PCR引物设计方法综述PCR(聚合酶链反应)是一种重要的分子生物学技术,在基因工程、生物医学研究、疾病诊断和基因表达分析等领域得到广泛应用。
PCR引物设计是PCR实验成功与否的重要因素之一,它对PCR反应的特异性和效果有着关键影响。
本文将综述PCR引物设计的一些常见方法。
1. 引物的长度和碱基组成合适的引物长度一般在18-30个碱基对之间,过短可能导致非特异性扩增,过长则可能降低扩增效率。
此外,引物中碱基的组成也要考虑。
GC含量通常应在40-60%之间,因为GC碱基对的热稳定性较高,有助于提高PCR反应的效果。
2. 引物的Tm值Tm值(熔解温度)是引物设计中重要的参数,表示DNA链的熔解温度。
引物对应的Tm值应该相似,一般在50-65℃之间。
Tm值的计算可以使用公式Tm= 2(A + T) + 4(G + C),其中A、T、G和C分别表示引物中的腺嘌呤、胸腺嘧啶、鸟嘌呤和胞嘧啶的个数。
3. 引物的特异性引物的特异性是PCR引物设计的关键。
在设计引物时,必须确保引物与目标序列的互补区域没有重复序列或能够和其他非目标DNA序列结合。
可以使用计算机软件进行引物特异性检查,如BLAST等。
此外,设计引物时应考虑引物的位置,尽可能避免设计在重复序列上。
4. 引物的杂交温度引物的杂交温度是PCR反应的另一个重要因素,影响PCR引物与模板DNA的结合和扩增效率。
一般情况下,引物的杂交温度应在Tm值的2-5℃之间。
较高的杂交温度有助于提高特异性,但也可能降低引物与模板DNA的结合能力。
5. 引物的设计工具和软件现代生物信息学提供了许多实用的工具和软件用于PCR引物设计。
比如,Primer3软件是一种常用的引物设计工具,能够自动设计引物,计算引物的Tm值和特异性等参数。
其他常用的软件还包括OligoAnalyzer、Beacon Designer和GeneFisher 等。
6. 引物的修饰在一些特殊的PCR反应中,引物的修饰可以提高PCR扩增效率和特异性。
(完整word版)PCR引物流程设计详解
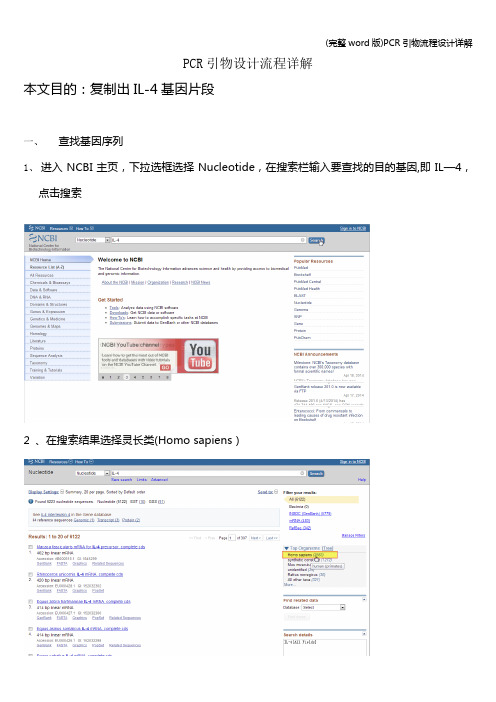
PCR引物设计流程详解本文目的:复制出IL-4基因片段一、查找基因序列1、进入NCBI主页,下拉选框选择Nucleotide,在搜索栏输入要查找的目的基因,即IL—4,点击搜索2 、在搜索结果选择灵长类(Homo sapiens)2、在灵长类IL-4基因中选择需要的mRNA序列3、查看基因的相关信息外显子区域CDs区域4、点击FASTA格式,并将序列保存到文档二、使用primer premier 5。
0设计引物1、建立新文件,将所得的序列复制进输入框内2、点击搜索按钮,搜索引物3、设置引物设计参数(因为在之前查找基因序列的时候获知,外显子区域分别为:1—200、201-248、249-425、426-618,又知在引物设计时引物位置最好跨越一个内含子,PCR产物长度通常为100—150bp,故设定上游引物位置为201—248,下游引物位置为249—425,产物长度为100-150bp)4、确认条件后,显示搜索结果4、双击选中得分最高的引物查看引物情况(上图为上游引物情况,下图为下游引物情况)5、将设计的上下游引物复制出来,保存到文档中三、使用oligo 6.0对设计的引物进行评价1、建立新文件,将从cnki上获得的cDNA复制进输入框,并点击accept接收2、接收后显示出该序列的相关信息3、点击edit按钮录入用primer设计的上游引物,每一次输入新数据后都需要点击accept按钮接收4、同理,录入下游引物5、分析上下游引物二聚体形成情况6、分析上下游引物发卡形成情况7、分析上下游引物GC%8、检测上下游引物与PCR模板其它位置错配情况9、分析PCR整体情况四、引物特异性检验(primer blast)1、进入NCBI主页,并选择blast2、选择primer blast3、在输入框内输入模板序列和上下游引物,并设定对比数据库,点击get primer进行对比4、查看blast结果Blast 结果显示,尽管IL—4与其它基因有相似区,但是引物的3’端没有完全互补。
设计RTPCR引物方法

设计RTPCR引物方法设计RT-PCR引物方法要鉴定某一个基因在mRNA水平上的表达,需要使用Q-PCR的方法。
引物设计是很重要的一环,要进行Q-PCR时,获得引物的途径有很多种,其中最为方便的是使用NCBI和primer bank,首先介绍这一方法:比如你要检测某药物对小鼠细胞中IL-2的表达影响,通过Q-PCR检测,要设计引物。
①登陆NCBI,选择gene,在选框中输入 IL-2 mus(搜索小鼠IL-2基因)点击搜索获得结果如下:选择第一个基因,打开,可以获得gene的ID利用primer bank和ncbi的gene ID就可以获得前人已经设计好的引物②登陆primer bank (),按下图选择NCBI gene ID 选择物种mouse ,在for text中输入前面获得的你要检测基因的ID (NCBI的编号)点击submit 查询,获得如下结果可以看到其它人设计的好几对引物,基本上是通过实验验证的,做Q-PCR的话直接拿来用就行了,点击绿色高亮的链接可以看到其他人使用这对引物做Q-PCR获得的结果。
二、自己设计Q-PCR引物方法比如要检测CXCR3的表达,做Q-PCR的扩增目的条带在100bp 左右,如果半定量的话(通过跑胶定量)扩增产物一般400bp左右①登陆NCBI,选择gene,在选框中输入 CXCR3 mus(搜索小鼠CXCR3基因)点击搜索点击打开将网页往下拖到NCBI reference sequence 选择箭头所示mRNA 序列打开链接往网页下方拖动,可以看到CDS,点击CDS(编码蛋白区)出现如下界面,高亮部分为CDS去ATG起始密码子TAA终止密码子复制CDS区及其附近部分序列,如果要提取基因的话需要完全包括CDS区,如果只是检测的话,比如半定量的话就没必要全部包括。
打开primer 5 软件,新建一个DNA序列将复制的序列粘贴到选项框中,点击oK序列就导入了primer 5中,然后单击箭头所指primer,进入引物设计界面界面如下,点击search,使用primer自动寻找引物功能进入界面后,分别按下图选取所需要的参数,PCR product size 使用默认值即可,primer length一般在20bp左右比较合适点击OK后进入如下界面,点击OK出现如下界面,点击rating,按打分排列,打分是100分制,分值越高引物越好,根据自己试验目的,选择打分高的,产物片段400bp左右的引物对,并且Tm温度最好在60以上,有义链和反义链Tm值靠的越近越好,最好不要超过2摄氏度,下图中Tm行中蓝色字体表示有义链的Tm值,红色字体表示反义链Tm值,PCR 退火温度一般设置比Tm值低5摄氏度。
PCR引物设计原理及方法

PCR引物设计基本思路1.根据实验需要,确定需要扩增的DNA序列,并知道其CDS区序列(编码结构基因区,即从起始密码子区至终止密码子区)ncbi网站查询RBS149..153/gene="eryF"CDS158..1372/gene="eryF"1ggatcccgat cgtgtcggag gaagaggcca agtcgcgccg ccccgaccag ctgctggtgc61tgccctggat ctaccgcgac gggttcgtcg aacgcgagca ggagttcctc gctggcggcg121gaaagctgat cttcccccta ccccgactgg aagtcgtatg acgaccgttc ccgatctcga181aagcgactcc ttccacgtcg actggtaccg cacctacgcc gagctgcgcg agaccgcgcc241ggtgacgccg gtgcgcttcc tcggccagga cgcgtggctg gtcaccggct acgacgaggc301gaaggccgcg ctgagcgacc tgcgcctgag cagcgacccg aagaagaagt acccgggcgt361ggaggtcgag ttcccggcat acctcggttt ccccgaggac gtgcggaact acttcgccac421caacatgggc accagcgacc cgccgaccca cacccggctg cgcaagctgg tgtcgcagga481gttcaccgtc cgccgcgtgg aggcgatgcg gccccgcgtc gagcagatca ccgcggagct541gctcgacgag gtgggcgact ccggcgtggt cgacatcgtc gaccgcttcg cccacccgct601gcccatcaag gtcatctgcg agctgctcgg cgtcgacgag aagtaccgcg gggagttcgg661gcggtggagc tcggagatcc tggtcatgga cccggagcgg gccgaacagc gcgggcaggc721ggccagggag gtcgtcaact tcatcctcga cctggtcgag cgccgccgca ccgagcccgg781cgacgacctg ctgtccgcgc tgatcagggt ccaggacgac gatgacggtc ggctcagcgc841cgacgagctg acctccatcg cgctggtgct gctgctggcc ggtttcgagg cgtcggtgag901cctcatcggg atcggcacct acctgctgct cacccacccg gaccagctcg cgctggtgcg 961gcgggacccg tcggcgctgc ccaacgccgt cgaggagatc ctgcgctaca tcgctccgcc 1021ggagaccacc acgcgcttcg ccgcggagga ggtggagatc ggcggtgtcg cgatccccca 1081gtacagcacg gtgctggtcg cgaacggcgc ggccaaccgc gacccgaagc agttcccgga 1141cccccaccgc ttcgacgtca cccgcgacac ccgcggccac ctgtcgttcg ggcagggcat 1201ccacttctgc atgggccggc cgctggccaa gctggagggc gaggtggcgc tgcgggcgct 1261gttcggccgc ttccccgctc tgtcgctggg aatcgacgcc gacgacgtgg tgtggcggcg 1321ttcgctgctg ctgcggggca tcgaccacct accggtgcgg ctcgacggat gagcacctgg 1381ctgcggcggt tcggtcctcc cgtcgagcac cgggcgcggc tggtgtgctt cccgcacgcg 1441ggagccgcgg ccgactccta cctcgacctc gcgcgcgcct tggcgcccga gatcgacgtg 1501cacgccgtgc agtacccggg gcgccaggac cgccgcgacg aggagcccct gggcaccgcc 1561ggcgagatcg ccgacgaggt ggccgccgtg ctgcgcgcgt cgggcggcga cggcccgttc 1621gccctgttcg ggcacagcat gggcgcgttg atcgcctacg agacggcgcg caggctcgaa 1681cgcgagcccg gcggcgggcc gctgcggctg ttcgtgtccg ggcagaccgc cccgcgcgtg 1741cacgagcgcc gcaccgacct gcccggcgac gacggtctgg tggacgagct gcgccggctc 1801ggcaccagcg aggcggcgct ggccgacgag gccctgctcg ccatgtcgct gccggtgctg 1861cgcgccgact accgcgtgct gcgctcctac gcctgggcgg acggaccacc gctgcgggcc 1921ggcatcaccg cgctgtgcgg cgacgccgac ccgctgaccg cgaccgggga cgccgagcgc 1981tggttgcagc actcggtcat ccccggccgg accaggacct tccccggcgg gcacttctac 2041ctgggtgaac aggtcaccga ggtggccggt gccgtgcgcc gggacctgct acgcgccggg 2101cttgcgggct gaggcgatca cgaagtcgag cgcgggcagc tcgcccttca tgcccgagtc 2161gctggtcagc gaccgcttga cctggctgta gaagagcctg ctcacgctct tcttgaacga2221ctcgtcctgc aggcacctgg ctg2.选择所需的载体,确定合适的酶切位点。
引物设计

无任何特异性
03
目的基因的获取-PCR引物设计
特异引物设计:
通用引物设计:
1、扩增的完整序列完全已知 1、扩增保守的同源基因
2、扩增序列长度要≥CDS
2、扩增序列长度一般<CDS
3、引物长度18-22,得分高
3、一般测序用
4、要blast
4、不用个人设计
5、测序时要提供引物,连到T载 5、测序不需要提供引物
03
PART THREE
引物设计原则和方法
03
目的基因的克隆
基因工程的主要目的是把经过遗传学和分子生物学前期 研究探明了结构与功能的靶基因(target gene)转导 宿主细胞中表达,以大量获取该基因的产物或改变宿主 性状。由此可见,如何分离得到靶基因是基因工程操作 的关键步骤之一,否则“巧妇难为无米之炊”。
序不好进行
3、 随机6或9个mers
4、要获得完整基因要做步移
PCR或反向PCR或测完整基因组
03
目的基因的获取-PCR引物设计
引物设计软件: 1、Oligo 6 2、Primer premier 5/6 3、Dnastar 4、Primer 3plus(在线)
03
目的基因的获取-PCR引物设计
引物二聚体
发夹结构
03
目的基因的获取-PCR引物设计
引物设计种类:
引物
克隆引物 表达引物
特异引物Specific
primers
只扩增某一类基因或生物
通用引物Universal 与基因两端基因匹配(载体)
Primers
兼并引物Degenerate 可以扩增某一类相似基因
Primers
随机引物
Random primers
引物设计方法(通用)

引物设计方法设计的目的是在两个目标间取得平衡:扩增特异性和扩增效率。
引物分析软件将试图通过使用每一引物设计变化的预定值在这两个目标间取得平衡。
设计引用有一些需要注意的基本原理:①引物长度一般引物长度为18~30碱基。
总的说来,决定引物退火温度(Tm值)最重要的因素就是引物的长度。
有以下公式可以用于粗略计算引物的退火温度。
在引物长度小于20bp时:[4(G+C)+2(A+T)]-5℃在引物长度大于20bp时:62.3℃+0.41℃(%G-C)-500/length-5℃另外有许多软件也可以对退火温度进行计算,其计算原理会各有不同,因此有时计算出的数值可能会有少量差距。
为了优化PCR反应,使用确保退火温度不低于54℃的最短的引物可获得最好的效率和特异性。
xml:namespace prefix = o ns ="urn:schemas-microsoft-com:office:office" /总的说来,每增加一个核苷酸引物特异性提高4倍,这样,大多数应用的最短引物长度为18个核苷酸。
引物长度的上限并不很重要,主要与反应效率有关。
由于熵的原因,引物越长,它退火结合到靶DNA上形成供DNA聚合酶结合的稳定双链模板的速率越小。
②GC含量一般引物序列中G+C含量一般为40%~60%,一对引物的GC含量和Tm值应该协调。
若是引物存在严重的GC倾向或AT倾向则可以在引物5’端加适量的A、T或G、C尾巴。
③退火温度退火温度需要比解链温度低5℃,如果引物碱基数较少,可以适当提高退火温度,这样可以使PCR的特异性增加;如果碱基数较多,那么可以适当减低退火温度,是DNA双链结合。
一对引物的退火温度相差4℃~6℃不会影响PCR 的产率,但是理想情况下一对引物的退火温度是一样的,可以在55℃~75℃间变化。
④避免扩增模板的二级结构区域选择扩增片段时最好避开模板的二级结构区域。
用有关计算机软件可以预测估计目的片段的稳定二级结构,有助于选择模板。
【原创】引物设计方法-丁香园论坛(移动版)

【原创】引物设计方法-丁香园论坛(移动版)naclexu wrote:最近在做一批细胞,打算要做PCR,因此引物设计这关逃不过了。
beta 2 (TGFB2)、TGF A、VEGFC、VEGI各位同仁,我通过向两个老师请教设计了两套引物,方法如下。
待各位同仁哪一种方法比较好。
引物是人工合成的两段寡核苷酸序列,一个引物与感兴趣区域一端的一条DNA模板链互补,另一个引物与感兴趣区域另一端的另一条DNA模板链互补。
设计原则:引物与模板的序列要紧密互补。
引物与引物之间避免形成稳定的二聚体或发夹结构。
引物不能在模板的非目的位点引发DNA 聚合反应(即错配)。
(1)引物长度引物的长度控制着产物的特异性和在PCR反应中的退火温度。
引物的长度一般为15-30bp,最好在18-24bp,但不能超过38bp。
引物每增加一个核苷酸, 引物特异性提高4 倍。
太短易形成错配(False Priming), 降低特异性; 长PCR引物能给扩增带来更好的特异性,长引物也允许通过降低退火温度来能提高反应的灵敏度,不过长引物通常更易于形成包括发卡结构、二聚体自身互补等二级结构。
(2)碱基分布的均衡性引物中四种碱基的分布最好是随机的,不要有聚嘌呤或聚嘧啶的存在。
尤其3′端不应超过3个连续的G或C,因这样会使引物在G+C 富集序列区错误引发。
GC含量一般40-60%—GC含量太低导致引物Tm值较低,使用较低的退火温度不利于提高PCR的特异性。
—GC含量太高也易于引发非特异扩增。
(3)引物Tm值:DNA熔解温度,指把DNA的双螺旋结构降解一半时的温度。
一般要求:55℃-65℃。
计算:—对于低于20个碱基的引物,Tm值可根据Tm=4(G+C)+2(A+T)来粗略估算—对于较长引物,Tm值则需要考虑热动力学参数,从“最近邻位”的计算方式得到,这也是现有的引物设计软件最常用的计算方式。
Tm = △H/(△S + R * ln (C/4)) + 16.6 log ([K+]/(1 + 0.7 [K+])) - 273.15(4)引物的二级结构引物二聚体(Dimer and Cross Dimer) —尽可能避免两个引物分子之间3’端有有较多碱基互补。
引物的设计及修饰最全教程

引物的设计及修饰最全教程引物的设计及修饰1. 引物设计的基本原则是什么?引物设计的下列原则供您参考:1) 引物最好在模板cDNA的保守区内设计。
2) 引物长度⼀般在15-30碱基之间。
3) 引物GC含量在40%-60%之间,Tm值最好接近72℃。
4) 引物3′端要避开密码⼦的第3位。
5) 引物3′端不能选择A,最好选择T。
6) 碱基要随机分布。
7) 引物⾃⾝及引物之间不应存在互补序列。
8) 引物5′端和中间△G值应该相对较⾼,⽽3′端△G值较低。
9) 引物的5′端可以修饰,⽽3′端不可修饰。
10) 扩增产物的单链不能形成⼆级结构。
11) 引物应具有特异性。
2. 常⽤引物设计软件有哪些?常⽤的软件有Oligo 6和Primer Premier 5.0。
引物设计软件是根据引物设计的指导意见设计⽽成。
其实,PCR扩增的成败最关键的是反应模板的制备和反应条件的控制。
引物设计软件的缺点是,有时判断为该基因没有⼀段区域满⾜标准引物的要求。
⾦斯瑞为您提供以下引物设计相关软件:引物计算⼯具引物设计⼯具测序引物设计软件Real-time PCR 引物设计软件3. ⽂献上找到的引物和探针序列能否直接使⽤?通常国外的⽂献可信度⽐较⾼,可直接使⽤;但为了保险起见,最好⽤blast对引物探针的序列进⾏必要的验证;或者再进⼀步⽤引物设计软件对引物探针的⼆级结构和退⽕温度进⾏分析,这样更有利于您对整个实验的把握。
4. 如何计算引物的Tm值?Tm值的概念:DNA熔解温度,指把DNA的双螺旋结构降解⼀半时的温度,亦即DNA 变性过程中,紫外吸收值达到最⼤值的50%时的温度称为 DNA 的解链温度(Tm)。
⾦斯瑞采⽤以下⽅法计算Tm值:长度为20mer及以下的引物,Tm计算公式为:Tm = 4℃(G + C)+ 2℃(A + T)。
但这个公式只适⽤于14~20个碱基的引物,引物的TM值还与引物长度、碱基组成、引物使⽤缓冲溶液的离⼦强度等有关。
关于引物设计

引物设计的方法1.Primer3(见手把手教你做PCR引物设计)2. Primer-BLAST是NCBI的引物设计和特异性检验工具。
(NCBI引物设计)Primer-Blast介绍Primer-BLAST,在线设计用于聚合酶链反应(PCR)的特异性寡核苷酸引物。
Primer-BLAST可以直接从Blast主页(/)找到,或是直接用下面的链接进入:/tools/primer-blast/这个工具整合了目前流行的Primer3软件,再加上NCBI的 Blast进行引物特异性的验证。
Primer-BLAST免除了用另一个站点或工具设计引物的步骤,设计好的引物程序直接用Blast进行引物特异性验证。
并且,Primer-BLAST能设计出只扩增某一特定剪接变异体基因的引物–an important feature for PCR protocols measuring tissue specific expression(注:没办法准确的翻译,只好作罢,汗!)。
Primer-BLAST有许多改进的功能,这样在选择引物方面比单个的用 Primer3和NCBI BLAST更加准确。
Primer-BLAST的输入Primer-BLAST界面包括了Primer3和BLAST的功能。
提交的界面主要包括三个部分:target template(模板区), the primers(引物区), 和specificity check(特异性验证区)。
跟其它的BLAST一样,点击底部的“Advanced parameters”有更多的参数设置。
在“PCR Template”下面的文本框,输入目标模板的序列,FASTA格式或直接用Accession Number。
如果你在这里输入了序列,是用于引物的设计。
Primer-BLAST 就会根据你输入的序列设计特异性引物,并且在目标数据库(在specificity check区选择)是唯一的。
RT-PCR引物设计原则和方法

RT-PCR引物设计原则和方法在NCBI上搜索到该基因,找到该基因的mRNA,在CDS选项中,找到编码区所在位置,在下面的origin 中,Copy该编码序列作为软件查询序列的候选对象。
打开Primer Premier5,点击File-New-DNA sequence, 出现输入序列窗口,Copy目的序列在输入框内(选择As),此窗口内,序列也可以直接翻译成蛋白。
点击Primer,进入引物窗口。
此窗口可以链接到―引物搜索‖、―引物编辑‖以及―搜索结果‖选项,点击Search按钮,进入引物搜索框,选择―PCR primers‖,―Pairs‖,设定搜索区域和引物长度和产物长度。
在Search Parameters里面,可以设定相应参数。
一般若无特殊需要,参数选择默认即可,但产物长度可以适当变化,因为100~200bp的产物电泳跑得较散,所以可以选择300~500bp。
点击OK,软件即开始自动搜索引物,搜索完成后,会自动跳出结果窗口,搜索结果默认按照评分(Rating)排序,点击其中任一个搜索结果,可以在―引物窗口‖中,显示出该引物的综合情况,包括上游引物和下游引物的序列和位置,引物的各种信息等。
对于引物的序列,可以简单查看一下,避免出现下列情况:3’端不要以A结尾,最好是G或者C,T也可以;3’不要出现连续的3个碱基相连的情况,比如GGG或CCC,否则容易引起错配。
此窗口中需要着重查看的包括:Tm应该在55~70度之间,GC%应该在45%~55%间,上游引物和下游引物的Tm值最好不要相差太多,大概在2度以下较好。
该窗口的最下面列出了两条引物的二级结构信息,包括,发卡,二聚体,引物间交叉二聚体和错误引发位置。
若按钮显示为红色,表示存在该二级结构,点击该红色按钮,即可看到相应二级结构位置图示。
最理想的引物,应该都不存在这些二级结构,即这几个按钮都显示为―None‖为好。
但有时很难找到各个条件都满足的引物,所以要求可以适当放宽,比如引物存在错配的话,可以就具体情况考察该错配的效率如何,是否会明显影响产物。
引物设计

在药物基因组学研究中,有一个非常重要的技术是引物设计,在此我转载一个引物设计的原则汇总。
一、引物设计step by step1、在NCBI上搜索到目的基因,找到该基因的mRNA,在CDS 选项中,找到编码区所在位置,在下面的origin中,Copy该编码序列作为软件查询序列的候选对象。
2、用Primer Premier5搜索引物①打开Primer Premier5,点击File-New-DNA sequence, 出现输入序列窗口,Copy目的序列在输入框内(选择As),此窗口内,序列也可以直接翻译成蛋白。
点击Primer,进入引物窗口。
②此窗口可以链接到“引物搜索”、“引物编辑”以及“搜索结果”选项,点击Search 按钮,进入引物搜索框,选择“PCR primers”,“Pairs”,设定搜索区域和引物长度和产物长度。
在Search Parameters里面,可以设定相应参数。
一般若无特殊需要,参数选择默认即可,但产物长度可以适当变化,因为100~200bp的产物电泳跑得较散,所以可以选择300~500bp.③点击OK,软件即开始自动搜索引物,搜索完成后,会自动跳出结果窗口,搜索结果默认按照评分(Rating)排序,点击其中任一个搜索结果,可以在“引物窗口”中,显示出该引物的综合情况,包括上游引物和下游引物的序列和位置,引物的各种信息等。
④对于引物的序列,可以简单查看一下,避免出现下列情况:3’不要出现连续的3个碱基相连的情况,比如GGG或 CCC,否则容易引起错配。
此窗口中需要着重查看的包括:Tm应该在55~70度之间,GC%应该在45%~55%间,上游引物和下游引物的Tm值最好不要相差太多,大概在2度以下较好。
该窗口的最下面列出了两条引物的二级结构信息,包括,发卡,二聚体,引物间交叉二聚体和错误引发位置。
若按钮显示为红色,表示存在该二级结构,点击该红色按钮,即可看到相应二级结构位置图示。
引物设计的详细步骤

引物设计的详细步骤引物设计是一项关键的实验技术,用于在分子生物学实验中扩增目标DNA片段。
该技术的成功与否直接影响到实验结果的准确性和可靠性。
以下是引物设计的详细步骤:1.确定目标DNA序列:首先,确定需要扩增的目标DNA序列。
这可以通过已知的参考序列、文献调研或基因数据库进行。
2.定位扩增区域:根据目标DNA序列,确定需要扩增的特定区域。
通常选择在该区域中的保守性较高的片段,以确保引物的特异性。
3. 确定引物长度:引物长度通常为18-25个核苷酸(nt),最好不超过30nt。
引物长度的选择是为了确保引物在反应温度下的特异性和稳定性,同时不会引起非特异扩增。
4.碱基组成与G/C含量:引物的碱基组成应平衡,避免过多的同质二聚物和结构异常。
G/C含量一般在40-60%之间,过高或过低的G/C含量可能会导致引物与模板DNA结合的效力降低。
5.特异性:使用基因序列数据库或引物设计软件进行引物BLAST比对,确保引物与目标DNA序列的独特性。
6. 避免互补引物间的二聚体形成:引物间不能有太多相互衔接的序列,以免引物自身形成二聚体。
通常应避免引物间的结合自由能低于-9 kcal/mol。
7.避免引物内部二聚体的形成:通过引物设计软件计算引物的内部二聚体结合自由能,避免过多的二聚体形成。
8.引物末端设计:通常引物的末端应设计在较保守的区域,以确保扩增的特异性。
9.引物的副产物与杂交:避免引物自身产生副产物以及与其他非特异目标DNA序列杂交。
10.引物设计验证:使用引物设计软件对设计的引物进行验证,包括引物特异性、二聚体和杂交等。
11.引物合成:通过合成引物的商业公司进行引物合成,选择信誉好的厂家。
12.引物纯化:使用聚丙烯酰胺凝胶电泳等方法对引物进行纯化。
13.引物浓度测定:使用紫外分光光度计等方法测定引物的浓度。
总之,引物设计是一项细致而复杂的步骤,需要考虑多个因素,如目标DNA序列,引物长度,碱基组成,特异性和二聚体等。
PCR引物设计方法和原理

实验试错法是一种相对原始的引物设计方法,通过实验尝试和调整来找到最佳的引物序列。该方法适用于实验条 件不明确或缺乏先验知识的情况,通过反复实验和调整来获得最佳的引物组合。虽然实验试错法效率较低,但对 于探索新领域和未知情况具有一定的价值。
04 pcr引物设计的原理
引物的特异性原理
引物的特异性原理是指引物与模板DNA的结合具有严格的特 异性和专一性。在PCR扩增过程中,引物与模板DNA的结合 位点是特定的,只有当引物的序列与模板DNA完全互补时, 引物才能与模板结合,进行有效的扩增。
为确保引物的特异性,通常要求引物在3'端具有18-20个与模 板DNA互补的序列,以形成稳定的氢键,同时要求引物之间 不存在互补性,以避免形成引物二聚体。
引物的长度和浓度原理
引物的长度和浓度是影响PCR扩增的重要因素。引物的长 度通常在18-30个核苷酸之间,过短可能无法有效结合模 板,过长则可能导致结合不稳定。
引物的浓度也需进行优化,过高可能导致非特异性结合,过 低则可能影响扩增效率。根据经验,一般将引物浓度设定在 0.1-0.5μM之间,具体浓度根据引物的长度和序列特性进行 适当调整。
引物的温度和平衡原理
引物的温度和平衡原理是指在PCR扩增过程中,需要选择合适的温度使引物与模板DNA结合,并保持引物与模板之间的平衡 状态。
详细描述
计算机辅助设计法利用专业的引物设计软件,根据输入的目标序列和实验条件,自动筛 选出符合要求的引物序列。该方法能够大大提高引物设计的效率和准确性,减少人工误
差和实验时间。常用的引物设计软件包括Primer3、Oligo等。
实验试错法
总结词
通过实验尝试和调整来找到最佳引物的方法,适用于未知情况和新领域。
引物设计总结word

引物设计总结寡核苷酸的优化设计郑仲承( 中国科学院上海生命科学院生物化学和细胞生物学研究所,上海200031 )在核酸分子杂交、DNA序列测定和通过PCR放大DNA片段等实验中,都需要使用寡核苷酸作为探针或引物,而对这些反应的质量起最重要影响作用的,就是这些寡核苷酸探针或引物。
用优化的寡核苷酸进行实验能够很快得到好的结果,而用不够合适的寡核苷酸时,常常得出似是而非的结果,不仅大大增加了后续实验的工作量,还可能一无所获。
怎样优化设计寡核苷酸呢?至少有下列几个方面的问题需要考虑。
1. 估测可能形成的DNA或RNA双链的稳定性寡核苷酸,无论是DNA的或者RNA的,都有形成双链结构的潜在可能性,正如下面反复提到的,这种结构对寡核苷酸的作用有很大影响。
所以,预测这种结构的稳定性对设计和优化寡核苷酸就很重要。
在一个双链结构中,碱基对的相对稳定性是由其邻近碱基决定的。
在热动力学中,这样的性质以双链形成时的自由能(ΔG)来表示。
现在,大多采用 Breslauer等人提出的,以最接近的相邻核苷酸的动力学数值(自由能)来预测双链稳定性的方法。
为简化起见,所有的计算都在25 ℃条件下进行。
此时,最接近的相邻核苷酸的自由能是:此主题相关图片如下:ΔG(kcal/mol)例如,双链d(ACGG/CCGT)的ΔG是:ΔG(ACGG)=ΔG(AC)+ΔG(CG)+ΔG(GG)=-(1.3+3.6+3.1)=-8.0 kcal/mol此计算方法特别适用于测定其3′末端会形成双链的引物的相容性。
也可以用来计算发夹环结构的ΔG。
不过,这时需要根据环区内核苷酸的数量添加一定的数值。
如3个核苷酸时为5.2 kcal/mol;4个时为4.5;5个为4.4;6个是4.3;7和8个为4.1 kcal/mo1。
2. 选择引物的一般规则设计和选择引物时有5个要素必需注意。
2.1 引物的3′末端不互补引物的3′末端一定不能有很大的互补性,因为它们的互补会形成引物二聚体,这就会带来很大的问题,例如合成出非专一的产物,极大地减少所期望产物的得量。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
Primer 5.0搜索引物:1.Primer Length我常设置在18-30bp,短了特异性不好,长了没有必要。
当然有特殊要求的除外,如加个酶切位点什么的。
2.PCR Product size最好是100-500bp之间,小于100bp的PCR产物琼脂糖凝胶电泳出来,条带很模糊,不好看。
至于上限倒也不必要求苛刻。
3.Search parameters还是选Manual吧,Search stringency应选High,GC含量一般是40-60%。
其它参数默认就可以了。
4.搜索出来的引物,按Rating排序,逐个送Oligo软件里评估。
当然,搜索出的引物,其扩增产物很短,你可以不选择它,或是引物3端≥2个A或T,或引物内部连续的G或C太多,或引物3端≥2个G或C,这样的引物应作为次选,没得选了就选它。
对于这样的引物,如果其它各项指标还可以,我喜欢在引物末端去掉一个不满意的或加上一个碱基,看看引物的评估参数有没有变好点。
Oligo 6.0评估引物:1.在analyze里,Duplex Formation不管是上游引物、下游引物还是上下游引物之间,The most stable 3’-Dimer绝对值应小于4.5kcal/mol, The most stable Dimer overall绝对值一般应小于多少kcal/mol跟PCR退火温度有关,我几次实验感觉在PCR退火温度在65°的时候,The most stable Dimer overall 6.7kcal/mol没有问题。
2.Hairpin Formation根据黄金法则3.False priming sites: Primer的priming efficiency应该是错配地方的4倍左右,更多当然更好。
4.在PCR栏,个人感觉其所显示的optimal annealing temperature数值值得参考。
在PCR摸索条件的时候,退火温度为其数值加减2的范围就可以了。
5.Internal stability很重要:我们希望引物的内部稳定性是中间高、两边低的弧形,最起码保证3端不要过于稳定。
下图1引物3端过于稳定,很容易导致不适当扩增。
△G参照黄金法则,这其实很好理解:把一滴水放到大海里,这滴水就会不停的扩散分布,扩散的越厉害越稳定,所以△G绝对值越大结构越稳定。
最后说一句,敢于尝试就会成功。
第二贴--科室工作很多,小医生了,没有办法,所以肯怕不能满足很多战友的要求(qq聊或帮助设计),在此表示抱歉。
就楼上的问题我试着回答一下,不一定正确,供参考吧。
--1、两个评价系统不一样,个人感觉oligo评价引物好点,primer出来的引物,我一般按效率排序,再结合退火温度和引物长度,选择引物到oligo测试。
这是初步的选择,其实引物到了oligo里,退火温度也不一样。
--2、3端的二聚体应该避免,这个要看你的退火温度决定,一个50°的退火温度肯定和65°对二聚体的影响不一样了,一般来讲尽量控制在-4.5kcal/mol以下(个人观点,很多东西真得还是需要自己摸索)。
--3、个人感觉3端有A无A影响不大,3端有T的没有经验。
有T是不是一定不行,个人感觉不见得。
软件是评估,法则也不是没有例外,不是1+1=2那么确定。
--4、错配和二聚体谁轻谁重,个人觉得“到致命的程度”谁都重要,我也说不好。
我设计的时候,尽量两个都不得罪。
--5、GC含量并非不重要,它直接影响引物各端稳定性,3端来两个G或C,稳定性就上去了,粘在模板上很牢。
所以我设计的时候,尽量避免这样的情况出现。
谈一下我学这个引物设计的过程吧:入门--前面我说过是我的大师兄把我领进门的,之后我就自己捣鼓。
一开始严格按照引物设计的黄金法则来,primer搜出来的引物oligo评估,我就这样一条引物一条引物地反复评估,不厌其烦,到处查阅资料,一做就是几个小时,幸运的是捣鼓出来一两条符合标准的引物。
提高--有了成功的经验后,就继续做,当时我们实验室要定很多引物,我就有了练手的素材,这也很重要--什么东西不是练出来?!设计多了,问题也多了。
有些引物按黄金法则来,行不通了,怎么办?1、我就把引物3端或5端延长或缩短一个或几个碱基,再反复评估2、有些引物还不行,我就把5端突变一个碱基,再反复评估3、再不行,我就放宽条件。
放宽到什么样可以,不好说,自己摸索。
有的一对引物,其中一条错配很厉害,而另一条很可以,在没有其它选择的情况下为什么不可以。
有的人担心,这不一定跑的出来啊。
对自己要有信心啊,原因有二:1.自己的引物谁有功夫帮你设计?所以啊,当时我就被“赶鸭子上架了”,反反复复搜索评估的基础上,出来的应该是目前最好的;2、还有,我经常看国外文章的引物,有的评估起来表面上差的要命,人家照用,结果还不是出来了。
我就分析这样的引物,得出经验,胆子也大了。
当然失败肯定是有一两次的。
当然,有人说我就设计那么一条引物,输不起。
那只好说Sorry了!总之,我的经验就是:立即上手,遇到问题再解决问题,不厌其烦,不就是个软件嘛,还征服不了?!在NCBI上搜索到该基因,找到该基因的mRNA,在CDS选项中,找到编码区所在位置,在下面的origin中,Copy该编码序列作为软件查询序列的候选对象。
打开Primer Premier5,点击File-New-DNA sequence, 出现输入序列窗口,Copy目的序列在输入框内(选择As),此窗口内,序列也可以直接翻译成蛋白。
点击Primer,进入引物窗口。
此窗口可以链接到“引物搜索”、“引物编辑”以及“搜索结果”选项,点击Search按钮,进入引物搜索框,选择“PCR primers”,“Pairs”,设定搜索区域和引物长度和产物长度。
在Search Parameters里面,可以设定相应参数。
一般若无特殊需要,参数选择默认即可,但产物长度可以适当变化,因为100~200bp的产物电泳跑得较散,所以可以选择300~500bp。
点击OK,软件即开始自动搜索引物,搜索完成后,会自动跳出结果窗口,搜索结果默认按照评分(Rating)排序,点击其中任一个搜索结果,可以在“引物窗口”中,显示出该引物的综合情况,包括上游引物和下游引物的序列和位置,引物的各种信息等。
对于引物的序列,可以简单查看一下,避免出现下列情况:3’端不要以A结尾,最好是G或者C,T也可以;3’不要出现连续的3个碱基相连的情况,比如GGG或CCC,否则容易引起错配。
此窗口中需要着重查看的包括:Tm应该在55~70度之间,GC%应该在45%~55%间,上游引物和下游引物的Tm值最好不要相差太多,大概在2度以下较好。
该窗口的最下面列出了两条引物的二级结构信息,包括,发卡,二聚体,引物间交叉二聚体和错误引发位置。
若按钮显示为红色,表示存在该二级结构,点击该红色按钮,即可看到相应二级结构位置图示。
最理想的引物,应该都不存在这些二级结构,即这几个按钮都显示为“None”为好。
但有时很难找到各个条件都满足的引物,所以要求可以适当放宽,比如引物存在错配的话,可以就具体情况考察该错配的效率如何,是否会明显影响产物。
对于引物具体详细的评价需要借助于Oligo来完成,Oligo自身虽然带有引物搜索功能,但其搜索出的引物质量感觉不如Primer5。
在Primer5窗口中,若觉得某一对引物合适,可以在搜索结果窗口中,点击该引物,然后在菜单栏,选择File-Print-Current pair,使用PDF虚拟打印机,即可转换为Pdf文档,里面有该引物的详细信息。
引物评价在Oligo软件界面,File菜单下,选择Open,定位到目的cDNA序列(在primer中,该序列已经被保存为Seq文件),会跳出来两个窗口,分别为Internal Stability(Delta G)窗口和Tm 窗口。
在Tm窗口中,点击最左下角的按钮,会出来引物定位对话框,输入候选的上游引物序列位置(Primer5已经给出)即可,而引物长度可以通过点击Change-Current oligo length 来改变。
定位后,点击Tm窗口的Upper按钮,确定上游引物,同样方法定位下游引物位置,点击Lower按钮,确定下游引物。
引物确定后,即可以充分利用Analyze菜单中各种强大的引物分析功能了。
Analyze中,第一项为Key info,点击Selected primers,会给出两条引物的概括性信息,其中包括引物的Tm值,此值Oligo是采用nearest neighbor method计算,会比Primer5中引物的Tm值略高,此窗口中还给出引物的Delta G和3’端的Delta G。
3’端的Delta G过高,会在错配位点形成双链结构并引起DNA聚合反应,因此此项绝对值应该小一些,最好不要超过9。
Analyze中第二项为Duplex Formation,即二聚体形成分析,可以选择上游引物或下游引物,分析上游引物间二聚体形成情况和下游引物间的二聚体情况,还可以选择Upper/Lower,即上下游引物之间的二聚体形成情况。
引物二聚体是影响PCR反应异常的重要因素,因此应该避免设计的引物存在二聚体,至少也要使设计的引物形成的二聚体是不稳定的,即其Delta G值应该偏低,一般不要使其超过4.5kcal/mol,结合碱基对不要超过3个。
Oligo此项的分析窗口中分别给出了3’端和整个引物的二聚体图示和Delta G值。
Analyze中第三项为Hairpin Formation,即发夹结构分析。
可以选择上游或者下游引物,同样,Delta G值不要超过4.5kcal/mol,碱基对不要超过3个。
Analyze中第四项为Composition and Tm,会给出上游引物、下游引物和产物的各个碱基的组成比例和Tm值。
上下游引物的GC%需要控制在40%~60%,而且上下游引物之间的GC%不要相差太大。
Tm值共有3个,分别采用三种方法计算出来,包括nearest neighbor method、%GC method和2(A+T)+4(G+C)method,最后一种应该是Primer5所采用的方法,Tm值可以控制在50~70度之间。
第五项为False Priming Sites,即错误引发位点,在Primer5中虽然也有False priming分析,但不如oligo详细,并且oligo会给我正确引发效率和错误引发效率,一般的原则要使错误引发效率在100以下,当然有时候正确位点的引发效率很高的话,比如达到400~500,错误引发效率超过100幅度若不大的话,也可以接受。
Analyze中,有参考价值的最后一项是“PCR”,在此窗口中,是基于此对引物的PCR反应Summary,并且给出了此反应的最佳退火温度,另外,提供了对于此对引物的简短评价。
