第6章 基因组分析2011
医学分子遗传学第6章 线粒体基因病

MERRF家系与多向性氧化磷酸化受损有关, 主要是呼吸链酶复合物Ⅰ和Ⅳ受到损伤。氧化磷 酸化酶缺陷程度与临床症状严重程度成正比。
80%~90%患者mtDNA的tRNA基因的第8344 位A→G突变,小部分在同一基因的8 356位T→C突 变。 主要影响线粒体呼吸链的酶复合物Ⅰ和Ⅳ。 该突变使tRNALys的TψC Loop区发生改变,蛋白 质合成受阻。
2)mtDNA4977缺失致聋
研究表明,mtDNA4977缺失的发生与老化有关; 内 耳 和 蜗 核 组 织 中 mtDNA4977 缺 失 与 老 年 聋 的 发 生有关。
由于细胞内线粒体有成百上千个mtDNA拷贝,在细胞分裂 时它们又被随机分配到子细胞中,这样线粒体遗传就不同 于核基因的孟德尔遗传。在正常组织中,所有的mtDNA分
子都是一致的,称为同质性。
如果mtDNA发生突变,造成在同一细胞或同一组织中两种 或两种以上mtDNA共存,一种为野生型,另一种为突变型,
生型漂变,经过无数次分裂后,细胞可达到同质性。
如果通过遗传瓶颈存活下来的一个线 粒体碰巧携带一种突变基因,那么这个基因 组就能够确保该线粒体类型在发育完成之后 的个体中的数量。
6.阈值效应
由mtDNA突变所致的表型表达与核基因的显 性或隐性表达不同,主要是由某种组织中野生型与 突变型mtDNA的相对比例以及该种组织对线粒体的 ATP产生的依赖程度所决定的。 突变mtDNA的数目需达到某种程度才足以引 起某种组织或器官功能的异常,这称为阈值效应。
复合体Ⅰ由30多个多肽组成,其中7个由mtDNA编码。 复合体Ⅱ由4个nDNA编码的多肽构成。 复合体Ⅲ由10个多肽构成;1个由mtDNA编码。 复合体Ⅳ由13个多肽构成,3个由mtDNA编码。 复合体Ⅴ由12个多肽组成,2个由mtDNA编码。
遗传学 第六章 真核生物遗传分析

1、单一序列(unique sequence)
➢ 真核生物的大多数基因在单倍体基因 组中都是单拷贝的。
➢ 单一序列所占的比例在不同生物基因 组中变化较大:
原核生物中一般只含有非重复序列;
较低等的真核生物中大部分DNA也 是单拷贝的;
动物中将近50%DNA是中度或高度 重复的;
植物和两栖类生物中单拷贝DNA序 列降低,而中度和高度重复序列增加, 如玉米的重复序列在80%以上。
(2)卫星DNA (satellite DNA)
➢ 其碱基组成不同于其他部份,可用 等密度梯度离心法将其与主体 DNA 分开,因而称为卫星DNA 或 随体DNA。
➢ 各类卫星DNA都由不同的重复序 列家族构成。
➢ 重复单位串联排列。 ➢ 卫星 DNA约占人基因组 5~6%。
卫星DNA 根据长度可将其分为3类:
➢ 基因组(genome):一个物种单倍体的染色体数 目及其所携带的全部遗传信息。
基因组DNA测序结果表明基因组中不仅包含着整 套基因的编码序列,同时还包含着大量非编码序列, 这些序列同样包含着遗传指令(genetic instruction)。 因此,基因组(应该)是整套染色体所包含的 DNA分子以及DNA分子所携带的全部遗传指令。
➢ 可用遗传学方法区分每个染色单 体。
顺序四分子分析( ordered tetrad analysis)
顺序四分子遗传分析的特殊意义在于: (1) 能从四分子不同类型出现的相对频率分析基因间的连
锁关系; (2) 能计算标记基因与着丝点之间的重组值,进行着丝粒
作图; (3) 子囊中子囊孢子严格的对称性质,表明减数分裂是一
Co = DNA concentration t1/2 = time for half reaction
6第六章细菌和噬菌体的遗传-PPT课件

(1)F-×F+
杂交时,F+的性纤毛在二者间形成接合管→F+中 的F质粒在O点处切开,以O为先导,F拖后,按 滚环复制的方式拷贝并转移到F-中→产生两个 F+→F+的染色体几乎没有进入F-→两种细菌的染 色体未发生重组。 O F F质粒
染 色 体
F质粒
接合
F+ F-
(2)Hfr× F-
杂交时,Hfr细菌的性纤毛在二者间形成接合 管→结合态的F质粒在O点处切开,形成两端- 一端为O点,一端为基因F→以O为先导,F拖后, 按滚环复制方式向F-转移→进入F-的Hfr菌染 色体上的基因与F-染色体间发生交换重组→重 组频率高于游离态1000倍,因此称高频重组菌 株。
·
这种通过不同时间分别阻断细菌的有性接合, 从而确定细菌染色体上的基因距离的方法,称 细菌阻断交配基因作图法。
3、重组方式
接合时,供体染色体片段(外基因子)进 入受体细胞→同受体染色体的同源区段 (内基因子)进行配对→形成部分二倍体 →发生交换重组: 单交换→产生不平衡的线性染色体 双交换→有活性的重组体和线性片段(在 细胞分裂中丢失。
第六章 细菌和病毒的遗传重组
第一节 第二节 细菌的遗传基础和遗传分析 噬菌体的遗传基础和和遗传分析
第一节 细菌的遗传基础和遗传分析
一、细菌的遗传基础
原核生物 真核生物
裸露的DNA分子 DNA呈环状 单倍体,基因单个存 在
DNA与蛋白质结合成染色体 DNA呈线状 二倍体,常染色体上基因成 对
(一)细菌细胞
整合过程 O F F质粒
主染色体
整合过程 O F F质粒 O F
a bHfr细菌 d
e
根据F因子,细菌分为: 雌性细菌(受体细菌,F-)-不含F因子,表面无性 纤毛。
第六章 真核生物的遗传分析

链孢霉的特点是它的四分体是顺序排列的。
不仅减数分裂的四个产物在子囊中仍连在 一起,而且代表减数分裂四个染色单体的子囊 孢子是直线排列的,排列的顺序跟减数分裂中 期板上染色单体的定向相同。
因此,我们用遗传学方法可以区分每个染 色单体及其基因型,而用细胞学检查方法是办 不到的。
四分体遗传分析的特殊意义:
接着在每条产囊菌丝中都发生下列过程: ①由每种交配型的一个核共同形成子囊原始细胞, ②这两个核在伸长的细胞中融合成二倍体细胞核; ③二倍体细胞核立即进行减数分裂; ④减数分裂的四个产物再进行一次有丝分裂,在一个
子囊中形成四对子囊孢子。 同时,其他菌丝形成了一个厚壁包围着产囊菌丝,构
成长颈瓶状的子囊壳。
的特异的碱基序列(单拷贝)的长度(或核苷数)之和来表示 复杂度(的大小) 。
DNA分子中无重复的核苷酸序列的最大长度.
病毒或细菌的基因组无重复序列,其基因组的复杂度与 C值(即基因组的大小)相等。
四、真核生物基因组DNA序列的复杂度
DNA复性动力学研究结果表明,真核生物基因组序列大致 可分为3种类型: 1、单拷贝序列(非重复序列):每个基因只有1-2个 拷贝。 2、中度重复序列:平均长度300bp,重复次数10-102。 3、高度重复序列:通常为6-200bp,重复次数在106。
第二次分裂分离: + - + - +--+
-++-
-+-+
每一个第二次分裂分离的子囊是供试位点与着丝点 之间发生一次交换的结果。
根据这种特殊情况,就有可能计算某一位点和着丝点之间的重组百分率。 重组百分率的标准公式如下:
A位点和着丝点之间重组 染色单体数 染色单体总数
100
交换值 (%)
重组型配子数 总配子数
遗传学第六章基因与发育

2
1
3种胚层经过了细胞分化生成各种器官的原基,如肢、眼、心等原基,这是器官生成。
原基先是生成微小而精确的结构,然后逐渐长大,在生物体的各个正确部位长成各种器官和组织,这是形态建成。
神经管胚形成期
器官生成 形态建成 人的发育过程
全能干细胞(totipotent):能够分化产生各种细胞直至个体的细胞。例如胚胎干细胞(embryonic stem cell)。
如果背部蛋白质没有进入细胞核,则细胞核的腹化基因不能激活,背化基因不被抑制——分化出背化细胞。
当合胞体细胞形成许多细胞时,背部蛋白质进入合胞体腹侧的细胞核,细胞核的腹化基因被激活,背化基因被抑制——分化出腹化细胞;
母体背部基因的RNA在卵受精后90min时才翻译产生背部蛋白质。
前端组织中心
后端组织中心
在8个卵裂球期,每个卵裂球在生化、形态和发育潜能上都没有差别,也就是在发育上是全能的。 可是当卵裂球成团结合时,细胞处在外层还是内层,会使以后生成的卵裂球出现了不同的生物学功能。处在外层的细胞生成滋养层,而处在内层的细胞则生成内细胞团而产生胚胎(几个细胞?)。
如果胚泡中只有一个细胞形成以后的胚胎,则生下小鼠的毛色或者全是黑色,或者全是白色。 如果胚泡中有两个细胞参与胚胎的生成,则异决表型小鼠的数目应占子代中的半数(1ww:2wB:1BB), 如果胚泡中的3个细胞生成胚胎,则异决表型小鼠应占75%(1www:3wwB:3wBB:1BBB); 如果胚泡中的4个细胞生成胚胎,则毛色黑白相间小鼠的概率为87.5%。
(2)成对规则基因(pairrule genes)
成对规则基因
2、分节基因
体节极性基因的转录图式是受成对规则基因pair-rule基因所调控,这类基因的功能是保持每一体节中的某些重复结构,体节极性基因在每个体节内部调控其发育模式,包括极性。 当这类基因发生突变后,会使每一体节的一部分结构缺失,而被该体节的另一部分的镜像结构所替代。例如:engrail基因是保持前后体节间的分界,engrail突变型胚胎则出现前后体节融合为一,即每一体节的后半部被后一个体节的前半部的重复结构所替代。
(整理)遗传学第六章课后习题解答
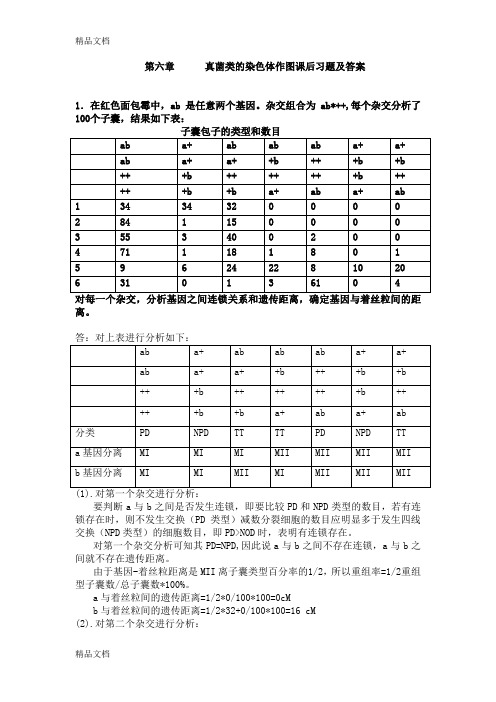
第六章真菌类的染色体作图课后习题及答案1.在红色面包霉中,ab是任意两个基因。
杂交组合为ab*++,每个杂交分析了100个子囊,结果如下表:子囊包子的类型和数目ab a+ab ab ab a+a+ab a+a++b+++b+b+++b+++++++b+++++b+b a+ab a+ab 134******** 2841150000 3553400200 4711181801 596242281020 6310136104对每一个杂交,分析基因之间连锁关系和遗传距离,确定基因与着丝粒间的距离。
答:对上表进行分析如下:ab a+ab ab ab a+a+ab a+a++b+++b+b+++b+++++++b+++++b+b a+ab a+ab分类PD NPD TT TT PD NPD TTa基因分离MI MI MI MII MII MII MII b基因分离MI MI MII MI MII MII MII(1).对第一个杂交进行分析:要判断a与b之间是否发生连锁,即要比较PD和NPD类型的数目,若有连锁存在时,则不发生交换(PD类型)减数分裂细胞的数目应明显多于发生四线交换(NPD类型)的细胞数目,即PD>NOD时,表明有连锁存在。
对第一个杂交分析可知其PD=NPD,因此说a与b之间不存在连锁,a与b之间就不存在遗传距离。
由于基因-着丝粒距离是MII离子囊类型百分率的1/2,所以重组率=1/2重组型子囊数/总子囊数*100%。
a与着丝粒间的遗传距离=1/2*0/100*100=0cMb与着丝粒间的遗传距离=1/2*32+0/100*100=16 cM(2).对第二个杂交进行分析:由于PD类型远大于NPD类型,即PD>NOD,表明a与b连锁。
a与b之间的遗传距离=50*(TT+6NPD)=50*(15/100+6*1/100)=10.5 cMa与着丝粒间的遗传距离=1/2*0/100*100=0 cMb与着丝粒间的遗传距离=1/2*15/100*100=7.5 cM由此可知a与b位于着丝粒的两端。
基因组信息分析

第5章基因组信息分析人类基因组计划的主要成果是得到一本“天书”,这本天书既简单又复杂。
说它简单,是因为这本天书仅仅由4个字母构成,这4个字母分别是A、T、C、G;说它复杂,是因为这本天书包括所有人类生长发育的信息,隐含人类生老病死的规律。
对于基因组序列,我们最关心的就是从序列之中找到基因及其表达调控信息。
寻找基因牵涉到两个方面的工作,一是识别与基因相关的特殊序列信号,如启动子、起始密码子,通过信号识别大致确定基因所在的区域。
另一个工作是预测基因的编码区域,或预测外显子所在的区域。
然后结合两个方面的结果确定基因的位置和结构。
绝大部分基因表达调控信息隐藏在基因序列的上游区域,在组成上具有一定的特征,可以通过序列分析识别这些特征。
5.1 关于遗传语言5.1.1 基因组DNA的奥秘生命是大自然最伟大的创造物,经过亿万年的进化,生命的形式从简单的有机物发展到现在高度复杂但有序的生物系统。
蛋白质是构造生命机器的基本元件,大量结构不同、功能各异的蛋白质在遗传信息的控制之下,被不断地合成出来,并有机地组成复杂的生物体。
遗传信息存贮在基因组中,具体说就是存贮在由4种字符组成的核酸序列中。
随着分子生物学中心法则的确立,人们逐渐认识到,遗传信息的载体主要是DNA(在少数情况下RNA也充当遗传信息载体),控制生物体性状的基因则是一系列DNA片段。
一方面,DNA通过自我复制,在生物体的繁衍过程中传递遗传信息,另一方面,基因通过转录和翻译,使遗传信息在生物个体中得以表达,并使后代表现出与亲代相似的生物性状。
在基因表达过程中,基因上的遗传信息首先通过转录从DNA传到RNA,然后再通过翻译从RNA传递到蛋白质。
基因控制着蛋白质的合成,基因的DNA序列到蛋白质序列存在着一种明确的对应关系,而这种对应关系就是我们所知道的遗传密码。
1961年,科学家Nirenberg使用信使RNA分子研究得到第一个遗传密码,1969年确定全部的遗传密码。
第六章 基因组解剖

杭州师范大学生命与环境科学学院 向太和
§6基因组解剖 § 6.1 真核生物基因组解剖
物种的染色体组
基因组学
杭州师范大学生命与环境科学学院 向太和
染色体结构
基因组学
杭州师范大学生命与环境科学学院 向太和
染色体显带或分带— 染色体显带或分带
chromosome banding
染色体带是指当染色体经一定的物理、 染色体带是指当染色体经一定的物理、化学因素处理并用特定的 染料染色后,在显微镜下可显示出特定的深浅不同的条纹, 染料染色后,在显微镜下可显示出特定的深浅不同的条纹,或在荧光 显微镜下看到不同强度的荧光节段。不同染色方法可分出Q 显微镜下看到不同强度的荧光节段。不同染色方法可分出Q、G、C、R、 Cd等几种类型的带 不同的染色体具有不同形态的带, 等几种类型的带。 N、T及Cd等几种类型的带。不同的染色体具有不同形态的带,称为 带型” 反映了染色体固有的结构。 “带型”,反映了染色体固有的结构。据此我们可以准确无误地识别每 一条染色体,并可用来分析染色体内部结构的变化。 一条染色体,并可用来分析染色体内部结构的变化。 染色体带显示的过程称为染色体显带或分带。1968年 染色体带显示的过程称为染色体显带或分带。1968年,科学家用 氮芥喹吖因使染色体不同部位分化染色,显示出清晰的带纹。 氮芥喹吖因使染色体不同部位分化染色,显示出清晰的带纹。自染色 体高分辨显带技术问世后, 体高分辨显带技术问世后,研究者可以在人体细胞的前中期染色体上 显示出1 256条带 在早前期染色体上可显示出3 000~ 000条带 条带, 条带。 显示出1 256条带,在早前期染色体上可显示出3 000~10 000条带。 显带技术不仅解决了染色体的识别问题, 显带技术不仅解决了染色体的识别问题,还为深入研究染色体的异常 及人类基因定位创造了条件。 及人类基因定位创造了条件。
第六章 基因组信息分析

说 明 腺嘌呤 鸟嘌呤 胸腺嘧啶 胞嘧啶 嘌呤 嘧啶 氨基 羧基 强氢键(3个氢键) 弱氢键(2个氢键) 非G 非A 非T(非U) 非C 任意碱基
核苷酸表示符号
共有序列构造过程:
(1)初始化共有序列为一系列可变位置,以“N”代表; (2)在可变位置寻找出现次数最多的核苷酸,并将该 位置转化为保守位置; (3)对当前所得到的共有序列进行特异性检查,若通 过检查,转(5),否则转(4); (4)形成与当前共有序列一致的位点子集,转(2); (5)从原位点集合中删除与当前共有序列一致的位点, 若还有剩余位点,则转(1),构造另外的共有 序列。
关联性分析
对于酵母基因组 PA=0.3248 PAA=0.1193
=0.1193/(0.3248*0.3248) =1.131 > 1 表明在两个连续位置上“A”的出现不是独立 的,而是相关的。 PAA’
同样,对于相隔一定距离k(k代表核苷酸个数) 的两个核苷酸,也可能具有一定的相关性。 假设Pij(k)代表核苷酸j出现在核苷酸i之后第k个位 置的频率,则可定义一个反应统计相关性的互信 息I(k)
一、 DNA序列分析步骤和分析结果评价
在DNA序列中,除了基因之外,还包含许多其它信息,这 些信息大部分与核酸的结构特征相关联,通常决定了DNA 与蛋白质或者DNA与RNA的相互作用。 存放这些信息的DNA片段称为功能位点 • 如启动子(Promoter)、基因终止序列(Terminator sequence)、剪切位点(Splice site)等。
2用感知矩阵分析功能位点用权系数描述功能位点各位置上每种核苷酸的相对重要性其大小为4n4代表碱基的种类数目n代表功能位点的长度矩阵的每一个元素maj的值代表第a种核苷酸在功能位点第j个位置上出现的得分a319916根据对应位置上核苷酸的类型取感知矩阵中对应的权值加和以后得到该序列的得分当人类基因组研究进入一个系统测序阶段时急需可靠自动的基因组序列翻译解释技术以处理大量已测定的但未知功能或未经注释的dna序列非翻译区域untranslatedregionsutr编码区域两端的dna有一部分被转录但是不被翻译这一部分称为非翻译区域对于任何给定的核酸序列单链dna或mrna根据密码子的起始位置可以按照三种方式进行解释
基因组数据分析

Protein Database
Nucleotide Database
6
程序名
搜索序列
数据库
内容
备注
blastp
Protein
Protein
比较氨基酸序列与蛋白 使用取代矩阵寻找较 质数据库 远的关系,进行SEG 过滤 比较核酸序列与核酸数 寻找较高分值的匹配, 据库 对较远的关系不太适 用 比较核酸序列理论上的 用于新的DNA序列和 六框架的所有转换结果 ESTs的分析,可转 和蛋白质数据库 译搜索序列 比较蛋白质序列和核酸 用于寻找数据库中没 序列数据库,动态转换 有标注的编码区,可 为六框架结果 转译数据库序列 比较核酸序列和核酸序 转译搜索序列与数据 列数据库,经过两次动 库序列 态转换为六框架结果
三、低复杂度区域(LCRs) •低复杂度区域过滤 •将该类区域转化为不明确字符(蛋白质用X,核酸用N)
35
多序列比对的目的
• 从物种的一些分子特性出发,从而了解物种之 间的生物系统发生的关系。 • 通过序列同源性的比较进而了解基因的进化以 及生物系统发生的内在规律。
36
• 分子钟 – 不同生物系统的同一血红蛋白分子的氨基酸随着时间的推移而以 几乎一定的比例相互量换着 (Zuckerkandl&Pauling,1962 ) – 蛋白质,基因序列在单位时间以大致恒定的速度进行置换
12-第06章 基因组解剖-真核生物基因组

基因 组等 高线
—
碱基 非均一
分布
基因组等高线系指连续分布的具有相似碱基组成的DNA区段,它们在 基因组中成片相嵌排列。按离心沉降区带分为为高比重(H)与 低比重(L)两类。H3组分的单拷贝比例高达50%~70%。H3, H1+H2和L1+L2所含基因的数量分别占整个基因组的28%,38% 和34%,说明大量的基因集中分布在人类基因组的有限区域。 Gene 100: 181-187,1991
第6章 基因组解剖--
真核生物基因组
染 色 体 结 构
基因组的结构成分
1) SAR和 MAR 2) CpG岛 3) 等 高 线 4) 基 因 区
SAR和MAR
SAR: Scaffold attehment region, 支架附着区,与染 色体骨架蛋白结合 的染色体DNA顺序.
MAR: matrix arrechment, region 基质附着区, 与细 胞核基质蛋白成分 结合的染色体DNA 顺序.
1) 等高线(isochore)系指基因组中具有较均一的 相似比例碱基组成的连续的DNA顺序.
2) 脊椎动物基因组中等高线DNA顺序具有相嵌 分布的特征.
3) 高GC比例区总是分布在基因密集区或常染 色 质区, 高AT比例区大多数分布在异染色质 区 或贫基因区.
4) 梯度密度离心可将不同碱基比例的具有均一 性组成的DNA顺序分离.
脊椎动物DNA甲基化只发生在双碱基-CpG-的胞嘧啶(C) 位置。胞嘧啶甲基化是基因沉默的信号,是基因表达 调控的结构基础。在人体癌变细胞中一些重要的功能 基因因甲基化修饰而沉默。绝大多数管家基因含有 CpG岛, 是基因注释的重要依据。
Genes & Dev. 25: 1010-102,2011
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
研究模式的改变——后基因组时代
• 传统模式: 功能-蛋白-基因-基因组
(结构基因组分析)
• 现代模式: 基因组-基因-蛋白-功能 (功能基因组分析)
Functional genomics
• --- the genome-wide study of the function of DNA, as well as its RNA and protein products, to understand the relationship between genotype and phenotype. • --- by analysis of SNP and epigenomics, (DNA function) expression variation, (RNA, protein function) protein interactions, (protein function) natural and experimental disruptions (disfunction function) ……
• Their locations in the genome can then be mapped,as landmarks.
—— NCBI.
STS and PCR (1)
• Operationally, an STS is defined by a pair of oligonucleotide primers that can be used in a PCR assay to detect a site that is unique in the genome.
• One could simply look up the STS in the database, synthesize the specified primers, and run the PCR to amplify the STS --- if the target DNA fragment contains the STS, than it can be localized in the genome along with the STS.
生物信息学
Bioinformatics
第六章 基因组分析
• 基因组和生物信息学
• 基因组特性
• 结构基因组分析 (基因组作图,STS *)
• 功能基因组分析 • 比较基因组分析 (COG *) • 基因组信息资源 (NCBI,UCSC*,Ensembl)
基因组(Genome)
• 基因组是指一个细胞生物体的单 倍细胞、细胞器或病毒 所包含的 所有基因与基因间序列的总体。
--功能基因组分析:方兴未艾而任重道远。
预测基因功能的方式
在确定ORF后—— • 通过序列比对寻找已知功能的同源序列; • 先预测结构,再通过结构相似性比较寻找 已知功能的大分子结构。 ——工作仍很粗浅,不能预测大部分(70%) ORF的功能。
非确定性读码框(URF)
• URF(unidentified reading frame, URF)是 指在DNA序列中识别出的一个可读框,但没 有相关的生物学功能信息(也没有已知的同 源物),它应该编码了一种蛋白质,但人们 从未发现或确定过该蛋白质的功能。
第六章 基因组分析
• 基因组和生物信息学
• 基因组特性
• 结构基因组分析 (基因组作图,STS *)
• 功能基因组分析 • 比较基因组分析 (COG *) • 基因组信息资源 (NCBI,UCSC*,Ensembl)
基因组分析 1. 基因组测序:基因组有什么样的序列? 2. 基因组解析:基因组各部分是什么? 它们具有什么样的生物学意义? 3. 基因组比较:基因组是怎样进化的?
• The significance of this technique can be seen by considering that it is possible to map a new sequence without performing a single experiment in the laboratory.
第六章 基因组分析
• 基因组和生物信息学
• 基因组特性
• 结构基因组分析 (基因组作图,STS *)
• 功能基因组分析 • 比较基因组分析 (COG *) • 基因组信息资源 (NCBI,UCSC*,Ensembl)
基因组学 1. 基因组测序:基因组有什么样的序列? 2. 基因组解析:基因组各部分是什么? 它们具有什么样的生物学意义? 3. 基因组比较:基因组是怎样进化的?
第六章 基因组分析
• 基因组和生物信息学
• 基因组特性
• 结构基因组分析 (基因组作图,STS *)
• 功能基因组分析 • 比较基因组分析 (COG *) • 基因组信息资源 (NCBI,UCSC*,Ensembl)
基因组的结构组成
• 基因组在形态结构上体现为染色体。原核 生物的“染色体”只有一条,且不大;而 真核生物的染色体通常很大,且数目众多 (尤其是在植物中)。 • 原核生物基因组中的非编码区很少,而真 核生物的基因组非编码DNA可能占大部分。
染色体上的重复序列
• 端粒区:随机的中度重复序列,和年龄 有关。 • 着丝粒:随机的高度重复序列。 • 核仁组织区:中度重复序列,编码核糖 体RNA的基因。
基因组中的遗传多态性
• 单核苷酸多态性(SNP):是指基因组内特 定核苷酸位点上存在不同的碱基,其中每 种在群体中的频率不小于1%。(派生 RFLP,SSCP)
NCBI --- COGs
Clusters of Orthologous Groups of proteins (COGs) were delineated by comparing protein sequences encoded in complete genomes, representing major phylogenetic lineages. Each COG consists of individual proteins or groups of paralogs from at least 3 lineages and thus corresponds to an ancient conserved domain.
借助STS定位目标序列
基因 组序 列 STS 标记 图 定位 目标 序列
模拟定位 —— E-PCR
• We could identify STSs in the target DNA sequence by searching for subsequences that match the PCR primers and are in the correct order, orientation, and spacing of a real PCR.
功能基因组分析的三个层次
染色体基因组,或简称基因组,即生物体内所有 细胞中的遗传信息。->DNA 表达基因组,或称转录基因组,即细胞某个特定 生长阶段中基因组的表达部分。->mRNA
蛋白质组,反映细胞特性和功能的所有蛋白质分 子。 ->蛋白质
功能基因组分析的发展
• 目前,已有大量的基因组DNA序列被储存 在数据库中,但对于其内容的理解,即 了解其功能,并不容易。所以,即使在 全基因组测序完成后,还需要相当长的 一段时间来了解生物体的全部复杂性。
• An STS is a relatively short sequence (200 to 500 bp) occurring only once in the genome. (--- can be specifically amplified by PCR in the presence of all genomic sequences)
分子进化中的同源性(2)
• 直系同源(orthology):由“物种分化”而产 生, 有功能一致性;可以反映物种血统上的同 源性,即物种进化的历史。 比如,小鼠和大鼠的肌红蛋白。
• 并系同源(paralogy):由基因“多重化 (duplicating)+功能分化” 而产生。 比如,人血红蛋白中的α、β亚基与肌红蛋白。
0.5% 1.5%
98%
99.5% Lunfish (dipnoi)
Human Coding (protein) RNA Non-coding
基因组的非编码区的意义
• 非编码区的意义还不很明确,现在已经 成为研究热点之一。
——至少它在进化中的作用是巨大的。由 于其非编码的特性,它为沉默突变的积 累提供了一个平台。这应该是真核生物 能够进化到如此复杂的一个重要原因。
基因组学(Genomics) 1. 基因组测序:基因组有什么样的序列? 2. 基因组解析:基因组各部分是什么? 它们具有什么样的生物学意义? 3. 基因组比较:基因组是怎样进化的? ——都离不开生物信息学。
基因组分析为生物信息学赋予了机会和挑战 • 机会:基因组序列信息的丰富性为生物信息 学提供了很好的研究对象;基因组序列信息 的物种完备性为生物信息学赋予了更高的研 究意义。 • 挑战:基因组所含信息量至少比单个基因高 几个数量级;草图序列中存在不少碱基甚至 基因组片段的缺失和错误。
基因组学 1. 基因组测序:基因组有什么样的序列? 2. 基因组解析:基因组各部分是什么? 它们具有什么样的生物学意义? 3. 基因组比较:基因组是怎样进化的?
结构基因组分析——基因组作图 基因组水平的研究最重要的特征之一是有位置 信息 ——
• 基因组序列图:什么位置是什么核苷酸?
• 基因组元素图:什么位置是什么元素 (elements:基因、疾病倾向位点、标记等)? ——终极目标:把所有图依照位置对应起来。genetic maps Genetic linkage maps Radiation Hybrid maps STS maps Clone-based maps ……
