pRetroX-PTuner2-C逆病毒载体使用说明
逆转录病毒试用装操作手册

汉恒逆转录病毒操作手册一、逆转录病毒的试用装分装与储存1.收到汉恒生物逆转录病毒产品后,请先对逆转录病毒产品进行分装处理,建议20-50μl/管。
如1-2天内使用,则留足够量病毒4℃保存,余下逆转录病毒置于-80℃长期保存。
2.病毒可以存放于-80℃ 6个月以上;但如果病毒储存时间超过6个月,建议在使用前重新测定病毒滴度。
3.反复冻融会降低逆转录病毒滴度:逆转录病毒滴度越低,冻融对滴度的影响越大,108IU/ml的逆转录病毒冻融2次滴度没有显著性差异,第3次冻融开始,每冻融一次滴度降低约10%;滴度107IU/ml滴度逆转录病毒,从第2次冻融开始,每次冻融病毒滴度下降10%-15%。
二、逆转录病毒试用装实验策略与关键知识点1.逆转录病毒感染策略:逆转录病毒本身的病毒滴度不高,感染细胞时病毒消耗量较大,试用装体积较小,建议使用96孔板进行MOI梯度感染测试。
逆转录病毒感染细胞后会反转录并插入基因组形成稳转,因此逆转录病毒感染一般采用感染少量细胞,然后将细胞放大化培养。
而不是直接大量感染。
注意:对于一些传代能力较差的原代细胞,比如BMSC等,建议采用腺病毒感染。
2.逆转录病毒感染关键知识点1)细胞准备:由于试用装体积有限,请使用96孔板准备细胞,逆转录病毒感染细胞前,请确保目的细胞状态良好、无支原体污染。
逆转录病毒感染的时候细胞汇合率为40%~60%间为宜。
2)感染细胞最佳MOI的测定:MOI(感染复数)是指每个细胞感染的病毒数。
逆转录病毒对于不同种类不同来源的细胞,其最适MOI各有差别,原则上最适MOI是感染效率较好的最低MOI。
MOI一般以3:10:30:100的比例递增进行梯度摸索,一般的细胞基础逆转录病毒感染MOI 可从3为开始,即设立4个常规的MOI摸索,即3,10,30,100。
3)助转剂polybrene的选择:实验证明,polybrene可提高逆转录病毒对大部分细胞的感染效率,但polybrene 有一定的细胞毒性,不同细胞对polybrene的敏感度不同,polybrene最常用的工作浓度为5~8μg/ml。
PRRSV2PCV2重组病毒的构建及PRRSV转录调控机制-微生物与感染

PRRSV 2PCV2重组病毒的构建及PRRSV 转录调控机制研究郑海红1,2 孙 志1 朱兴全2 袁世山1 【摘要】 目的 本研究旨在探讨猪繁殖与呼吸综合征病毒(PRRS V )感染性cDNA 克隆作为猪圆环病毒(PC V2)的主要免疫原开放阅读框架2(ORF2)表达载体的可行性,以及利用重组病毒对PRRS V 复制转录过程进行解剖。
方法 以北美株PRRS V 感染性克隆pAPRRS 为平台进行反向遗传操作,分别在pAPRRS 的ORF1和ORF2间,ORF5和ORF6间,ORF6和ORF7间插入PC V2ORF2,且对拯救病毒vPC V 进行了病毒学及分子生物学鉴定。
结果 vPC V 的sgmRNA211利用了PRRS V mRNA2的转录调控序列(TRS ),形成由PRRS V G P2和PC V2衣壳蛋白组成的sgmRNA211。
外源基因的插入同时也导致重组病毒启用新的TRS 而产生3条新亚基因组,其中sgmRNA212和sgmRNA213采用PC V2上的序列来取代PRRS V RNA2本身的TRS;另一条sgmRNA214则为非经典型亚基因组,其TRS 在PRRS V 相应的AUG 下游。
结论 2株PRRS V -PC V2重组病毒可以稳定传代,为进一步研发PRRS V 遗传标疫苗奠定了基础;插入片段上一些类似TRS 序列的引入及插入片段(718bp )过长导致PRRS V ORF2TRS 本身和其两翼序列的RNA 结构变化是引起重组病毒遗传不稳定性的最主要原因;PRRS V 可以利用TRS 样外源序列作为转录启动子,这为进一步解剖PRRS V 复制转录过程奠定了基础。
【关键词】 猪繁殖与呼吸道综合征病毒;猪圆环病毒Ⅱ型;载体表达;转录调控序列Construction of recombinant porcine reproductive and respiratory syndrome virus expressing porcine circovirustype Ⅱ(PRRSV 2PCV 2)antigen and study of PRRSV transcription mech anisms with this novel viral vectorZHENG Hai 2hong 1,2,SUN Zhi 1,ZH U Xing 2quan 2,YUAN Shi 2shan 1(Department o f Animal Infectious Diseases ,Shanghai Veterinary Research Institute ,China Academy o f Agricultural Sciences ,Shanghai 200241,China )【Abstract 】 Objective The g oal of the current study is to develop in fectious porcine reproductive and respiratory syndrome virus (PRRS V )cDNA clones as expression vectors for porcine circovirus type Ⅱ(PC V2)ORF2antigens and to use this new vector to dissect the replication and transcription abilities of PRRS V 1Methods ORF2from PC V2was inserted into our previously established in fectious clone of PRRS V ,designated pAPRRS ,at three locations :1)between ORF1b and ORF2a ;2)between ORF5and ORF6;and 3)between ORF6and ORF71The three full length mutant clones were then trans fected into M ARC 2145cells ,resulting in three recombinant viruses 1The genetic instability of recombinant virus was further examined via PRRS V ’s mRNA22specific RT 2PCR sequence analysis of sgmRNA2,which enabled us to determine the origin of the sequence at the junction site 1R esults Our results dem onstrated that the sgmRNA2of this recombinant PRRS V 2PC V2has one TRS derived from the PRRS V ORF2TRS ,and tw o TRS segments were derived from the sequence of PC V21One TRS was derived from the PRRS V ORF2noncanonical TRS located at the downstream of PRRS V ORF2initiation cordon 1Conclusion T w o recombinant PRRS V 2PC V viruses are stable for m ore than ten passages ,which makes them useful as marker vaccines 1The m ost comm on reas on for genetic instability of the PRRS V 2PC V recombinant virus is an insertion of a sequence that resembles TRS and a subsequent change of the RNA structure for PRRS V ORF2TRS and its flanking sequence 1PRRS V can use a PC V2sequence as its own TRS ,which is useful for dissecting the mechanism behind PRRS V replication and transcription 1【K ey w ords 】 P orcine reproductive and respiratory syndrome virus ; P orcine circovirus type Ⅱ; Express vector ;T ranscription 2regulation sequence 基金项目:国家自然科学基金重点项目资助(编号:30530580);上海市浦江人才项目(2006P J48114) 作者单位:11中国农业科学院上海兽医研究所动物传染病研究室,农业部动物寄生虫病重点实验室,上海200241;21华南农业大学兽医学院,广州510642 通讯作者:袁世山,E 2mail :shishanyuan @shvri 1ac 1cn 猪繁殖与呼吸道综合征病毒(porcine reproductive and respiratory syndrome virus ,PRRS V )和猪圆环病毒Ⅱ型(porcine circovirus type Ⅱ,PC V2)是引起仔猪断奶后多系统衰竭综合征(postweaningmultisystemic wasting syndrome ,PMWS )的主要病原,给全世界养猪业带来了巨大经济损失。
罗氏(Roche)公司Tunel试剂盒操作说明书

罗氏(Roche)公司Tunel试剂盒操作说明书一、原理:TUNEL(TdT-mediated dUTP nick end labeling)细胞凋亡检测试剂盒是用来检测组织细胞在凋亡早期过程中细胞核DNA的断裂情况。
其原理是荧光素(fluorescein)标记的dUTP在脱氧核糖核苷酸末端转移酶(TdT Enzyme)的作用下,可以连接到凋亡细胞中断裂DNA的3’-OH末端,并与连接辣根过氧化酶(HRP,horse-radish peroxidase)的荧光素抗体特异性结合,后者又与HRP底物二氨基联苯胺(DAB)反应产生很强的颜色反应(呈深棕色),特异准确地定位正在凋亡的细胞,因而在光学显微镜下即可观察凋亡细胞;由于正常的或正在增殖的细胞几乎没有DNA断裂,因而没有3‘-OH形成,很少能够被染色。
本试剂盒适用于组织样本(石蜡包埋、冰冻和超薄切片)和细胞样本(细胞涂片)在单细胞水平上的凋亡原位检测。
还可应用于抗肿瘤药的药效评价,以及通过双色法确定细胞死亡类型和分化阶段。
二、器材与试剂器材:光学显微镜及其成像系统、小型染色缸、湿盒(塑料饭盒与纱布)、塑料盖玻片或封口膜、吸管、各种规格的加样器及枪头等;试剂:试剂盒含TdT 10×、荧光素标记的dUTP 1×、标记荧光素抗体的HR P;自备试剂:PBS、双蒸水、二甲苯、梯度乙醇(100、95、90、80、70%)、DAB工作液(临用前配制,5 μl 20×DAB+1μL 30%H2O2+94 μl PBS)、Proteinase K工作液(10-20 μg/ml in 10 mM Tris/HCl,pH 7.4-8)或细胞通透液(0.1% Triton X-100 in 0.1% sodium citrate,临用前配制)、苏木素或甲基绿、DNase 1(3000 U/ml– 3 U/ml in 50 mM Tris-HCl,pH 7. 5,10 mM MgCl2,1 mg/ml BSA)等。
pRetroX-SG2M-Cyan逆病毒载体使用说明

pRetroX-SG2M-Cyan
pRetroX-SG2M-Cyan 逆病毒载体基本信息:
载体名称:
pRetroX-SG2M-Cyan 质粒类型: 逆病毒表达载体;荧光报告载体 高拷贝/低拷贝: 高拷贝 克隆方法: 限制性内切酶,多克隆位点
启动子:
CMV IE 载体大小:
8061 bp 5' 测序引物及序列:
-- 3' 测序引物及序列:
-- 载体标签:
-- 载体抗性: 氨苄青霉素 筛选标记: Neomycin1.html' target='_blank'>新霉素(Neomycin )
克隆菌株:
DH5α, HB101 宿主细胞(系): 常规细胞系,293、CV-1、CHO 等
备注: pRetroX-SG2M-Cyan 载体是逆病毒载体,组成型表达青色
荧光蛋白AmCyan1-hGeminin ,用于构建青色荧光标记的
细胞系。
稳定性: 稳表达
组成型/诱导型:
-- 病毒/非病毒: 逆转录病毒
pRetroX-SG2M-Cyan 载体质粒图谱和多克隆位点信息:
pRetroX-SG2M-Cyan载体简介:pRetroX-SG2M-Cyan载体序列:。
猪圆环病毒 PCR 检测试剂盒 说明书

猪圆环病毒PCR检测试剂盒用途猪圆环病毒(PCV)的聚合酶链反应(PCR)试验用于检测猪血清和组织中的PCV,适用于PCV 的检测、诊断和流行病学调查。
原理提取病料DNA作为模板,高温使模板的一条双链DNA变性后形成两条单链,低温使引物与互补的模板形成双链,中温时,在TaqDNA聚合酶作用下,以dNTP为原料,以引物为复制的起点,沿模板合成一条新链。
每个循环包括:高温变性、低温退火、适温延伸三个过程。
每一次循环使扩增的DNA 片段拷贝数放大一倍。
经过35次循环,使扩增的DNA片段放大了数百万倍。
将扩增产物进行电泳,经染色后,在紫外灯照射下,肉眼可见到DNA片段的扩增带。
试剂1. 试剂盒组成名称10头份50头份0.2 mL薄壁PCR管 15个 60个PCV-1阳性对照350 µL 1 mLPCV-2阳性对照350 µL 1 mLA液:消化液 6 mL 30 mLB液:蛋白酶K 110 µL 600 µLC液:酚/氯仿/异戊醇混合液7 mL 34 mLD液:异丙醇 6 mL 30 mLE液:70%乙醇12 mL 60 mLF液:灭菌去离子水650 µL 2 mLJ液:PCR反应液200 µL 1 mLK液:Taq DNA聚合酶25 µL 120 µLL液:矿物油300 µL 1.2 mLM液:50倍TAE电泳缓冲液(50倍稀释后使用)20 mL 100 mLO液:上样缓冲液50 µL 250 µL染色液20 µL 50 µL2. 贮藏条件:塑料袋内红盖试剂(阳性对照、B、E、J、K液)-20 ℃冻存。
其他室温保存。
3. 试剂盒保存期:6个月需要自备的器材1.仪器:分析天平、水浴锅、台式高速离心机、真空干燥器、PCR扩增仪、电泳仪、电泳槽、紫外凝胶成像仪(或紫外分析仪)、液氮或-70 ℃冰箱、微波炉、组织研磨器、-20 ℃冰箱、可调移液器(2 µL、20 µL、200 µL、1000 µL)。
pCL-Ampho逆病毒载体使用说明

pCL-Ampho 载体简介: 逆病毒包装载体 pCL-Ampho 的使用方法——逆病毒包装与转染方法 293T Growth Medium: 450 ml DMEM (JRH Biosciences) 50 ml FBS (JRH Sciences) 5 ml Glutamine (200 mM in saline, JRH Sciences) 2.5 ml Gentamycin(10 mg/ml in water, Sigma) HC11 Growth Medium: 5 ml L-Glutamine (200 mM in saline, JRH Sciences) 2.5 ml Bovine Insulin (1 mg/ml in 0.01 N HCL, Sigma) 2.5 ml Gentamycin (10 mg/ml in water, Sigma) 0.5 ml EGF (10 μg/ml in water, Gibco) 50 ml FBS (JRH Sciences) 440 ml RPMI (JRH Scieቤተ መጻሕፍቲ ባይዱces) Day 0: Split 293T cells for transfection: Wash cells off plate with 10 ml Hanks and put into 15 ml Falcon tube Add 1 ml 10X trypsin and mix by inversion for about 1-2 minutes Spin at 1000 rpm in clinical centrifuge for 1-2 minutes Resuspend cells in 10 ml growth medium Count cells Plate cells at 1X106 cells per 100 mm dish Day 2: Transfect 293T cells:
金拓思慢病毒产品说明书

金拓思慢病毒产品说明书一、产品简介慢病毒载体是一类重组逆转录病毒载体,由于其结构和功能的特点,慢病毒载体作为一种重要的基因转移工具应用于基因治疗和细胞分子生物学研究领域。
区别于一般的逆转录病毒载体,它对分裂细胞和非分裂细胞均具有感染能力。
该载体可以将外源基因有效地整合到宿主染色体上,从而达到持久性表达。
在感染能力方面可有效地感染神经元细胞、肝细胞、心肌细胞、肿瘤细胞、内皮细胞、干细胞等多种类型细胞,达到良好的基因治疗效果。
我公司生产的重组慢病毒均以国际通用的第三代载体四质粒体系生产,通过重组改造后将含有目的基因的慢病毒骨架及其相应的作用元件组合为新质粒,并通过辅助质粒将病毒包装的元件组成重组病毒。
通过自我灭活的方式,阻止病毒自我复制。
保证了慢病毒使用过程中的良好生物安全性。
二、重要说明2.1安全操作说明1.应在Ⅱ级及以上级别生物安全柜中使用慢病毒产品。
2.虽经过改造后病毒安全性极大提高,操作中仍需佩戴口罩、手套等安全防护措施以免产生潜在危害。
3.操作中所有接触慢病毒试剂的样品、耗材、器皿等均需通过84消毒液(1:20)浸泡后,高温121℃灭活15min以上。
4.实验过程中有病毒液洒落的情况时,应用纸巾将病毒液吸干后喷洒70乙醇,并将擦干的纸巾一并高温处理以免造成其它伤害、污染环境。
2.2使用注意事项所有慢病毒产品均通过干冰低温运输,请收到产品后立即转入-80℃冰箱保存。
每次使用时提前取出病毒液放置在4℃冰箱待融化,并保存于4℃冰箱。
每次融化后请尽快使用,病毒液应尽量避免反复冻融降低病毒滴度。
三、慢病毒制备与使用3.1实验材料细胞:人贴壁细胞293T培养基:高糖DMEM培养基血清:胎牛血清抗生素:青链霉素转染试剂:Transfection-mate增强剂:Polybrene3.2病毒制备方法3.2.1细胞准备细胞复苏:1.将液氮保存细胞取出后,迅速放入37℃水浴锅内,应及时轻柔摇动加快解冻速度。
2.将完全溶解的细胞离心,1500rpm,3min。
反转录试剂盒说明书

行业文档TaKaRa Code:DRR014APrimeScript™ RT-PCR Kit(50次量)目录内容页码●制品说明 1●制品内容 1●保 存 2●原 理 2●试剂盒特点 3●RNA样品制备 3●使用注意 4●反转录引物的选择 5●实验操作 5●实验例 7 ●Q&A8●制品说明PCR(Polymerase Chain Reaction;聚合酶链式反应)是一种体外扩增DNA的简单而有效的方法。
虽然原理上PCR法是扩增DNA,RNA不能直接被扩增,但是经过反转录酶的作用把RNA反转录成cDNA 后,PCR法便可应用于RNA的解析了。
迄今为止,此方法已广泛应用于RNA的构造解析、cDNA的克隆及RNA水平上的表达解析等多种领域。
PrimeScript™ RT-PCR Kit是具有良好的延伸性能与高扩增效率的2 Step RT-PCR试剂盒。
反转录反应使用了TaKaRa独自开发的新型反转录酶PrimeScript™ RTase;PCR反应使用了扩增性能良好的Hot Start 型DNA聚合酶TaKaRa Ex Taq TM HS。
本试剂盒具有以下优点:1.进行高效的RT-PCR扩增。
2.在标准的RNA反转录反应温度(42℃)下,便可使具有复杂结构的RNA进行良好的延伸,可以避免RNA在高温条件下的降解。
3.能有效抑制非特异性的PCR扩增。
4.使用本试剂盒扩增得到的目的产物3′端附有一个A碱基,可以直接克隆于T-Vector中。
本试剂盒含有反转录反应及PCR扩增反应所需的全部试剂。
●制品内容(50次量*1)1.PrimeScript™ RTase(for 2 Step)2.5×PrimeScript™ Buffer3.RNase Inhibitor(40 U/μl)4.dNTP Mixture(10 mM each)5.Oligo dT Primer(2.5 μM)6.Random 6 mers(20 μM)7.TaKaRa Ex Taq TM HS(5 U/μl)8.10×PCR BufferⅡ9.Control F-1 Primer*2(20 μM)10.Control R-1 Primer*3(20 μM)11.Positive Control RNA(2×105 copies/μl)12. RNase Free dH2O25 μl 200 μl 25 μl 150 μl 50 μl 50 μl 25 μl 250 μl 10 μl 10 μl 20 μl 1 ml*1 反转录反应20 μl 、PCR反应50 μl体系时可使用50次。
慢病毒生产及使用操作手册

慢病毒生产及使用操作手册一、实验流程制备慢病毒穿梭质粒及其辅助包装原件载体质粒,三种质粒载体分别进行高纯度无内毒素抽提,共转染293T细胞,转染后6 h 更换为完全培养基,培养48和72h后,分别收集富含慢病毒颗粒的细胞上清液,病毒上清液通过超离心浓缩病毒。
以下内容由汉恒生物科技(上海)有限公司精心整理总结。
二、实验材料(一)慢病毒载体、包装细胞和菌株该病毒包装系统为三质粒系统,组成为pspax2, pMD2G,pHBLV TM系列质粒.1、载体信息(见附录)2、细胞株 293T,慢病毒的包装细胞,为贴壁依赖型成上皮样细胞,生长培养基为DMEM (含10% FBS).贴壁细胞经培养生长增殖形成单层细胞。
3、菌株大肠杆菌菌株DH5α。
用于扩增慢病毒载体和辅助包装载体质粒。
三、包装细胞293T细胞的培养(一) 293T细胞的冻存随着传代的次数增加,293T细胞会出现生长状态下降、突变等。
为了防止此类现象的出现,我们需要在开始就对细胞进行大量冻存,以保证实验的稳定性和持续性。
在细胞对数生长期进行冻存,增加细胞复苏成活率。
1、去掉上清液,加入PBS洗去残留的培养基;2、加入0。
25%的胰酶,消化1~2min后,镜下观察细胞变圆,细胞间间隙加大时,去除胰酶,加入新鲜培养基吹打混匀,移入离心管中.3、细胞计数,将细胞全部晃下,加入 3mL 37 ℃预热的 10% DMEM,用 10mL 移液管进行吹打,较大力吹打 6~8 次即可,不留死角,之后,将所有细胞吸出,置于15mL 离心管中,取 50ul 混匀后的细胞于 1.5mL eppendorf 管中,加入 450ul 10% DMEM,即为 10 倍稀释,混匀,取 10ul 细胞于计数板中计数。
计数板上共 4 大格,每大格 16 小格。
计数时,4 大格均计数,总数除以 4(得每大格细胞数),再乘以 10(10 倍稀释),即为实际 n万/mL 细胞浓度.4、细胞离心,1000rpm,5min。
非洲猪瘟病毒荧光PCR核酸检测试剂盒风途说明书

非洲猪瘟病毒荧光PCR核酸检测试剂盒说明书【兽药名称】通用名称:非洲猪瘟病毒荧光PCR核酸检测试剂盒汉语拼音:Feizliouzhuwen Bingdu Yingguang PCR Hesuan Jianceshijihe英文名称:African swine Fever Virus (ASFV) PCR Nucleic Acid Diagnostic Kit 商品名称:无【用法与判定】1 用法1.1待检样品釆集、保存及运输1.1.1样品釆集(1)活猪样品无菌采集抗凝血或血清5mL(2)病死猪剖检样品或屠宰场剖检样品无菌采集死猪的牌、肺、肾、扁桃体、淋巴结、肌肉等组织样品。
2〜8°C低温运至实验室用于检测。
(3)病猪污染的周边环境采集与病猪相关场所的粪便、饲料、污水样品。
2〜8°C低温运至实验室用于检测。
1.1.2样品保存采集的样品在2〜8°C保存应不超过24小时,-70°C条件下保存,避免反复冻融。
1.1.3样品运输泡沫箱加冰袋后密封进行运输。
包装和运输应符合农业农村部《高致病性动物病原微生物菌(毒)种或者样品运输包装规范》和交通运输部门关于危险品运输管理的有关规定。
1.2样品处理1.2.1血液样品处理抗凝血样品置于离心管中,8000 r/min离心2分钟取上层血浆,编号待检。
非抗凝血样品置于离心管中,待凝固后,8000 r/min离心2分钟取200μL上层血清,编号待检。
1.2.2组织样品处理取适量脾脏、肝脏、淋巴结、扁桃体、肌肉等组织,置于研磨器或研磨管中研磨,再加适量生理盐水混匀,制成约10%的组织匀浆,8000 r/min离心2分钟,取200μL上清于RNase/DNase-free无菌离心管中,编号备用。
1.2.3环境样品处理1.2.3.1粪便、饲料样品处理方法取适量的粪便、饲料放入盛有PBS缓冲液的研磨管中研磨混匀,制成约10%的匀浆液.8000 r/min离心2分钟,取200μL上清于RNase/DNase-free无菌离心管中,编号备用。
反转录PCR操作手册1

RT-PCR实验步骤一.实验器具:1.移液枪:1ml、200μl、20μl、10μl、2.5μl2.吸头:1ml、200μl、20μl3.匀浆管:5ml4.EP管:1.5ml、500μl、200μl5.试剂瓶:棕色试剂瓶(广口,放75%乙醇)6.量筒:100ml7.容量瓶:1000ml8.试管架:5ml、1.5ml、20μl9.铝制饭盒:1-2个10.大瓷缸:1个11.锡泊纸:一卷12.卷纸:2卷13.三角烧瓶:带盖,稍大二.实验器具处理1.塑料制品:(包括枪头、EP管、匀浆管等)先将DEPC水从容量瓶中倒入瓷缸中,将塑料制品逐个浸泡其中(注意:小枪头要充分浸泡,必要时需要针筒打入DEPC水),过夜后取出(注意:DEPC水很难自然晾干,所以建议先将刚取出的DEPC物品甩干,枪头装入枪头盒后甩干,EP管和匀浆管装入饭盒后甩干,然后在37度孵箱烘干)。
高压后烤干备用。
如果DEPC处理过的物品时间已超过一个星期,再次使用前应再次高压。
2.玻璃制品:泡酸过夜,冲洗干净,先泡1‰DEPC过夜,再蒙锡纸烤干备用。
3.匀浆器:(包括剪刀、镊子)先洗净后,超声消毒即可(不需要泡DEPC)。
三.试剂配制1.DEPC水:吸出1ml DEPC放在1000ml容量瓶中加双蒸水定容至1000ml,配成1‰DEPC水,并充分振荡混匀备用。
2.75%乙醇:用无水乙醇+DEPC水配,然后放-20℃保存(DEPC水需先高压,高压时注意:1.装DEPC水的瓶子在盖子和瓶之间要放上线头;2.高压时间在40-45分钟,所以水要适当多加一点;3.高压结束后,不要强行放气,要让压力自然下降,不然水会喷出)。
3.异丙醇:放入棕色瓶中。
4.氯仿:放入棕色瓶中。
5.琼脂糖四.缓冲液配制1.电泳缓冲液(5×TBE贮存液):Tris 54g、硼酸 27.5g、0.5M EDTA 20ml pH8.0、加蒸溜水至1000ml。
使用时,将5×TBE稀释10倍成0.5×TBE就可以在电泳时使用(工作浓度) 2.上样缓冲液(6×缓冲液,4℃保存):0.25%溴酚蓝、0.25%二甲苯青FF、30%甘油五.琼脂糖凝胶配制1.1.0%:1.0g琼脂糖+100ml电泳缓冲液,微波炉加热至沸腾(如果首次煮胶,一定要煮透,以不出现泡沫为标志),熔化的琼脂物冷却至60℃时加入10mg/ml溴化乙锭2.5μl,充分混匀,将温热的凝胶倒入已置好梳子的胶膜中,在室温下放置30-45min(可以放入4度冰箱加快琼脂糖凝固)后现进行电泳。
小反刍兽疫病毒(PPRV) 核酸扩增检测试剂盒说明书

小反刍兽疫病毒(PPRV)核酸扩增检测试剂盒说明书【名称】中文名:小反刍兽疫病毒(PPRV)核酸检测试剂盒英文名:Peste des Petits Ruminants Virus Fluorescence Quantitative Polymerase Chain Reaction(PCR)Diagnostic Kit汉语拼音:xiao fan chu shou yi bing du he suan jian ce shi ji he【目的】本试剂盒适用于检测动物肺、淋巴结、血清等标本中小反刍兽疫病毒(PPRV)mRNA,适用于小反刍兽疫病毒感染的辅助诊断。
其检测结果仅供参考。
【原理】本试剂盒用一对小反刍兽疫病毒特异性引物,Trizol提取RNA,经逆转录酶作用将RNA转录成cDNA,后者在Taq酶作用下,配以SD-Buffer(内含Mg2+、Tris-HCl等)、四种核苷酸单体(dNTPs)等成分,用RT-PCR体外扩增法对小反刍兽疫病毒RNA进行扩增,产物经凝胶电泳,溴化乙锭(EB)染色,紫外灯观察,从而达到快速检测之目的。
【组成】名称数量规格PPRV RT MIX2管140μl/管逆转录酶系1管40μl/管PPRV-PCR反应管40管50μl/管PPRV阳性质控品1管50μl/管阴性质控品1管250μl/管DEPC水1管2000μl/管备注:Trizol等另行配备【标本采集】1.适用标本类型:动物肺、淋巴结、血清或其他组织等2.标本采集动物肺、淋巴结:无菌条件下取动物肺、淋巴结或其他组织等100mg左右,置入1.5ml洁净EP管,立即送检。
血清:用一次性无菌注射器抽取受检动物耳静脉或者前腔静脉静脉血2-5ml,静置斜放待血清析出,1,0000rpm离心3分钟,可取上清立即用于测试;或吸取上部血清(约200µl)转入无菌1.5ml Eppendoff管,保存待检。
【保存和运输】上述标本短期内可保存于-20℃,长期保存可置-70℃。
pCold-ProS2载体说明

pCold-ProS2编号 载体名称北京华越洋生物VECT4580 pCold-‐ProS2pCold-‐ProS2载体基本信息载体名称: pCold-‐ProS2质粒类型: 大肠杆菌表达载体高拷贝/低拷贝: 高拷贝启动子: C SPA克隆方法: 多克隆位点,限制性内切酶载体大小: 5025 b p5' 测序引物及序列: pCold-‐F:5'-‐ACGCCATATCGCCGAAAGG-‐3' 3' 测序引物及序列: pCold-‐R:5'-‐GGCAGGGATCTTAGATTCTG-‐3' 载体标签: ProS2载体抗性: 氨苄青霉素(Ampicillin)筛选标记: -‐-‐备注: -‐-‐稳定性: -‐-‐组成型: 诱导型表达病毒/非病毒: 非病毒pCold-‐ProS2载体质粒图谱和多克隆位点信息pCold-‐ProS2载体简介pCold P roS2 D NA 是一种融合的冷休克表达载体,使用来源于革兰氏阴性菌Myxococcus xanthus中的Protein S作为可溶性标签。
这种Protein S存在于粘细菌的孢子衣中,由173个氨基酸残基组成,是一种非常稳定的可溶性蛋白质。
将两个Protein S的N-‐末端结构域串联,再结合ProS2 T ag的作用,就能使融合目的蛋白的稳定性和可溶性得到提高。
此外,由于目的蛋白和ProS2-‐Tag的相互作用性很低,因此可以将二者有效分离。
pCold P roS2 DNA Vector在cspA启动子的下游插入了5’非编码区(5’-‐UTR)、翻译增强元件(TEE)、His-‐Tag序列、ProS2-‐Tag和多克隆位点(MCS)。
猪托克特诺病毒 核酸扩增检测试剂盒说明书
Cat SA-42121TTV(DNA)2010第四版1/2猪托克特诺病毒(TTVTTV))核酸扩增检测试剂盒说明书(PCR-荧光探针法)【名称】中文名:猪托克特诺病毒(TTV)核酸扩增检测试剂盒英文名:Porcine Torque teno virus Fluorescence Quantitative Polymerase Chain Reaction (PCR)Diagnostic Kit汉语拼音:zhu tuo ke te nuo bing du he suan kuo zeng jian ce shi ji he【目的】本试剂盒适用于检测猪血清或淋巴结等可疑组织标本中猪托克特诺病毒(TTV)DNA ,适用于猪托克特诺病毒(包括TTV1型和TTV2型)感染的辅助诊断及流行病学调查。
其检测结果仅供参考。
【原理】本试剂盒选取两对猪托克特诺病毒特异性引物和两条特异性荧光探针(包括TTV1型和TTV2型),通过碱裂解法提取猪托克特诺病毒(TTV)DNA ,后者在耐热DNA 聚合酶(Taq 酶)作用下,配以FQ-Buffer(内含Mg 2+、Tris-HCl 等)、四种核苷酸单体(dNTPs)等成分,通过PCR-荧光探针体外扩增法对猪托克特诺病毒(TTV)进行扩增,从而达到实时快速定量检测之目的。
【组成】名称数量规格核酸提取液B4管500μl/管Taq 酶系1管80μl/管TTV PCR MIX1管960μl/管TTV 阳性质控品1管50μl/管(1×107Copies/ml)阴性质控品1管500μl/管【标本采集、保存和运输】1.适用标本类型:猪血清或淋巴结等组织。
2.标本采集组织:无菌条件下取猪疑似组织等1g 左右,置入1.5ml 洁净EP 管,立即送检。
血清:用一次性无菌注射器抽取受检猪耳静脉或者前腔静脉静脉血2-5ml ,静置斜放待血清析出,1,0000rpm 离心3分钟,可取上清立即用于测试;或吸取上部血清(约200µl)转入无菌1.5ml Eppendoff 管,保存待检。
TB Green Premix Ex Taq II (Tli RNaseH Plus) 说明书

Code No. RR82LR 研究用TB Green®Premix Ex Taq™ II(Tli RNaseH Plus), ROX plus说明书v202203Da目录内容页码●制品说明 1 ●试剂盒原理 1 ●制品内容 1 ●保存 2 ●试剂盒外必备主要试剂和仪器 2 ●使用注意 2 ●操作方法 2 ●附录 4 ●关联产品7●制品说明本制品是采用TB Green嵌合荧光法进行Real Time PCR的专用试剂。
制品中含有Real Time PCR反应的最适浓度TB Green和ROX Reference Dye,是一种2X浓度的Premix Type试剂,进行实验时,PCR反应液的配制十分方便简单。
本制品中还添加了Tli RNaseH(耐热性RNaseH),以cDNA作为模板进行PCR反应时,可以很好抑制由于cDNA中残存mRNA对PCR反应造成的阻害作用。
本制品Buffer经过改良,反应特异性更高,能抑制非特异性反应,可以在宽广的范围内进行更加准确的定量。
本Buffer和Hot Start法用DNA聚合酶TaKaRa Ex Taq® HS组合使用,可以进行重复性好、可信度高的Real Time PCR解析。
适用的Real Time PCR扩增仪◆需要使用ROX Reference Dye进行信号校正的仪器*∙ StepOnePlus Real-Time PCR System (Thermo Fisher Scientific)◆不需要使用ROX Reference Dye进行信号校正的仪器∙ Thermal Cycler Dice™ Real Time System III (Code No. TP950/TP970/TP980/TP990)∙ Thermal Cycler Dice Real Time System II(Code No. TP900/TP960:终卖)∙ Thermal Cycler Dice Real Time System Lite (Code No. TP700/TP760:终卖)(注)使用Smart Cycler System/Smart Cycler II System (Cepheid)时,建议使用TB Green Premix Ex Taq (Tli RNaseH Plus) (Code No. RR420A/B/L/W、RR42LR/WR) *:对于Applied Biosystems 7500/7500 Fast Real-Time PCR System (Thermo Fisher Scientific),需要使用ROX Reference Dye II校正,推荐使用TB Green Premix Ex TaqII(Tli RNaseH Plus), Bulk (Code No. RR820L/W)。
碧云天 L31600 pLenti-TLR2-sgRNA 产品说明书
pLenti-TLR2-sgRNA产品简介:pLenti-TLR2-sgRNA (TLR2基因敲除质粒)是一种在动物细胞中可以同时表达Cas9、目的基因的sgRNA 和puromycin 抗性基因的质粒。
用于在动物细胞中直接基于CRISPR/Cas9技术敲除目的基因,或者通过包装慢病毒后基于CRISPR/Cas9技术敲除目的基因。
本质粒中sgRNA 的有效性已经通过T7EI 法的验证。
本质粒在细菌中为Amp 抗性,全长约13,000bp 。
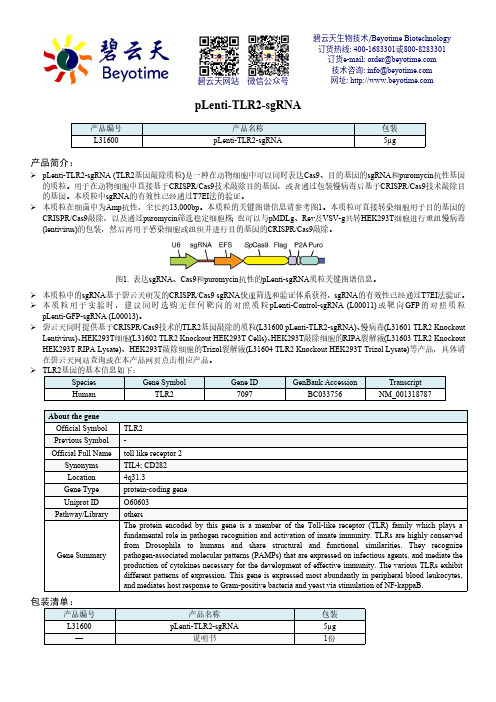
本质粒的关键图谱信息请参考图1。
本质粒可直接转染细胞用于目的基因的CRISPR/Cas9敲除,以及通过puromycin 筛选稳定细胞株;也可以与pMDLg 、Rev 及VSV-g 共转HEK293T 细胞进行重组慢病毒(lentivirus)的包装,然后再用于感染细胞或组织并进行目的基因的CRISPR/Cas9敲除。
图1. 表达sgRNA 、Cas9和puromycin 抗性的pLenti-sgRNA 质粒关键图谱信息。
本质粒中的sgRNA 基于碧云天研发的CRISPR/Cas9 sgRNA 快速筛选和验证体系获得,sgRNA 的有效性已经通过T7EI 法验证。
本质粒用于实验时,建议同时选购无任何靶向的对照质粒pLenti-Control-sgRNA (L00011)或靶向GFP 的对照质粒pLenti-GFP-sgRNA (L00013)。
碧云天同时提供基于CRISPR/Cas9技术的TLR2基因敲除的质粒(L31600 pLenti-TLR2-sgRNA)、慢病毒(L31601 TLR2 Knockout Lentivirus)、HEK293T 细胞(L31602 TLR2 Knockout HEK293T Cells)、HEK293T 敲除细胞的RIPA 裂解液(L31603 TLR2 Knockout HEK293T RIPA Lysate)、HEK293T 敲除细胞的Trizol 裂解液(L31604 TLR2 Knockout HEK293T Trizol Lysate)等产品,具体请在碧云天网站查询或在本产品网页点击相应产品。
Premix Ex Taq TM(Probe qPCR)说明书宝生物工程公司Premix Ex Taq TM(Probe qPCR)说明书
Takara Code :DRR390APremix Ex Taq TM(Probe qPCR) (200次量)宝生物工程(大连)有限公司目录内容页码●制品说明1●制品内容 1 ●适用的Real Time PCR扩增仪1●保存 1 ●试剂盒原理 1 ●试剂盒特长 2 ●操作注意 2 ●操作方法 2 ◆应用Thermal Cycler Dice Real Time System扩增仪的操作方法2◆应用ABI PRISM 7000/7700/7300 Real-Time PCR System,StepOnePlus Real-Time PCR System的操作方法4◆应用ABI PRISM 7500/7500 Fast Real-Time PCR System的操作方法 5 ◆应用LightCycler Real Time PCR扩增仪的操作方法7◆应用Smart Cycler II System Real Time PCR扩增仪的操作方法8 ●PCR反应条件说明9 ●进行RT-PCR反应时的实验方法9 ●引物设计说明10 ●特别提示:本公司提供用于基因表达定量分析的引物设计及合成服务11 ●问答11●制品说明本制品是采用探针法(TaqMan®,Molecular Beacon等)进行Real Time PCR(qPCR)反应的专用试剂。
是一种2×浓度的Premix Type试剂,进行实验时,PCR反应液的配制十分方便简单。
Mix中添加了Tli RNaseH(耐热性RNaseH),以cDNA作为模板进行PCR反应时,可以最大限度抑制由于cDNA中残存mRNA对PCR反应造成的阻害作用。
制品中的DNA聚合酶使用了改良后的Hot Start法用DNA聚合酶TaKaRa Ex Taq HS,与Takara精心研制的Real Time PCR用Buffer组合使用,可以有效抑制非特异性的PCR扩增,大大提高PCR的扩增效率,进行高灵敏度的Real Time PCR扩增反应。
invitrog逆转录试剂盒说明书

ThermoScript ™ RT-PCR SystemCatalog nos. (25 reactions): Catalog nos. (100 reactions): 11146-02411146-01611146-057 (w/ Platinum ® Taq DNA polymerase) 11146-032 (w/ Platinum ® Taq DNA polymerase)11146-040 (w/ Platinum ® Taq DNA polymerase High Fidelity)Store at -20°C (stability can be extended by storing at -70°C)DescriptionThe ThermoScript ™ RT-PCR System is designed for the sensitive and reproducible detection and analysis of RNA molecules in a two-step process. ThermoScript ™ RT, an avian reverse transcriptase with reduced RNase H activity, is engineered to have higher thermal stability,produce higher yields of cDNA, and produce more full-length cDNA transcripts than AMV RT. cDNA synthesis is performed in the first step using either total RNA or poly(A)+-selected RNA primed with oligo(dT), random primers or a gene-specific primer, at 50-65°C. In the second step, PCR is performed in a separate tube using primers specific for the gene of interest. RNA targets from 100 bp to >12 kb can be detected with this system, using 10 pg to 5 Yg of total RNA. PCR is carried out with Platinum ® Taq DNA Polymerase or Platinum ® Taq DNA Polymerase High Fidelity. Platinum ® Taq DNA Polymerase High Fidelity is suitable for templates from 100 bp to >12 kb. Platinum ® Taq DNA polymerase (1) provides automatic hot-start conditions for increased specificity up to 3 kb.Reagents are provided for 25 or 100 cDNA synthesis reactions of 20 μl each and 25 or 100 amplification reactions of 50 μl each.Component 25 rxn kit 100 rxn kit ThermoScript ™ RT (15 U/Yl) 25 μl 100 μl 5X cDNA Synthesis Buffer* 500 μl 500 μl 0.1 M DTT 250 μl 250 μl 10 mM dNTP Mix 100 μl 2 × 250 μlRNaseOUT ™ (40 U/Yl) 25 μl 100 μlOligo (dT)20 (50 YM) 25 μl 100 μlRandom Hexamers (50 ng/Yl) 50 μl 250 μlDEPC-Treated Water 1.25 ml 1.25 mlE . coli RNase H (2 U/Yl) 50 μl 2 × 50 μl*250 mM Tris acetate (pH 8.4), 375 mM potassium acetate, 40 mMmagnesium acetate, stabilizerCatalog numbers 11146-057 (25 rxns) and 11146-032 (100 rxns) include the following, in addition to the components to the left:Component 25 rxn kit 100 rxn kitPlatinum ® Taq DNA polymerase (5 U/Yl) 100 units 250 units10X PCR buffer Minus Mg 1.0 ml 1.0 ml 50 mM MgCl 2 1.0 ml 1.0 ml Catalog number 11146-040 (100 rxns) includes the following, in addition to the components to the left: Component 100 rxn kit Platinum ®Taq DNA Polymerase High Fidelity (5 U/μl) 100 units 10X High Fidelity PCR Buffer 1.0 ml 50 mM MgSO 4 1.0 ml Quality ControlThe Certificate of Analysis (CofA) provides detailed quality control information for each product. The CofA is available on our website at/cofa, and is searchable by product lot number, which is printed on each box .Summary of ProcedurePart no. 11146.pps MAN0000941 Rev. date: 11 Jun 201020 GSPr andom hexame r C, 30-60 min 50-65°C, 30-60 min 25°C, 10 min ↓ 50-60°C, 20-50 min↓85°C, 5 min↓ Add 1 μl RNase H 37°C, 20 min ↓ Remove 2 μl aliquot for PCRWater 1 *If less than 1 ng of RNA is used, reduce the amount of ThermoScript RT in the reaction to 0.5 μl. For technical support, email tech_support@. For country-specific contact information, visit .Page 2 of 4 Important Parameters to ConsiderRNA•High quality intact RNA is essential for successful full-length cDNA synthesis and successful long RT-PCR.•RNA should be devoid of any RNase contamination and aseptic conditions should be maintained. •Recommended methods of total RNA isolation include the Micro-to-Midi Total RNA PurificationSystem (Catalog no. 12183-018) and TRIzol® Reagent(Catalog no. 15596-026) (2, 3). Oligo(dT)-selection forpoly(A)+ RNA is typically not necessary, althoughincorporating this step may improve the yield ofspecific cDNAs.cDNA Synthesis Primers•Oligo(dT)20 (50 pmoles/reaction) is recommended for priming polyadenylated RNA. Use of Oligo(dT)20allows the detection of multiple transcripts from asingle first-strand reaction.•Random hexamers (50-250 ng/reaction) are efficient primers for the detection of multiple short RT-PCRtargets. Use of more than 50-100 ng primer/Yg ofRNA can increase the yield of short products but may inhibit detection of long targets (>3kb) or raretranscripts. If random hexamers are used, the first-strand reaction must be incubated at 25°C for 10 minto extend the primers prior to increasing the reactiontemperature for synthesis.•Gene-specific primers (GSP) should be used at 10 to20 pmol/reaction. Specificity of priming may beimproved by optimizing annealing/reactiontemperature.•Treatment of cDNA with RNase H to remove the complementary RNA prior to PCR is optional. RNaseH digestion will improve the RT-PCR signal of manytargets and is required for the efficient and consistentamplification of long RT-PCR templates.cDNA Synthesis Reaction•Denaturation of the RNA template and primer by incubating at 65°C for 5 min is optional. Most targetscan be reverse transcribed efficiently without this step.However, heating the RNA in the absence of reactionbuffer and enzyme prior to cDNA synthesis canremove secondary structure that may impede full-length cDNA synthesis.•ThermoScript™ RT can be used at 50-65°C. We recommend incubation at 50-55°C for most RT-PCRtargets. However, incubation at 50-60°C for oligo(dT)and 50-65°C for gene-specific primers can beemployed to reduce secondary structure or to improve specificity.•Most targets can be amplified after only a 30-min incubation for the first-strand reaction. Rare RNAs,long transcripts, or targets at the 5′ end of longtranscripts benefit from longer incubation times (50-60 min).PCR Primers• A final primer concentration of 0.2 - 0.4 μM for each primer is generally optimal; however, a primertitration is recommended for best results.•Design primers that anneal to sequence in exons on both sides of an intron or exon/exon boundary of the mRNA to allow differentiation between amplification of cDNA and potential contaminating genomic DNA. •Primers should not be self-complementary or complementary to each other at the 3′ ends.PCR Reactions•Most targets will be efficiently amplified using 2 Yl or less of the cDNA synthesis reaction.•The optimum magnesium concentration varies from1.5 to 3 mM. Generally, 1.82 mM magnesium chloridefor Platinum® Taq DNA polymerase and 2.32 mMmagnesium sulfate for Platinum® Taq DNAPolymerase High Fidelity is effective for most primer sets. However, titration of the magnesiumconcentration is recommended for the best result.Each Yl of the cDNA synthesis reaction adds 0.16 mM to the final magnesium concentration in a 50-μl PCRreaction.•Assemble the PCR reactions on ice, transfer them to a pre-heated thermal cycler (85-95°C) and immediately start the PCR amplification program.•The annealing temperature should be 10°C below the melting temperature of the primers used.•The optimum extension temperature for Platinum®Taq DNA Polymerase High Fidelity is 68°C. Theextension time varies with the size of the amplicon(approximately 1 min per 1 kb of amplicon).Page 3 of 4cDNA Synthesis1. In a 0.2- or 0.5-ml tube, combine primer (50 μM Oligo(dT)20,50 ng/μl random primer or 10 μM gene-specific primer), RNA, and dNTP mix and adjust volume to 12 Yl withDEPC-treated water.Component AmountPrimer 1 YlRNA (10 pg -5 Yg) x Yl10 mM dNTP Mix 2 YlDEPC-treated water to 12 Yl2. Denature RNA and primer by incubating at 65°C for 5 minand then place on ice (optional).3. Vortex the 5X cDNA Synthesis Buffer for 5 s just prior touse.4. Prepare a master reaction mix on ice and vortex gently.Component 1 Reaction 10 Reactions5x cDNA Synthesis Buffer 4 Yl 40 Yl0.1 M DTT 1 Yl 10 YlRNaseOUT™ (40 U/Yl) 1 Yl 10 YlDEPC-treated water 1 Yl 10 YlThermoScript™ RT (15 units/Yl)1 Yl* 10 Yl**NOTE: If less than 1 ng of template RNA is used, reduce the amount of ThermoScript™ RT in the reaction to 0.5 μl/reaction(5 μl/10 reactions). Increase the amount of DEPC-treatedwater in the master reaction mix to 1.5 μl/reaction (15 μl/10 reactions).5. Pipet 8 Yl of master reaction mix into each reaction tube on ice.6. Transfer the sample to a thermal cycler preheated to the appropriate cDNA synthesis temperature and incubate as follows.Oligo(dT)20 primed: 30-60 min at 50°C (or 50-60°C)Gene-specific primed 30-60 min at 50°C (or 50-65°C) Random-hexamer primed: 25°C for 10 min, followed by 20-50 min at 50°C (or 50-65°C)7. Terminate the reaction by incubating at 85°C for 5 min.8. Add 1 Yl of RNase H and incubate at 37°C for 20 min (optional).9. cDNA synthesis reactions can be stored at -20°C or used for PCR immediately.PCR with Platinum®Taq DNA Polymerase High FidelityUse only 10% of the cDNA synthesis reaction (2 Yl) for PCR.Use of 2 μl of 50 mM MgSO4 and 2 Yl of cDNA (0.32 mM magnesium in a 50-Yl PCR) results in a final concentration of2.32 mM magnesium, which is effective for most primer sets. However, titration of the magnesium concentration with the provided 50 mM MgSO4 is recommended for best results.1. Add the following to a 0.2- or 0.5-ml, thin-walled, PCR tube: Component 1 Reaction 10 Reactions10X High Fidelity PCR Buffer 5 Yl 50 Yl50 mM MgSO4 2μl 20μl10 mM dNTP Mix 1 Yl 10 Yl10 YM sense primer 1 Yl 10 Yl10 YM antisense primer 1 Yl 10 YlPlatinum®Taq High Fidelity 0.2 Yl 2 YlcDNA (from cDNA synthesis reaction) 2 Yl 20 YlDEPC-treated water 37.8 Yl 378 YlFinal volume 50 Yl 500 μl 2. Mix gently and overlay with silicone oil or mineral oil if thethermal cycler lacks a heated lid.3. Incubate at 94°C for 2 min, then perform 20 to 40 cycles ofPCR with optimized conditions for your sample (1 min/kb extension time at 68°C).4. Analyze 10 Yl of the amplified sample by agarose gelelectrophoresis.PCR with Platinum® Taq DNA PolymeraseUse only 10% of the cDNA synthesis reaction (2 Yl) for PCR. Use of 50 mM MgCl2 and 2 Yl of cDNA will result in a final magnesium concentration of 1.82 mM, which is adequate for most primers and targets. However, titration of magnesium concentration is recommended for best results.1. Add the following to a 0.2- or 0.5-ml, thin-walled, PCR tube: Component 1 Reaction 10 Reactions 10X PCR Buffer Minus Mg 5 Yl 50 Yl50 mM MgCl2 1.5 Yl 15 Yl10 mM dNTP Mix 1 Yl 10 Yl10 YM sense primer 1 Yl 10 Yl10 YM antisense primer 1 Yl 10 YlPlatinum®Taq DNA polymerase 0.4 Yl 4 Yl(5 U /Yl)cDNA (from cDNA synthesis reaction) 2 Yl 20 Yl DEPC-treated water 38.1 Yl 381 Yl Final volume 50 Yl 500 μl 2. Mix gently and overlay with silicone oil or mineral oil if thethermal cycler lacks a heated lid.3. Incubate at 94°C for 2 min, then perform 20 to 40 cycles ofPCR with optimized conditions for your sample (1 min/kb extension time at 68-72°C)4. Analyze 10 Yl of the amplified sample by agarose gelelectrophoresis.Control ReactionsAn RT-PCR Primer and Control Set is available separately for monitoring the performance of the system (Cat. Number 10929-016).1. Use 1 ng of the Control RNA in the cDNA SynthesisReaction.2. Perform the PCR using Platinum® Taq DNA Polymerase, as described above.Page 4 of 4 Troubleshooting GuideProblem Possible cause Possible solutionNo RT-PCR product No cDNA synthesis (temperature too high) For the cDNA synthesis step, incubate at 45-50°C.Incomplete synthesis of target cDNA (secondary structure of RNA blocks synthesis) For the cDNA synthesis step, incubate at 50-70°C. For long mRNAs, increase cDNA synthesis incubation time (up to 50 min)RNase contamination Maintain aseptic conditions; add RNaseOUT™ (RNaseinhibitor).Concentration of template RNA in reaction is too low Increase the concentration of template RNA; use 1-5 μg of total RNA or reduce the volume of ThermoScript™ RT used in the reaction.RNA has been damaged or degraded Replace RNA.RT inhibitors are present in RNA Remove inhibitors in the RNA preparation by anadditional 70% ethanol wash after ethanolprecipation.Note: Inhibitors of RT include SDS, EDTA,guanidinium chloride, formamide, sodiumphosphate and spermidine (4).Cycle number is too low Increase cycle number.Low yield/low specificity in PCR Reaction conditions not optimal Optimize magnesium concentration.Optimize the primer concentrationOptimize the annealing temperature and extensiontime.Increase temperature of RT reaction to 50-60°C.Unexpected bands after electrophoresis RNA contamination with genomic DNA Pre-treat RNA with DNase I.Redesign PCR primers to anneal to sequence in exonson both sides of an intron in the target gene.References1.Westfall, B., Sitaraman, K., Solus, J., Hughes, J., and Rashtchian, A. (1997) Focus®19, 46.2.Chomczynski, P and Sacchi, N. (1987) Anal. Biochem. 162, 156.3.Chirgwin, J.M., Przybyla, A.E., MacDonald, R.J., and Rutter, W.J. (1979) Biochemistry 18, 5294.4.Gerard, G.F (1994). Focus®16, 102.Limited Use Label License No. 1: Thermostable Polymerases (applies to 11146-057, 11146-032, and 11146-040)Use of this product is covered by one or more of the following US patents and corresponding patent claims outside the US: 5,789,224, 5,618,711, and 6,127,155. The purchase of this product includes a limited, non-transferable immunity from suit under the foregoing patent claims for using only this amount of product for the purchaser’s own internal research. No right under any other patent claim, no right to perform any patented method, and no right to perform commercial services of any kind, including without limitation reporting the results of purchaser's activities for a fee or other commercial consideration, is conveyed expressly, by implication, or by estoppel. This product is for research use only. Diagnostic uses under Roche patents require a separate license from Roche. Further information on purchasinglicenses may be obtained by contacting the Director of Licensing, Applied Biosystems, 850 Lincoln Centre Drive, Foster City, California 94404, USA.Limited Use Label License No. 4: Products for PCR that include no rights to perform PCR (applies to 11146-016 and 11146-024)This product is compatible for use in the Polymerase Chain Reaction (PCR) process claimed in patents owned by Roche Molecular Systems, Inc. and F. Hoffmann-La Roche Ltd. No license under these patents is conveyed expressly, by implication, by estoppel or otherwise to the purchaser by the purchase of this product. A license to use these patented processes for certain research and development activities accompanies the purchase of certain reagents of Applied Biosystems and other licensed suppliers when used in conjunction with an authorized thermal cycler, or is available from Applied Biosystems. Further information on purchasing licenses may be obtained by contacting the Director of Licensing, Applied Biosystems, 850 Lincoln Centre Drive, Foster City, California 94404, USA.Limited Use Label License No: 5: Invitrogen Technology (applies to all)The purchase of this product conveys to the buyer the non-transferable right to use the purchased amount of the product and components of the product in research conducted by the buyer (whether the buyer is an academic or for-profit entity). The buyer cannot sell or otherwise transfer (a) this product (b) its components or (c) materials made using this product or its components to a third party or otherwise use this product or its components or materials made using this product or its components for Commercial Purposes. The buyer may transfer information or materials made through the use of this product to a scientific collaborator, provided that such transfer is not for any Commercial Purpose, and that such collaborator agrees in writing (a) not to transfer such materials to any third party, and (b) to use such transferred materials and/or information solely for research and not for Commercial Purposes. Commercial Purposes means any activity by a party for consideration and may include, but is not limited to: (1) use of the product or its components in manufacturing; (2) use of the product or its components to provide a service, information, or data; (3) use of the product or its components for therapeutic, diagnostic or prophylactic purposes; or (4) resale of the product or its components, whether or not such product or its components are resold for use in research. For products that are subject to multiple limited use label licenses, the terms of the most restrictive limited use label license shall control. Life Technologies Corporation will not assert a claim against the buyer of infringement of patents owned or controlled by Life Technologies Corporation which cover this product based upon the manufacture, use or sale of a therapeutic, clinical diagnostic, vaccine or prophylactic product developed in research by the buyer in which this product or its components was employed, provided that neither this product nor any of its components was used in the manufacture of such product. If the purchaser is not willing to accept the limitations of this limited use statement, Life Technologies is willing to accept return of the product with a full refund. For information about purchasing a license to use this product or the technology embedded in it for any use other than for research use please contact Out Licensing, Life Technologies, 5791 Van Allen Way, Carlsbad, California 92008; Phone (760) 603-7200 or e-mail:outlicensing@.Limited Use Label License No: 14: Direct Inhibition by Anti-Polymerase Antibodies(applies to 11146-057, 11146-032, and 11146-040)Licensed to Life Technologies Corporation, under U.S. Patent Nos. 5,338,671; 5,587,287; and foreign equivalents for use in research onlyLimited Use Label License No. 17: AFLP® Products (applies to 11146-040)The AFLP® technique is covered by patents or patented applications owned by Keygene n.v and this product is sold under license from Keygene n.v. This kit may be used for research purposes only. For use of this kit in plant breeding, contact the Licensing Department, Life Technologies Corporation, 5791 Van Allen Way, Carlsbad, California 92008. Phone (760) 603-7200. Fax (760) 602-6500. The use of this kit for any other purpose, including but not limited to the use for clinical, diagnostic, and/or therapeutic purposes; or for providing services to third parties, requires a license from Keygene n.v., P.O. Box 216, 6700 AE Wageningen, The Netherlands.©2010 Life Technologies Corporation. All rights reserved.For research use only. Not intended for any animal or human therapeutic or diagnostic use.The trademarks mentioned herein are the property of Life Technologies Corporation or their respective owners.。
北京四正柏生物科技有限公司 两步法 RT-PCR 试剂盒 产品说明书 MRT6103-20

两步法RT-PCR试剂盒产品编号规格MRT6103-2020rxn保存条件及效期:-20℃恒温保存一年。
产品描述:本产品是高灵敏度的RT-PCR反应系统,可以从极低量的总RNA或poly(A)mRNA中高效率的合成第一链cDNA,并能够通读GC含量高、二级结构复杂的RNA模板。
该试剂盒使用重组表达的M-MμlV Reverse Transcriptase(RTase),通过定点突变去除了RNase H的活性(RNase H-),并增加了该酶与模板-引物的亲和力,具有更好的热稳定性,因此其DNA聚合酶的活性和持续合成能力均高于野生型M-MμlV RTase,合成的cDNA产量更多、更长。
本试剂盒中的2×HiFiTaq PCR Mix含PCR反应所需的HiFiTaq DNA Polymerase、dNTPs、Mg2+、反应缓冲液和稳定剂等成分并对体系进行了优化,能有效扩增的10kb以下片段,同时使用更快速简便。
产品组分:组分MRT6103-20RTase(200U/μl)20μl5×RTase buffer*200μlRNase inhibitor(RNasin)16μlOligo(dT)18(50M)20μlRandom Primer(50M)20μl10mM dNTP Mix30μl2×HiFiTaq PCR StarMix500μlDNase/RNase-free ddH2O1ml*5×RTase buffer:250mM Tris-HCl(pH8.3),375mM KCl,15mM MgCl2,50mM DTT注意事项:1.实验过程中请注意避免RNase污染。
2.除酶以外的各种试剂,使用之前请完全溶解并充分混匀,以防因盐离子浓度不均影响实验结果。
3.RNA模板的完整性对cDNA合成效率起着决定性作用,因此请选择可靠的RNA提取/纯化方法。
4.由于所有的RNA提取方法都不能完全去除基因组DNA,所以应根据后续实验的需求,选择是否需要在反转录反应前去除残留基因组DNA。
整理)慢病毒稳转细胞株步骤

稳转慢病毒一、所需试剂1、慢病毒载体(详细信息见附录及《质粒的扩增提取》)(大肠杆菌-80℃保存2-3年,质粒-20℃保存2-3年,病毒液-80℃保存1年)(1)载体质粒:两端的LTR、剪切位点、包装信号Ψ以及抗性或荧光基因、gag基因5′端350bp的序列及位于env序列中的RRE,含宿主RNA聚合酶识别部分(2)包装质粒(psPAX2):包含了pol、gag包装成分(3)包膜质粒(pMD2.G):用其他病毒的包膜蛋白代替了env基因.三种质粒共同转染产生不具有自我复制能力的病毒载体。
2、包装细胞:293T细胞3、菌株:大肠杆菌,用于提取质粒4、转染试剂:XTREME-GENE(-20℃保存,不可分装),一种脂质与其他组份构成的混合物5、浓缩试剂(配好后4℃保存,原材料室温保存):5X PEG8000/NaCl溶液(聚乙二醇):NaCl 8.766 g; PEG8000 50g溶解在200ml Milli-Q纯水中,高压蒸汽灭菌**也可直接从公司买来病毒液(-80℃封口膜封口冻存管保存,4℃保存3天):滴度一般为108TU/ml6、10mg/ml polybrene(-20℃分装保存):溴化己二甲铵。
是带正电的小分子,与细胞表面的阴离子结合,提高慢病毒对细胞的感染效率,通常加入polybrene 能提高感染效率2~10 倍。
有一定细胞毒性,需要摸索浓度(1~10μg/ml)7、无血清培养基:optimen8、贴壁细胞(复苏后3代以上的细胞)9、puromycin:嘌呤霉素,用于筛选稳转细胞二、具体步骤<一>病毒包装与收集(中皿,转染步骤类似于瞬转)第一天1、种板,10×105个293T细胞,加入全培养基双抗DMEM 4-5ml,过夜2、配制5X PEG8000/NaCl溶液称取NaCl 8.766 g; PEG8000 50g溶解在200ml Milli-Q纯水中;121摄氏度 30min 湿热灭绝 30min;保存在4℃第二天2、加入2ml全培养基DMEM3、将1加入2,孵育10h,换成5ml全培养基第四天第五天:1、9:00和17:00各收取一次5ml培养液,共20ml(-80℃保存)2、过滤:用孔径为0.45mm的过滤器除去上清中的293T细胞3、加入5ml 5XPEG8000/NaCl溶液,每30min-1h上下摇匀一次4、4℃过夜第六天1、4℃,3500rpm,20min2、弃上清,倒扣纸上静置1-2min,吸干残余液体3、加入120-150μl PBS,缓慢吹打,以防形成气溶胶4、50μl分装,-80℃保存。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
pRetroX-PTuner2-CpRetroX-PTuner2-C 逆病毒载体基本信息:载体名称:pRetroX-PTuner2-C 质粒类型: 逆病毒载体;双顺反子载体高拷贝/低拷贝: 高拷贝克隆方法: 限制性内切酶,多克隆位点启动子:CMV IE 载体大小:7356 bp 5' 测序引物及序列:-- 3' 测序引物及序列:-- 载体标签: DD tag (C-端)载体抗性: 氨苄青霉素筛选标记: 嘌呤霉素(Puromycin )克隆菌株:DH5α , Stellar 宿主细胞(系): 常规细胞系(293、CV-1、CHO 等)备注: 逆病毒载体pRetroX-PTuner2-C 载体表达C 端DD 标签融合蛋白;DD 标签是一个大小为12KD 的FKBP (L106P)去稳定结构域,带有DD 标签的目的蛋白将很快被降解;向培养基中加入Shield1配体后,情况将发生逆转,细胞中目的蛋白的量则迅速提高。
稳定性: 稳表达组成型/诱导型: 组成型病毒/非病毒: 逆转录病毒pRetroX-PTuner2-C 载体质粒图谱和多克隆位点信息:pRetroX-PTuner2-C载体简介pRetroX-PTuner2-C载体序列:ORIGIN1 TCCGCGTTAC ATAACTTACG GTAAATGGCC CGCCTGGCTG ACCGCCCAAC GACCCCCGCC61 CATTGACGTC AATAATGACG TATGTTCCCA TAGTAACGCC AATAGGGACT TTCCATTGAC121 GTCAATGGGT GGAGTATTTA CGGTAAACTG CCCACTTGGC AGTACATCAA GTGTATCATA 181 TGCCAAGTAC GCCCCCTATT GACGTCAATG ACGGTAAATG GCCCGCCTGG CATTATGCCC 241 AGTACATGAC CTTATGGGAC TTTCCTACTT GGCAGTACAT CTACGTATTA GTCATCGCTA301 TTACCATGGT GATGCGGTTT TGGCAGTACA TCAATGGGCG TGGATAGCGG TTTGACTCAC 361 GGGGATTTCC AAGTCTCCAC CCCATTGACG TCAATGGGAG TTTGTTTTGG CACCAAAATC 421 AACGGGACTT TCCAAAATGT CGTAACAACT CCGCCCCATT GACGCAAATG GGCGGTAGGC 481 GTGTACGGTG GGAGGTCTAT ATAAGCAGAG CTCAATAAAA GAGCCCACAA CCCCTCACTC 541 GGCGCGCCAG TCTTCCGATA GACTGCGTCG CCCGGGTACC CGTATTCCCA ATAAAGCCTC 601 TTGCTGTTTG CATCCGAATC GTGGTCTCGC TGTTCCTTGG GAGGGTCTCC TCTGAGTGAT661 TGACTACCCA CGACGGGGGT CTTTCATTTG GGGGCTCGTC CGGGATTTGG AGACCCCTGC 721 CCAGGGACCA CCGACCCACC ACCGGGAGGT AAGCTGGCCA GCAACTTATC TGTGTCTGTC 781 CGATTGTCTA GTGTCTATGT TTGATGTTAT GCGCCTGCGT CTGTACTAGT TAGCTAACTA841 GCTCTGTATC TGGCGGACCC GTGGTGGAAC TGACGAGTTC TGAACACCCG GCCGCAACCC 901 TGGGAGACGT CCCAGGGACT TTGGGGGCCG TTTTTGTGGC CCGACCTGAG GAAGGGAGTC 961 GATGTGGAAT CCGACCCCGT CAGGATATGT GGTTCTGGTA GGAGACGAGA ACCTAAAACA 1021 GTTCCCGCCT CCGTCTGAAT TTTTGCTTTC GGTTTGGAAC CGAAGCCGCG CGTCTTGTCT 1081 GCTGCAGCGC TGCAGCATCG TTCTGTGTTG TCTCTGTCTG ACTGTGTTTC TGTATTTGTC 1141 TGAAAATTAG GGCCAGACTG TTACCACTCC CTTAAGTTTG ACCTTAGGTC ACTGGAAAGA 1201 TGTCGAGCGG ATCGCTCACA ACCAGTCGGT AGATGTCAAG AAGAGACGTT GGGTTACCTT 1261 CTGCTCTGCA GAATGGCCAA CCTTTAACGT CGGATGGCCG CGAGACGGCA CCTTTAACCG 1321 AGACCTCATC ACCCAGGTTA AGATCAAGGT CTTTTCACCT GGCCCGCATG GACACCCAGA 1381 CCAGGTCCCC TACATCGTGA CCTGGGAAGC CTTGGCTTTT GACCCCCCTC CCTGGGTCAA 1441 GCCCTTTGTA CACCCTAAGC CTCCGCCTCC TCTTCCTCCA TCCGCCCCGT CTCTCCCCCT1501 TGAACCTCCT CGTTCGACCC CGCCTCGATC CTCCCTTTAT CCAGCCCTCA CTCCTTCTCT1561 AGGCGCCGGA ATTGAAGATC ATAGTTATTA ATAGTAATCA ATTACGGGGT CATTAGTTCA 1621 TAGCCCATAT ATGGAGTTCC GCGTTACATA ACTTACGGTA AATGGCCCGC CTGGCTGACC 1681 GCCCAACGAC CCCCGCCCAT TGACGTCAAT AATGACGTAT GTTCCCATAG TAACGCCAAT 1741 AGGGACTTTC CATTGACGTC AATGGGTGGA GTATTTACGG TAAACTGCCC ACTTGGCAGT 1801 ACATCAAGTG TATCATATGC CAAGTACGCC CCCTATTGAC GTCAATGACG GTAAATGGCC 1861 CGCCTGGCAT TATGCCCAGT ACATGACCTT ATGGGACTTT CCTACTTGGC AGTACATCTA 1921 CGTATTAGTC ATCGCTATTA CCATGGTGAT GCGGTTTTGG CAGTACATCA ATGGGCGTGG 1981 ATAGCGGTTT GACTCACGGG GATTTCCAAG TCTCCACCCC ATTGACGTCA ATGGGAGTTT 2041 GTTTTGGCAC CAAAATCAAC GGGACTTTCC AAAATGTCGT AACAACTCCG CCCCATTGAC 2101 GCAAATGGGC GGTAGGCGTG TACGGTGGGA GGTCTATATA AGCAGAGCTG GTTTAGTGAA 2161 CCGTCAGATC CGCTAGCGCT ACCGGACTCA GATCTCGAGC TCAAGCTTCG AATTCTGCAG 2221 TCGACGGTAC CGCGGGCCCG GGATCCATCA GTCTGATTGC GGCGTTAGCG GTAGATCACG 2281 TTATCGGCAT GGAAAACGCC ATGCCGTGGA ACCTGCCTGC CGATCTCGCC TGGTTTAAAC 2341 GCAACACCTT AAATAAACCC GTGATTATGG GCCGCCATAC CTGGGAATCA ATCGGTCGTC 2401 CGTTGCCAGG ACGCAAAAAT ATTATCCTCA GCAGTCAACC GAGTACGGAC GATCGCGTAA 2461 CGTGGGTGAA GTCGGTGGAT GAAGCCATCG CGGCGTGTGG TGACGTACCA GAAATCATGG2521 TGATTGGCGG CGGTCGCGTC TATGAACAGC TCCTGCCAAA AGCGCAAAAA CTGTATCTGA 2581 CGCATATCGA CGCAGAAGTG GAAGGCGACA CCCATTTCCC GGATTACGAG CCGGATGACT 2641 GGGAATCGGT ATTCAGCGAA TTCCACGATG CTGATGCGCA GAACTCTCAC AGCTATTGCT 2701 TTGAGATTCT GGAGCGGCGA TGAGCGGCCG CGACTCTAGA TAATTCTACC GGGTAGGGGA 2761 GGCGCTTTTC CCAAGGCAGT CTGGAGCATG CGCTTTAGCA GCCCCGCTGG GCACTTGGCG 2821 CTACACAAGT GGCCTCTGGC CTCGCACACA TTCCACATCC ACCGGTAGGC GCCAACCGGC 2881 TCCGTTCTTT GGTGGCCCCT TCGCGCCACC TTCTACTCCT CCCCTAGTCA GGAAGTTCCC 2941 CCCCGCCCCG CAGCTCGCGT CGTGCAGGAC GTGACAAATG GAAGTAGCAC GTCTCACTAG 3001 TCTCGTGCAG ATGGACAGCA CCGCTGAGCA ATGGAAGCGG GTAGGCCTTT GGGGCAGCGG 3061 CCAATAGCAG CTTTGCTCCT TCGCTTTCTG GGCTCAGAGG CTGGGAAGGG GTGGGTCCGG 3121 GGGCGGGCTC AGGGGCGGGC TCAGGGGCGG GGCGGGCGCC CGAAGGTCCT CCGGAGGCCC 3181 GGCATTCTGC ACGCTTCAAA AGCGCACGTC TGCCGCGCTG TTCTCCTCTT CCTCATCTCC 3241 GGGCCTTTCG ACCTGCAGCC CAAGCTTACC ATGACCGAGT ACAAGCCCAC GGTGCGCCTC 3301 GCCACCCGCG ACGACGTCCC CAGGGCCGTA CGCACCCTCG CCGCCGCGTT CGCCGACTAC 3361 CCCGCCACGC GCCACACCGT CGATCCGGAC CGCCACATCG AGCGGGTCAC CGAGCTGCAA 3421 GAACTCTTCC TCACGCGCGT CGGGCTCGAC ATCGGCAAGG TGTGGGTCGC GGACGACGGC 3481 GCCGCGGTGG CGGTCTGGAC CACGCCGGAG AGCGTCGAAG CGGGGGCGGT GTTCGCCGAG 3541 ATCGGCCCGC GCATGGCCGA GTTGAGCGGT TCCCGGCTGG CCGCGCAGCA ACAGATGGAA 3601 GGCCTCCTGG CGCCGCACCG GCCCAAGGAG CCCGCGTGGT TCCTGGCCAC CGTCGGCGTC 3661 TCGCCCGACC ACCAGGGCAA GGGTCTGGGC AGCGCCGTCG TGCTCCCCGG AGTGGAGGCG 3721 GCCGAGCGCG CCGGGGTGCC CGCCTTCCTG GAGACCTCCG CGCCCCGCAA CCTCCCCTTC 3781 TACGAGCGGC TCGGCTTCAC CGTCACCGCC GACGTCGAGG TGCCCGAAGG ACCGCGCACC 3841 TGGTGCATGA CCCGCAAGCC CGGTGCCTGA CGCCCGCCCC ACGACCCGCA GCGCCCGACC 3901 GAAAGGAGCG CACGACCCCA TGCATCGGCG TCTCGATCGA GATATCAGTG GTCCAGGCTC 3961 TAGTTTTGAC TCAACAATAT CACCAGCTGA AGCCTATAGA GTACGAGCCA TAGATAAAAT 4021 AAAAGATTTT ATTTAGTCTC CAGAAAAAGG GGGGAATGAA AGACCCCACC TGTAGGTTTG 4081 GCAAGCTACT AGCTTAAGTA ACGCCATTTT GCAAGGCATG GAAAAATACA TAACTGAGAA 4141 TAGAGAAGTT CAGATCAAGG TCAGGAACAG ATGGAACAGG GTCGATCGAC CCTAGAGAAC 4201 CATCAGATGT TTCCAGGGTG CCCCAAGGAC CTGAAATGAC CCTGTGCCTT ATTTGAACTA 4261 ACCAATCAGT TCGCTTCTCG CTTCTGTTCG CGCGCTTCTG CTCCCCGAGC TCAATAAAAG 4321 AGCCCACAAC CCCTCACTCG GGGCGCCAGT CCTCCGATTG ACTGAGTCGC CCGGGTACCC 4381 GTGTATCCAA TAAACCCTCT TGCAGTTGCA TCCGACTTGT GGTCTCGCTG TTCCTTGGGA 4441 GGGTCTCCTC TGAGTGATTG ACTACCCGTC AGCGGGGGTC TTTCATTTGG GGGCTCGTCC 4501 GGGATCGGGA GACCCCTGCC CAGGGACCAC CGACCCACCA CCGGGAGGTA AGCTGGCTGC 4561 CTCGCGCGTT TCGGTGATGA CGGTGAAAAC CTCTGACACA TGCAGCTCCC GGAGACGGTC 4621 ACAGCTTGTC TGTAAGCGGA TGCCGGGAGC AGACAAGCCC GTCAGGGCGC GTCAGCGGGT 4681 GTTGGCGGGT GTCGGGGCGC AGCCATGACC CAGTCACGTA GCGATAGCGG AGTGTAGATC 4741 CGGCTGTGGA ATGTGTGTCA GTTAGGGTGT GGAAAGTCCC CAGGCTCCCC AGCAGGCAGA 4801 AGTATGCAAA GCATGCATCT CAATTAGTCA GCAACCAGGT GTGGAAAGTC CCCAGGCTCC 4861 CCAGCAGGCA GAAGTATGCA AAGCATGCAT CTCAATTAGT CAGCAACCAT AGTCCCGCCC 4921 CTAACTCCGC CCATCCCGCC CCTAACTCCG CCCAGTTCCG CCCATTCTCC GCCCCATGGC 4981 TGACTAATTT TTTTTATTTA TGCAGAGGCC GAGGCCGCCT CGGCCTCTGA GCTATTCCAG 5041 AAGTAGTGAG GAGGCTTTTT TGGAGGCCTA GGCTTTTGCA AAAAGCTTAC TGGCTTAACT 5101 ATGCGGCATC AGAGCAGATT GTACTGAGAG TGCACCATAT GCGGTGTGAA ATACCGCACA5161 GATGCGTAAG GAGAAAATAC CGCATCAGGC GCTCTTCCGC TTCCTCGCTC ACTGACTCGC 5221 TGCGCTCGGT CGTTCGGCTG CGGCGAGCGG TATCAGCTCA CTCAAAGGCG GTAATACGGT 5281 TATCCACAGA ATCAGGGGAT AACGCAGGAA AGAACATGTG AGCAAAAGGC CAGCAAAAGG 5341 CCAGGAACCG TAAAAAGGCC GCGTTGCTGG CGTTTTTCCA TAGGCTCCGC CCCCCTGACG 5401 AGCATCACAA AAATCGACGC TCAAGTCAGA GGTGGCGAAA CCCGACAGGA CTATAAAGAT 5461 ACCAGGCGTT TCCCCCTGGA AGCTCCCTCG TGCGCTCTCC TGTTCCGACC CTGCCGCTTA 5521 CCGGATACCT GTCCGCCTTT CTCCCTTCGG GAAGCGTGGC GCTTTCTCAT AGCTCACGCT 5581 GTAGGTATCT CAGTTCGGTG TAGGTCGTTC GCTCCAAGCT GGGCTGTGTG CACGAACCCC 5641 CCGTTCAGCC CGACCGCTGC GCCTTATCCG GTAACTATCG TCTTGAGTCC AACCCGGTAA 5701 GACACGACTT ATCGCCACTG GCAGCAGCCA CTGGTAACAG GATTAGCAGA GCGAGGTATG 5761 TAGGCGGTGC TACAGAGTTC TTGAAGTGGT GGCCTAACTA CGGCTACACT AGAAGGACAG 5821 TATTTGGTAT CTGCGCTCTG CTGAAGCCAG TTACCTTCGG AAAAAGAGTT GGTAGCTCTT 5881 GATCCGGCAA ACAAACCACC GCTGGTAGCG GTGGTTTTTT TGTTTGCAAG CAGCAGATTA 5941 CGCGCAGAAA AAAAGGATCT CAAGAAGATC CTTTGATCTT TTCTACGGGG TCTGACGCTC 6001 AGTGGAACGA AAACTCACGT TAAGGGATTT TGGTCATGAG ATTATCAAAA AGGATCTTCA 6061 CCTAGATCCT TTTAAATTAA AAATGAAGTT TTAAATCAAT CTAAAGTATA TATGAGTAAA6121 CTTGGTCTGA CAGTTACCAA TGCTTAATCA GTGAGGCACC TATCTCAGCG ATCTGTCTAT 6181 TTCGTTCATC CATAGTTGCC TGACTCCCCG TCGTGTAGAT AACTACGATA CGGGAGGGCT 6241 TACCATCTGG CCCCAGTGCT GCAATGATAC CGCGAGACCC ACGCTCACCG GCTCCAGATT 6301 TATCAGCAAT AAACCAGCCA GCCGGAAGGG CCGAGCGCAG AAGTGGTCCT GCAACTTTAT 6361 CCGCCTCCAT CCAGTCTATT AATTGTTGCC GGGAAGCTAG AGTAAGTAGT TCGCCAGTTA 6421 ATAGTTTGCG CAACGTTGTT GCCATTGCTG CAGGCATCGT GGTGTCACGC TCGTCGTTTG 6481 GTATGGCTTC ATTCAGCTCC GGTTCCCAAC GATCAAGGCG AGTTACATGA TCCCCCATGT 6541 TGTGCAAAAA AGCGGTTAGC TCCTTCGGTC CTCCGATCGT TGTCAGAAGT AAGTTGGCCG 6601 CAGTGTTATC ACTCATGGTT ATGGCAGCAC TGCATAATTC TCTTACTGTC ATGCCATCCG6661 TAAGATGCTT TTCTGTGACT GGTGAGTACT CAACCAAGTC ATTCTGAGAA TAGTGTATGC 6721 GGCGACCGAG TTGCTCTTGC CCGGCGTCAA CACGGGATAA TACCGCGCCA CATAGCAGAA 6781 CTTTAAAAGT GCTCATCATT GGAAAACGTT CTTCGGGGCG AAAACTCTCA AGGATCTTAC 6841 CGCTGTTGAG ATCCAGTTCG ATGTAACCCA CTCGTGCACC CAACTGATCT TCAGCATCTT 6901 TTACTTTCAC CAGCGTTTCT GGGTGAGCAA AAACAGGAAG GCAAAATGCC GCAAAAAAGG 6961 GAATAAGGGC GACACGGAAA TGTTGAATAC TCATACTCTT CCTTTTTCAA TATTATTGAA 7021 GCATTTATCA GGGTTATTGT CTCATGAGCG GATACATATT TGAATGTATT TAGAAAAATA7081 AACAAATAGG GGTTCCGCGC ACATTTCCCC GAAAAGTGCC ACCTGACGTC TAAGAAACCA 7141 TTATTATCAT GACATTAACC TATAAAAATA GGCGTATCAC GAGGCCCTTT CGTCTTCAAG7201 AATTAGCTTG GCCATTGCAT ACGTTGTATC CATATCATAA TATGTACATT TATATTGGCT7261 CATGTCCAAC ATTACCGCCA TGTTGACATT GATTATTGAC TAGTTATTAA TAGTAATCAA7321 TTACGGGGTC ATTAGTTCAT AGCCCATATA TGGAGT//pRetroX-PTuner2-C其他相关逆病毒载体:pVSV-G pMSCV-FLIP-puro-dsRed-GFP-miRNApCLXSNpMKO.1-GFP pBABE-puro-IRES-EGFP pBABEpBABE-hygro-hTERT pBABE-neo-hTERT pBABE-neo-SV40LTpBABE-GFP pBABE-hygro pBABE-neopBABE-Puro pVPack-GP pVPack-10A1pVPack-Eco pVPack-Ampho pCMV-Gag-PolpCL-Eco pUMVC pCMV-VSV-GpMSCV-PIG pQCXIN pFB-ERVpFB-hrGFP pFB-Luc pFB-NeopCFB pRetro-Lib pRetroX-Tight-Pur-Luc pRetroX-TetOne pRetroX-PTuner pRetroX-PTuner2 pRetroX-PTuner-IRES pRetroX-TRE3G pRetroX-TRE3G-Luc pRetroX-Tet-On Advanced pRetroX-Tet-Off Advanced pRetroX-Tight-Hyg pRetroX-Tight-Pur pRetroX-G1-Red pRetroQ-mCherry-N1 pRetroQ-DsRed-Monomer-C1 pRetroQ-DsRed-Monomer-N1 pRetroQ-AcGFP1-C1pRetroX-SG2M-Cyan pRetroX-IRES-DsRedExpressRNAi-ReadypSIREN-RetroQ-DsRed-ExpresspLXSN pLNCX2 pQCXIXpSuper.Retro.Puro pSilencer5.1-U6 Retro RNAi-ReadypSIREN-RetroQ-ZsGreen1pRetroX-IRES-ZsGreen1 RNAi-Ready pSIREN-RetroQ pMSCVhyg pMSCVpuro pQCXIH pSuper.Retro.Neo pMKO.1-puro pCFB-EGSH pRetroQ-AcGFP1-N1 pBABE-HAII pFB pLXINpBABE-Puro SV40LT pRetroX-TetOne-luc pSuper.Retro.Neo+GFPpBABE-zeo pRetroX-PTuner2-C pRetroX-IRES-DsRed ExpresspVPack-VSV-G pRetroX-Tet3G pMSCVneopCL-Ampho pRetroX-Tight-Hyg-Luc pSilencer5.1-H1 Retro pCMV-VSVGpRetroQ-mCherry-C1。
