pENTR-D-TOPO载体手册
测序通用引物序列

测序通用引物序列常见载体的测序引物:Primer of Vector:Vector:Primer(F);Primer(R)pACT T7 T3pACT2 GAL4 AD pACT2-RpAS2-1 GAL4 BD pAS2-1.RpB42AD pB42ADF pB42ADRpBACPAK8 BAC1 BAC2pBK-CMV T7 T3PBS(SK/KS)/M13- M13F/T7 T3/M13RpBV220 PBV220F PBV220RpCAMBIA 1301(1300) P1 P2pCAMBIA 2300 M13R(-48) M13F(-47)PCANTB5E S1/M13R S6pCAT3-enhancer RVP3 此载体无反向引物pcDNA3.0 CMV-F/T7 SP6/BGHpcDNA3.1 T7 BGHpcDNA4 T7/CMV-F BGHpcDNA6 T7/CMV-F(J21025在T7前面)BGHpcDNAII T7 SP6pCE2.1 M13F M13RpCEP4 pCEP-F EBV-RpCF-T M13F M13RpCI T7(17Base)此载体无反向引物pCI-neo T7(17Base)T3pCMS-EGFP T7 T3pCMV-3Tag-4A T3 /PFLAG-CMV-F T7pCMV5 pCMV5F pCMV5RpCMV5-Flag pCMV5F pCMV5RpCMV-MYC/HA pCMV-F pCMV-RpCMV-Sport M13F/T7 SP6/M13RpCMV-Tag(KAN+) T3 T7pCR2.1-TOPO M13F/T7 M13RpCR3.1 T7 BGHpCS2 SP6 T7pDonar M13F M13RpDONR221 M13F M13RpDrive T7 SP6pDRIveR(KAN+) T7 SP6pDsRED1-C1 pDsRED-ex-C1-F pEGFP-N-3’(距离很近,一般不用) pDsRED2-C1(KAN+) pDsRED-ex-C1-F pEGFP-N-3’pDSRED-N1(KAN+) pEGFP-N-5’ PDSRED-N-RpECFP-C pEGFP-C-5’ pECFP-C-3′pECFP-N1 pEGFP-N-5’ pEGFP-N-3’pEF/myc/ER pEF-F pCDNA3.1RpEGFP-C(KAN+) pEGFP-C-5’ pEGFP-C-3′pEGFP-N(KAN+) pEGFP-N-5’ pEGFP-N-3’pENTR/D-TOPO(KAN+) M13F M13RpET-*(His)(KAN+) T7 T7 TerpET22(a,b,c)(AMP+) T7 T7 TerpET28,30,24,49(KAN+) T7 T7 TerpET32(a,b,c)(AMP+) T7/S.tag T7 TerpET50(amp+) T7/s.tag T7TERpET44(AMP+) T7/s.tag T7 TerpEYFP-N1 pEGFP-N-5’ pEGFP-N-3’pEYFP-C1 pEGFP-C-5’ pEGFP-C-3′pFLAG-CMV pFLAG-CMV -F pFLAG-CMV -R pGAD424 GAL4 AD 3′ADpGADGH GAL4 AD 3′ADpGADT7-Rec GAL4 AD/T7 3′ADpGAPZa-A a-FACTOR 3′AOXpGBKT7(Kana+) T7 3′BDpGEM-T(-Easy) M13F/T7 SP6/M13RpGEX-(*)T pGEX-4T-5’ pGEX-4T-3’pGL2 GLP1 GLP2pGL3 RVP3 GLP2(RVP4)(没有特别说明用GLP2) PinpointTM Vector Pinpoint Primer SP6pIRES PIRES-F/T7 T3/PIRES-R (J40720)pIRES2-EGFP pIRES2-EGFP.P5’ pIRES2-EGFP.P3’pLXSN pLXSN-F pLXSN-RpMAL-c2E MalE primer M13F(-47)pMAL-C2x P5’ P3’pMAL-p2X MalE primer M13F(-47)PMD18-T M13R(-48) M13F(-47)PMD19-T M13F(-47) M13R(-48)pPIC9K(AMP+、ZEO+)5’AOX/a-factor 3’AOX pPICZa 5′AOX/PPICZa-F 3′AOXpPROEXHTA M13R(-48) 无反向引物pQE30or40 pQE30-F pQE-R:pRSET T7 T7TERpSilencer3.1-H1 hygro M13F(-47) 3.0REV pSILENCEV2.0-U6 T7 2.0REVpSK01-T M13F M13RpSK-CMV(KAN+) T7 T3pSP72 T7 SP6pSport1 SP6/M13F(-47) T7/M13RpSUPER T7 T3pTA2 M13F/T7(优先使用M13F)T3/M13RpT-Adv M13F /T7 M13RpTARGET TM pTarget.F(在T7前面)/T7 pTarget.R pTO-T7 T7 T7TERpTRC99a-c pTRC99C-F pTRC99C-RpTriPLEx2 5′pTriPLEx2 T7pTWIN-1 T7 T7TER(客户确认再用)pUC18(19)/118(119) M13F(-47) M13R(-48)pUCm-T M13F/T7 M13RpVAX1 T7 BGHpWSK29 T7/M13F/M13F(-47) T3/M13R/M13R(-48)pXT7 T7 SP6pYES2 T7 pYes2.RpLEXA pLEXA-F pLEXA-RpLNCX pLNCX-F pLNCX-RpRECEIVER CMV-F 此载体无反向引物pSHUTTLE-CMV PSHUTTLE-CMV-F PSHUTTLE-CMV-R pSP64/65 SP6 此载体无反向引物pSTBlue-1 T7 SP6pT7Blue(R) T7 M13F(-47)引物名称序列(5′-3′):M13R:CAG GAA ACA GCT ATG ACCM13F:TGT AAA ACG ACG GCC AGTM13F(-47):CGC CAG GGT TTT CCC AGT CAC GACM13R(-48):AGC GGA TAA CAA TTT CAC ACA GGAM13(-96):CCC TCA TAG TTA GCG TAA CGSP6:ATT TAG GTG ACA CTA TAGT7:TAA TAC GAC TCA CTA TAG GGT7 terminator:TGC TAG TTA TTG CTC AGC GGT3:ATT AAC CCT CAC TAA AGG GApGEX-4T-5′:GGG CTG GCA AGC CAC GTT TGG TG pGEX-4T-3′:CCG GGA GCT GCA TGT GTC AGA GG GLp1:TGT ATC TTA TGG TAC TGT AAC TGGLp2:CTT TAT GTT TTT GGC GTC TTC CARVp3:CTA GCA AAA TAG GCT GTC CCRVp4:GAC GAT AGT CAT GCC CCG CGpcDNA3.1R:TAG AAG GCA CAG TCG AGGPinPoint primer:CGT GAC GCG GTG CAG GGC G pCMV-F:TCT AAA AGC TGC GGA ATT GTpCMV-R:TCCAAACTCATCAATGTATCpTRC99C-F: TTG CGC CGA CAT CAT AACpTRC99C-R: CTGCGTTCTGATTTAATCTGpCEP-F: AGA GCT CGT TTA GTG AAC CGEBV-R : GTG GTT TGT CCA AAC TCA TCpIRES2-EGFP.P5’: GTA GGC GTG TAC GGT GGG AG pIRES2-EGFP.P3’: AAC GCA CAC CGG CCT TAT TC3′AD: AGA TGG TGC ACG ATG CAC AGCMV -F:CGC AAA TGG GCG GTA GGC GTGS1:CAA CGT GAA AAA ATT ATT ATT CGCS6:GTA AAT GAA TTT TCT GTA GTA GG5`AOX1:GAC TGG TTC CAA TTG ACA AGC3`AOX1:GCA AAT GGC ATT CTG ACA TCCα-Factor:TAC TAT TGC CAG CAT TGC TGCGAL4 AD:TAC CAC TAC AAT GGA TGpACT2-R:GTGCACGATGCACAGTTGAApB42ADF:CCA GCC TCT TGC TGA GTG GAG ATGpB42ADR:AAG CCG ACA ACC TTG ATT GGA GpEGFP-N-5’:TGG GAG GTC TAT ATA AGC AGA G pEGFP-N-3’:CGT CGC CGT CCA GCT CGA CCA G pEGFP-C-5’:CAT GGT CCT GCT GGA GTT CGT G pEGFP-C-3′ :TAT GGC TGA TTA TGA TCA GTPBV220F:AAG AAG GGC AGC ATT CAA AGPBV220R:CTG CGT TCT GAT TTA ATC TGU6:ATG GAC TAT CAT ATG CTT ACC GTA2.0rev primer:AGG CGA TTA AGT TGG GTA3.0rev:GAG TTA GCT CAC TCA TTA GGCS.tag:GAA CGC CAG CAC ATG GAC5′TriplEx2:CTC CGA GAT CTG GAC GAG CRVP4:GAC GAT AGT CAT GCC CCG CGRVP3:CTA GCA AAA TAG GCT GTC CCpQE30-R:GTT CTG AGG TCA TTA CTG GpQE30-F:TGA GCG GAT AAC AAT TTC ACpEF-F:TCA AGC CTC AGA CAG TGG TTCpDSRED-N1-R:TGA AGC GCA TGA ACT CCT TG pDsRED-express-C1-F:TCC CAC AAC GAG GAC TAC AC pCMV5R:ATT ATA GAG GAC ACC TAG TCpCMV5F:TTC CAA AAT GTC GTA ATA ACP5′:TGC GTA CTG CGG TGA TCA ACP3′:CTG CAA GGC GAT TAA GTT GGBAC2:ACG CAC AGA ATC TAG CGC TTBAC1:AAC CAT CTC GCA AAT AAA TA3′BD:TAA GAG TCA CTT TAA AAT TTG TAT AC pTarget.F:CCA GGA TTT TCC CAG TCA CpTarget.R:GGC TTT ACA CTT TAT GCT TCT7(17base):ACA TCC ACT TTG CCT TTC TCpYes2.R:TCG GTT AGA GCG GAT GTGGAL4 BD:TCA TCG GAA GAG AGT AGpAS2-1.R:AAG AGT TAC TCA AGA ACA AGA ApFlag-CMV-F: GGT AGG CGT GTA CGG TGGpFlag-CMV-R: GCA CTG GAG TGG CAA CTTpIRES-F: CTT ACT GAC ATC CAC TTT GCpIRES.R: CAC TGC ATT CTA GTT GTG GTP1:CCA GGC TTT ACA CTT TAT GCP2:GCG ATT AAG TTG GGT AAC GCpLEXA-F:CGT CAG CAG AGC TTC ACC ATpLEXA-R:TAA AAC CTA AGA GTC ACT TTpLNCX-F:AGC TCG TTT AGT GAA CCG TCA GAT CG pLNCX-R:ACC TAC AGG TGG GGT CTT TCA TTC CC pLXSN-F:CTT GAA CCT CCT CGT TCG ACpLXSN-R:GTT GCT GAC TAA TTG AGA TGpSHUTTLE-CMV-F:GGT CTA TAT AAG CAG AGG TG pSHUTTLE-CMV-R:GTG GTA TGG CTG ATT ATG ATC AG MalE Primer:GGT CGT CAG ACT GTC GAT GAA GCC。
☆如何阅读质粒图谱

如何阅读质粒图谱最近由于实验需要,需要查阅载体图谱,到园子里搜罗一番,发现虽然有人问载体图谱阅读的问题,也有前辈回答,但都不详细,借自己也在琢磨这个问题的机会,将我学到的东西整理一下,于大家分享。
载体主要有病毒和非病毒两大类,其中质粒DNA是一种新的非病毒转基因载体。
一、一个合格质粒的组成要素#复制起始位点Oril 即控制复制起始的位点。
原核生物DNA分子中只有一个复制起始点。
而真核生物DNA分子有多个复制起始位点。
#抗生素抗性基因可以便于加以检测,如Amp+l ,Kan+#多克隆位点MCS 克隆携带外源基因片段l#P/E 启动子/增强子l#Termsl 终止信号#加poly(A)信号l 可以起到稳定mRNA作用二、如何阅读质粒图谱第一步:首先看Ori的位置,了解质粒的类型(原核/真核/穿梭质粒)第二步:再看筛选标记,如抗性,决定使用什么筛选标记。
(1)Ampr 水解β-内酰胺环,解除氨苄的毒性。
(2)tetr 可以阻止四环素进入细胞。
(3)camr 生成氯霉素羟乙酰基衍生物,使之失去毒性。
(4)neor(kanr)氨基糖苷磷酸转移酶使G418(卡那霉素衍生物)失活(5)hygr 使潮霉素β失活。
第三步:看多克隆位点(MCS)。
它具有多个限制酶的单一切点。
便于外源基因的插入。
如果在这些位点外有外源基因的插入,会导致某种标志基因的失活,而便于筛选。
决定能不能放目的基因以及如何放置目的基因。
第四步:再看外源DNA插入片段大小。
质粒一般只能容纳小于10Kb的外源DNA片段。
一般来说,外源DNA片段越长,越难插入,越不稳定,转化效率越低。
第五步:是否含有表达系统元件,即启动子-核糖体结合位点-克隆位点-转录终止信号。
这是用来区别克隆载体与表达载体。
克隆载体中加入一些与表达调控有关的元件即成为表达载体。
选用那种载体,还是要以实验目的为准绳。
启动子-核糖体结合位点-克隆位点-转录终止信号#启动子-促进DNA转录的DNA顺序,这个DNA区域常在基因或操纵子编码顺序的上游,是DNA分子上可以与RNApol特异性结合并使之开始转录的部位,但启动子本身不被转录。
Gateway

Gateway™技术提供以下可能:∙通过去除冗长的亚克隆步骤节省您的时间∙将您的基因转入到多个表达系统∙在任何您选择的系统――体外,细菌,酵母,昆虫,或哺乳动物――分析表达Gateway™技术能够克隆一个或多个基因进入到任何蛋白表达系统(图1)。
这项强大的体外技术大大地简化了基因克隆和亚克隆的步骤,而同时典型的克隆效率高达95%或更高。
当基因在目的表达载体之间快速简便的穿梭时,还可以保证正确的方向和阅读框。
Gateway™也有助于进行带不同数目纯化和检测标签的表达。
Gateway™利用了位点特异重组,所以在构建入门载体后,不再需要使用限制性内切酶和连接酶。
一旦您拥有了一个入门克隆,就可以多次使用它,转移您感兴趣的基因到Gateway™改造过的的各种表达载体(目的载体)。
此外,由于在重组时DNA片段的阅读框和方向保持不变,因而您不必再为新的表达克隆的测序担心。
在使用每一种新的表达系统时,将会节省您更多的时间。
图1-Gateway™技术的灵活性*目的基因克隆进入门载体后,可以同时转移目的基因到多个目的载体。
一种强大而可靠的技术Gateway™技术是克隆和亚克隆DNA序列的一项新颖的通用系统,便于功能基因的分析和蛋白质的表达。
一旦进入这个多功能的操作系统,DNA片段可以通过位点特异的重组在载体之间转移。
Gateway™技术是基于已研究的非常清楚的λ嗜菌体位点特异重组系统(attB x attP →attL x attR)。
BP和LR两个反应就构成了Gateway™技术(表1和图2)。
BP反应利用一个attB DNA片段或表达克隆和一个attP供体载体之间的重组反应,创建一个入门克隆。
LR反应是一个attL入门克隆和一个attR目的载体之间的重组反应。
LR反应用来在在平行的反应中转移目的序列到一个或更多个目的载体。
Gateway™技术也利用了ccdB选择方法,确保高效率的分离重组克隆。
典型的效率是>95%。
Gateway

Gateway也可以被视为一种克隆操作平台:把目的基因克隆到入门载体(Entry Vector)后,就不用依赖限制性内切酶,而靠载体上存在的特定重组位点和重组酶,高效、快速地将目的基因克隆到其它的受体载体(Destination Vector,目的载体)上。
Gateway的原理也是建立在噬菌体DNA定点整合到细菌宿主基因组上。
在噬菌体和细菌的整合因子(INF、Int)的作用下,lambda的attP位点和大肠杆菌基因组的attB位点可以发生定点重组,lambda噬菌体DNA整合到大肠杆菌的基因组DNA中,两侧产生两个新位点:attL和attR。
这是一个可逆的过程,如果在一个噬菌体编码蛋白Xis和IHF、Int的共同介导下这两个新位点可以再次重组回复为attB和attP位点,噬菌体从细菌基因组上裂解下来。
这一过程的方向是受控于两个重要因素:存在的介导蛋白和重组位点。
在Gateway系统中,入门载体包含两个重组位点序列attL1和attL2,大小均为100bp,中间夹着一个自杀基因——ccdB基因。
由于ccdB基因的表达产物能抑制普通的E.coli生长,在克隆时没有切开或者自身环化的载体在转化时不能生长。
在构建含目的基因的入门载体时必须切掉这个基因,接入目的基因。
ccdB基因两端可以选择的酶切位点有限(2个),同时还必须考虑读码框架、启动子、终止密码等问题,因此Gateway系统提供了5种不同的入门载体以供选择。
需要特别注意的是转化用的菌株必须是不含F附加体的,因为它表达的一种产物能阻断ccdB基因,影响筛选结果。
同样,目的载体(Destination Vector)也必须和Gateway系统配套,即目的载体的表达调控元件下游有两个重组位点attR1和attR2,大小均为125bp,同样也夹着一个ccdB自杀基因。
当需要将目的基因从入门载体(Entry Vector)转移到目的载体(Destination Vector)时,只要将两种质粒混合(线性化能有效提高重组率),加入含有Int、IHF、Xis等重组因子的LR重组酶混合物,attR2序列和attL2序列发生重组,生成一个融合质粒。
常见载体引物表

无
pDSRED-PRESS-C1
pDSRED -C1-F
pDSRED-C1-R
pCR-Blunt
M13R
M13F/T7
pMT/V5-His_A (_B,_C)
MT-Profor
BGHrev
pEASY-T3
T7/M13F
SP6/M13R
PSILENCE-1.0-U6
T7
2.0REV(同P2(pCAMBIA 1301))
pMSCV
MSCV5'
MSCV-rev
pBABE
pBABE5’
pBABE3’
pFastBac HT_A (_B, _C)
pFastBac-F
pFastBac-R
pFastBac1
PH-Profor/BaculoVfor
pFastBac-R
pACYCDuet-1 (MCS I)
ACYCDuetUP 1
T7/S.tag
T7 Ter
pET-42a (-b, -c)(+)
Stag
T7 Ter
pET50(amp+)
T7/s.tag
T7TER
pET44(AMP+)
T7/s.tag
T7 Ter
pEYFP-N1
pEGFP-N-5’
pEGFP-N-3’
PETBlue
PETBlueT7UP(太靠前,在T7前)/ PETBlueUP
pLEXA-R
pCDH
pCDH-EF1
无
PVX
LBA
LBB
pAcGP67 (_A, _B, _C)
PH
pAcGP67rv
pAD-Track
gateway重组技术

Gateway也可以被视为一种克隆操作平台:把目的基因克隆到入门载体(Entry Vector)后,就不用依赖限制性内切酶,而靠载体上存在的特定重组位点和重组酶,高效、快速地将目的基因克隆到其它的受体载体(Destination Vector,目的载体)上。
Gateway的原理也是建立在噬菌体DNA定点整合到细菌宿主基因组上。
在噬菌体和细菌的整合因子(INF、Int)的作用下,lambda的attP位点和大肠杆菌基因组的attB位点可以发生定点重组,lambda噬菌体DNA整合到大肠杆菌的基因组DNA中,两侧产生两个新位点:attL和attR。
这是一个可逆的过程,如果在一个噬菌体编码蛋白Xis和IHF、Int的共同介导下这两个新位点可以再次重组回复为attB和attP位点,噬菌体从细菌基因组上裂解下来。
这一过程的方向是受控于两个重要因素:存在的介导蛋白和重组位点。
在Gateway系统中,入门载体包含两个重组位点序列attL1和attL2,大小均为100bp,中间夹着一个自杀基因——ccdB基因。
由于ccdB基因的表达产物能抑制普通的E.coli生长,在克隆时没有切开或者自身环化的载体在转化时不能生长。
在构建含目的基因的入门载体时必须切掉这个基因,接入目的基因。
ccdB基因两端可以选择的酶切位点有限(2个),同时还必须考虑读码框架、启动子、终止密码等问题,因此Gateway系统提供了5种不同的入门载体以供选择。
需要特别注意的是转化用的菌株必须是不含F附加体的,因为它表达的一种产物能阻断ccdB基因,影响筛选结果。
同样,目的载体(Destination Vector)也必须和Gateway系统配套,即目的载体的表达调控元件下游有两个重组位点attR1和attR2,大小均为125bp,同样也夹着一个ccdB自杀基因。
当需要将目的基因从入门载体(Entry Vector)转移到目的载体(Destination Vector)时,只要将两种质粒混合(线性化能有效提高重组率),加入含有Int、IHF、Xis等重组因子的LR重组酶混合物,attR2序列和attL2序列发生重组,生成一个融合质粒。
常用载体测序引物列表

常用载体测序引物列表 载体名称 正向引物 反向引物pACT T7 T3pACT2 GAL4 AD(5’AD) 3'AD/pACT2-R PACYCDUET-1(MCSII) DuetUP2 T7TERpAD/pl-DEST CMV-FpAD-Track pAD-Track-F 无pAS2-1 5’BD M13-26/48 PB42AD PB42ADF PB42ADR PBAD PBAD-F PBAD-R pBABE pBABE5’ pBABE3’ PBACPAK8 BAC1BAC2 pBK-CMV T7/M13-20 T3/M13-26 PBS(SK/KS)/M13- M13F/T7T3/M13R pBV220 PBV220F PBV220R pBI121需根据酶切位点确认 需根据酶切位点确认 pCAMBIA 1301(1300)需根据酶切位点确认 需根据酶切位点确认 pCAMBIA 2300 需根据酶切位点确认 需根据酶切位点确认PCANTB5E S1/M13R S6pCAT3-enhancer pGL3+pcDNA3.0 pEGFP-N5/T7 SP6/BGHpcDNA3.1 pEGFP-N5/T7 BGHpcDNA4 T7/pEGFP-N5 BGHpcDNA6 T7/pEGFP-N5 BGHpcDNAII T7SP6 pCE2.1 M13FM13R pCEP4 pCMV5R/PCEP-F pEGFP-N5/EBV-R pCF-T M13FM13R pCIT7(17Base ) pCMV5R pCI-neo T7(17Base ) T3pCMS-EGFP T7T3 pCMV-3Tag-4A T3 /pFlag-CMV-F T7pCMV5 pCMV5F pCMV5R pCMV5-Flag pCMV5FpCMV5R pCMV-MYC/NUC pCMV5FpCMV5R PCMV-HA pCMV5FpCMV5R pCMV-SportSP6/M13RpCMV-Tag T3T7 pCMV-TNT T7/SP6pCMV5R pCR2.1-TOPO M13F/T7M13R pCR3.1 T7BGH pCS2 SP6T7PDNR-LIB M13F M13RpDonar M13F M13R pDONR201 pDONR-F pDONR-R pDONR221 M13F M13RpDrive T7 SP6 pDRIveR T7 SP6pDsRED1-C1 pDsRED-C1-F pECFP-C-3 pDsRed1-N1 pEGFP-N-5 RFP-NrevpDsRed-Monomer-N1 pEGFP-N-5 pDsRed1-NpDsRED2-C1 pDsRED-ex-C1-F pEGFP-N-3’pEGFP-N-5 pDsRed1-N pDSRED2-N1pDSRED-N1 pEGFP-N-5’ PDSRED-N-R pEASY-BLUNT M13F/T7 M13RTerpEASY-E1 T7 T7pEASY-T1 M13F/T7 M13RpEASY-T3 M13F/T7 M13R/SP6 pECFP-C pEGFP-C-5’ pECFP-C-3' pECFP-N1 pEGFP-N-5’ pEGFP-N-3’pEF/myc/ER pEF-F pCDNA3.1R pEGFP-C pEGFP-C-5’ pEGFP-C-3' pEGFP-N pEGFP-N-5’ pEGFP-N-3’ pENTR/D-TOPO M13F M13RTerpET-*(His) T7 T7pET22(a,b,c) T7 T7TerTerpET28,30,24,49 T7 T7TerpET32(a,b,c) T7/S.tag T7pET-42a (-b, -c)(+) Stag T7 TerpET50 T7/s.tag T7TERpET44 T7/s.tag T7Ter pEYFP-N1 pEGFP-N-5’ pEGFP-N-3’ PETBlue M13-20 PETBlueDOWN PETDUET-1(MCSI) PBRrevBam DuetDOMN1PETDUET-1(MCSII) DuetUP2 T7TERpEYFP-C1 pEGFP-C-5’ pEGFP-C-3' pEYFP-N1 pEGFP-N-5’ pEGFP-N-3’ pFastBac pFastBac-F pFastBac-R pFastBac HT_A,B,C pFastBac-PH pECFP-C-3 pFastBac1 pFastBac-PH pECFP-C-3 pFastBac Dual (MCSI) pFastBac-PH pECFP-C-3 pFLAG-CMV CMV30/pFLAG-CMV-F CMV-24/pFLAG-CMV-Rp3xFlag-CMV-7.1 CMV-F/CMV24 CMV30pGAD424 5’AD 3'ADpGADGH 5’AD 3'ADpGADT7-Rec 5’AD/T7 3'ADpGAPZa-A/B/C a-FACTOR 3'AOXforward 3'AOXpGAPZ-A/B/C pGAPpGBKT7 T7 3'BDpGEM-T(-Easy) M13F/T7 SP6/M13RpGEX-(*)T pGEX-5’ pGEX-3’pGL2 pCMV5R PGL3-Pgl3-BASIC pGL3+ PGL3-pGL3-Enhancer pGL3+ PGL3-PinPoint TM PinPoint Primer SP6pIRES PIRES-F/T7 T3/PIRES-RpIRES2-EGFP pIRES2-EGFP.P5’ pIRES2-EGFP.P3’ pLenti6/v5-Dest pEGFP-N-5’PLEXA PLEXA-F PLEXA-R PLNCX PLNCX-F PLNCX-R pLXSN pLXSN-F pLXSN-RPjc1-tac T7pJet.1.21.Blunt T7primer M13F(-47) pMAL-c2E MalEP3/PMAL-C2X-R pMAL-C2x P5/PMAL-C2X-Fprimer M13F(-47) pMAL-p2X MalEPMD18-T M13R(-48) M13F(-47) PMD19-T M13F(-47) M13R(-48) pMIR-report M13FPPC86 PPC86-F PPC86-R3’AOXpPIC9K 5’AOX/a-factor3'AOX pPICZa 5'AOX/PPICZa-FpPROEXHTA M13R(-48)pQE30or40 pQE30+ pQE30- pRECEIVER CMV-FpRSET T7 T7TER PRSFDUET-1(MCSI) DuetUP1 DuetDOWN1 PRSFDUET-1(MCSII) DuetUP2 T7TERpShuttle-CMV pShuttle-CMV-F pShuttle-CMV-R PSILENCE-1.0-U6 T7 T3pSilencer3.1-H1 hygro M13F(-47) 3.0revpSILENCEV2.0-U6 T7 2.0revpSK01-T M13F M13RpSK-CMV T7 T3Psos H1PSP64/65 SP6pSP72 T7 SP6T7/M13RpSport1 SP6/M13F(-47)pSTBlue-1 T7 SP6pSUPER T7 T3pT7Blue(R) T7 M13F(-47)pTA2 M13F/T7 T3/M13R/T7 M13RpT-Adv M13FpTARGET TM pTarget-F/T7 arget-RPTHIOHISA,B,C TRX-FORWARD TRX-REVERSEpTO-T7 T7 T7TERPBV220-/PTRC99C-R pTRC99a-c PQE30+/PTRC99C-FpTriPLEx2 PT5/5'TriplEx2 T7pTz57r/t M13+ M13-pTWIN-1 T7 T7TERpUC18(19)/118(119) M13F(-47) M13R(-48)M13F(-47)/M13F pUC57 M13R(-48)/M13RpUCm-T M13F/T7 M13RpVAX1 T7 BGHT3/M13R/M13R(-48) pWSK29 T7/M13F/M13F(-47)pXT7 T7 SP6pYES2 T7 pYes2.R。
Gateway技术

Gateway™技术提供以下可能:•通过去除冗长的亚克隆步骤节省您的时间•将您的基因转入到多个表达系统•在任何您选择的系统――体外,细菌,酵母,昆虫,或哺乳动物――分析表达一种更好的克隆方法Gateway™技术能够克隆一个或多个基因进入到任何蛋白表达系统(图1)。
这项强大的体外技术大大地简化了基因克隆和亚克隆的步骤,而同时典型的克隆效率高达95%或更高。
当基因在目的表达载体之间快速简便的穿梭时,还可以保证正确的方向和阅读框。
Gateway™也有助于进行带不同数目纯化和检测标签的表达。
Gateway™利用了位点特异重组,所以在构建入门载体后,不再需要使用限制性内切酶和连接酶。
一旦您拥有了一个入门克隆,就可以多次使用它,转移您感兴趣的基因到Gateway™改造过的的各种表达载体(目的载体)。
此外,由于在重组时DNA片段的阅读框和方向保持不变,因而您不必再为新的表达克隆的测序担心。
在使用每一种新的表达系统时,将会节省您更多的时间。
图1-Gateway™技术的灵活性*目的基因克隆进入门载体后,可以同时转移目的基因到多个目的载体。
一种强大而可靠的技术Gateway™技术是克隆和亚克隆DNA序列的一项新颖的通用系统,便于功能基因的分析和蛋白质的表达。
一旦进入这个多功能的操作系统,DNA片段可以通过位点特异的重组在载体之间转移。
Gateway™技术是基于已研究的非常清楚的λ嗜菌体位点特异重组系统(attB x attP →attL x attR)。
BP和LR两个反应就构成了Gateway™技术(表1和图2)。
BP反应利用一个attB DNA片段或表达克隆和一个attP供体载体之间的重组反应,创建一个入门克隆。
LR反应是一个attL入门克隆和一个attR目的载体之间的重组反应。
LR反应用来在在平行的反应中转移目的序列到一个或更多个目的载体。
Gateway™技术也利用了ccdB选择方法,确保高效率的分离重组克隆。
HTP -pENTR-D-TOPO定向克隆载体

Methods.............................................................................................................................5
Designing PCR Primers ........................................................................................................................................... 5 Producing Blunt-End PCR Products...................................................................................................................... 9 HTP TOPO® Cloning and Transformation with Bulk Cells.............................................................................. 11 HTP TOPO® Cloning and Transformation with MultiShot™ Cells.................................................................. 13 Analyzing Transformants...................................................................................................................................... 15 Guidelines to Perform the LR Recombination Reaction ................................................................................... 16
常见载体引物表
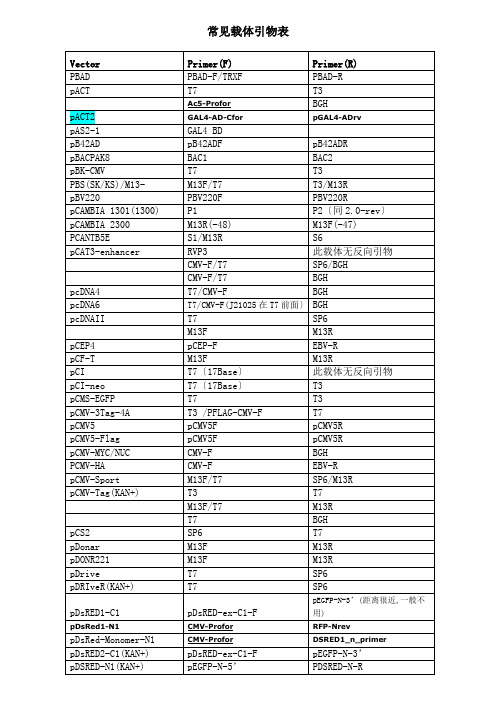
pLXSNfw
pLXSNrv
pLEXA
pLEXA-F
pLEXA-R
pCDH
pCDH-EF1
无
PVX
LBA
LBB
pAcGP67 (_A, _B, _C)
PH
pAcGP67rv
pAD-Track
pAD-Track-F
无
PLPC
PLPC-F/CMV-F
PLPC-R
pHAGE-CMV-MCS-IZsGreen
pLVX-IRES-ZsGreen1
PIRES2-EGFP-F
PIRES2-EGFP-R
pDC316
pDC316F
pDC316R
PLXSN-5
PLXSN-3
PVAX1
T7/CMV-F
BGH
PSHUTTLE-CMV
pShuttle-CMV-F
pShuttle-CMV-R
pSilencer™ 4.1-CMV neo
此载体无反向引物
CMV-F/T7
SP6/BGH
CMV-F/T7
BGH
pcDNA4
T7/CMV-F
BGH
pcDNA6
T7/CMV-F(J21025在T7前面〕
BGH
pcDNAII
T7
SP6
M13F
M13R
pCEP4
pCEP-F
EBV-R
pCF-T
M13F
M13R
pCI
T7〔17Base〕
此载体无反向引物
T7
pTWIN-1
T7
T7TER(客户确认再用〕
pUC18(19)/118(119)
M13F(-47)
常见载体引物表

pMIR-report
M13F
PSI-CHECK2
PSI-CHECK2-F
PSICHECF-2-R1
PLK0.1
LK0.1-5
p3xFlag-CMV-7.1
CMV-F
CMV-F
无
pDSRED-PRESS-C1
pDSRED -C1-F
pDSRED-C1-R
pCR-Blunt
M13R
M13F/T7
pMT/V5-His_A (_B,_C)
MT-Profor
BGHrev
pEASY-T3
T7/M13F
SP6/M13R
PSILENCE-1.0-U6
T7
2.0REV(同P2(pCAMBIA 1301))
T7/S.tag
T7 Ter
pET-42a (-b, -c)(+)
Stag
T7 Ter
pET50(amp+)
T7/s.tag
T7TER
pET44(AMP+)
T7/s.tag
T7 Ter
pEYFP-N1
pEGFP-N-5’
pEGFP-N-3’
PETBlue
PETBlueT7UP(太靠前,在T7前)/ PETBlueUP
常见载体引物表
Vector
Primer(F)
Primer(R)
PBAD
PBAD-F/TRXF
PBAD-R
pACT
T7
T3
pAc5.1/V5-His
Ac5-Profor
BGH
pACT2
GAL4-AD-Cfor
pGAL4-ADrv
pAS2-1
常用载体测序引物列表

常⽤载体测序引物列表常⽤载体测序引物列表载体名称正向引物反向引物pACT T7 T3pACT2 GAL4 AD(5’AD) 3'AD/pACT2-R PACYCDUET-1(MCSII) DuetUP2 T7TERpAD/pl-DEST CMV-FpAD-Track pAD-Track-F ⽆pAS2-1 5’BD M13-26/48 PB42AD PB42ADF PB42ADR PBAD PBAD-F PBAD-R pBABE pBABE5’ pBABE3’ PBACPAK8 BAC1BAC2 pBK-CMV T7/M13-20 T3/M13-26 PBS(SK/KS)/M13- M13F/T7T3/M13R pBV220 PBV220F PBV220R pBI121需根据酶切位点确认需根据酶切位点确认 pCAMBIA 1301(1300)需根据酶切位点确认需根据酶切位点确认 pCAMBIA 2300 需根据酶切位点确认需根据酶切位点确认PCANTB5E S1/M13R S6pCAT3-enhancer pGL3+pcDNA3.0 pEGFP-N5/T7 SP6/BGHpcDNA3.1 pEGFP-N5/T7 BGHpcDNA4 T7/pEGFP-N5 BGHpcDNA6 T7/pEGFP-N5 BGHpcDNAII T7SP6 pCE2.1 M13FM13R pCEP4 pCMV5R/PCEP-F pEGFP-N5/EBV-R pCF-T M13FM13R pCIT7(17Base ) pCMV5R pCI-neo T7(17Base ) T3pCMS-EGFP T7T3 pCMV-3Tag-4A T3 /pFlag-CMV-F T7pCMV5 pCMV5F pCMV5R pCMV5-Flag pCMV5FpCMV5R pCMV-MYC/NUC pCMV5FpCMV5R PCMV-HA pCMV5FpCMV5R pCMV-SportSP6/M13RpCMV-Tag T3T7 pCMV-TNT T7/SP6pCMV5R pCR2.1-TOPO M13F/T7M13R pCR3.1 T7BGH pCS2 SP6T7PDNR-LIB M13F M13RpDonar M13F M13R pDONR201 pDONR-F pDONR-R pDONR221 M13F M13RpDrive T7 SP6 pDRIveR T7 SP6pDsRED1-C1 pDsRED-C1-F pECFP-C-3 pDsRed1-N1 pEGFP-N-5 RFP-NrevpDsRed-Monomer-N1 pEGFP-N-5 pDsRed1-NpDsRED2-C1 pDsRED-ex-C1-F pEGFP-N-3’pEGFP-N-5 pDsRed1-N pDSRED2-N1pDSRED-N1 pEGFP-N-5’ PDSRED-N-R pEASY-BLUNT M13F/T7 M13RTerpEASY-E1 T7 T7pEASY-T1 M13F/T7 M13RpEASY-T3 M13F/T7 M13R/SP6 pECFP-C pEGFP-C-5’ pECFP-C-3' pECFP-N1 pEGFP-N-5’ pEGFP-N-3’pEF/myc/ER pEF-F pCDNA3.1R pEGFP-C pEGFP-C-5’ pEGFP-C-3' pEGFP-N pEGFP-N-5’ pEGFP-N-3’ pENTR/D-TOPO M13F M13RTerpET-*(His) T7 T7pET22(a,b,c) T7 T7TerTerpET28,30,24,49 T7 T7TerpET32(a,b,c) T7/S.tag T7pET-42a (-b, -c)(+) Stag T7 TerpET50 T7/s.tag T7TERpET44 T7/s.tag T7Ter pEYFP-N1 pEGFP-N-5’ pEGFP-N-3’ PETBlue M13-20 PETBlueDOWN PETDUET-1(MCSI) PBRrevBam DuetDOMN1 PETDUET-1(MCSII) DuetUP2 T7TERpEYFP-C1 pEGFP-C-5’ pEGFP-C-3' pEYFP-N1 pEGFP-N-5’ pEGFP-N-3’ pFastBac pFastBac-F pFastBac-R pFastBac HT_A,B,C pFastBac-PH pECFP-C-3 pFastBac1 pFastBac-PH pECFP-C-3 pFastBac Dual (MCSI) pFastBac-PH pECFP-C-3 pFLAG-CMV CMV30/pFLAG-CMV-F CMV-24/pFLAG-CMV-Rp3xFlag-CMV-7.1 CMV-F/CMV24 CMV30pGAD424 5’AD 3'ADpGADGH 5’AD 3'ADpGADT7-Rec 5’AD/T7 3'ADpGAPZa-A/B/C a-FACTOR 3'AOXforward 3'AOXpGAPZ-A/B/C pGAPpGBKT7 T7 3'BDpGEM-T(-Easy) M13F/T7 SP6/M13RpGEX-(*)T pGEX-5’ pGEX-3’pGL2 pCMV5R PGL3-Pgl3-BASIC pGL3+ PGL3-pGL3-Enhancer pGL3+ PGL3-PinPoint TM PinPoint Primer SP6pIRES PIRES-F/T7 T3/PIRES-RpIRES2-EGFP pIRES2-EGFP.P5’ pIRES2-EGFP.P3’ pLenti6/v5-Dest pEGFP-N-5’PLEXA PLEXA-F PLEXA-R PLNCX PLNCX-F PLNCX-R pLXSN pLXSN-F pLXSN-RPjc1-tac T7pJet.1.21.Blunt T7primer M13F(-47) pMAL-c2E MalEP3/PMAL-C2X-R pMAL-C2x P5/PMAL-C2X-Fprimer M13F(-47) pMAL-p2X MalEPMD18-T M13R(-48) M13F(-47) PMD19-T M13F(-47) M13R(-48) pMIR-report M13FPPC86 PPC86-F PPC86-R3’AOXpPIC9K 5’AOX/a-factor3'AOX pPICZa 5'AOX/PPICZa-FpPROEXHTA M13R(-48)pQE30or40 pQE30+ pQE30- pRECEIVER CMV-FpRSET T7 T7TER PRSFDUET-1(MCSI) DuetUP1 DuetDOWN1 PRSFDUET-1(MCSII) DuetUP2 T7TER pShuttle-CMV pShuttle-CMV-F pShuttle-CMV-R PSILENCE-1.0-U6 T7 T3pSilencer3.1-H1 hygro M13F(-47) 3.0revpSILENCEV2.0-U6 T7 2.0revpSK01-T M13F M13RpSK-CMV T7 T3Psos H1PSP64/65 SP6pSP72 T7 SP6T7/M13RpSport1 SP6/M13F(-47)pSTBlue-1 T7 SP6pSUPER T7 T3pT7Blue(R) T7 M13F(-47)pTA2 M13F/T7 T3/M13R/T7 M13RpT-Adv M13FpTARGET TM pTarget-F/T7 arget-RPTHIOHISA,B,C TRX-FORWARD TRX-REVERSE pTO-T7 T7 T7TERPBV220-/PTRC99C-R pTRC99a-c PQE30+/PTRC99C-F pTriPLEx2 PT5/5'TriplEx2 T7pTz57r/t M13+ M13-pTWIN-1 T7 T7TERpUC18(19)/118(119) M13F(-47) M13R(-48)M13F(-47)/M13F pUC57 M13R(-48)/M13RpUCm-T M13F/T7 M13RpVAX1 T7 BGHT3/M13R/M13R(-48) pWSK29 T7/M13F/M13F(-47)pXT7 T7 SP6pYES2 T7 pYes2.R。
一个杨树GDSL基因组织表达的特性及其在拟南芥异源的表达

一个杨树GDSL基因组织表达的特性及其在拟南芥异源的表达作者:奈婕菲程玉祥来源:《江苏农业科学》2014年第03期摘要:GDSL酯酶为脂肪水解酶家族的一个分支,参与植物生长发育和防御反应等多种功能。
半定量RT-PCR分析结果显示,毛果杨GDSL酯酶基因Potri.002G253400在顶端茎组织中高丰度特异性表达;构建Potri.002G253400-GFP融合的植物表达载体,转化模式植物拟南芥并获得其过量表达的转基因株系7个;激光共聚焦显微镜检测结果显示,GFP荧光蛋白高丰度表达于转基因植株根的细胞壁区域,说明Potri.002G253400蛋白可能定位于细胞壁。
关键词:毛果杨;拟南芥;GDSL酯酶;载体构建;转基因中图分类号: S792.110.4 文献标志码: A 文章编号:1002-1302(2014)03-0016-03GDSL酯酶是脂肪水解酶超家族的一个亚家族,它具有广泛的底物特异性和专一性,能够水解多种酯类物质[1]。
它因具有GDS(L)保守区域(pfam,PF00657)简称GDSL,其中G、D和S分别代表甘氨酸、天冬氨酸和丝氨酸等氨基酸残基[2]。
近年来,人们从水稻、向日葵、拟南芥和玉米等多种植物体内分离出GDSL酯酶,并鉴定出它具有脂肪酰酯水解酶活性[3]。
植物GDSL酯酶是一个多基因家族,在12个不同的植物物种内发现GDSL酯酶成员超过1 100个,如苔藓、葡萄、高粱、水稻、拟南芥和杨树基因组各存在57、96、130、144、108、126个GDSL家族成员[4-5]。
GDSL酯酶参与植物发育、形态发生、次级代谢合成及多种防御反应[6-8]。
最近,Dharmawardhana等报道了杨树茎由初级到次级生长转变中转录组变化模式[9],一个GDSL基因Potri.002G253400是逐渐降低转录组聚类成员之一,这一信息初步暗示它可能参与了杨树茎的初级生长。
本研究通过半定量RT-PCR手段鉴定Potri.002G253400在不同木质化程度茎节中转录表达模式,构建Potri.002G253400融合绿色荧光蛋白基因GFP的植物表达载体,在拟南芥中过量表达Potri.002G253400-GFP并分析该蛋白细胞内的定位情况。
哺乳动物病毒表达系统选择指南

哺乳动物病毒表达系统选择指南三、慢病毒表达系统选择指南1 ViraPower™ 慢病毒表达系统的优势2 ViraPower™慢病毒表达系统操作流程3 ViraPower™慢病毒表达系统选择依据及相关产品选择指南4 ViraPower™慢病毒表达系统使用常见问题及解决办法1 ViraPower™ 慢病毒表达系统的优势ViraPower™慢病毒表达系统可用于任何哺乳动物细胞的稳定表达,不管是难转染的细胞,还是动物模型或者为了更有效的将外源基因导入细胞,采用ViralPower慢病毒表达系统,可以获得非复制型的病毒颗粒,它们可用于有效转导几乎所有分裂性或非分裂型细胞类型。
并可准确调控被转导细胞的数量和整合到细胞中的基因拷贝数,同时实验具有可重复性。
与传统的逆转录病毒比较,慢病毒可以通过顺式作用元件整合到非分裂细胞的胞核中,可以在几乎任何细胞类型中实现稳定的、长达几周甚至几个月的高水平表达。
慢病毒表达系统既可以进行瞬时表达,也可以通过杀稻瘟霉素(Blasticidin)或Zeocin等选择试剂进行筛选,将目的基因稳定整合到靶细胞的基因组中,在CMV或UbC启动子的作用下,获得外源基因的组成型或可调节的稳定表达。
与传统的逆转录病毒不一样的是,ViralPower慢病毒表达系统可以直接转导非分裂细胞,而不需要病毒复制和细胞复制。
即使是生长抑制或药物抗性的细胞、非分裂原代细胞、干细胞等,都可用慢病毒进行转染。
ViraPower™慢病毒表达系统与逆转录病毒相同的是,病毒包装是顺式进行的,由于去除了病毒的关键蛋白,因而对人体是安全的。
以下几点设计确保了安全性:●在ViraPower™ 包装混合物(ViraPower™ Packaging Mix)中缺失了LTRs,从而使用于包装的载体旨在用于表达的细胞系中表达而不会被包装成病毒颗粒;●病毒颗粒是复制缺陷型,因而不会产生其它病毒类型●已被改造为自身失活性,病毒转导并整合到细胞后不再具有产生具有包装能力的病毒基因组ViraPower™慢病毒表达系统除了安全性以外,还具有以下特点●有效转导分裂和非分裂细胞●可进行长期的基因表达●可重复性高●可根据需要选择组织特异性或细胞特异性启动子●可以采用荧光报告基因优化和调节蛋白表达2 ViraPower™慢病毒表达系统操作流程采用ViraPower™慢病毒表达系统仅需几个步骤即可获得稳定的基因表达(如图3)。
2个不同番茄品系fasciated基因克隆及表达载体构建

与植物生长调节物质、温度、光照、营养等条件有关,各种外界 环境均是通过影响番茄本身的遗传基因的表达而对番茄心室 形成起作用的 。 [3-5]
在实验室经多代自交获得,二者心室数及果实大小差异明显。 1.1.2 入门克隆载体 pENTR/D-TOPO 、卡那霉素抗性、 pCR 8/GW/TOPO 、壮 观 霉 素 抗 性,统 一 购 自 美 国 英 杰
20世纪以来,我国番茄生产中畸形果发生率高达 20%以 FL1番茄,心室数差异明显,但 fasciated的序列未知。为了明
上,特别是冬春季节温室番茄生产中畸形果发生率更高。在 确该基因在 2个供试材料中的序列差异,明确供试番茄材料
大果型番茄品种中,每一花序最先开的花畸形果发生最多,以 的心室数差异是否由 fasciated序列差异或是转录水平差异引
第一花序畸形果发生率最高,这严重影响番茄的食用品质,且 起,同时为了进一步通过该基因的过表达和沉默植株研究其
果实畸形,外观品质欠佳,亦影响销售,在生产中造成严重的 调控机理,进行了本试验。
经济损失[1]。因此,许多科技工作者就如何减少番茄畸形果 的发生进行了一系列的研究,目前已经明确了番茄心室数是
1 材料与方法
pB7GWIWG2(Ⅰ)(RNAi载体),分别以抗潮霉素 hyg基因和 抗除草剂 bar基因作为筛选标记,2个载体均由笔者所在实验
交已完全纯合 的 材 料,分 别 命 名 为 多 心 室 MLK1和 少 心 室 室保存备用。
1.1.4 菌种 pGEM -T载体及大肠杆菌 Escherichiacoli感
收稿日期:2018-01-10
受态细胞购自北京天根生化科技有限公司。
基金项目:福建省自然科学基金面上项目(编号:2015J01609)。
关于TOPO和GATEWAY

Gateway®克隆的强大性和灵活性
在目的基因(go或者开放阅读框(ORF)克隆进Gateway®入门载体后,可以很容易将它转移到任何目的载体,创建各种各种各样的表达克隆。当每一个基因具有进行Gateway®重组的att序列时它们就可能在载体间穿梭。
Gateway技术原理
Gateway克隆技术是利用λ噬菌体与大肠杆菌的染色体之间发生的λ嗜菌体位点特异重组系统的重组整合与切出反应(attB x attP →attL x attR)BP和LR两个反应构成的(表2和图4)。BP反应利用一个attB DNA片段或表达克隆和一个attP供体载体之间的重组反应,创建一个入门克隆。LR反应是一个attL入门克隆和一个attR目的载体之间的重组反应。LR反应用来在在平行的反应中转移目的序列到一个或更多个目的载体。
6)用10-50μl,涂于平板上,正放5min后,37摄氏度倒置培养过夜
7)直接进行PCR检测,或挑取菌落在50μl含有50μg/ml氨苄或25μg/ml Zeocin™的LB培养基(置于200μl微量离心管中)培养4h
或者:
Fresh PCR product1.33 µl(from Step 1)
Salt Solution0.33µl(in kit(试剂盒)in-20°Cfreezer)
TOPO Vector(载体)0.33 µl(in kit(试剂盒)in-20°Cfreezer)
FINAL VOLUME2.00µl
1)连接:按上表进行配制溶液后,轻轻混匀,室温静置5min
2)转化:将试管置于冰盒中进行冰浴5-30min
(图2)
TOPO克隆高效性原理
Topo连接反应有两个分子参与,而传统的连接反应有三个分子参与,其热力学上的优势导致5分钟的快速连接。
利用GFP-Trap和酵母双杂交技术筛选TaPR1互作靶标

第44卷第5期2021年9月河北农业大学学报JOURNAL OF HEBEI AGRICULTURAL UNIVERSITYVol.44 No.5Sep.2021利用GFP-Trap和酵母双杂交技术筛选TaPR1互作靶标申松松,王 菲,耿怀民,孟麟硕,冯 燕,王海燕(河北农业大学 植物保护学院/河北省农作物病虫害生物防治工程技术研究中心,河北 保定 071000)摘要:病程相关蛋白PR1(pathogenesis related PR1 proteins, PR1)在小麦抗病防御反应中发挥重要作用。
本试验是在前期研究基础上,借助烟草成功表达TaPR1蛋白,利用 GFP-Trap 技术结合质谱分析获得与TaPR1互作的蛋白。
同时,利用酵母双杂交技术(Yeast two-hybrid, Y2H)在接种叶锈菌生理小种PHNT的‘中国春’小麦酵母文库中筛选获得与TaPR1互作的寄主蛋白。
经过序列比对分析,2种方法共同筛选出谷氨酰氨合成酶(Glutamine synthetase)、细胞色素b6(Cytochrome b6)、应激蛋白(Stress protein)等可能与TaPR1互作的蛋白,为进一步深入研究TaPR1参与小麦叶锈病防御反应的分子机制提供了理论基础。
关 键 词:小麦叶锈菌;病程相关蛋白PR1;GFP-Trap;酵母双杂交;互作蛋白中图分类号:S432.1 开放科学(资源服务)标识码(OSID):文献标志码:AScreening of interacting target of TaPR1 using GFP-Trapand yeast two-hybrid techniqueSHEN Songsong, WANG Fei, GENG Huaimin, MENG Linshuo,FENG Yan, WANG Haiyan(College of Plant Protection, Hebei Agricultural University / Biological Control Technological Innovation Center ofPlant Diseases and Plant Pests of Hebei Province, Baoding 071000, China)Abstract: Pathogenesis related PR1 (pathogenesis related PR1 proteins, PR1) proteins played an important role inwheat disease defense response. On the basis of the previous study, the TaPR1 protein was successfully expressedby tobacco, the proteins interacting with TaPR1 were obtained by GFP-Trap technique combined with massspectrometry. At the same time, host proteins interacting with the TaPR1 were screened from the yeast libraryof‘Chinese Spring’wheat inoculated with physiological race PHNT of leaf rust by yeast two-hybrid technique(Yeast two-hybrid, Y2H). Sequence analysis showed that Glutamine synthetase, Cytochrome B6, Stress protein andother proteins that might interact with the TaPR1 were screened by both two methods. The study laid a theoreticalfoundation for exploring the molecular mechanism of the TaPR1 participating in wheat leaf rust disease defenseresponse.Keywords: wheat leaf rust; pathogenesis related PR1 proteins; GFP-Trap; yeast two-hybrid; interacting proteins收稿日期:2021-03-19基金项目: 河北省自然科学基金(C2020204028).第一作者:申松松(1996-),男,河北邯郸人,硕士研究生,主要从事分子植物病理学研究.E-通信作者: 王海燕(1977-),女,河北遵化人,博士,教授,主要从事分子植物病理学研究.E-本刊网址:http: //hauxb. hebau. edu. cn文章编号:1000-1573(2021)05-0064-07DOI:10.13320/ki.jauh.2021.008565第5期作为世界上最重要的粮食作物之一,小麦的产量受多个方面的影响,其中对产量影响最大的是小麦病害。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
Vector Map: pENTR/D-TOPONAME: pENTR/D-TOPORESISTANT MARKER: Kanamycin resistant; 25 µg/mlTechnologies SOURCE: InvitrogenLifevectorV_TYPE: GatewayentrySEQUENCING PRIMERS:M13(-21), M13 reverse, T7POLYLINKER SEQUENCE:TAACGCTAGCATGGATGTTTTCCCAGTCACGACGT TGTAAAACGACGGCCAGT CTTAAGCTCGGGCCCCAAATAATGATTTTATTTTGACTGATAGTGACCTGTTCGTTGCAACAAATTGATGAGCAATGCTTTTTTATAATGCCAACTTTGTACAAAAAAGCAGGCTCCGCGGCCGCCCCCTTCACC-ORF-AAGGGTGGGCGCGCCGACCCAGCTTTCTTGTACAAAGTTGGCATTATAAGAAAGCATTGCTTATCAATTTGTTGCAACGAACAGGTCACTATCAGTCAAAATAAAATCATTATTTGCCATCCAGCTGATATC CCCTATAGTGAGTCGTATTAC ATGGTCATAGCTGTTTCCTGGCAGCTCTGSequence:1 CTTTCCTGCG TTATCCCCTG ATTCTGTGGA TAACCGTATT ACCGCCTTTG AGTGAGCTGA 6061 TACCGCTCGC CGCAGCCGAA CGACCGAGCG CAGCGAGTCA GTGAGCGAGG AAGCGGAAGA 120121 GCGCCCAATA CGCAAACCGC CTCTCCCCGC GCGTTGGCCG ATTCATTAAT GCAGCTGGCA 180181 CGACAGGTTT CCCGACTGGA AAGCGGGCAG TGAGCGCAAC GCAATTAATA CGCGTACCGC 240241 TAGCCAGGAA GAGTTTGTAG AAACGCAAAA AGGCCATCCG TCAGGATGGC CTTCTGCTTA 300301 GTTTGATGCC TGGCAGTTTA TGGCGGGCGT CCTGCCCGCC ACCCTCCGGG CCGTTGCTTC 360361 ACAACGTTCA AATCCGCTCC CGGCGGATTT GTCCTACTCA GGAGAGCGTT CACCGACAAA 420421 CAACAGATAA AACGAAAGGC CCAGTCTTCC GACTGAGCCT TTCGTTTTAT TTGATGCCTG 480481 GCAGTTCCCT ACTCTCGCGT TAACGCTAGC ATGGATGTTT TCCCAGTCAC GACGTTGTAA 540541 AACGACGGCC AGTCTTAAGC TCGGGCCCCA AATAATGATT TTATTTTGAC TGATAGTGAC 600601 CTGTTCGTTG CAACAAATTG ATGAGCAATG CTTTTTTATA ATGCCAACTT TGTACAAAAA 660661 AGCAGGCTCC GCGGCCGCCC CCTTCACCAA GGGTGGGCGC GCCGACCCAG CTTTCTTGTA 720721 CAAAGTTGGC ATTATAAGAA AGCATTGCTT ATCAATTTGT TGCAACGAAC AGGTCACTAT 780781 CAGTCAAAAT AAAATCATTA TTTGCCATCC AGCTGATATC CCCTATAGTG AGTCGTATTA 840841 CATGGTCATA GCTGTTTCCT GGCAGCTCTG GCCCGTGTCT CAAAATCTCT GATGTTACAT 900901 TGCACAAGAT AAAAATATAT CATCATGAAC AATAAAACTG TCTGCTTACA TAAACAGTAA 960961 TACAAGGGGT GTTATGAGCC ATATTCAACG GGAAACGTCG AGGCCGCGAT TAAATTCCAA 10201021 CATGGATGCT GATTTATATG GGTATAAATG GGCTCGCGAT AATGTCGGGC AATCAGGTGC 10801081 GACAATCTAT CGCTTGTATG GGAAGCCCGA TGCGCCAGAG TTGTTTCTGA AACATGGCAA 11401141 AGGTAGCGTT GCCAATGATG TTACAGATGA GATGGTCAGA CTAAACTGGC TGACGGAATT 12001201 TATGCCTCTT CCGACCATCA AGCATTTTAT CCGTACTCCT GATGATGCAT GGTTACTCAC 12601261 CACTGCGATC CCCGGAAAAA CAGCATTCCA GGTATTAGAA GAATATCCTG ATTCAGGTGA 13201321 AAATATTGTT GATGCGCTGG CAGTGTTCCT GCGCCGGTTG CATTCGATTC CTGTTTGTAA 13801381 TTGTCCTTTT AACAGCGATC GCGTATTTCG TCTCGCTCAG GCGCAATCAC GAATGAATAA 14401441 CGGTTTGGTT GATGCGAGTG ATTTTGATGA CGAGCGTAAT GGCTGGCCTG TTGAACAAGT 15001501 CTGGAAAGAA ATGCATAAAC TTTTGCCATT CTCACCGGAT TCAGTCGTCA CTCATGGTGA 15601561 TTTCTCACTT GATAACCTTA TTTTTGACGA GGGGAAATTA ATAGGTTGTA TTGATGTTGG 16201621 ACGAGTCGGA ATCGCAGACC GATACCAGGA TCTTGCCATC CTATGGAACT GCCTCGGTGA 16801681 GTTTTCTCCT TCATTACAGA AACGGCTTTT TCAAAAATAT GGTATTGATA ATCCTGATAT 17401741 GAATAAATTG CAGTTTCATT TGATGCTCGA TGAGTTTTTC TAATCAGAAT TGGTTAATTG 18001801 GTTGTAACAC TGGCAGAGCA TTACGCTGAC TTGACGGGAC GGCGCAAGCT CATGACCAAA 18601861 ATCCCTTAAC GTGAGTTACG CGTCGTTCCA CTGAGCGTCA GACCCCGTAG AAAAGATCAA 19201921 AGGATCTTCT TGAGATCCTT TTTTTCTGCG CGTAATCTGC TGCTTGCAAA CAAAAAAACC 19801981 ACCGCTACCA GCGGTGGTTT GTTTGCCGGA TCAAGAGCTA CCAACTCTTT TTCCGAAGGT 20402041 AACTGGCTTC AGCAGAGCGC AGATACCAAA TACTGTCCTT CTAGTGTAGC CGTAGTTAGG 21002101 CCACCACTTC AAGAACTCTG TAGCACCGCC TACATACCTC GCTCTGCTAA TCCTGTTACC 21602161 AGTGGCTGCT GCCAGTGGCG ATAAGTCGTG TCTTACCGGG TTGGACTCAA GACGATAGTT 22202221 ACCGGATAAG GCGCAGCGGT CGGGCTGAAC GGGGGGTTCG TGCACACAGC CCAGCTTGGA 22802281 GCGAACGACC TACACCGAAC TGAGATACCT ACAGCGTGAG CATTGAGAAA GCGCCACGCT 23402341 TCCCGAAGGG AGAAAGGCGG ACAGGTATCC GGTAAGCGGC AGGGTCGGAA CAGGAGAGCG 24002401 CACGAGGGAG CTTCCAGGGG GAAACGCCTG GTATCTTTAT AGTCCTGTCG GGTTTCGCCA 24602461 CCTCTGACTT GAGCGTCGAT TTTTGTGATG CTCGTCAGGG GGGCGGAGCC TATGGAAAAA 25202521 CGCCAGCAAC GCGGCCTTTT TACGGTTCCT GGCCTTTTGC TGGCCTTTTG CTCACATGTT 2580All ORF clones are provided in Entry Vectors for the Invitrogen Gateway® cloning system. Invitrogen offers a whole range of Destination Vectors for a wide range of applications. Refer to the homepage of Invitrogen for further information on the Gateway® cloning system and related Invitrogen products at:/content.cfm?pageid=4072For further information on the ORFeome Collaboration, visit their homepage at /html/index.shtml.For furhter technical information visit our homepage at: http://www.dnaform.jp or contact us under: ORF@dnaform.jp.。
