荧光原位杂交技术和流式细胞仪用于环境样品中产毒及非产毒微囊藻的定量监测
微囊藻毒素的荧光定量 PCR检测法

微囊藻毒素的荧光定量 PCR检测法摘要:荧光定量 PCR检测方法具有灵敏度高、精确度高、检测范围宽、重复性好优势,本文就其方法对水体中的微囊藻进行检测。
关键词:荧光定量PCR;微囊藻毒素;检测随着我国工农业的迅猛发展以及城市化进程的加快,生活污水、工业废水中污染物的排放量日益增加,环境污染越来越严重,造成水体富营养化及污染日益严重。
水中过量的氮、磷等营养性物质,使水中蓝藻等藻类过渡繁殖,蓝藻水华暴发频繁。
藻类水华爆发严重威胁城市水源水安全。
因此,及时监测城市源水蓝藻发生情况,对保证城市供水安全具有重要意义。
蓝藻监测,传统的方法主要通过显微镜镜检法,对水体中的藻细胞计数及分类,后来又有通过藻类的16S rDNA 序列分析进行藻种分类,然而这些方法不能区分产毒微囊藻及非产毒微囊藻,并在蓝藻暴发之前对其进行准确预测。
实时荧光定量PCR(Real- time fluorescence quantitative PCR,qPCR)方法是一种特异性强、敏感性高、准确性好的分子检测方法,已被用于产毒微囊藻的监测。
微囊藻毒素由一些藻毒素合成酶基因簇控制,该基因簇由 mcyA~mcyJ等10个基因构成。
通过检测微囊藻毒素的产毒基因 mcyA mcyBmcyD 可以用于监测水体中的产毒微囊藻细胞。
mcyE 基因可以用于设计种特异性引物,用于监测水体中的产生微囊藻毒素的不同产毒藻种。
Ouahid 等利用 mcyE 等藻毒素合成酶基因监测水体中的产毒微囊藻[5]。
Al- Tebrineh 等利用 mcyE 和 ndaF 基因设计的实时荧光定量 PCR 方法,同时监测水体中的不同微囊藻毒素产毒藻种[3]。
由于不同水域环境中水体的产毒微囊藻的种类及分布不同,本文利用荧光定量 PCR技术,对水体中的微囊藻进行检测。
1 材料与方法1.1 藻种来源和培养条件实验用藻种购买于中国科学院典型培养物保藏委员会淡水藻种库,藻种编号为鱼腥藻(Anabaena sp)FACHB-82水华束丝藻( Aphanizomenon flos-aque)FACHB-1039铜绿微囊藻FACHB-315柱胞鱼腥藻(Ana-baena cylindrica)FACHB-170 席藻(Phormidium sp.)FACHB- 1099 水华束丝藻 FACHB- 245 颤藻(Oscillatoriasp.)FACHB- 1053,于 BG- 11 培养基中扩大培养,颤藻 FACHB-528在 SE培养基中扩大培养。
荧光原位杂交(FISH)技术检测水体中大肠菌群研究

荧光原位杂交(FISH)技术检测水体中大肠菌群研究王建龙【期刊名称】《中国生物工程杂志》【年(卷),期】2004(24)2【摘要】大肠菌群广泛用作饮用水的细菌学检测指标。
传统的检测方法有多管发酵法和滤膜法。
这些方法存在一些缺点 ,如检测时间长、专一性差、干扰因素多。
因此 ,迫切需要一些快速、灵敏的检测方法。
FISH(fluorescentinsituhybridization)技术利用寡核苷酸探针检测特定细胞内的互补核苷酸序列。
针对于Enterobacteriaceae的 1 6SrRNA分子区域专门设计的探针可以用于饮用水样品的微生物检测。
FISH技术可以用于饮用水中大肠菌群的检测 ,并且在较短的时间内 ( 6~ 8h)给出定量分析结果 ,但该技术用于日常检测尚需更深入的研究。
【总页数】4页(P70-73)【关键词】饮用水;检测;FISH;荧光原位杂交;大肠菌群【作者】王建龙【作者单位】清华大学核能技术研究院环境技术研究室【正文语种】中文【中图分类】R123【相关文献】1.荧光原位杂交(FISH)技术在转基因小鼠检测中的应用 [J], 谢建云;邵伟娟;潘漪清;高诚2.荧光原位杂交(FISH)技术与免疫组织化学(IHC)技术应用于乳腺癌患者Her2基因检测的评价 [J], 黄俊;邓明凤;郭华雄;陈登峰;王昌富3.工业化学品的致突变研究部分:—用荧光原位杂交(FISH)检测接触二硫化碳CS2工人精子X染色体非整倍体率的初步研究 [J], 郑履康;邓丽霞;等4.用三色荧光原位杂交(Three-Color FISH)检测小鼠精子中染色体数目异常的研究[J], 余龙;张坚宣;史庆华;I.-D.Adler因版权原因,仅展示原文概要,查看原文内容请购买。
实时荧光定量PCR方法检测南太湖入湖口产毒微囊藻

实时荧光定量PCR方法检测南太湖入湖口产毒微囊藻刘洋;胡佩茹;马思三;叶金云【摘要】南太湖入湖口产毒微囊藻的丰度对于周边县市取水口的水质安全和太湖水质有着重要影响.以藻毒素合成酶基因mcyE/ndaF为靶基因,建立实时荧光定量PCR检测产毒微囊藻的方法,并对南太湖入湖口7个监测点水样中产毒微囊藻的丰度进行检测,结果表明:该实时荧光定量PCR方法的特异性强及准确性、重复性较好.建立铜绿微囊藻(Microcystis aeruginosa)的标准曲线方程为y=-3.454x+49.88,斜率为-3.454,R2=0.991,扩增效率E为94.6%,定量检测区间为1.689×104~1.689×108拷贝数/μl.对南太湖入湖口7个监测点检测表明,夹浦和合溪2个监测点的产毒微囊藻数量最高,预测产毒微囊藻浓度分别为(1.99±0.35) ×105和(1.47±0.23) ×105 cells/ml.7个监测点的产毒微囊藻的种类较为一致,均为铜绿微囊藻.该方法可以快速准确地检测水体中微囊藻毒素产毒藻种种类和数量,为蓝藻水华监测、预警提供技术依据.【期刊名称】《湖泊科学》【年(卷),期】2016(028)002【总页数】7页(P246-252)【关键词】实时荧光定量PCR;产毒微囊藻;入湖口;南太湖;铜绿微囊藻【作者】刘洋;胡佩茹;马思三;叶金云【作者单位】湖州师范学院生命科学院,湖州313000;浙江省水生生物资源养护与开发技术研究重点实验室,湖州313000;中国水产科学研究院水生动物繁育与营养重点实验室,湖州313000;湖州师范学院生命科学院,湖州313000;湖州师范学院生命科学院,湖州313000;湖州师范学院生命科学院,湖州313000;浙江省水生生物资源养护与开发技术研究重点实验室,湖州313000;中国水产科学研究院水生动物繁育与营养重点实验室,湖州313000【正文语种】中文©2016 by Journal of Lake Sciences微囊藻毒素(Microcystin,MC)是一些产毒微囊藻产生的单环七肽,共有100多种异构体.它是一种肝毒素,长期饮用有微囊藻毒素的水,会引发肝损伤甚至肝癌[1-3].微囊藻毒素主要由微囊藻属(Microcystis)、鱼腥藻属(Anabaena)、束丝藻属(Aphanizomenon)、念珠藻属(Nostoc)、席藻属(Phormidium)和浮丝藻属(Planktothrix)等一些藻种产生[4].产毒微囊藻的暴发,对于人类的公共安全、水生动物及牲畜造成极大的危害[5].在我国大部分富营养化水体中,铜绿微囊藻(Microcystis aeruginosa)在数量和发生频率上均占优势.太湖是我国第三大淡水湖泊,水域面积约2428 km2.近年来,由于太湖流域经济的飞速发展,向水体中排放了大量污染物,造成水体污染和富营养化日益严重,蓝藻水华暴发频繁[6].南太湖位于太湖南岸,一般指浙江省内沿岸部分湖面,管辖水域面积约300 km2,湖岸线长64 km.苕溪水系和长兴水系是南太湖主要的入湖水系,这2条水系共有大钱港、新港口、小梅口、杨家浦、合溪、新塘和夹浦7个入湖口.南太湖入湖水系与太湖水域季节性水交换频繁,洪水期由入湖水系向太湖蓄水,河道枯水期,太湖水体倒灌入入湖河道[7-8].南太湖附近几个县市饮用水取水口位于这几条入湖溪流内.因太湖水倒灌,太湖蓝藻对南太湖入湖河道水质的影响巨大,严重威胁该区域供水安全[8].因此,及时监测该区域的蓝藻发生情况,对保证该区域的供水安全具有重要意义.蓝藻水华监测,传统方法主要通过显微镜镜检法,对水体中的藻细胞计数及分类,后来又有通过藻类的16S rDNA序列分析进行藻种分类,然而这些方法不能区分产毒微囊藻及非产毒微囊藻,并在蓝藻暴发之前对其进行准确预测[9-10].实时荧光定量PCR(Real-time fluorescence quantitative PCR,qPCR)方法是一种特异性强、敏感性高、准确性好的分子检测方法,已被用于产毒微囊藻的监测[9].微囊藻毒素由一些藻毒素合成酶基因簇控制,该基因簇由mcyA~mcyJ等10个基因构成.通过检测微囊藻毒素的产毒基因mcyA、mcyB、mcyD可以用于监测水体中的产毒微囊藻细胞[11-14].mcyE基因可以用于设计种特异性引物,用于监测水体中的产生微囊藻毒素的不同产毒藻种[15-16].Ouahid等利用mcyE等藻毒素合成酶基因监测水体中的产毒微囊藻[17].Al-Tebrineh等利用mcyE和ndaF基因设计的实时荧光定量PCR方法,同时监测水体中的不同微囊藻毒素产毒藻种[15].由于不同水域环境中水体的产毒微囊藻的种类及分布不同,本研究利用mcyE/ndaF基因为目标模版,建立实时荧光定量PCR方法,对南太湖入湖口7个水质监测点水体中的产毒微囊藻进行检测,为该区域的蓝藻水华监测、预警和水质安全提供依据.1.1 藻种来源和培养条件实验用藻种购买于中国科学院典型培养物保藏委员会淡水藻种库,藻种编号为鱼腥藻(Anabaena sP.)FACHB-82、水华束丝藻(Aphanizomenon flos-aque)FACHB-1039、铜绿微囊藻 FACHB-315、柱胞鱼腥藻(Anabaena cylindrica)FACHB-170、席藻(Phormidium sP.)FACHB-1099、水华束丝藻FACHB-245、颤藻(Oscillatoria sP.)FACHB-1053,于BG-11培养基中扩大培养,颤藻FACHB-528在SE培养基中扩大培养.培养温度为25℃,光照强度为30 μE/(m2·s),光暗比12 h:12 h.1.2 藻细胞基因组DNA的提取1.2.1 基因组DNA提取分别采用2种方法提取藻细胞基因组DNA:方法1按植物基因组DNA提取试剂盒(SK8203,上海生工)的操作程序,提取藻细胞基因组DNA.取藻液10 ml,12000转/min离心15 min,弃上清,取沉淀用于基因组DNA提取;方法2基因组DNA的快速提取,参考文献[18].1.2.2 DNA浓度与纯度检测分别取藻类基因组的DNA各5 μl,加入495 μl TE缓冲液稀释,用微量核酸蛋白测定仪(Beckman coulter DU-530,美国)测定DNA浓度,测定A260和A280,DNA浓度计算公式为:[DNA]= A260×稀释倍数100×40 μg/ml.1.3 PCR引物设计与PCR扩增根据mcyE和ndaF基因设计普通PCR和荧光定量PCR引物,具体参考文献[15],引物序列见表1.16S rDNA和mcyE/ndaF基因的PCR反应体系:10 μmol/L引物-F 1 μl,10μmol/L引物-R 1 μl,10×PCR buffer 2 μl,10 μmol/L dNTP 2 μl,25 mmol/L MgCl21.5 μl,模版DNA 1 μl,Tag酶(5U/μl)0.5 μl,dd H2O 17μl,总体积25 μl,PCR反应条件如下:94℃预变性4 min,94℃变性30 s,退火温度分别为50℃和56℃,30 s,72℃延伸30 s,30个循环,72℃,5 min.将PCR产物在3%琼脂糖凝胶进行电泳.1.4 荧光定量PCR在Bio-rad CFX3700仪器上进行荧光定量PCR扩增,PCR反应体系:Hotstart fluo-PCR mix(SK2956A,上海生工)10 μl,10 μmol/L cyano-real mcyE-F 0.5 μl,10 μmol/L cyano-real mcyE-R 0.5 μl,0.1%(w/v)BSA 2 μl,10%(w/v)PVP-30 2 μl,模版DNA 1 μl,dd H2O 4 μl,总体积20 μl,PCR反应条件如下:95℃预变性10 min,95℃变性15 s,56℃退火30 s,72℃延伸30 s,40个循环,退火过程中收集荧光.每个样品重复3次.熔解曲线的过程:95℃ 15 s,从60℃开始升高至95℃,每30 s温度升高0.5℃,进行熔解曲线分析.1.5 特异性检测提取不同藻种基因组DNA,进行普通PCR扩增和荧光定量PCR扩增,并以16S rDNA为对照,检测引物的特异性.1.6 荧光定量PCR标准曲线建立分别以铜绿微囊藻FACHB-315基因组DNA进行10倍稀释,测定DNA浓度,以基因拷贝数的对数值与反应循环数(Ct)值之间建立标准曲线.基因拷贝数的计算公式为:DNA的拷贝数=6.02×1023(coPies/mol)× DNA浓度(g/μl)/基因组DNA分子量,微囊藻基因组为4.70 Mb,1 bP碱基分子量为660 g/mol.扩增效率E=10-1/S-1,S为标准曲线线性方程的斜率.1.7 环境样品检测在南太湖入湖口的7个监测点进行采样,采样时间为2014年8月3日,水温26℃.用采水器采集表层20 cm水样.每升水样加15 ml左右鲁哥试剂.1000 ml水样直接静置沉淀24 h,用虹吸管小心抽掉上清液,余下20~25 ml沉淀物转入50 ml离心管中,4℃保存,带回实验室后利用显微镜镜检法进行藻细胞检测.用0.45 μm滤膜过滤收集藻细胞,置于-20℃条件下保存水样和藻细胞.按基因组DNA快速提取法提取DNA,并进行荧光定量PCR,检测样品中产毒微囊藻.每个样品重复3次.2.1 引物特异性检测提取不同产毒藻种的基因组DNA进行微囊藻毒素产毒基因mcyE/ndaF和16S rDNA基因PCR扩增,其中铜绿微囊藻FACHB-315为微囊藻毒素产毒藻种,鱼腥藻 FACHB-82、颤藻FACHB-528、水华束丝藻FACHB-245、颤藻FACHB-1053、席藻FACHB-1099为鱼腥藻毒素产毒藻种,柱胞鱼腥藻FACHB-1039为柱胞藻毒素产毒藻种.仅在微囊藻毒素产毒藻种铜绿微囊藻FACHB-315扩增出125 bP左右目标条带,其他产毒藻种未扩增出目标条带,说明引物特异性较好;所有藻种的16S rDNA基因均扩增出80 bP左右的目标条带,表明藻种基因组DNA提取及PCR扩增体系无问题(图1).对这些产毒藻种的荧光定量PCR结果表明,只有其中铜绿微囊藻FACHB-315有荧光检测信号,其余藻种均未能扩增出信号.2.2 荧光定量PCR灵敏度与重复性2.2.1 标准曲线的建立以铜绿微囊藻FACHB-315基因组DNA 10倍梯度稀释后作为荧光定量PCR模版DNA,进行qPCR测定.以标准品稀释后浓度的对数值为横坐标、Ct为纵坐标建立实时定量PCR的标准曲线,结果见图2.利用该qPCR反应条件检测微囊藻毒素产毒藻种,标准曲线斜率为-3.454,R2=0.991,扩增效率E=101/3.454-1=94.6%,定量检测区间为1.689×104~1.689×108拷贝数/μl.2.2.2 熔解曲线以铜绿微囊藻FACHB-315基因组DNA为模板进行荧光定量PCR,并进行熔解曲线分析,由图3可见,熔解曲线平稳,峰尖且窄,熔解温度为80±0.5℃,表明该PCR扩增产物特异,无非特异性扩增.2.2.3 重复性将不同浓度的铜绿微囊藻FACHB-315基因组DNA进行荧光定量PCR测定,结果如表2所示.基因组标准品的循环数变异系数分别为0~5.62%,表明基因组DNA的标准曲线稳定性良好,符合制备实时荧光定量PCR标准曲线的要求.2.3 全细胞荧光定量PCR检测将铜绿微囊藻FACHB-315藻细胞进行10倍系列稀释,分别取1 ml藻样,提取基因组DNA,进行qPCR,结果如表3,可检测的藻细胞浓度最低为3.26×104,mcyE/ndaF基因拷贝数为5.415×105,藻细胞浓度与mcyE/ndaF基因的相关性良好(图2),可以根据样品中的mcyE/ndaF基因拷贝数,利用y=-3.454x+49.88方程推测藻细胞浓度.2.4 南太湖入湖口产毒微囊藻检测南太湖入湖口水质对于周边县市的饮用水安全及太湖水质有着重要影响,本研究选取南太湖入湖口7个监测点,在太湖蓝藻容易暴发的8月进行取样,利用荧光定量PCR方法检测产毒微囊藻丰度,结果见表4,夹浦和合溪监测点的产毒微囊藻丰度最高.通过荧光定量PCR溶解曲线分析表明,这7个监测点的产毒微囊藻的熔解温度为80.5℃,表明产毒微囊藻的种类较为一致,均为铜绿微囊藻.在蓝藻水华暴发影响水质安全之前,快速准确地对产毒微囊藻进行监控是非常重要的.传统监测方法不能区分蓝藻水华中的产毒微囊藻与非产毒微囊藻.以微囊藻毒素产毒和节球藻毒素基因mcyE/ndaF为靶基因,应用实时荧光定量PCR方法可以快速准确的检测水体中的产毒微囊藻,可以同时检测多种产微囊藻毒素和节球藻素的不同藻种细胞,特异性强,灵敏度高、准确性好[15-16].本研究以微囊藻属、鱼腥藻属、柱孢藻属等8种不同藻种,以mcyE/ndaF为靶基因进行常规PCR和荧光定量PCR检测,表明引物特异性非常高.该结果与Al-Tebrineh等[15]的研究结果一致.通过对产毒微囊藻铜绿微囊藻FACHB-315进行灵敏度和重复性检测,发现利用qPCR对铜绿微囊藻的范围为1.689×104~1.689×108拷贝数/μl,灵敏度低于Vaitomaa等的报道[19],其对于铜绿微囊藻的检测范围为6.6×102~6.6×106mcyE基因拷贝/反应体系,可能与藻基因组提取质量有关.本研究采用了一种DNA快速提取方法,该方法可以高效快速地提取藻细胞DNA,整个过程仅需20 min,DNA提取质量经检测,纯度未达到A260/A280大于1.8,但可以用于高通量的普通PCR反应和荧光定量PCR反应.在荧光定量PCR反应中需加入2种试剂,即聚烙吡咯烷酮(PVP-30)和牛血清蛋白BSA,该试剂可以显著增强反应的灵敏度,降低反应对于模版质量的要求,与Xin等[18]的研究结果一致,对于高通量的荧光定量PCR反应十分重要.全细胞荧光定量PCR表明,藻细胞浓度与mcyE/ndaF基因的相关性良好.因此应用该方法不仅可以用于水华发生时对铜绿微囊藻定量检测,而且可以用于低密度时对该藻进行实时监控,并及时做出预警.本研究通过实时荧光定量PCR方法检测太湖蓝藻水华高发季节,南太湖入湖口产毒微囊藻的数量,研究结果表明,南太湖入湖口产毒微囊藻主要由铜绿微囊藻构成,在夹浦和合溪2个监测点铜绿微囊藻丰度较高,分别为(1.99±0.35)×105和(1.47±0.23)×105cells/ml,这与杨晓红等[8]的研究结果一致.该方法可以快速准确地对产毒微囊藻进行监测、预警,对于保证该区域的饮用水安全及了解南太湖入湖水系对太湖水质的影响具有十分重要的意义.建立了以mcyE/ndaF基因为靶基因的产毒微囊藻实时荧光定量PCR检测方法,其标准曲线方程为:y= -3.454x+49.88,斜率为-3.454,R2=0.991,扩增效率E=101/3.454-1=94.6%,定量检测区间为1.689×104~1.689×108拷贝数/μl.对南太湖入湖口7个监测点的产毒微囊藻进行了检测,其中夹浦和合溪2个监测点的产毒微囊藻丰度最高,分别为(1.99±0.35)×105和(1.47±0.23)×105cells/ml.7个监测点的产毒微囊藻种类较为一致,均为铜绿微囊藻.【相关文献】[1]张萍,钟儒刚,崔海信等.微囊藻毒素诱发癌症的作用机制研究进展.癌变·畸变·突变,2009,21(1):70-73.[2]许川,舒为群,曹佳.我国水环境微囊藻毒素污染及其健康危害研究.癌变·畸变·突变,2007,19(3):202-205.[3]陈刚,俞顺章.微囊藻毒素LR和黄曲霉毒素B1对肝脏促癌作用的实验研究.癌变·畸变·突变,1996,8 (3):129-132.[4]张敬平,肖付刚,赵晓联等.微囊藻毒素分析检测技术.北京:化学工业出版社,2009.[5] Meneely JP,Elliott CT.Microcystins:measuring human exPosure and the imPact on human health.Biomarkers,2013,18 (8):639-649.[6]李大命,孔繁翔,于洋等.太湖蓝藻水华期间水体和底泥中产毒微囊藻与非产毒微囊藻种群丰度研究.环境科学学报,2011,31(2):292-298.[7]韩志萍,邵朝纲,张忠山等.南太湖入湖口蓝藻生物量与TN、TP的年变化特征及相关性研究.海洋与湖沼,2012,43(5):911-918.[8]杨晓红,陈江,周李等.南太湖入湖口蓝藻水华时空分布规律及相关响应因子分析.中国环境监测,2011,27(2):92-96.[9] Pearson LA,Neilan BA.The molecular genetics of cyanobacterial toxicity as a basis for monitoring water quality and Public health risk.Current Opinion in Biotechnology,2008,19(3):281-288.[10] Sivonen K.Emerging high throughPut analyses of cyanobacterial toxins and toxic cyanobacteria.Advances in Experimental Medicine and Biology,2008,619:539-557. [11]何恩奇,钮伟民,吴庆刚等.产毒微囊藻mcyA基因荧光定量PCR方法的建立.环境科学与技术,2011,34 (12):66-70.[12] Fortin N,Aranda-Rodriguez R,Jing H et al.Detection of microcystin-Producing cyanobacteria in Missisquoi bay,Quebec,Canada,using quantitative PCR.Applied and Environmental Microbiology,2010,76(15):5105-5112.[13] Hautala H,Lamminmaki U,SPoof L.Quantitative PCR detection and imProved samPle PreParation of microcystin-Producing Anabaena,Microcystis and Planktothrix.Ecotoxicology and Environmental Safety,2012,87(1):49-56.[14] Via-ordorika L,Fastner J,Kurmayer R et al.Distribution of Microcystin-Producing and non-microcystin-Producing Microcystis sP.in EuroPean freshwater bodies:Detection of microcystins and microcystin genes in individual colonies.Systematic and Applied Microbiology,2004,27(5):592-602.[15] Al-tebrineh J,Gehringer M,Akcaalan R et al.A new quantitative PCR assay for the detection of hePatotoxigenic cyanobacteria.Toxicon,2011,57(4):546-554.[16] Rantala A,Rizzi E,Castiglioni B et al.Identification of hePatotoxin-Producing cyanobacteria by DNA-ChiP.Environmental Microbiology,2008,10(3):653-664. [17] Ouahid Y,del CamPo FFD.TyPing of toxinogenic Microcystis from environmental samPles by multiPlex PCR.Applied Microbiology and Biotechnology,2009,85(2):405-412.[18] Xin ZG,Jeff PV,Melvin Q et al.High-throughPut DNA extraction method suitable for PCR.Biotechniques,2003,34(4):820-824.[19] Vaitomaa J,Rantala A,Halinen K et al.Quantitative Real-time PCR for determination of microcystin synthetase E coPy numbers for Microcystis and Anabaena in lakes.Applied and Environmental Microbiology,2003,69(12):7289-7297.。
荧光原位杂交(fish)

荧光原位杂交技术荧光原位杂交技术(fluorescence in situ hybridization),简称FISH。
是利用荧光标记的特异核酸探针与细胞内相应的靶DNA分子或RNA分子杂交,通过在荧光显微镜或共聚焦激光扫描仪下观察荧光信号,来确定与特异探针杂交后被染色的细胞或细胞器的形态和分布,或者是结合了荧光探针的DNA区域或RNA分子在染色体或其他细胞器中的定位。
中文名fish外文名fluorescent in situ hybridization建立时间1986年发展历程1969年,Pardue和John等两个研究小组开始采用放射性标记DNA或28S RNA发明了原位杂交技术(ISH)。
尽管当时原位杂交技术已经具有较高的特异性和灵敏度,但鉴于放射性同位素自身特性的局限,如安全性、空间分辨率低、不稳定性等问题,这项技术仅限于实验室研究方面的应用。
1986年科研工作者开始利用异硫氰酸盐荧光素来标记探针,并在荧光显微镜下进行观察分析,建立了荧光原位杂交技术(FISH)。
1989年,Delong首次使用荧光标记寡核苷酸探针检测单个微生物细胞。
由于FISH技术具有敏感度高、信号强、背景低、快速等优点,该方法在环境微生物的检测中得到了广泛的应用。
随着科技的迅速发展,FISH探针标记物越来越多,不仅从单一荧光发展到多色荧光检测,而且应用范围也进一步扩大,不仅可以用于分裂相细胞而且可以用于间期细胞检测,为FISH技术的临床应用打下了坚实的基础。
操作步骤编辑播报(1)样品的固定;(2)样品的制备和预处理;(3)预杂交;(4)探针和样品变性;(5)用不同的探针杂交以检测不同的靶序列;(6)漂洗去除未结合的探针;(7)检测杂交信号,进行结果分析·荧光信号观察:将处理好的样品置于荧光显微镜下,选择分散较好的区域来观察。
三色(或者更多)荧光激发下,观察到不同颜色的荧光图像。
通常选用20X物镜来扫描样品杂交区域,40X或100X物镜下观察样品,从一定的方向进行计数,并对计数情况进行分析。
应用荧光原位杂交方法检测亚历山大藻

应用荧光原位杂交方法检测亚历山大藻梁斌;王欢;陈斌;郭皓【期刊名称】《海洋环境科学》【年(卷),期】2009(28)1【摘要】采用PCR方法对三株亚历山大藻核糖体DNA大亚基部分序列和ITS区序列进行了特异性扩增和序列测定。
ITS区测序及BLAST比对结果显示,三株藻类分别为塔玛亚历山大藻(Alexandrium tamarense),链状亚历山大藻(A. catenella)和微小亚历山大藻(A. minimum)。
在此基础上,针对中国沿海分布的有毒亚历山大藻,根据其核糖体DNA大亚基(LSU rD-NA)序列信息,设计了特异性的荧光标记探针,建立了基于荧光原位杂交技术(FISH)上的有毒亚历山大藻检测方法。
结果表明,探针Alexp1较理想地标记选定的目标藻——塔玛亚历山大藻(YA藻株),经探针标记的藻细胞在荧光显微镜下可以明显区分于其他非目标藻。
【总页数】5页(P80-83)【关键词】亚历山大藻;rDNA序列分析;分子探针;荧光原位杂交【作者】梁斌;王欢;陈斌;郭皓【作者单位】国家海洋环境监测中心,辽宁大连116023;大连水产学院生命科学与技术学院,辽宁大连116023【正文语种】中文【中图分类】Q331【相关文献】1.应用荧光原位杂交方法检测鼻咽癌细胞系中EGFR基因拷贝数变化 [J], 彭培建;廖海;李苏;张星;潘振奎;张力2.肽核酸荧光原位杂交技术检测生肉中沙门氏菌方法的建立及应用 [J], 杨彤;吴姗;李可;帅江冰;叶子弘;张晓峰3.应用实时荧光定量PCR方法检测水库水体中微囊藻毒素 [J], 陈蕾4.应用荧光原位杂交方法检测赤潮异弯藻 [J], 陈斌;梁斌;郭皓5.应用荧光原位杂交方法检测中国沿海塔玛/链状亚历山大藻复合种(亚洲温带基因型) [J], 于仁成;唐祥海;张清春;陈洋;王云峰;颜天;周名江因版权原因,仅展示原文概要,查看原文内容请购买。
实时荧光定量PCR方法检测南太湖入湖口产毒微囊藻

D0I 1 0 .1 8 3 07 /2 01 6 . 02 0 2
@2 0 1 6 b y J o u r n a l o fL ak e S c i e n c e s
最高 , 预测 产 毒 微 囊 藻 浓 度 分 别 为 ( 1 . 9 9  ̄ 0 . 3 5 ) ×1 0 和( 1 . 4 7  ̄ 0 . 2 3 ) × 1 0 c e l l s / m1 . 7个 监 测 点 的产 毒 微 囊 藻 的种 类 较 为
一
致, 均为铜绿微囊藻 . 该 方法可 以快速准确地检测水体中微囊藻毒素产毒藻种种类 和数量 , 为蓝藻水华监测 、 预警 提供
Ab s t r ac t :Th e a mo u n t o f Mi c r o s y s t i s c e l l s a t t h e e n t r a n c e o f iv r e r s t o s o u t h e m L a k e T a i h u h a s i mp o r t a n t i n lu f e n c e o n t h e d in r k i n g
实 时荧 光 定 量 P C R方法 检 测 南 太 湖 入 湖 口产 毒微 囊 藻
刘 洋 ' , 胡佩 茹 , 马思三 , 叶金 云 ,
( 1 : 湖州师范学 院生命科学院 ,湖州 3 1 3 0 0 0)
( 2 : 浙 江 省水 生 生 物 资 源 养 护 与 开 发 技术 研 究 重 点 实 验 室 , 湖州 3 1 3 0 0 0 )
( 3 : 中国水产科 学研究 院水生动物繁育与营养重点实验室 , 湖州 3 1 3 0 0 0 )
荧光标记原位分子杂交定位mrna

荧光标记原位分子杂交定位mrna荧光标记原位杂交(fluorescent in situ hybridization,FISH)是一种常用的分子生物学技术,用于在细胞或组织水平上检测和定位特定的RNA或DNA序列。
在这种技术中,通过标记RNA或DNA探针的荧光物质,可以在显微镜下直接观察到目标序列的位置,从而实现对RNA或DNA的定位和定量分析。
在细胞生物学和分子生物学研究中,经常需要对mRNA进行定位和定量分析,以了解基因的表达水平和分布情况。
荧光标记原位分子杂交定位mRNA技术可以用于检测和定位细胞内的特定mRNA序列,帮助研究人员深入了解基因的表达模式和功能。
在进行荧光标记原位分子杂交定位mRNA实验时,首先需要设计和合成适当的RNA探针。
RNA探针通常是由DNA序列合成而来,通过体外转录的方法获得RNA标记。
在合成RNA探针的过程中,可以选择荧光标记物(如荧光素、荧光蛋白等)进行标记,以便在显微镜下观察到。
接下来,将标记好的RNA探针与待检测的细胞或组织样品进行杂交反应。
在杂交过程中,RNA探针与目标mRNA序列发生互补配对,形成稳定的杂交复合物。
通过充分洗涤去除非特异性结合的RNA探针后,可以使用荧光显微镜观察样品,检测目标mRNA的位置和信号强度。
荧光标记原位分子杂交定位mRNA技术具有高灵敏度和高特异性的优势,可以在单个细胞水平上检测目标mRNA的存在和分布情况。
通过对不同细胞类型和组织样品进行mRNA定位分析,研究人员可以了解基因表达的时空动态变化,探究基因调控机制和信号传导途径。
总的来说,荧光标记原位分子杂交定位mRNA技术为细胞生物学和分子生物学研究提供了一种重要的工具,可用于检测和定位mRNA在细胞或组织中的分布和表达情况,为深入理解基因功能和调控机制提供了有力支持。
荧光原位杂交技术在产前诊断中应用的专家共识

荧光原位杂交技术(FISH)在产前诊断中的应用一直备受关注。
近年来,随着该技术在临床实践中的不断深入和发展,专家共识也逐渐形成。
在本文中,将从深度和广度上对荧光原位杂交技术在产前诊断中的专家共识进行全面评估,并撰写一篇有价值的文章,以便读者能更深入地理解这一领域的最新进展和专家观点。
1. 荧光原位杂交技术概述- 荧光原位杂交技术是一种基于DNA的细胞遗传学技术,能够定位和检测细胞中特定DNA序列的存在和定位。
该技术通过使用标记了荧光物质的探针,使得特定的DNA序列在细胞或组织的显微镜下呈现出荧光信号,从而实现对细胞遗传信息的定量和定位检测。
在产前诊断中,荧光原位杂交技术能够用于检测胎儿染色体异常、基因突变等遗传性疾病,具有高灵敏度和特异性的优势。
2. 专家共识的形成- 随着荧光原位杂交技术在产前诊断中的广泛应用,越来越多的专家学者参与其中,并在临床实践和科研工作中积累了大量的经验和数据。
通过学术会议、专家讨论会、文献研究等形式,专家们逐渐达成了对荧光原位杂交技术在产前诊断中应用的共识。
这些共识涵盖了该技术的临床适应症、操作规范、质控要求、结果解读等方面,为该领域的规范化和标准化提供了重要指导。
3. 专家共识的内容和意义- 在产前诊断中,荧光原位杂交技术的专家共识主要包括对该技术的临床应用范围和标准化操作流程的制定。
专家们一致认为,该技术在胎儿染色体异常、染色体结构异常、基因突变等方面具有重要应用意义,可以为胎儿遗传疾病的早期筛查和诊断提供可靠依据。
专家共识还对该技术的样本采集、实验操作、结果解读等方面提出了具体要求,以确保临床应用的准确性和可重复性。
4. 个人观点和理解- 就我个人来说,我认为荧光原位杂交技术在产前诊断中的应用具有重要的临床意义和发展前景。
通过该技术,我们可以更准确地了解胎儿遗传信息,及时发现和诊断潜在的遗传疾病,为家庭和社会减少遗传疾病的发病率和负担提供了可能。
专家共识的形成也为该技术在临床实践中的标准化和规范化提供了重要支持,有助于推动该领域的进一步发展和应用。
primeflow原理

PrimeFlow技术是一种基于荧光原位杂交(FISH)的细胞核酸检测技术,能够实现对细胞内RNA和DNA的高灵敏度、高特异性的检测。
PrimeFlow技术结合了FISH技术和流式细胞仪的优势,广泛应用于细胞生物学、免疫学、肿瘤学等领域,为研究人员提供了强大的工具来探索细胞内的基因表达和基因组变化。
本文将从PrimeFlow技术的原理、关键步骤和应用领域等方面进行详细介绍。
一、PrimeFlow技术原理1. 荧光原位杂交(FISH)原理荧光原位杂交是一种通过与靶标DNA或RNA互补的荧光标记探针结合来检测细胞内特定核酸序列的技术。
在FISH过程中,靶标核酸序列与荧光标记的探针发生互补杂交,形成稳定的双链结构,然后利用激光共聚焦显微镜等设备观察并分析。
2. PrimeFlow技术原理PrimeFlow技术在FISH技术的基础上引入了RNA或DNA的逆转录(RT)和PCR扩增步骤,以提高对低丰度核酸的检测灵敏度。
具体步骤如下:(1)细胞固定和透化:将待检测的细胞进行固定和透化处理,使得细胞内的RNA或DNA得以保持稳定并对探针具有较好的通透性。
(2)逆转录:在细胞内对RNA进行逆转录反应,将RNA转录成cDNA,同时引入RNA标记物,如荧光素或生物素。
(3)PCR扩增:对逆转录产物进行PCR扩增,增加目标核酸序列的拷贝数,提高检测灵敏度。
(4)FISH杂交:使用荧光标记的探针与PCR扩增产物进行FISH 杂交,形成稳定的双链结构。
(5)流式细胞仪检测:利用流式细胞仪对荧光信号进行检测和分析,实现对细胞内RNA或DNA的高灵敏度、高特异性检测。
二、PrimeFlow技术关键步骤1. 细胞样品准备:包括细胞培养、固定和透化处理等。
2. 逆转录和PCR扩增:通过逆转录和PCR扩增步骤提高对RNA 或DNA的检测灵敏度。
3. FISH杂交:利用荧光标记的探针与PCR扩增产物进行FISH 杂交,形成稳定的双链结构。
荧光原位杂交技术及其在微生物生态学中的应用_呼庆

第24卷第5期2004年5月生 态 学 报AC TA ECOLOGICA SIN ICAV o l.24,N o.5M ay ,2004荧光原位杂交技术及其在微生物生态学中的应用呼 庆,齐鸿雁*,张洪勋(中国科学院生态环境研究中心环境生物技术研究室,北京 100085)基金项目:国家“十五”科技攻关重点资助项目(2001BA903B)收稿日期:2003-10-21;修订日期:2004-01-10作者简介:呼庆(1977~),男,内蒙古呼和浩特市人,博士生,主要从事环境微生物分子生态学研究.*通讯作者Author fo r co rrespond ence 。
E-mail :qih y @Foundation item :th e National “Ten th F iv e-year Plan ”Key Techn ologies R &D Prog rame(No.2001BA903B)Received date :2003-10-21;Accepted date :2004-01-10Biography :HU Qing,Ph.D.candidate,mainly engaged in molecular ecology of environmental microorganism.摘要:综述了荧光原位杂交技术(fluor escence in situ h ybridization FIS H)在微生物生态学领域的各种应用,同时就其发展过程、原理及种类做了介绍。
关键词:荧光原位杂交;微生物生态;16Sr RN A;探针Fluorescence in situ hybridization (FISH )and its applications in microbial ecologyHU Qing ,QI Hong -Yan *,ZHAN G Hong -Xun (Environmental Biotechnolog y Lab .,Res earch Center for Ec o -envir onmentalSciences ,Ch ines e Acad emy of Sciences ,B eijin g 100085,Ch ina ).Acta Ecolog ica Sinica ,2004,24(5):1048~1054.Abstract :During r ecent y ears,molecular tech niques such as PC R and denaturing g radient g el electro pho r esis (D GG E)o r DN A sequencing hav e rev olutio nized all fields of micr obio lo gy ,a nd sensitiv e detectio n and ex act identifica tio n o f bacteria a re po ssible.Fluo rescence in situ hy bridization (F ISH)using 16Sr RN A pro bes do es no t rely o n PCR amplificatio n,a nd as such prov ides a useful complementa ry tech nique to DG GE for the ana ly sis of o rg anisms.Because of FI SH allo wing nucleic acid sequences to be exa mined inside cells witho ut a ltering the cell 's mo rpho lo g y o r the integ rity o f its v arious compar tments and pr oviding info r matio n abo ut number,spatial distributio n and cellular env ironment,it has beco me a po w erful too l fo r phylog enetic,eco lo gic,diag no stic and enviro nmenta l studies in micr obio lo gy.Fluo rescence in situ hy bridization ca n detect nucleic acid sequences by a fluor escently labeled pr obe that hybridizes specifically to its complementa ry ta rge t sequence within the intact cell .Its g ener al procedure in FISH a na ly sis ofmicr oo rg anisms is as follow s :(1)fix ation of the specimen ;(2)pr epa ration of th e sam ple ,possibly including specificpret reatment steps ;(3)hy bridization w ith the respectiv e pro bes fo r detecting the respectiv e tar ge t sequences ;(4)w ashing steps to r emov e unbound pr obes ;(5)mounting ,v isualization and documentatio n of r esults .Because F ISH g iv es a detailed picture of the micro envir oments without any selectiv e purificatio n o r amplificatio n steps ,ther e is a la rg e sco pe o f FISH applica tio ns .It ha s bee n ex tensiv ely used in the field of envir onmental micr oo rg anisms div e rsity such a s the inv estiga tio n o f micro bia l communities o f aqua tic ha bitats and soil ha bitats.M o st o bliga te mic ro bia l symbio nts a re as-y et uncultur ed.U sing the 16Sr RN A approa ch ,they can be identified and ph ylog ene tica lly ing different FI SH stra teg ies,bacte rial endo symbio nts w er e detected in many micro or ganisms.Po pulation analysis by F ISH has prov en par ticula rly useful fo r descriptio n o f the no rma l flo ra o r that o f mixed micro bia l infectio ns.This has been sho wn fo r medicine resea rch such as ora l cavity ,g astro -intestina l flo ra,respira tor y t ract infectio ns.It sh ould be pointed o ut that FISH is no t f ree o f bia ses a s with o ther mo lecula r methods.T he mo st st riking pr oblem is auto fluo rescence of micro org anisms themselv es.In additio n,accur acy and reliability of FIS H is highly dependent o n the specificity of the o lig o nucleo tide pro be.that FI SH as a pow er ful to ol will make mo r e co ntributionscommunities.Key words:fluo rescence in situ hy bridization;micro bial eco lo gy;16Sr RN A;pro be文章编号:1000-0933(2004)05-1048-07 中图分类号:Q93-3,Q938 文献标识码:A微生物是地球生物圈的重要组成部分。
流式-荧光原位杂交(Flow-FISH)
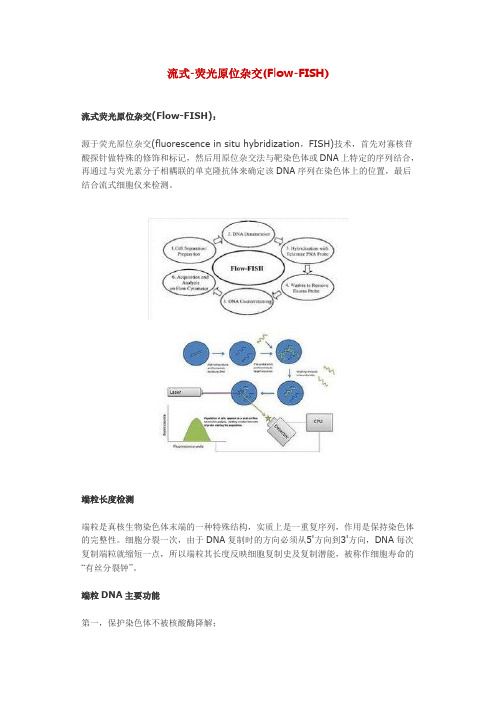
流式-荧光原位杂交(Flow-FISH)流式荧光原位杂交(Flow-FISH):源于荧光原位杂交(fluorescence in situ hybridization,FISH)技术,首先对寡核苷酸探针做特殊的修饰和标记,然后用原位杂交法与靶染色体或DNA上特定的序列结合,再通过与荧光素分子相耦联的单克隆抗体来确定该DNA序列在染色体上的位置,最后结合流式细胞仪来检测。
端粒长度检测端粒是真核生物染色体末端的一种特殊结构,实质上是一重复序列,作用是保持染色体的完整性。
细胞分裂一次,由于DNA复制时的方向必须从5'方向到3'方向,DNA每次复制端粒就缩短一点,所以端粒其长度反映细胞复制史及复制潜能,被称作细胞寿命的“有丝分裂钟”。
端粒DNA主要功能第一,保护染色体不被核酸酶降解;第二,防止染色体相互融合;第三,为端粒酶提供底物,解决DNA复制的末端隐缩,保证染色体的完全复制。
主要检测方法:DNA印迹法(Southern blot, SB):使用限制性核酸内切酶消化DNA, 然后琼脂糖电泳分离不同大小的片段, 转移到硝酸纤维或尼龙膜上。
用同位素或生物素、碱性磷酸酯酶标记的端粒特异探针与其杂交。
末端限制酶切片段(TRF)通过光密度计定量测量。
杂交保护分析法(hybridization protection assay, HPA) :需要制备基因组DNA、细胞或组织溶胞产物同吖啶酯(AE)标记端粒的探针进行杂交, 检测发光强度, 确定端粒在Alu序列中比例。
荧光原位杂交(FISH):FISH直接将寡核苷酸探针标记在端粒序列上. 标准的FISH包括制作分裂中期的染色体以及DNA变性, 与FITC或Cy3标记的寡核苷酸探针杂交, 用DAPI或PI复染, 最后用荧光显微镜检测信号。
流式-荧光原位杂交(Flow-FISH):包括6个基本步骤: 细胞分离, DNA变性并与PNA 探针杂交, 洗去多余探针, 复染后用流式细胞计量术采集和分析。
荧光原位杂交和流式细胞术结合(FISH-FCM)对肉食品中肠杆菌科细菌的快速定量检测

荧光原位杂交和流式细胞术结合(FISH-FCM)对肉食品中肠杆菌科细菌的快速定量检测汪沐;谢鹏;唐梦君;卜柱【期刊名称】《江苏农业科学》【年(卷),期】2016(044)007【摘要】为建立一种新的荧光原位杂交与流式细胞术相结合(FISH-FCM)的方法,以快速鉴定肉食品中的肠杆菌科细菌,采用与肠杆菌科细菌16S rRNA保守序列互补的探针Enter1432并验证其特异性。
结果表明:6种肠杆科细菌与探针Enter1432杂交率均在90%以上,而5种非肠杆菌科细菌与探针杂交率均低于1%;与SN/T 0738—1997《出口食品中肠杆菌科检验方法》传统培养法相比,FISH-FCM定量检测法全程约需4~5 h,检测8份猪肉样品中肠杆菌科细菌数为1.4万~640万个/g,4份鸡肉样品中肠杆菌科细菌数为5.0万~21万个/g,检出率为100%。
综上所述,研究建立的FISH-FCM法可快速检测肉品中的肠杆菌科细菌,适于食品卫生、临床实验室推广使用。
【总页数】3页(P336-338)【作者】汪沐;谢鹏;唐梦君;卜柱【作者单位】江苏省淮安市农业委员会,江苏淮安223001;中国农业科学院家禽研究所,江苏扬州225125;中国农业科学院家禽研究所,江苏扬州225125;中国农业科学院家禽研究所,江苏扬州225125【正文语种】中文【中图分类】TS207.4【相关文献】1.流式细胞术定量检测食管癌组织中P53、cyclin D1和 Rb蛋白表达的意义 [J], 丁卫军;刘志辉;常建华;黄朝晖;李莉华2.流式细胞术定量检测胃癌组织中Fas基因表达和DNA倍体及临床意义 [J], 庞伟;于明俊;马丽;丛春莉3.流式细胞术定量检测胃癌、癌旁组织中凋亡调控基因的表达 [J], 周永列;邱莲女4.核酸适配体功能化近红外量子点结合流式细胞术快速检测白血病细胞 [J], 汤进录;石慧;何晓晓;王柯敏;李朵;颜律安;雷艳丽;刘剑波5.荧光原位杂交技术和流式细胞仪在菌斑中致龋性变形链球菌定量检测中的应用[J], 姜云涛;梁景平;李超伦;叶冬霞因版权原因,仅展示原文概要,查看原文内容请购买。
流式细胞术对生乳中微生物检测的应用研究

流式细胞术对生乳中微生物检测的应用研究杨莉婷;何丽;何海宁;吴琼【摘要】Flow cytometry (FCM) is a rapid technique for cell-by-cell multiparameter analysis,which is often used in combination with fluorescent labelling for fast viability assessment.FCM analysss with fastand accurate protocol appeared to be workable for food bacteria detection.This article applied enzymatic treatment and membrane filtration method to digest proteins and treatment of milk by centrifugation to remove lipids.Treated milk samples were detected and enumerated by FCM.The bacteria were stained with 7-AAD and counted by fluorescent microspheres.The detection limit of 101CFU/mL was achieved.Raw milk data showed a significant correlation (P<0.01) and a goodagreement(r=0.980 2) between FCM and standard plate count methods.%流式细胞术(flow cytometry,FCM)是一种对流动的单细胞或小颗粒分子进行快速、多参数检测的新兴技术,其以快速、精确的特点,被应用于食品微生物的检测中.本研究以牛乳为材料,使用蛋白酶消化和尼龙膜过滤的方法来去除生乳中的大颗粒物质;利用7-AAD进行染色,结合荧光微球,对细菌总数进行计数;在FACS AriaⅡ flow cytometer (BD)流式细胞仪的操作系统上建立微生物的检测程序检测细菌总数;检测限达到101CFU/mL 数量级.检测结果与平皿菌落计数法进行比较分析,结果表明,流式细胞术与平皿菌落计数法的结果呈显著相关(P<0.01).【期刊名称】《广西师范大学学报(自然科学版)》【年(卷),期】2017(035)002【总页数】5页(P112-116)【关键词】流式细胞术;食品检测;微生物;牛乳【作者】杨莉婷;何丽;何海宁;吴琼【作者单位】甘肃省动物源性制品安全分析与检测技术重点实验室,甘肃兰州730000;广西师范大学生命科学学院,广西桂林541006;广西师范大学生命科学学院,广西桂林541006;甘肃省动物源性制品安全分析与检测技术重点实验室,甘肃兰州730000;甘肃省动物源性制品安全分析与检测技术重点实验室,甘肃兰州730000;广西师范大学生命科学学院,广西桂林541006【正文语种】中文【中图分类】Q932流式细胞术(flow cytometry, FCM)是一种对高速流动的单行细胞或颗粒进行快速、多参数、定量分析和分选的技术。
荧光原位杂交和流式细胞术结合FISH—FCM对肉食品中肠杆菌科细菌的

荧光原位杂交和流式细胞术结合FISH—FCM对肉食品中肠杆菌科细菌的快速定量检测作者:汪沐谢鹏唐梦君来源:《江苏农业科学》2016年第07期摘要:为建立一种新的荧光原位杂交与流式细胞术相结合(FISH-FCM)的方法,以快速鉴定肉食品中的肠杆菌科细菌,采用与肠杆菌科细菌16S rRNA保守序列互补的探针Enter1432并验证其特异性。
结果表明:6种肠杆科细菌与探针Enter1432杂交率均在90%以上,而5种非肠杆菌科细菌与探针杂交率均低于1%;与SN/T 0738—1997《出口食品中肠杆菌科检验方法》传统培养法相比,FISH-FCM定量检测法全程约需4~5 h,检测8份猪肉样品中肠杆菌科细菌数为1.4万~640万个/g,4份鸡肉样品中肠杆菌科细菌数为5.0万~21万个/g,检出率为100%。
综上所述,研究建立的FISH-FCM法可快速检测肉品中的肠杆菌科细菌,适于食品卫生、临床实验室推广使用。
关键词:荧光原位杂交;流式细胞术;肠杆菌科;快速定量检测中图分类号: TS207.4 文献标志码: A 文章编号:1002-1302(2016)07-0336-03肠杆菌科(Enterobacteriaceae)细菌是一类分布极为广泛的革兰氏阴性菌,包括对人致病性较强的埃希菌属、志贺菌属、沙门菌属、耶尔森菌属细菌[1]。
肠杆菌作为欧盟各国食品卫生检查中重要的指标细菌,是国际间食品微生物学实验室质量控制、水平测试的必测项目之一。
目前,我国仍以大肠菌群、粪大肠菌群作为食品卫生指标菌。
以肠杆科菌替代大肠菌群、粪大肠菌群作为检测指标可消除因后者产气特性随检验方法、试验条件不同而造成结果的不准确性[2]。
由于我国农产品生产管理不当,出口畜禽产品中肠杆菌科细菌检测超标现象时有发生,易被欧盟发达国家市场拒绝,进而形成所谓的贸易壁垒。
我国进出口商检局经过多次修订,制定了针对畜禽等农产品中肠杆菌检测的行业标准SN/T 0738—1997《出口食品中肠杆菌科检验方法》。
原位杂交和荧光定量

原位杂交和荧光定量
原位杂交是一种将特定标记的已知顺序核酸为探针与细胞或组织切片中核酸进行杂交,从而对特定核酸顺序进行精确定量定位的过程。
基本原理是两条核苷酸单链片段,在适宜的条件下,能过氢键结合,形成DNA-DNA、DNA-RNA或RNA-RNA双键分子的特点,应用带有标记的(放射性同位素、荧光素生物素、地高辛等非放射性物质)DNA或RNA片段作为核酸探针,与组织切片或细胞内待测核酸(RNA或DNA)片段进行杂交,然后可用放射自显影等方法予以显示,在光镜或电镜下观察目的mRNA或DNA 的存在并定位。
荧光定量原位杂交技术则是一种分子遗传学实验技术,将直接与荧光素结合的寡聚核苷酸探针或采用间接法标记的寡聚核苷酸探针与变性后的染色体、细胞或组织中的核酸按照碱基互补配对原则进行杂交,经变性-退火-复性-洗涤后即可形成靶DNA 与核酸探针的杂交体,直接检测或通过免疫荧光系统检测,最后在荧光显微镜下显影,即可对待测DNA进行定性、定量或相对定位分析。
总的来说,原位杂交和荧光定量原位杂交都是一种在细胞或组织切片中定位特定核酸序列的技术,其中荧光定量原位杂交技术可以提供更多的定量和定性信息。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
中国科学: 生命科学2010年 第40卷 第3期: 250 ~ 259 SCIENTIA SINICA Vitae 英文版见: Gan N Q, Huang Q, Zheng L L, et al. Quantitative assessment of toxic and nontoxic Microcystis colonies in natural environments using fluorescence in situhybridization and flow cytometry. Sci China Life Sci, 2010, 53: doi: 10.1007/s11427-010-0074-8《中国科学》杂志社SCIENCE CHINA PRESS论 文荧光原位杂交技术和流式细胞仪用于环境样品中产毒及非产毒微囊藻的定量监测甘南琴, 黄群, 郑凌凌, 宋立荣*中国科学院水生生物研究所, 淡水生态与生物技术国家重点实验室, 武汉 430072 * 联系人, E-mail: lrsong@收稿日期: 2009-10-10; 接受日期: 2009-11-17国家重点基础研究发展计划(批准号: 2008CB418000)、国家自然科学基金(批准号: 20407017)、国家自然科学基金-云南省联合基金(批准号: U0833604)和中国科学院重大交叉项目(批准号: KZCX1-YW-14-1)资助摘要 全球范围内, 高频次、大范围暴发的蓝藻水华对淡水水体环境造成严重影响. 微囊藻因其在生长特别是衰亡过程中向水体释放微囊藻毒素而威胁人类健康. 因此, 分析其产毒株及非产毒株在环境样品中的组成, 建立产毒蓝藻的预报及评价体系显得极为重要. 本文采用荧光原位杂交技术结合流式细胞技术实现对环境样品中产毒藻株的鉴别与定量. 针对目标基因mcyA 设计的、以地高辛标记的双链DNA 探针可有效应用于产毒微囊藻FACHB 905和PCC7806的鉴别. 分别对来自滇池、太湖和关桥的11个样品进行分析显示, 该方法与传统的形态学鉴定及PCR 方法有较好的匹配. 荧光原位杂交技术与流式细胞相结合可有效鉴别产毒与非产毒微囊藻, 尤其可以对野外样品中产毒与非产毒藻株进行简便、可视化地鉴别, 从而达到对产毒微囊藻水华早期预警的目的.关键词蓝藻水华 产毒微囊藻荧光原位杂交技术流式细胞术随着经济的快速发展等诸多因素, 藻类水华已成为全球淡水湖泊面临的严重问题. 有毒蓝藻水华对人类健康及地表水生态质量造成威胁. 各种蓝藻毒素中, 肝毒素较神经毒素等更为常见, 其在微囊藻(Microcystis )、鱼腥藻(Anabaena )和浮颤藻(Plankto- thrix )等蓝藻中均有发现. 在中国, 最常见的优势种是微囊藻, 其产生的微囊藻毒素为环肽肝毒素. 有毒蓝藻水华引起的中毒事件已有报道[1,2]. 微囊藻毒素在淡水湖泊中的广泛存在、急性毒性及促肿瘤等特性迫切要求建立快速预测有毒蓝藻水华的有效方法.目前测定微囊藻毒素的方法有多种[3], 但用于定量鉴别野外样品中产毒、非产毒微囊藻的方法却鲜有报道. 基质辅助激光解析电离飞行时间质谱(MALDI-TOF MS)可有效应用于单群体中微囊藻毒素的鉴别并能提供有毒蓝藻水华的早期预警, 但该仪器价格十分昂贵不能满足高频次的监测[4]. 聚合酶链式反应(PCR)已成功应用于微囊藻毒素合成基因丛mcy 的检测, 同时能够区分实验室培养和野外样品中有毒株及无毒株[5~7]. 此外, 定量PCR 技术(QRT-PCR)也已应用于微囊藻和颤藻的鉴别[8~10]. Ingmar 等人[11]成功地将rRNA ITS-DGGE(rRNA-internal transcribed spacer denaturing gradient gel electrophoresis)用于湖泊中产毒、非产毒微囊藻群体的鉴定.在众多的分析微生物的分子生态学方法中, 原中国科学: 生命科学 2010年 第40卷 第3期251位杂交(ISH)及荧光原位杂交(FISH)技术已广泛应用于复杂体系中微生物的监测[12,13]. 目前, ISH 和FISH 技术已成功用于蓝藻(Cyanobacteria)、原绿球藻(Pro- chlorococcus )和微微型真核藻(Picoeukaryotes)的研 究[14~16]. 而近年来, 流氏细胞术(FCM)因可以快速、准确地检测小颗粒物质, 从而存在潜在定性及定量检测环境中微生物种群的功能而倍受关注[17]. 尽管ISH 和FISH 技术在复杂的微生物群落中适合蓝藻的定量检测, 但仍需对一些新的技术进行尝试, 它们与FCM 相结合可用于高通量分析.本文采用荧光原位杂交技术结合流式细胞技术,实现对环境样品中产毒藻株的鉴别与定量. 首先, 以实验室培养的已知产毒株和非产毒株, 评估FISH 鉴别产毒株和非产毒株的能力; 其次, 测试实验室培养样品及野外样品以解决样品收集及杂交过程中细胞丢失的问题; 最后, FISH 和FCM 相结合用于评估湖泊中产毒微囊藻含量, 选取的湖泊有: 滇池(昆明), 太湖(无锡)和官桥池塘(武汉). 该方法可准确解决自然环境中哪些微囊藻产毒及产毒量的问题.1 材料与方法1.1 藻株与培养基藻种相关信息见表 1. 实验选用3株纯微囊藻 藻株来自中国科学院水生生物研究所淡水藻种库(FACHB-collection, 武汉, 中国), 一株来自法国巴士德藻种库(Pasteur Culture Collection, PCC, 法国). 各藻株在光强20 µE/m 2·s, 温度(25±1)℃, 光周期12 h ︰12 h 的BG11完全培养基中培养.1.2 仪器与试剂高效液相色谱仪(LC-10AS, 日本岛津), 96孔酶标板(Costar, USA), 酶标仪(Bio-Rad 550, USA), 超声波细胞粉碎机(JY92-2D, 宁波), 荧光显微镜(NIKON, Eclipse E600, Japan), 流式细胞仪(Epics Altra, Coulter, Beckman, USA).单克隆抗体于本实验室研制[18]. 高效DNA 地高辛标记和检测试剂盒Ⅰ(DIG high prime DNA Labeling and Detection Starter Kit Ⅰ)及FITC 标记抗地高辛Fab 片段(Anti-Digoxigenin-Fluorescein (Fab fragments))均购自Roche 公司(USA). 超纯水(18.2 M Ω·cm, Millipore, Molsheim, France). 其他试剂均为色谱纯及分析纯.1.3 样品采集及微囊藻形态特征分析以浮游植物网(20 µm 孔径)采集2007年夏季湖泊藻样, 进行群体分离及形态特征分析, 发现其优势种为微囊藻(表1). 部分样品以5%甲醛固定, 黑暗保存, 进一步检测其群体组成, 胶鞘特征, 其余样品用于原位杂交.1.4 微囊藻毒素检测高效液相色谱法: 流动相采用甲醇-磷酸盐缓冲液(0.05 mol/L KH 2PO 4用20%磷酸调节pH 至3.0)按表1 藻种相关信息编号来源采集时间 形态 Microcystis aeruginosa FACHB 905 滇池, 中国– 单细胞 Microcystis aeruginosa PCC7806 巴斯德研究所, 法国 – 单细胞 Microcystis aeruginosa FACHB469 – – 单细胞Microcystis wesenbergii FACHB 929 滇池, 中国 – 单细胞 Dianchi525 滇池, 昆明 2007-05-25 群体 Dianchi611 滇池, 昆明 2007-06-11 群体 Dianchi709 滇池, 昆明 2007-07-09 群体 Dianchi805 滇池, 昆明 2007-08-05 群体 Dianchi819 滇池, 昆明 2007-08-19 群体 Taihu708 太湖, 无锡 2007-07-08 群体 Taihu728 太湖, 无锡 2007-07-28 群体 Taihu806 太湖, 无锡 2007-08-06 群体 Taihu819 太湖, 无锡 2007-08-19 群体 Guanqiao713 官桥池塘, 武汉 2007-07-13 群体 Guanqiao822官桥池塘, 武汉2007-08-22群体甘南琴等: 荧光原位杂交技术和流式细胞仪用于环境样品中产毒及非产毒微囊藻的定量监测25260︰40(体积比)混合, 流速1 mL/min, 紫外检测波长238 nm.酶联免疫法(ELISA): 采用间接竞争酶联免疫吸附实验法[19].1.5 DNA 模板的制备用于PCR 扩增的特异引物由Rantala 等人[20]设计: mcyA-Cd1F(5′-AAAATTAAAAGCCGTATCAAA-3′), mcyA-Cd1R(5′-AAAAGTGTTTTATTAGCGGCTCAT-3′). 非特异探针引物设计如下: Forward: 5′-TCCAG CAACACCAATAGGA-3′; Reverse: 5′-AGGTCGTTG TGGTTATCCT-3′. PCR 反应条件为: 94℃反应30 s, 94℃变性10 s, 56℃退火30 s, 72℃延伸1 min, 35个循环, 72℃ 3 min, 4℃保存. PCR 产物用PCR clean-up kit 进行纯化以清除过量的引物及核苷, 并以琼脂糖凝胶电泳进行分析. 260 nm 波长光吸收值定量检测PCR 片段, 以这些片段作为地高辛标记的模板.1.6 DNA 模板的随机引物地高辛标记使用高效DNA 地高辛标记和检测试剂盒Ⅰ进行PCR 片段的地高辛标记, 具体步骤如下: 1 µg PCR 产物与无菌双蒸水加入反应管中使终体积为16 µL, DNA 在沸水中变性10 min 后立即置于冰水浴中. 4 µL 变性PCR 产物加入高效地高辛引物、混合并低速离心, 混合物在37℃下反应1 h, 65℃加热10 min 终止反应. 标记的DNA 片段−20℃保存.1.7 细胞预处理培养细胞: 将1 mL 培养藻细胞(约1×105 个细胞)转移至 1.5 mL 离心管中. 野外样品: 群体细胞以超声波方法解聚成单细胞. 藻细胞以5000×g 离心3 min, 吸掉上清, 用1 mL 缓冲液(pH 7.8)洗细胞3次, 然后用2%多聚甲醛和0.5%戊二醛(Sigma)固定细胞1 h. 将固定的细胞涂于以1%多聚赖氨酸(Sigma)处理过15 min 并晾干的盖玻片上, 细胞在45℃下干燥5 min 后以冰乙醇骤冷逐级脱水[50%, 60%和96%(体积比)], 每步3 min. 空气干燥再以30 µL 溶菌酶(终浓度为0.25 g/mL) 37℃消化细胞壁30 min. 蒸馏水淋洗后细胞再以乙醇逐级脱水.1.8 原位杂交地高辛标记探针用于全细胞杂交的方法基于Amann 等人[21]的相关描述. 经预处理干燥过的标本中加入20 µL 杂交缓冲液[0.9 mol/L NaCl, 20 mmol/L Tris-HCl, pH 7.5, 0.1% SDS(重量/体积)于37℃培养30 min. 然后加入2 µL/片变性地高辛标记的探针(终浓度为25 ng/mL). 杂交反应在37℃反应4 h 或过夜. 杂交反应结束后以洗液淋洗, 然后用100 mL 封闭液37℃封阻30 min; 加入20 µL 的抗体溶液37℃反应30 min, 用洗涤液在室温黑暗下洗涤2次, 每次15 min. 以20 mL 反应缓冲液平衡反应2~5 min, 再加入10 µL Anti-DIG-Fluorescein 黑暗中反应3 h. TE 缓冲液终止反应, 指甲油封片. 荧光显微镜观察并照相.1.9 流式细胞技术用流式细胞术检测荧光原位杂交标记好的样品, 激发波长为488 nm, 通过530/30 nm(绿色荧光, FITC 标记)和650 nm(红色荧光, 自发荧光)带通滤片检测荧光强度信号, 为使样品达到相近浓度, 样品以PBS 稀释. FCM 图谱以CYTOWIN 软件进行分析.2 结果2.1 探针设计及实验室培养纯藻株的应用 为建立原位鉴别有毒及无毒微囊藻藻株的方法, 设计合适的探针是必要的, 本实验设计的探针以微囊藻毒素合成基因mcy 为目标. 基于应用范围及标记策略, 目前已有多种探针可用于原位杂交, 随机引物合成及切口平移法是目前应用最多的双链DNA 片段标记的方法. 采用随机引物标记双链DNA 为探针, 模拟实验室培养系统中有毒及无毒微囊藻藻株. 特异性探针及非特异性探针分别用于杂交实验, 以非特异性探针标记的藻株将显示红色自发荧光, 而以特异性探针标记的产毒微囊藻株将显示明亮的绿色荧光. 铜绿微囊藻(Microcystis aeruginosa )FACHB905和PCC7806作为阳性对照, 惠氏微囊藻(Microcystis wesenbergii )FACHB929及铜绿微囊藻FACHB469作为阴性对照, 高效液相色谱(HPLC)及ELISA 结果显示了上述微囊藻的产毒及非产毒特性(表2).图1显示了以特异性探针标记的产毒微囊藻FACHB905和PCC7806的明亮绿色荧光(图1(A)和(B)), 及非产毒微囊藻FACHB929的自发红色荧光(图1(C)).中国科学: 生命科学 2010年 第40卷 第3期2532.2 FISH 用于野外样品中产毒及非产毒微囊藻藻株的鉴别群体微囊藻源于滇池、太湖和关桥池塘, 样品采集时间大多集中于微囊藻为优势种季节, 即2007年7~8月, 微囊藻的优势种主要包括惠氏微囊藻、绿色微囊藻(Microcystis viridis )和铜绿微囊藻, 不同湖泊、 不同阶段优势种的比例有所差别(表3). 其中除Dianchi525样品外, 滇池水华中藻类主要优势种为惠氏微囊藻和绿色微囊藻, 太湖水体中惠氏微囊藻为主要优势种, 其次为铜绿微囊藻. 图2显示了不同微囊藻的形态差异.由于微囊藻常以群体形式存在[22,23], 采用超声表2 毒素含量HPLC ELISA藻种(毒素类型) 单位体积(µg/mL)细胞干重(µg/mg)单位体积(µg/mL)细胞干重(µg/mg)M. aeruginosa FACHB 905MC-RR 4.73 2.45MC-LR 2.03 1.01总毒素 6.98 3.60 5.38 2.88M. aeruginosa PCC7806MC-RR 3.75 0.54 MC-YR0.24 0.03MC-LR 11.06 2.13总毒素14.92 2.67 18.96 3.40 M. wesenbergii FACHB 929 未检测到 未检测到 M. aeruginosa FACHB 469 未检测到未检测到图1 培养样品杂交后荧光图像(A) 铜绿微囊藻FACHB905; (B) 铜绿微囊藻PCC7806; (C) 惠氏微囊藻FACHB 929. 比例尺: 5 µm甘南琴等: 荧光原位杂交技术和流式细胞仪用于环境样品中产毒及非产毒微囊藻的定量监测254表3 滇池、太湖、官桥池塘样品中不同优势种群微囊藻比例样品惠氏微囊藻(%)绿色微囊藻(%)铜绿微囊藻(%)其他(%)Dianchi525 60 20 20 <1Dianchi611 50 50 <1 <1Dianchi709 50 50 <1 <1Dianchi806 45 50 5 <1Dianchi819 5050 <1 <1Taihu708 70 <1 30 <1Taihu728 70 <1 30 <1Taihu806 95 <1 5 <1 Taihu819 70 <1 20 10Guanqiao713 80 <1 20 <1 Guanqiao822 50<1 40 10图2 优势微囊藻形态特征(A) 鱼害微囊藻(Microcystis ichthyoblabe )来自太湖; (B) 惠氏微囊藻来自太湖; (C) 史密斯微囊藻(Microcystis smithii )来自官桥池塘; (D)绿色微囊藻来自官桥; (E) 绿色微囊藻来自滇池; (F) 水华微囊藻(Microcystis flos-aquae 来自太湖; (G) 铜绿微囊藻来自太湖; (H) 鱼害微囊藻来自官桥池塘. 比例尺: 10 µm中国科学: 生命科学 2010年 第40卷 第3期255方法将群体细胞解聚为单细胞. 图3显示了FISH 标记滇池、太湖及关桥样品的结果, 对照及非特异性探针结果显示无交叉反应, 而特异性探针标记观察到象征性信号.2.3 流式细胞检测首先对实验室培养的已知产毒特性的藻株进行研究, 测试FISH 与流式细胞术相结合检测有毒及无毒微囊藻的可行性. 选择两株产毒藻株(M. aeruginosa PCC7806, M. aeruginosa FACHB 905)和两株非产毒株(M. aeruginosa FACHB 469, M. wesenbergii FACHB 929), 将产毒株与非产毒株按1︰1混合(即M. aeru- ginosa PCC7806: M. aeruginosa FACHB 469=1︰1, M.aeruginosa FACHB 905︰M. wesenbergii FACHB 929=1︰1), 然后以FISH 与流式细胞术相结合检测产毒比例, 结果产毒比例分别为46.42%和45.69%, 接近50%.接着进行野外样品中产毒微囊藻的检测, 来自滇池、太湖、关桥池塘的样品以特异性探针进行杂交, 阳性信号(绿色荧光)显示了高于阴性对照6~56倍的信号强度(图4), 这种特异、非特异荧光比例达到6~56倍的差异足以区分阳性及阴性信号, 也就是说这种信号的差别足以区分有毒及无毒微囊藻藻株. 表4清楚地显示了不同样品来源的微囊藻产毒情况.上述结果表明, FISH 及流式细胞术相结合能有效地鉴别实验室培养及野外样品中产毒及非产毒微囊藻.图3 野外样品杂交后荧光图像(A) Dianchi819; (B) Taihu819; (C) Quanqiao822; (D) Dianchi806. 比例尺: 5 µm甘南琴等: 荧光原位杂交技术和流式细胞仪用于环境样品中产毒及非产毒微囊藻的定量监测2563 讨论迄今, 国内外检测MCYSTs 的方法已有多种, 包括小鼠生物体法、高效液相色谱法(HPLC)、液-质联用法(HPLC-MS)、薄层层析法(TLC)、毛细管电泳法(CE)、酶联免疫法(ELISA)、蛋白磷酸酶抑制-PP1/ PP2A 法, 以及以产毒基因为基础的产微囊藻毒素藻株的分子鉴别法等. 有关产毒蓝藻的鉴别, 常规形态学方法已基本失效. 1996年, Meissner 等人[5]首次利用聚合酶链式反应(PCR)方法鉴别产毒和非产毒微囊藻, 目前已成功应用于不同类型样品产毒微囊藻与非产毒微囊藻的鉴别, 其可简单、快速、准确地检测2000个细胞/mL 水样. 该方法根据毒素合成酶mcy基因丛设计引物, 特异性地扩增产毒微囊藻的基图4 野外样品杂交后经流式细胞检测效果(A) Dianchi709; (B) Dianchi806; (C) Taihu708中国科学: 生命科学 2010年 第40卷 第3期257因片段. 研究发现, 铜绿微囊藻和绿色微囊藻多表现为产毒特性, 而惠氏微囊藻、挪氏微囊藻(Microcystis novaceckii )及鱼害微囊藻多表现为非产毒特性[24~26].但也有研究发现, 在少数微囊藻株中虽检测到mcy 基因阳性, 但并未检测到微囊藻毒素[27,28]. 即使在相同的条件下, 不同藻株产毒量差别也较大, 另外环境因子如营养盐浓度、光照、温度等也可能影响细胞内微囊藻毒素的含量. 因此了解藻华暴发早期产毒微囊藻的组成及比例具有重要意义. 原位杂交及荧光原位杂交技术已被成功应用于蓝藻、原绿球藻及微微型真核藻等[14~16]. 针对有毒及无毒株的鉴别, 免疫荧光分析已成功应用于Pseudonitzschia pungens [29]和Prymnesium parvum [30]. 但尚未见有关产毒与非产毒微囊藻鉴别的相关报道. 本文采用FISH 标记方法鉴别有毒及无毒微囊藻株, 该方法具有以下优势: (1) 克服了交叉反应的问题; (2) 可快速、准确区分野外样品中产毒及非产毒微囊藻; (3) 可视化加以鉴别.流式细胞术是快速鉴定水环境中细菌数目的有效方法, 通过单细胞或微粒产生的散射光和发射荧光强度从而对细胞或亚细胞进行快速定量测定和分析[31]. 本文选取流式细胞术检测产毒微囊藻与非产毒微囊藻的数量及比例, 利用未标记上的微囊藻的自发红色荧光与标记上的微囊藻发出的绿色荧光可以明显地在流式细胞仪上加以鉴别, 方法简单、明了(图3). 如样品Dianchi819和Tianhu819在荧光显微镜下显示阳性结果分别为60%和33%(图3(A)和(B), 而流式细胞术显示阳性结果分别为41.28%和20.65% (表4), 可能原因是有些细胞聚集, 采用流式细胞检测时只能记录为一个细胞, 而用荧光显微镜计数时则记录的是两个细胞(图3(C)和(D)).比较表3和4, FISH 及流式细胞术相结合检测样品Dianchi819, Taihu728, Taihu819, 结果显示产毒微囊藻比例分别为41.28%, 30.85%, 20.65%, 而根据不同形态特性及PCR 结果显示, 样品Dianchi819, Taihu728, Taihu819产毒微囊藻比例分别为50%, 30%和20%, 两种方法结果吻合. 样品Dianchi525和Dianchi611则有差别, 可能存在两种原因: 一是这两个样品中的绿色微囊藻有部分不产毒, 二是在进行间接免疫荧光标记时藻类自发红色荧光背景较强掩盖了绿色荧光强度.表4 FISH 与FCM 相结合检测实验室培养及野外样品中产毒及非产毒微囊藻含量流式细胞技术数据(细胞/mL)样品发绿色荧光细胞数a)发红色荧光细胞数a)总细胞数 非产毒比例(%)产毒比例(%) M.a. PCC7806 41376 598 42074 1.42 98.34 M.a. FACHB 905 39633 479 40212 1.19 98.56 M.a. FACHB469 510 20775 24285 85.54 2.10M.w. FACHB 929736206342437084.663.02M.a.PCC7806 and M.a.FACHB 469b 9433 10087 20320 49.64 46.42 M.a FACHB905 and M.w. FACHB929c11271 12397 24668 50.25 45.69Dianchi525 11608 55594 68202 81.51 17.02Dianchi611 7911 13254 22165 59.79 35.69Dianchi709 3165 16521 20686 79.86 15.30Dianchi806 3078 16401 20479 80.08 15.03Dianchi819 9150 12015 22165 54.2 41.28Taihu708 12905 48437 62342 77.69 20.70Taihu728 20830 45690 67520 67.66 30.85Taihu806 2577 18114 21691 83.5 11.88Taihu819 13098 49330 63428 77.77 20.65Guanqiao713 11785 55415 68200 81.25 17.28 Guanqiao822 17729 42300 61029 69.31 29.05a) 鉴别后发荧光数量; b: 50% M.a. PCC7806和50% M.a. FACHB 469; c : 50% M. a. FACHB 905和50% M. w. FACHB 929甘南琴等: 荧光原位杂交技术和流式细胞仪用于环境样品中产毒及非产毒微囊藻的定量监测258 总之, 免疫原位杂交与流式细胞术相结合的方法是进行产毒微囊藻与非产毒微囊藻的定量检测的有效方法, 该方法对野外样品中产毒微囊藻百分率进行预计, 对产毒微囊藻水华的暴发也可起到早期预警的作用.参考文献1 Ueno Y, Nagata S, Tsutsumi T, et al. Detection of microcystins, a blue-green algal hepatotoxin, in drinking water sample in Haimen and Fusui,endemic areas of Primary Liver Cancer in China, by highly sensitive immunoassay. Carcinogenesis, 1996, 17: 1317—13212 Jochimsen E M, Carmichael W W, Jisi A. Liver failure and death after exposure to microcystins at a hemodialysis center in Brazil. New Engl JMed, 1998, 338: 873—8783 Rapala J, Erkomaa K, Kukkonen J, et al. Detection of microcystins with 2protein phosphatase inhibition assay, high-performance liquidchromatography-UV detection and enzyme-linked immunosorbent assay comparison of methods. Analytica Chimica Acta, 2002, 466: 213—231 4 Kurmayer R, Dittmann E, Fastner J, et al. Diversity of microcystin genes within a population of the toxic cyanobacterium Microcystis spp. in LakeWannsee(Berlin, Germany). Microb Ecol, 2002, 43: 107—1185 Meissner K, Dittmann E, Börner T.Toxic and non-toxic strains of the canobacterium Microcystis aeruginosa contain sequences homologous topeptide synthetase genes. FEMS Microbiol Lett, 1996, 135: 295—3036 Pan H, Song L, Liu Y, et al. Detection of hepatotoxic Microcystis strains by PCR with intact cells from both culture and environmental samples.Arch Microbiol, 2002, 178: 421—4277 Oberholster P J, Myburqh J G, Govender D, et al. Identification of toxigenic Microcystis strains after incidents of wild animal mortalities in KrugerNational Park, South Africa. Ecotoxicol Environ Saf, 2009, 72: 1177—11828 Vaitomaa J, Rantala A, Halinen K, et al. Quanitative real-time PCR for determination of microcystin synthetase e copy numbers for Microcystisand Anabaena in lakes. Appl Environ Microbiol, 2003, 69: 7289—72979 Kurmayer R, Kutzenberger T.Application of real-time PCR for quantification of microcystin genotypes in a population of the toxiccyanobacterium Microcystis sp. Appl Environ Microbiol, 2003, 69: 6723—673010 Ha J H, Hidaka T, Tsuno H. Quantification of toxic Microcystis and evaluation of its dominance ratio in blooms using real-time PCR. Environ SciTechnol, 2009, 43: 812—81811 Ingmar J, Kardinaal W, Marion M, et al. Toxic and nontoxic Microcystis colonies in natural populations can be differentiated on the basis of rRNAgene internal transcribed spacer diversity. Appl Environ Microbiol, 2004, 70: 3979—398712 Franks A H, Harmsen H, Raangs G C, et al. Variations of bacterial populations in human feces measured by fluorescent in situ hybridization withgroup-specific 16S rRNA-targeted oligomucleotide probes. Appl Environ Microbiol, 1998, 64: 3336—334513 Manz W, Amann R, Ludwig W, et al. Application of a suite of 16S rRNA-specific oligonucleotide probes designed to investigate bacteria of thephylum cytophaga-flavobacter-bacteroides in the natural environment. Microbiology, 1996, 142: 1097—110614 Wilhelm S, Boris Z, Stella E, et al. In situ identification of cyanobacteria with Horseradish Peroxidase-labeled, rRNA-Targeted Oligonucleotideprobes. Appl Environ Microbiol, 1999, 65: 1259—126715 Nyree J W, Wilhelm A S, Nicholas J F, et al. Closely related Prochlorococcus genotypes show remarkably different depth distributions in twooceanic regions as relealed by in situ hybridization using 16S rRNA-targeted oligonucleotides. Microbiology, 2001, 147: 1731—174416 Isabelle C B, Fabrice N, Daniel V, et al. Quantitative assessment of Picoeukaryotes in the natural environment by using taxon-specificoligonucleotide probes in association with tyramide signal amplification-fluorescence in situ hybridization and flow cytometry. Appl Environ Microbiol, 2003, 69: 5519—552917 Sckar R, Fuchs B M, Amann R, et al. Flow sorting of marine bacterioplankton after fluorescence in situ hybridization. Appl Environ Microbiol,2004, 70: 6210—621918 Lei L M, Wu Y S, Gan N Q, et al. An ELISA-like time-resolved fluorescence immunoassay for microcystin detection. Clinica Chimica Acta, 2004,348: 177—18019 Hu C L, Gan N Q, He Z K, et al. A novel chemiluminescent immunoassay for microcystin(MC) detection based on gold nanoparticles label and itsapplication to MC analysis in aquatic environmental samples. Int J Environ Anal Chem, 2008, 88: 267—27720 Rantala A, Fewer D P, Hisbergues M, et al. Phylogenetic evidence for the early evolution of microcystin synthesis. Proc Natl Acad Sci USA, 2004,101: 568—57321 Amann R I, Zarda B, David A S, et al. Identification of individual prokaryotic cells by using enzyme-labeled, rRNA-Targeted Oligonucleotideprobes. Appl Environ Microbiol, 1992, 58: 3007—301122 Asai R, McNiven S, Ikebukuro K, et al. Development of a fluorometric sensor for the measurement of phycobilin pigment and application tofreshwater phytoplankton. Field Anal Chem Technol, 2000, 4: 53—61中国科学: 生命科学 2010年第40卷第3期23 Lyck S. Simultaneous changes in cell quotas of microcystin, chlo rophyll a, protein and carbohydrate during different growth phases of a batchculture experiment with Microcystis aeruginosa. J Plankton Res, 2004, 26: 727—73624 Kaya K, Watanabe M M. Microcystin composition of an axenic clonal strain of Microcystis viridis and Microcystis viridis-containingwater-blooms in Japanese freshwaters. J Appl Phycol, 1990, 2: 173—17825 Vasconcelos V M, Sivonen K, Evans W R, et al. Isolation and characterization of microcystins(heptapeptide hepatotoxins) from Portuguese strainsof Microcystis aeruginosa Kutz. Emend Elekin Arch Hydrobiol, 1995, 134: 295—30526 Otsuka S, Suda S, Li R, et al. Phylogenetic relationships between toxic and nontoxic strains of the genus Microcystis based on 16S to 23S internaltranscribed spacer sequences. FEMS Microbiol Lett, 1999, 172: 15—2127 Neilan B A, Dittmann E, Rouhiainen L, et al. Nonribosomal peptide synthesis and toxigenicity of cyanobacteria. J Bacteriol, 1999, 181: 4089—409728 Mikalsen B, Boison G, Skulberg O M, et al. Natural variation in the microcystin synthetase operon mcyABC and impact on microcystin productionin Microcystis strains. J Bacteriol, 2003, 185: 2774—278529 Bates S S, Leger C, Keafer B A, et al. Discrimination between domoic-acid-producing and non-toxic forms of the diatom Pseudonitzschia pungensusing immunofluorrescence. Mar Ecol Prog Ser, 1993, 100: 185—19530 West N J, Bacchieri R, Hansen G, et al. Rapid quantification of the toxic alga Prymnesium parvum in natural samples by use of a specificmonoclonal antibody and solid-phase cytometry. Appl Environ Microb, 2006, 72: 860—86831 Monfort P, Baleux B. Comparison of flow cytometry and epifluorescence microscopy for counting bacteria in aquatic ecosystems. Cytometry,1992, 13: 188—192259。
