oligo7官方说明 中文版
Oligo_7_最新个人总结使用教程
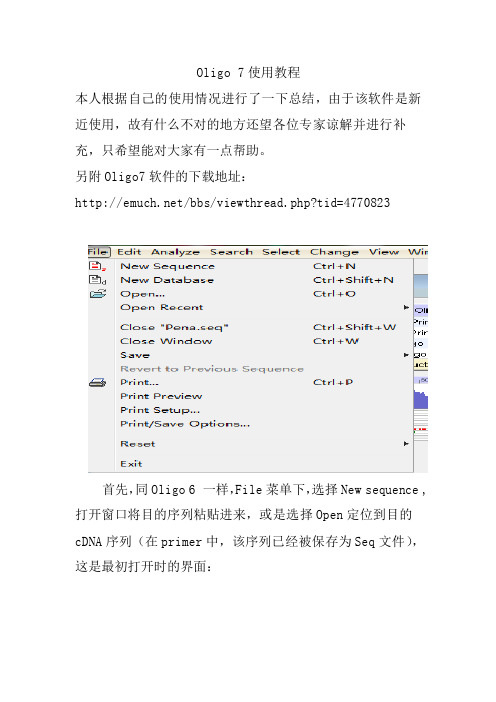
Oligo 7使用教程本人根据自己的使用情况进行了一下总结,由于该软件是新近使用,故有什么不对的地方还望各位专家谅解并进行补充,只希望能对大家有一点帮助。
另附Oligo7软件的下载地址:/bbs/viewthread.php?tid=4770823首先,同Oligo 6 一样,File菜单下,选择New sequence ,打开窗口将目的序列粘贴进来,或是选择Open定位到目的cDNA序列(在primer中,该序列已经被保存为Seq文件),这是最初打开时的界面:然后就是进行引物的设计了。
Search 菜单下,选择for primes &probes ,即出现引物搜寻窗口:根据自己的实际情况选择Parameters 或Ranges设置引物的相关参数和范围。
然后选择Search即开始进行引物的搜索,之后会出现软件所列出的依据得分(Score)高低排列设计的引物。
双击每一行所列出的引物会弹出该对引物的具体信息,以及软件对该对引物的相关评价。
双击之后在最初的Sequence 窗口中就会出现下面的窗口:点击绿色方形图标前面的i标志可了解对应的具体信息。
之后便是对该引物的具体分析了,这部分的分析同Oligo 6基本上是一样的。
选择Analyze菜单,如下图:(1)Analyze中,第一项为Key info,点击Selected primers,会给出两条引物的概括性信息,其中包括引物的Tm值,此值Oligo是采用nearest neighbor method计算,会比Primer5中引物的Tm值略高,此窗口中还给出引物的Delta G和3’端的Delta G.3’端的Delta G过高,会在错配位点形成双链结构并引起DNA聚合反应,因此此项绝对值应该小一些,最好不要超过9。
(2)Analyze中第二项为Duplex Formation,即二聚体形成分析,可以选择上游引物或下游引物,分析上游引物间二聚体形成情况和下游引物间的二聚体情况,还可以选择Upper/Lower ,即上下游引物之间的二聚体形成情况。
oligo7评价引物标准

oligo7评价引物标准
Oligo 7是一款引物设计软件,可以对引物进行评价。
以下是Oligo 7评价引物的标准:
1. 引物的特异性:评价引物与非特异性序列的结合能力,以及在基因组中的潜在结合位点。
2. 引物的长度:引物的长度会影响其特异性,太长或太短的引物可能导致不准确的PCR产物。
3. 引物的GC含量:GC含量过高或过低可能导致PCR过程中退火温度的选择和产物稳定性出现问题。
4. 引物二聚体和发卡结构:这些结构可能导致引物自身结合,影响PCR扩增效率。
5. 引物间的互补性:评价引物之间是否存在互补区域,这可能导致引物二聚体的形成。
6. 引物与模板的互补性:评价引物与模板DNA的结合能力,以确保PCR 产物的一致性。
7. 引物的突变和多态性:评价引物对基因突变和多态性的敏感性,以确保引物适用于不同基因型。
8. 引物的热稳定性:评价引物的解离温度(Tm),以确保PCR过程中选择合适的退火温度。
9. 引物的安全性:评价引物是否可能产生任何安全性问题,如对实验者的毒性或对环境的污染。
这些标准可以帮助您评估Oligo 7设计的引物质量,以确保PCR实验的成功和准确性。
泓格以太网分布式IOtGW700用户使用手册

tGW-700系列中文使用手册(版本1.2, 2011年7月)目录检查配件 (1)更多信息 (1)1. 产品介绍 (2)1.1 何谓E THERNET 解决方案 (3)1.2 何谓W EB S ERVER技术 (4)2.硬件信息 (5)2.1 规格 (5)2.2 特色 (6)2.3 选型指南 (6)2.4 T GW-700配置图 (7)2.5 机构图 (9)2.6 脚位定义 (11)2.6.1 tGW-712 脚位定义 (11)2.6.2 tGW-722 脚位定义 (12)2.6.3 tGW-732 脚位定义 (13)2.6.4 tGW-715 脚位定义 (14)2.6.5 tGW-725 脚位定义 (15)2.6.6 tGW-735 脚位定义 (16)2.6.7 tGW-718 脚位定义 (17)2.6.8 tGW-724 脚位定义 (18)2.6.9 tGW-734 脚位定义 (19)2.7 RS-232/422/485接线 (20)2.7.1 RS-232 接线 (20)2.7.2 RS-422 接线 (21)2.7.3 RS-485 接线 (21)3. 启动TGW-700模块 (22)步骤1:连接电源和计算机主机 (22)步骤2:安装M ODBUS U TILITY及E S EARCH U TILITY 到您的计算机 (23)步骤3:以太网络配置设定 (24)步骤4:测试 T GW-700 (25)4. 配置网页 (29)4.1 登入T GW-700网页服务器 (29)4.2 H OME 首页 (31)4.3 N ETWORK S ETTING (32)4.3.1 Network and Miscellaneous Settings (32)4.3.2 IP Address Selection (32)4.3.3 General Configuration Settings (35)4.3.4 Restore Factory Defaults (36)4.4 P ORT1设定 (37)4.4.1 Port1 Settings (37)4.4.2 Port Settings (37)4.4.3 Pair-Connection Settings (38)4.5C HANGE P ASSWORD (39)4.6L OGOUT (39)5. TGW-700 应用 (40)5.1 M ODBUS G ATEWAY (40)5.2 P AIR-C ONNECTION应用 (41)步骤1:连接至网络、电源和计算机主机 (42)步骤2:以太网络配置设定 (43)步骤3:在 T GW-700#1网页服务器配置P AIR-C ONNECTION (43)步骤4:在 T GW-700#2网页服务器配置P AIR-C ONNECTION (45)步骤5:测试P AIR-C ONNECTION 功能 (45)附录: 相关名词 (48)1. ARP(A DDRESS R ESOLUTION P ROTOCOL) (48)2. C LIENTS/S ERVERS (48)3. E THERNET (48)4. F IRMWARE (48)5. G ATEWAY (49)6. ICMP(I NTERNET C ONTROL M ESSAGES P ROTOCOL) (49)7. I NTERNET (49)8. IP(I NTERNET P ROTOCOL) ADDRESS (49)9. MAC(M EDIA A CCESS C ONTROL) ADDRESS (49)10. P ACKET (50)11. P ING (50)12. RARP(R EVERSE A DDRESS R ESOLUTION P ROTOCOL) (50)13. S OCKET (50)14. S UBNET M ASK (50)15. TCP(T RANSMISSION C ONTROL P ROTOCOL) (51)16. TCP/IP (51)17. UDP(U SER D ATAGRAM P ROTOCOL) (51)附录: FAQ (52)1. 使用IE浏览器进入T GW-700网页服务器时,如IE浏览器画面显示为空白,请执行下列步骤。
oligo7中文说明

Oligo使用方法介绍作为目前最好、最专业的引物设计软件,Oligo的功能很强,在这里我们介绍它的一些主要功能:如:普通引物对的搜索、测序引物的设计、杂交探针的设计以及评估引物对质量等等。
在正式进行引物设计前,我们首先面临的一个任务就是向Oligo程序导入模板序列,根据不同的实验情况,导入模板有三种方法:1,直接用键盘输入:a,点击file菜单中的New Sequence 浮动命令,或直接点击工具栏中的New Sequence命令,进入序列展示窗口;b,此时即可键入DNA序列;c,如果需要的话,Oligo提供碱基回放功能,在边键入时边读出碱基,防止输入错误。
点击Edit菜单中的“Readback on”即可。
2,利用复制和粘贴:当我们序列已经作为TXT文件存在或其它oligo不能直接open的文件格式,如word文件.html格式,这个功能就显得很有用了。
在相应文件中复制序列后在序列展示窗口粘贴,oligo会自动去除非碱基字符。
当序列输入或粘贴完成后,点击Accept/Discard菜单中的Accept浮动命令,即可进入引物设计模式。
3,如果序列已经保存为Seq格式或者FASTA,GenBank格式时,oligo就可以直接打开序列文件。
点击File菜单中的“Open”浮动命令,找到所需文件,打开即可。
进入引物设计模式后,oligo一般会弹出三个窗口,分别是6-碱基频率窗口,碱基退火温度窗口以及序列内部碱基稳定性窗口,其中的退火温度窗口是我们引物设计的主窗口,其它的两个窗口则在设计过程中起辅助作用,比如6-碱基频率窗口可以使我们很直观地看到所设计引物在相应物种基因组中的出现频率,如果我们的模板是基因组DNA或混合DNA时,该信息就显得有用了,而内部稳定性窗口则可以显示引物的5’端稳定性是否稍高于3’端等。
一,普通引物对的搜索:以Mouse 4E(cDNA序列)为例。
我们的目的是以Mouse 4E(2361 bp)为模板,设计一对引物来扩增出600-800bp长的PCR产物。
Z00MG7说明书

ZOOMG7中文说名书G7说明书G7说明书Selecting Patches for Playing(Play mode) P14~15一.选择音色(Selcting patch)下面介绍一下在演奏模式下的音色选择。
1.从1-4踏板中将LED等不亮的小踏板踩下,踩下的小踏板灯会亮,同时会叫出一个新的音色。
提示(HINT):●在演奏模式下,转动参数旋钮1可选择音色2.要选择别的音色时,踩下BANK[▲/▼]小踏板,再踩下1-4小踏板提示(HINT):在演奏模式下,可以通过旋钮[TYPE],选择音色库。
注意(NOTE):●在用BANK[▲/▼]踏板选择音色时,踩下后请立刻释放。
●持续踩住小踏板BANK[▼]一秒以上,G7.1UT就会被切换为菜单模式。
P18●持续踩住小踏板BANK[▲]一秒以上,G7.1UT就会被切换为[BYPASS]状态持续踩住不放,就会进入[MUTE]状态。
P20二:调节音色(Adjusting the sound)在演奏模式下,可以通过面板上的快捷键对音箱模拟的主要参数和输出水平进行调节。
1.在演奏模式下选择音色。
2.按下[CHANNEL A/B]中的一个键,来选择音箱模拟的输出通道。
G7.1UT有两条分别设定的A,B输出通道,通过[CHANNEL A/B] 键,可实现通道的瞬间转换。
提示(HINT):通过[FUNCTON]的1和2两个功能小踏板,也可进行A,B输出通道的切换。
P343.旋转[AMP TYPE] 旋钮可调节失真类型。
[AMP TYPE] 旋钮是调节失真类型(音箱模拟和压缩效果)的旋钮。
旋转该钮,液晶屏会显示新效果的类型名称。
提示(HINT):●调节失真旋钮时,液晶屏会显示E符号,[STORE/SW AP]键指示灯会亮。
●E符号表示设定值和原值不同,设定值返回原值后指示灯就会不亮。
●[STORE/SWAP]键指示灯亮表示,现在显示和不显示的参数同原值不同,返回原值灯就亮。
关于LEICA LGO7使用注意事项

关于LEICA LGO7.0的使用注意事项
1、天线不匹配
由于新的LGO7.0软件的问题,在新版本出现前,存在新天线无法有效识别的问题,所以在输出RINEX文件前时,请注意以下操作事项。
1、在天线中选择当前的天线,按“右键”,选择“属性”。
2、在天线属性中,将名称复制
3、将复制的天线类型粘贴到IGS名中,注意粘贴的天线名称时,不需要粘贴GNSS后的部分。
4、点击“确定”。
重复以上操作将天线中有三角感叹号图标的天线修改完毕,后输出为RINEX文件。
5、RINEX输出时,仅仅输出GPS的设置。
在输出RINEX时,在GNSS类型中选择“仅GPS”,即可。
2、量高的问题
由于新的LGO7.0软件的问题,在新版本出现前,存在新天线无法有效识别的问题,同时由于标准的RINEX文件中的天线高度是,量高尺高度+改正数的后高度(观测墩的改正数是0,三脚架的改正数是0.36),所以在配置集天线(GPS1200+系列)选择使用三脚架模式是会出现RINEX文件中的高度和量高尺高度差0.36的现象,所以建议在外业手工手簿记录时注明量高的方式,记录的天线高=量高尺读数+0.36,在LGO软件中输入是请输入量高尺的读数值。
同时在当前项目的天线中的A TX1230+ GNSS Tripod的垂直偏差从0.00改为0.36。
也可以将天线设置为观测墩的模式,在手工记录手簿中天线高记录为量高尺+0.36米后的高度,在这种模式下,在LGO软件中输入的天线高为手工记录手簿的高。
同时不需要改正当前项目的天线的垂直偏差。
L7N使用手册中文

注意
▪ 接通电源前,请再次确认输入电压(AC200 ~ 230[V])及电源接线。 ▪ 初期接通电源时,请务必在使能关闭的状态下接通。
操作及运行注意事项
▪ 运行前,请确认各参数并进行设置。 ▪ 运行中,请勿接触电机旋转部分。 ▪ 运行中,请勿接触散热片部位。
注意
使用注意事项
注意
▪ 请在外部安装急停回路,以便发生异常状况时能够及时停止运行。 ▪ 请在使能关闭的状态下进行报警复位,如在使能状态下报警复位时,会导致伺服马上重新启动。 ▪ 请使用干扰滤波器及直流电抗,尽可能屏蔽电子干扰,以免对周围的电子设备造成电子干扰。 ▪ 请正确选用指定配套的伺服驱动器和伺服电机。 ▪ 伺服电机的刹车只能用于停机或停电保护,请勿适用于一般的运行制动。 ▪ 刹车因其寿命或机械结构不同(用同步带连接电机和滚珠丝杆时)有可能无法停止设备,为了确
iii
安全使用注意事项
安全使用注意事项
.本使用手册根据安全注意事项,分为“危险”和“注意”等级。
注意事项
意义
危险
不当操作时可能会导致死亡或重伤等危险状况发生
注意
不当操作时可能会导致轻伤或机器损坏等危险状况发生
▪ 标记为“注意”的事项,视情况也可能会导致严重的后果,请留意。
防止触电注意事项
危险 ▪ 接线作业和检查工作,请在关闭电源 15 分钟后,在充电(CHARGE)指示灯灭灯的状态下,确认
▪ 没有灰尘、铁粉、腐蚀性气体、爆发性气体等物质的场所 务必遵守正确的安装方向。 ▪ 避免跌落或碰撞。 ▪ 请勿将本产品安装在潮湿、有腐蚀性气体、易燃性气体和可燃性物质附近。 ▪ 请在可以承重的地方安装本产品。 ▪ 请勿攀爬本产品,也不可在本产品上放置重物。 ▪ 伺服驱动器的安装间距要确保规定距离。 ▪ 伺服驱动器与伺服电机内部请勿混入导电性物质或可燃性物质。 ▪ 伺服电机必须牢固安装在机器上。 ▪ 装有减速机的伺服电机必须安装在指定的方向。 ▪ 避免运行中触摸伺服电机的转动部分。 ▪ 伺服电机的轴上安装联轴器时,请避免碰撞。 ▪ 请勿施加超过伺服电机轴的额定负载。
OLIGO软件应用简介

oligo软件应用简介在专门的引物设计软件中,“Oligo”是最著名的。
它的使用并不十分复杂,但初学者容易被其复杂的图表吓倒。
Oligo 5.0的初始界面是两个图:Tm图和ΔG图;Oligo 6.0的界面更复杂,出现三个图,加了个Frq图。
“Oligo”的功能比“Premier”还要单一,就是引物设计。
但它的引物分析功能如此强大以至于能风靡全世界。
oligo的下载和安装我就不多说了,打开oligo相信也无需多讲。
打开oligo的页面如下:单击file菜单再点open或点击“打开”快捷图标或者用快捷键“CTrl+O”可打开下面的窗口:在打开的OPEN窗口内选择FreqSeq再点“打开”出现以下窗口:选择drosfr或者其它一个文件点击“打开”出现以下窗口,点击“window”再点击“Tile”.出现以下窗口,图中显示的三个指标分别为Tm、ΔG和Frq,其中Frq是6.0版本的新功能,为邻近6至7个碱基组成的亚单位在一个指定数据库文件中的出现频率。
该频率高则可增加错误引发的可能性。
因为分析要涉及多个指标,起动窗口的cascade排列方式不太方便,可从windows菜单改为tile方式。
如果觉得太拥挤,可去掉一个指标,如Frq,这样界面的结构同于Oligo 5.0,只是显示更清楚了。
∆G值反映了序列与模板的结合强度,最好引物的∆G值在5’端和中间值比较高,而在3’端相对低(如图:)Tm值曲线以选取72℃附近为佳,5’到3’的下降形状也有利于引物引发聚合反应。
Frq 曲线为“Oligo 6”新引进的一个指标,揭示了序列片段存在的重复机率大小。
选取引物时,宜选用3’端Frq值相对较低的片段。
再点击Search再点“Fo'r Primers and probes”或使用快捷键F3.出现以下窗口,点OK就OK了出现Search Status窗口,点“OK”。
GE_LOGIQ7操作手册

用户自定义条件
未激活应用条件 显示激活的应用条件 此符号表示 通过旋钮调节
左右三角 可调整参数 显示多列图谱 可任意选择 功能开关键
应用条件
腹部 保存 腹部 重设置 肾脏 产科 保存 中.晚孕 重设置 早孕 胎儿心脏
应用条件
妇科 保存 妇科 重设置 卵泡 保存 成人 心脏 重设置 儿科 冠脉
应用条件
右键
确认
L 左键
R 右键
常用键使用说明
Patient: 输入或显示患者资料信息 Freeze: 冻结图像 L和R键: 双幅图像显示及左右图像选择,彩色模式时两键同时按即一幅二维及一幅彩色同步实时显示 3D功能,LOGIQ View 功能启动时: L 键开始 ,R 键结束。 B Pause: 多普勒模式下B-模式暂停键 Auto: ATO 自动优化图像功能,右键开启功能,左键关闭 B-Flow: 启动 B-Flow 功能 PDI: 彩色多普勒能量图 P 1: 存储动态或静态图像进入剪贴板 Function key : Logiq View 3D Contrast Harmonic 特殊功能键: 宽景成像 三维 造影剂成像 组织谐波 Depth: 调整深度 Gain增益: B ; M ; CFM ; PW 的增益 均在各自模式键,旋转即可调整增益大小 Set: 确认键,常用于测量后确认的应用 Scan Area: 在B-模式状态下改变扇扫角度;在CFM模式下改变彩色取样框大小 M/D Cursor: 调出M -mode 和 PWD- mode 取样线 Zoon: 局部放大(在冻结,实时状态下均可),使细小结构显示清晰 Measure: 测量键 Body Pattern: 体表标记可选择不同的体位标识及探头标记 Comment: 注释 F 2: 键头 Clear: 测量清除键 TGC: 时间增益补偿调节不同深度的增益 Audio Volume: 扬声器控制多普勒声音大小
GELOGIQ7LOGIQ7Pro基本用户手册-4

B Flow Color (BFC)描述 B Flow Color 目的在于通过伪彩血流和作为背景的 B 模式图像再现二维灰阶血流。
尽管 BFC 基于二维灰阶血流技术,但 BFC 图像由彩色血流处理器处理和制作,因此同时具有二维灰阶血流和彩色血流的优势。
相应地,成像参数和预设在功能上也与彩色血流 PDI 相同。
有关每一参数的具体信息,请参阅“优化彩色血流”。
注意: 您可以通过 Utility(实用程序)--> Imaging(成像)--> BFC 预设置BFC 参数。
激活激活 B Flow Color:1. 激活探头。
2.在 B 模式中,按下 BF 激活二维灰阶血流。
然后按下触摸面板上的 Color(彩色)。
如果您为特定应用和探头将BF控件赋值为 BFC,则您可以通过按下BF控件激活 BFC。
3. 调整参数以获得最佳图像。
将 BF 控件赋值为 BFC:1. 按下 Utility(实用程序)--> Imaging(成像)--> General(常规)。
2.从 BF/BFC 按钮下的应用特定下拉菜单中,选择 BF 或 BFC。
3. 按下Save(保存),然后Exit(退出)。
SRI(斑点噪声减少成像)(仅限 LOGIQ 7)描述 SRI(斑点噪声减少成像)是一种减少超声图像中不必要的斑点噪声影响的适应算法。
图像斑点噪声通常以粒状纹理出现在本应一致的组织区。
它的外观与图像系统特征而非组织特征关联,因此系统设置中的更改(如探头类型、频率、深度等)可能会改变斑点外观。
斑点噪声太多可能会降低图像质量,导致难以看清图像中所需的细节。
同样,斑点噪声过滤太多可能会掩盖所需要的图像细节或使其变暗。
必须倍加小心选择最佳的 SRI 级别。
SRI 在 B 模式成像中有效,如果图像斑点噪声似乎干扰所需的图像细节时,SRI 可与任何传感器或临床应用配合使用图 5-4. SRI 之前的 B 模式图像(左侧)和 SRI 之后的 B 模式图像(右侧)SRI(斑点噪声减少成像)(仅限 LOGIQ 7) (继续)调节要激活 SRI,请调节 SRI 触摸面板。
oligo使用说明
liz
发表于 06-12-30 10:00 只看该作者
Re: 【分享】PCR引物设计软件oligo应用简介
谢谢楼主分享
TOP 小 中 大 7#
热心酷友
个人空间 加为好友
发短消息 当前离线
‹‹ 上一主题 | 下一主题 ››
相关主题 标题
TOP
作者
本功能由奇虎搜索实现 最后发表
引物设计 PCR 引物设计及软件使用技巧 分子生物学几个相关软件简介 生物信息学简介及分子生物学相关重要数据库 点击阅读更多关于PCR oligo 引物 软件 简介 的相关帖子
oligo的下载和安装我就不多说了,打开oligo相信也无需多讲。打开 oligo的页面如下:
单击file菜单再点open或点击“打开”快捷图标或者用快捷键“CTrl +O”可打开下面的窗口
在打开的OPEN窗口内选择FreqSeq再点“打开” 选择drosfr或者其它一个文件点击“打开”
出现以下窗口,点击“window”再点击“Tile”
TOP 小 中 大 5#
热血酷友 个人空间 加为好友
kaige88
发短消息 当前离线
TOP
发表于 06-12-29 13:59 只看该作者
小 中 大 6#
Re: 【分享】PCR引物设计软件oligo应用简介 好象在另外网站看过且下载了,不错的分享,能标明去处更好!
版主
个人空间 加为好友
发短消息 当前离线
点击任一行出现“PCR”窗口,告知你扩增片断的位置,最合适的退 火温度等等信息。
关掉“PCR窗口”和“primer Pairs窗口”回到原来的窗口你就能看 到你引物的序列和位置,图中手型鼠标所指即为引物序列。
至此引物设计已经完成,你可以用“Analyse”菜单分析你的引物:有 无引物二聚体、发卡结构等等。
oligo7中文说明

Oligo使用方法介绍作为目前最好、最专业的引物设计软件,Oligo的功能很强,在这里我们介绍它的一些主要功能:如:普通引物对的搜索、测序引物的设计、杂交探针的设计以及评估引物对质量等等。
在正式进行引物设计前,我们首先面临的一个任务就是向Oligo程序导入模板序列,根据不同的实验情况,导入模板有三种方法:1,直接用键盘输入:a,点击file菜单中的New Sequence 浮动命令,或直接点击工具栏中的New Sequence命令,进入序列展示窗口;b,此时即可键入DNA序列;c,如果需要的话,Oligo提供碱基回放功能,在边键入时边读出碱基,防止输入错误。
点击Edit菜单中的“Readback on”即可。
2,利用复制和粘贴:当我们序列已经作为TXT文件存在或其它oligo不能直接open的文件格式,如word文件.html格式,这个功能就显得很有用了。
在相应文件中复制序列后在序列展示窗口粘贴,oligo会自动去除非碱基字符。
当序列输入或粘贴完成后,点击Accept/Discard菜单中的Accept浮动命令,即可进入引物设计模式。
3,如果序列已经保存为Seq格式或者FASTA,GenBank格式时,oligo就可以直接打开序列文件。
点击File菜单中的“Open”浮动命令,找到所需文件,打开即可。
进入引物设计模式后,oligo一般会弹出三个窗口,分别是6-碱基频率窗口,碱基退火温度窗口以及序列内部碱基稳定性窗口,其中的退火温度窗口是我们引物设计的主窗口,其它的两个窗口则在设计过程中起辅助作用,比如6-碱基频率窗口可以使我们很直观地看到所设计引物在相应物种基因组中的出现频率,如果我们的模板是基因组DNA或混合DNA时,该信息就显得有用了,而内部稳定性窗口则可以显示引物的5’端稳定性是否稍高于3’端等。
一,普通引物对的搜索:以Mouse 4E(cDNA序列)为例。
我们的目的是以Mouse 4E(2361 bp)为模板,设计一对引物来扩增出600-800bp长的PCR产物。
欧真喷水织机电控7寸触摸屏使用手册V2.2

欧真喷水织机电控7寸触摸屏使用手册V2.21.界面结构主界面如图(1.1)所示,上部为常用功能按钮区域,左侧为菜单栏,底部为状态信息栏,中间部分显示织机运行主要信息。
(图1.1)左侧菜单栏可进入下一级菜单或界面,菜单总体结构如(图1.2)、(图1.3)、(图1.4)所示。
左下角为系统状态栏,为保护参数误修改,影响运行,某些参数必须在显示为“停止状态”时才能修改,开机运行、点动或刹车解除时是不能修改的。
(图1.2)主编码器方向等主编码器方向、电机方向、卷取减速比等主编码器方向、电机方向、送经减速比等主编码器方向、电机方向、送经减速比等恢复织机电控数据为出厂默认值。
恢复储纬主控数据为出厂默认值。
恢复储纬头数据为出厂默认值。
恢复卷取和送经数据为出厂默认值。
主编码器方向、慢速反转定位、自动反向寻纬功能等(图1.3)(图1.4)2. 登录和HMI 配置(图2.1)主页点“登陆”按钮进入登陆界面。
此界面可以设置显示使用语言。
(图2.2)系统权限分三级:初级用户级、调机人员用户级、专业人员用户级。
初级用户级:不需要登陆,供挡车工使用,只可查看主界面数据、停机信息、生产管理数据、警告信息等基本信息。
当前用户为“无”时,即为初级用户级。
调机人员用户级:需要输入密码登陆。
调机人员用户级可以设置除系统配置菜单下的其他所有数据,供调试工、一般维修人员使用。
专业人员用户级:需要输入密码登陆。
专业人员用户级可设置修改所有数据,包括系统配置数据,供电控厂家、主机厂或终端用户专业人员使用。
专业人员用户级还可以设置默认用户级别,即重新上电或退出专业人员登陆后,系统可自动以设定的默认用户级别登陆,以供厂家管理人员设定开放权限。
默认用户级别选择“无”,则默认为初级用户级。
专业人员用户进入登录页面后选择“专业人员”用户,输入密码后点“登录”,注意当前用户显示为“专业人员”才表示登录成功;(图2.3)以专业人员用户登录后,进入“系统配置”项,选择“HMI配置”来设定系统实际配置,要根据实际配置来设定,否则可能会报通讯错误或者相关参数没有显示。
反转录试剂盒说明书

行业文档TaKaRa Code:DRR014APrimeScript™ RT-PCR Kit(50次量)目录内容页码●制品说明 1●制品内容 1●保 存 2●原 理 2●试剂盒特点 3●RNA样品制备 3●使用注意 4●反转录引物的选择 5●实验操作 5●实验例 7 ●Q&A8●制品说明PCR(Polymerase Chain Reaction;聚合酶链式反应)是一种体外扩增DNA的简单而有效的方法。
虽然原理上PCR法是扩增DNA,RNA不能直接被扩增,但是经过反转录酶的作用把RNA反转录成cDNA 后,PCR法便可应用于RNA的解析了。
迄今为止,此方法已广泛应用于RNA的构造解析、cDNA的克隆及RNA水平上的表达解析等多种领域。
PrimeScript™ RT-PCR Kit是具有良好的延伸性能与高扩增效率的2 Step RT-PCR试剂盒。
反转录反应使用了TaKaRa独自开发的新型反转录酶PrimeScript™ RTase;PCR反应使用了扩增性能良好的Hot Start 型DNA聚合酶TaKaRa Ex Taq TM HS。
本试剂盒具有以下优点:1.进行高效的RT-PCR扩增。
2.在标准的RNA反转录反应温度(42℃)下,便可使具有复杂结构的RNA进行良好的延伸,可以避免RNA在高温条件下的降解。
3.能有效抑制非特异性的PCR扩增。
4.使用本试剂盒扩增得到的目的产物3′端附有一个A碱基,可以直接克隆于T-Vector中。
本试剂盒含有反转录反应及PCR扩增反应所需的全部试剂。
●制品内容(50次量*1)1.PrimeScript™ RTase(for 2 Step)2.5×PrimeScript™ Buffer3.RNase Inhibitor(40 U/μl)4.dNTP Mixture(10 mM each)5.Oligo dT Primer(2.5 μM)6.Random 6 mers(20 μM)7.TaKaRa Ex Taq TM HS(5 U/μl)8.10×PCR BufferⅡ9.Control F-1 Primer*2(20 μM)10.Control R-1 Primer*3(20 μM)11.Positive Control RNA(2×105 copies/μl)12. RNase Free dH2O25 μl 200 μl 25 μl 150 μl 50 μl 50 μl 25 μl 250 μl 10 μl 10 μl 20 μl 1 ml*1 反转录反应20 μl 、PCR反应50 μl体系时可使用50次。
7500 1.4中文说明书 基因鉴定
Part Number 4347964 Rev. B 3/2004
ii
7300/7500 实时定ቤተ መጻሕፍቲ ባይዱ PCR 仪等位基因鉴别实验入门指南
3 30, 2004 8:25 œ¬ŒÁ, AD_GSG_FrontCover.fm
þæ¿″Œƒµµ
ķȉ
జ1
1 ˫ǎ 7300/7500 PCR ˫ǎķȉ ደ ˫ǎķȉ ደ ҔᏄ ˫ǎķȉ ደ 7ҔᏄ
ྜྷඡᒎฉ
等位基因鉴别实验
美国应用生物系统公司 7300/7500 实时定量 PCR 仪
简介和示例实验
设计等位基因 鉴别实验
设定反应板
读取等位基因鉴 别实验扩增前 信号
生成 扩增数据
读取等位基因鉴 别实验扩增后 信号
© Copyright 2004, Applied Biosystems. All rights reserved. For Internal Use Only. For Research Use Only. Not for use in diagnostic procedures.
第2章
设计等位基因鉴别实验
13
工作流程 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . .13 使用 TaqMan® 探针法的序列检测化学试剂 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . .14 选择探针和引物 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . .15
SP EVO7手动拉伸工具产品说明说明书

Manual tensioning tool plastic housing : SP EVO7 Blade Replacement blade for manual tensioning tool (spare part),Article number: 110-70021Features•Ergonomic, slip-proof handle for a comfortable and secure grip•Extremely low-maintenance•Fast and precise application with minimum effort (TLC mechanism)•Convenient and simple tension adjustment•Extended, slim nose for use in narrow spaces•Housing made of resilient and lightweight glass fibre-reinforced polyester•Standardgrip span (90 mm) and short grip span (80 mm) availableHellermannTyton's EVO7 mechanical hand tool was ergonomically designed to reduce the risks of repetitive stress injuries to operators while concurrently increasing productivity. The EVO7 Tension/Lock/Cut Technology delivers performance, safety and comfort for the operators. Available in a standard grip span (EVO7) or short grip span (EVO7SP), for smaller hands. Additional accessories are available.Color Various (MIX)Features and Benefits The new EVO family protects muscles and joints and increaseseffectiveness • Less recoil shock reduces impact to operator hand• Application of force is significantly reducedOperation manuallyPack Cont.1pc.PART DESCRIPTION SP EVO7 Blade-MIXProduct Family EVO7 up to 4.8 mm strap widthProduct Group Manual tensioning tool plastic housingShort Description Replacement blade for manual tensioning tool (spare part) Tension Setting Tools AdjustableTool property Manual clipping。
elgo说明书部分翻译资料

4. Controller in Operation4。
控制器的操作4.1 Single operation4.1单操作(A/I and Q windows without function)(没有A / I和Q 窗口功能)Select Single by pressing F sequentially选择单一顺序按下FPress: Keys 1-9 to enter the desired position into Demand Window按:1 - 9键需求所需的位置进入命令窗口Press:, for positioning to take place按: Start键,定位4.2 Manual inching4.2手动微调Select the manual mode by pressing F sequentially选择手动模式按顺序按下FUse Buttons > 7 8 9 for inching then使用按钮> 7 8 9进行缓慢Button > Slow backwards (forwards)按钮>缓慢向后(向前)Button 7 Fast backwards (forwards)按钮7快速向后(向前)Button 8 Fast forwards (backwards)按钮8快速向前(向后)Button 9 Slow forwards (backwards)按钮9缓慢向前(向后)4.3 Program line operation4.3程序行操作Select Program mode by pressing F.选择程序模式按下F。
Press Start to activate each move in a programmed sequence. Alternatively, pressing F again,按Start键激活程序序列中的每个步骤。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
稳定性较差的引物与产物相差30°Tm。一组警告并不会取消它的资格,而只是表明它可能不是最优的选择。 当警告是“前向起爆药的终端稳定性太高”时,并不意味着反应会受到任何损害。这意味着当使用复杂的 底物时,如基因组DNA或逆转录的总mRNA,你可能会有错误的产物形成问题。至少有必要仔细检查预测产品 的大小。
The next slide shows the calculation graphically (data for temperature of 37° ).
DG 计算示例
Total DG = 1+1+0.3+0.5-1.3-1.3-2.2 = -2.0 kcal/mol
这是一种罕见情况。通常情况下,悬挂的末端正在稳定下来。这是在提醒你,有时添加一个基 地可能会降低双相熔化温度,后面展示了一个即使添加两个碱基也不会影响TM的例子
在搜索引物和探针之后,您可 以打开选定的寡核苷酸窗口。 在此示例中,在所选寡核苷酸 窗口底部列出的文件中执行了 对一致性引物的排序搜索(要以 此方式搜索,您需要单击 Search for Primer&Probe(搜 索引物和探针)窗口的 “SubSearches”(子搜索)选项 卡,然后单击“Consensus Primers”(一致性引物)检查 框中,选择文件并开始搜索。)。选中“选择共识寡核 苷酸”框时,如右图所示,当您双击显示有关给定引 物信息的行时,Oligo 7会选择共识引物。
引物位置的列表可以通过点击(或按住Option键单击)窗口的标题来按分数、寡头位置或/和窗 口的任何其他列进行排序。左上角的下拉菜单允许您选择正向或反向引物显示。在搜索T aqMan集合或嵌套引物之后,您将有更多的选择。
寡核苷酸设置窗口
搜索PCR引物(上图)、TaqMan探针 (2ndimage)或嵌套引物(两幅下图)后,将 打开寡核苷酸集窗口。通过单击此窗口中 的任何行,您就选择了一个集合。双击会 导致打开PCR分析窗口。窗口显示引物和探 针的位数、收到的分数、PCR产物的长度和 最适退火温度以及GC含量。单击列标题时 ,可以对集合进行重新排序。 如果是嵌套引物结果,您可以通过单击 “U/L产品”按钮将外部引物PCR数据的显 示更改为内部显示,如下图所示。 下一张幻灯片上显示的PCR窗口中提供了有 关任何给定集合的更多信息。
PCR窗口
在手动选择引物或搜索PCR引物、TaqMan探 针或嵌套引物之后,您可以打开PCR窗口。 选择套式引物后,您可以检查外部引物PCR 产物(“F/R产品”)或套式引物产品数据 (“U/L产品”)。该窗口以图形方式显示 PCR产物和引物的整个序列和位置。此窗口 列出最佳和最高退火温度,以及产品和底 漆的Tmand GC%含量。还给出了分数(质量 度量)。在下表中,您可以看到可以随时编 辑的盐和底漆浓度。当底漆形成二聚体、 Tmor太低或与底漆相比产品TMIS过高时, 注释框会显示警告。在后一种情况下,预 计PCR产量会比正常情况低,但这本身通常 不会完全抑制反应;此警告的阈值是
• 3. 搜索选项 - 搜索引物和探针 - 其它搜索 - 批处理
• 4. 寡核苷酸数据库 - 复合
• 5. 结束语
引物稳定性计算
最邻近法介绍
引物最重要的特性之一是其熔点和各区域的稳定性.最邻近法提供最精确的 Tm 计算. 引物 Tm 值计算比较复杂:
Tm (°C) = DH/(DS + R * ln[C]) + 16.6 log [K+]/(1+0.7*[K+]) – 273.15 其中 DH and DS是螺旋形成的焓和熵。R是摩尔气体常数(1.987 cal/°C * mol),c是引物 探针的浓度(通常用于比目标DNA多出几倍的情况),可用电脑进行计算. 为了理解双相稳定 性计算的复杂性,通过展示自由能 (DG, 另一种衡量DNA稳定性的方法), 公式更简单:
熔化温度图形窗口,续
当点击“DG”行时,将显示没有悬垂末端的自由能图。如果您有退化序列文件,也可以选择GC%内容显示或退 化图。请注意,TM、DG GC%或简并度数据是针对当前寡核苷酸大小的DNA片段给出的(以红色字母显示,并可使 用更改当前寡核苷酸长度命令进行更改)。在窗口的顶部也有放大和缩小图标,类似于放大镜。您可以选择不 同的放大样式。例如,缩小操作将创建此类型的窗口:
窗口的底部显示了序列的放大部分,在Tm图形中以黄色高亮显示(此图的左上角)。光标位置和 将从光标位置开始的oligo的Tm显示在右侧,刚好位于位置数字刻度的上方。在标度以下你可 以看到DNA序列,其中正链是红色的。如果你选择引物,它们会出现在序列的上面和下面。翻 译后的蛋白序列以各种颜色显示。颜色象征密码子的频率(黑色是最频繁的,看到打开的阅读 框窗口)。下面的行和它们的颜色象征着相同的特征,但是放大了,显示在Tm graph下面。更 详细的解释在下一张幻灯片。
影响PCR反应的其它非引物因素
除了上一页列出的引物属性外,PCR效率还取决于模板。具有高二级结构 和GC岛的区域扩增较差。寡核苷酸7惩罚了那些会扩增强大发夹结构的引 物,从而降低了PCR引物集的总得分。
如果您必须使扩增产物变得困难,请选择得分较高的引物(通常Tm and GC含量高于标准),并通过添加溶剂、添加剂或减少/改变盐来调整PCR组 合的组成。由于非特异性引物/引物延伸,不建议将引物浓度提高到1 mmol以上(即使软件没有显示任何3‘-二聚体)。
二聚体信息窗口
双链形成窗口显示最 强的3‘-双链,不一 定在3’端的最稳定的 双链,以及最强的发 夹。DG数据是在考虑 悬挂端和起始DG值的 情况下给出。MIXED OLOOS DUPLEX窗口不 显示发夹
发卡结构窗口
发夹形成窗口显示给定寡 核苷酸中所有可能的发夹 ,从最强到最弱列出。当 给定的管段涉及许多不同 的结构时,由于结构之间 的竞争,特定发夹的实际 熔化温度比计算的要低
如何设计好引物
1. 一个好引物应具有以下几点
- 具有相当高的 Tm, - 没有二聚体,特别是在它们的3’端没有发卡结构(防治自引发) - 缺乏重复序列以确保快速和正确的退火。 -同一孵化混合物中的所有引物/探针之间不应形成显著的3‘二聚体。
2. 高特异性引物设计要点
- 足够长,以增加特异性 -唯一,特别是在其3‘-端,以避免假引发 -在其3’-端适度稳定(而不是高度富含GC),以确保非常短的片段不会初始化延 伸(太低的3‘-端稳定性会损害引发效率)。
同源性窗口
同源窗口显示探针的同源位点, 并从相似性最高到最低列出位点 。在这种情况下,3‘端是不相关 的,探针(或引物)的同源性与实 际的DNA链对齐,而不是与其补体 对齐,就像在假启动位点窗口中 一样。当在杂交实验中使用较短 的DNA片段时,同源性低于90%通 常不显著,也不会产生错误信号 。
选定的寡核苷酸窗口
这是一个屏幕截图显示选择的引物和探针
上游引物Tm 是56.7°, 下游引物 Tm 是 56.2° (差别不大). 两端各加一个碱基,选择片段为上下两个 核苷酸,上核苷酸Tm 为 56.7° (no change in Tm) 下核苷酸 Tm 上升至 61° (from 56.2°).在不考 虑垂悬末端的情况下,增加引物长度会增加 Tm.您将在关键信息窗口说明中看到这些特定寡头的更全面 的分析数据。
彩色矩形中的图形在缩放区域中展开
使用Oligo可以选择最多4个引物或探针
序列窗口中其他符号的说明
模板中更强的发卡结构
潜在引物中弱茎形成中涉及的核苷酸
回文
搜索字符串找到的结果
高亮功能
关键信息窗口
关于这些寡糖的更多统计数据提供了它们的组成和tm窗口,以及浓度窗 口。
关键信息窗口显示所选 引物或探针的基本信息 .这些是在前一节中讨 论的寡聚体,其中较长 的13mer寡聚体(左下) 具有与上面显示的较短 的11 mer寡聚体相同的 Tm。tm是在不考虑悬垂 端影响的情况下计算出 的熔点温度,该温度增 加了近7°。值得注意 的是,同长度的互补寡 核苷酸如右图所示,其 tm与预期相同,但tm不 同,尤其是13-mers。
DG = DH – TDS (是温度,单位是K). 虽然看起来简单,但还是比较复杂这是一个计算4-mer TCGA长链的例 子:
DG 可用如下公式计算:
DG (TC) + DG (CG) + DG (GA) + initiation DG for T + initiation DG for A + DG of 3’-dangling end AC + DG of 5’-dangling end GT
Oligo 7 Primer Analysis Software
PCR引物选择和软件演示规则
Ⓒ 2010 Molecular Biology Insights, Inc.
目录
• 1. PCR 引物选择标准 - 引物稳定性计算 - 如何设计好的引物 - 影响引物特性的其他因素
• 2. Oligo 7 分析特征 - 序列窗口 - 一般信息 (关键信息) - 二聚体和发卡 - 引物相关数据(组成和融化温度) - 假引物位点和同源性分析 -寡核苷酸稳定性图 (Tm, DG) - 内部稳定性及意义 - 序列频率 - 其它DNA分析选项
组成及Tm 窗口
成分和Tm窗口显示用不同的方法计算的熔化温度,即
最近邻法(TDI是归一化的Tmi1M盐条件,而TM是一个变
量值,取决于在搜索参数窗口中设置的盐和DNA浓度)
。
使用%GC方法计算的
TmCalculated不依赖
于搜索参数设置。文
中还给出了最简单的
Tm值计算方法(A或T为
2°,C或G为4°),然
The LCR Window
这个窗口帮助设计连接酶链式反应的探针。当您单击左上角的任何突变按钮时, 会设计一个特殊的寡核苷酸来通过LCR检测此突变。当这种酶用尾巴连接一个较长 的寡核苷酸时,你就可以通过凝胶电泳检测到它。
