Vector NTI 使用中文说明
Vector NTI的使用经验交流

Thank you!
4. 加入特征:点击工具栏中的add feature按钮,出现右图框体,可 以选择添加的类型,长度并对其 命名
序列的添加
选中需要添加序列的位置,点击 “ Edit-New-Insert sequence at…”
将需要插入的序列复制进去,点击OK便可以完成序列的插入,随 之再利用之前所说到的add feature功能对其添加特征
图谱显示的变更 右键pMV2614488,选择”Molecule Properties…”
勾选图谱的各项属性,由linear改为circular
数据的保存
存为文件
保存至本地数据库
图谱的导出
在图谱空白处点击右键--camera
可以选择复制到剪贴板(clipboard)然后粘贴到PS或画图进行进一步编辑 或者可以直接输出为图片文件(file),不过只能选择jpg格式
文字窗口
图谱窗口 序列窗口
载体序列的操作
点击File-Open打开如下界面选择从GenBank上下载的文件
该序列的常规信息
以图形+注释的形式显示序列的特征:可编辑
序列信息:默认以双链形式显示,复制时默认复制正链
限制性酶切位点分析
选择工具栏中的Restriction site
选择需要添加或移除的酶切位点
Web of Science
检索高被引文章 检索近期高被引文章
位于页面左侧的筛选方式相较于NCBI更加可视化
文献清晰图片的导出方法ቤተ መጻሕፍቲ ባይዱ
直接去下载文章的官方网站获取图片
找到图片的位置并点击弹出新窗口 点击放大以获取最大的图片
点击download slide会下载一张ppt,其中带有矢量图片
Vector_NTI_10_使用说明书(繁体中文)-2

第十一章 AlignX BlocksAlignX Blocks可以用在蛋白質序列比對,和AlignX不同的地方在於比對的序列會以圖形表示比對的結果。
啟動AlignX Blocks的方式和AlignX是一樣的,可直接開啟或是從主程式上方開啟(圖11.1-2):圖11.1 直接於程式集中開啟AlignX Blocks圖11.2 於Vector NTI程式內開啟AlignX Blocks開啟程式後會看見和AlignX很類似的操作界面(圖11.3):圖11.3 開啟AlignX程式後的介面首先使用者要把序列檔案載入程式裡面,可以點選或者從左上方的Project→Add Files把序列檔案載入,序列只能分析蛋白質序列(圖11.4):圖11.4 利用Project→Add Files將序列載入到左上視窗進行比對前先將要比對的序列用滑鼠反白(圖11.5),操作和AlignX相同:圖11.5 全選欲進行比對的序列比對前可以依各人需求調整序列比對的條件跟參數(圖11.6)。
可點選或是從Blocks→AlignX Blocks Setup進行調整:圖11.6 參數的設定會影響程式的運算,要適當的調整得到所要的結果參數的設定會影響到程式的運算,並造成不同的比對結果。
使用者可針對這三個參數進行調整,直到得到滿意的結果為止。
Blocks'colors項目(圖11.7)表示比對後各序列相似區域所顯示的顏色,預設值為紅色,可以依照喜好設定顏色。
圖11.7 Blocks' colors改變顯示的顏色和AlignX相同,使用者也可以設定Score matrix的參數(圖11.8),但是不建議一般使用者更改。
圖11.8 更改Score matrix的參數,設定好了以後按下或者是選取Blocks→Search for blocks程式進行運算,經運算完成之後會出現下面的畫面:圖11.9 運算的結果:左下為相似的區域,右上為序列的圖形顯示,右下為比對的畫面在左下的視窗(圖11.9)中所顯示的是程式依照蛋白質序列和設定的參數所比對出相似的區域,此區域稱之為block;右上方的視窗是把比對的序列以圖形顯示,可以判別block之間的位置關係;右下方則是序列的比對畫面。
vector nti 使用教程 多重序列比对
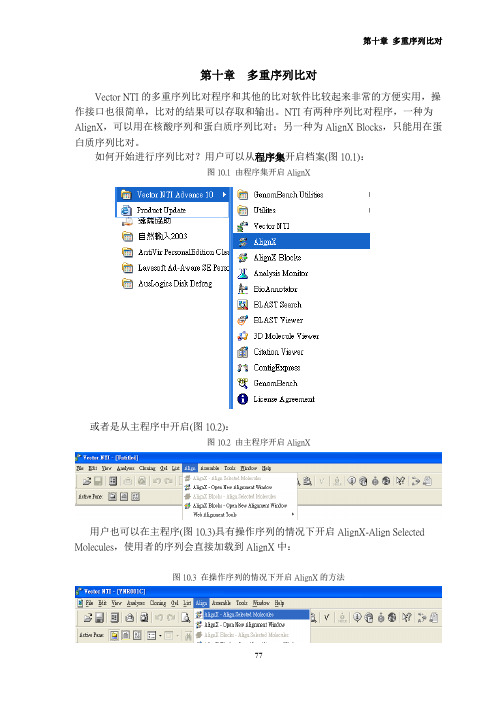
第十章多重序列比对 Vector NTI的多重序列比对程序和其他的比对软件比较起来非常的方便实用,操作接口也很简单,比对的结果可以存取和输出。
NTI有两种序列比对程序,一种为AlignX,可以用在核酸序列和蛋白质序列比对;另一种为AlignX Blocks,只能用在蛋白质序列比对。
如何开始进行序列比对?用户可以从程序集开启档案(图10.1):图10.1 由程序集开启AlignX 或者是从主程序中开启(图10.2):图10.2 由主程序开启AlignX 用户也可以在主程序(图10.3)具有操作序列的情况下开启AlignX-Align Selected Molecules,使用者的序列会直接加载到AlignX中:图10.3 在操作序列的情况下开启AlignX的方法 开启AlignX之后,使用者会见到图10.4的画面:图10.4在操作序列的情况下开启AlignX首先用户要把序列加载Vector NTI程序中,可以点选或者从左上方的Project →Add Files把序列档案加载,请注意文件名不可以过长,檔名过长会造成程序进行比对时无法完全显示文件名(图10.5):图10.5 输入的档名注意不可过长 选取档案后按下开启就可以加载程序中,若比对的序列很多时可以用鼠标圈选欲分析的序列后选择开启。
序列档案加载的时候程序会询问该序列为核酸序列或是蛋白质序列,点选好以后再点选Import就可以了(图10.6):图10.6 载入时,会询问序列的性质,核酸序列或蛋白质序列接下来程序的左上方会出现使用者加载的序列(图10.7),序列加载完成以后就可以开始进行比对的操作:图10.7 成功载入序列的画面进行比对前,先把欲比对的序列用鼠标进行圈选(图10.8):图10.8 选取欲比对之序列只要按下或是从上方Align→Align Selected Sequence(图10.9)就会进行比对运算:图10.9按下Align→Align Selected Sequence进行比对运算好以后就会出现下面的画面(图10.10);图10.10 比对完的结果 分析完成后画面(图10.11)会出现比对的相关结果,最下方是序列比对的图形,左边中间的区块所显示的图形为导引树(Guide tree),用来表示序列之间的关连性。
vector nti 11 使用教程 第十三章_蛋白质结构

第十三章蛋白质结构在进行蛋白质结构分析之前,用户首先要准备具有蛋白质结构的档案。
蛋白质结构的档案可以在/pdb/home/home.do之中寻找,找到欲分析的结构后即可直接下载。
下载的结构档案称之为PDB檔,除了NTI之外也可以用其他的软件进行观察。
NOTE:Vector NTI无法进行未知蛋白质结构的仿真,若要进行结构仿真必须使用其他软件。
在档案下载回来后请确认扩展名是否为.pdb,如果不是请将扩展名更改为.pdb。
只要扩展名的名称正确就可以直接点选开启,也可以从Vector NTI的程序中选择3D Molecule Viewer(图13.1):图13.1 利用程序集内的3D Molecule Viewer仿真蛋白质结构开启以后再选择或者是File→Open打开档案(图13.2):图13.2 选择PDB的档案,开启蛋白质序列打开档案后就可以看到一个蛋白质结构的图案(图13.3):图13.3 右边的窗口为蛋白质的结构,下方则是此蛋白质的序列在这个程序中的下方窗口为蛋白序列;左边中间的窗口是整个蛋白质结构的组成信息;左上方窗口为该结构的文件名。
使用者可以从上方这三个按钮来决定是否显示这些数据。
使用者可以进行翻转、移动及缩放蛋白质结构图形:只要按住键盘Shift键跟鼠标左键就可以将结构翻转;按住键盘Ctrl键跟鼠标左键拖曳就可以将结构移动;按住键盘Shift键、Ctrl键跟鼠标左键拖曳图形就可以将结构缩放。
结构的图片可以利用作输出。
图形的操作结果可以用或者是File→Save Project储存。
PDB档案打开来以后所呈现的图形都是未经过整理的,想要呈现较美观的图形必需将图形简化和改变显示方式。
首先用户可以先把显示原子的图形先关闭,只留下骨干的架构(图13.4)。
使用者可以在右上方的工具栏中点选或者从View→Hide→All atoms将显示原子图形的功能给关闭:图13.4 蛋白质的骨干架构用户可以看到图形只剩下骨干的构造,变得很单纯。
Vector NTI Suite使用简介

Vector NTI Suite使用简介资料来源:丁香园 Vector NTI Suite是一套功能强大、界面美观而又友好的分子生物学应用软件包。
它主要包括四个组件,分别对DNA、RNA和蛋白质进行各种分析和操作。
一、Vector NTI作为Vector NTI Suite的核心组成部分,它可以在各种分子生物学研究项目的全过程中提供数据组织、编辑和分析支持。
(一)对分子序列的操作我们以一个DNA序列为例,进行一系列的常规分析;最后将此DNA序列翻译成氨基酸序列,并对此氨基酸序列进行各种分析。
A,DNA序列为猪生长激素的cDNA序列,长为761bp。
首先使用Vector NTI的Create New命令将此序列导入到Vector NTI的数据库中:1,第一种方法:如果只知道序列时,点击Molecule才菜单中的Create New——Using Sequence Editor(DNA/RNA……);2,在出现的“New DNA/RNA Molecule”对话框中,首先在General填入导入序列的名称——PGH;3,在DNA/RNA Molecule活页中,选中Linear DNA, Animal/other Eukaryotes,Replicon Type中选Chromosome;4,Description中填入:S.Scrofa Growth hormone mRNA;5,在Sequence and Maps中点击“Edit Sequence”按钮,将DNA序列复制后,点“Paste”按钮-点“OK”-确认后就可以完成序列导入。
B,如果是一个从GenBank上下载的序列文件,则:点击“Molecule”菜单-Open-Molecule files命令,找到序列文件,在File format中选中GenBank Files;点击OK。
(二)常规操作:当序列导入完成后,在桌面出现三个窗口,上左侧的窗口中显示的是该序列的常规信息,上右侧窗口则以图形的格式展示序列的特征区及酶切图谱等。
vector NTI 11 使用教程 第12章 序列串接

第十二章 序列串接 使用Vector NTI中的ContigExpress程序(图12.1)可利用序列相似度,将小片段序列进行串连。
此程序可以将单纯序列文本文件或是由定序出来的讯号档案直接进行分析,串接后的片段称为Contigs,在进行长片段的序列定序或是genomic library定序时非常实用。
图12.1 使用ContigExpress将小片段序列串接将小片段的序列串接成完整大片段序列的方式如下之操作过程。
可以直接开启或是从主程序项目开启ContigExpress 程序(图12.2-3):图12.2 利用程序集开启ContigExpress程序图12.3 利用主程序开启ContigExpress程序开启此程序后会出现一个操作的画面,此画面会分为左右两个区块:图12.4 ContigExpress程序产生的两个区块在此窗口中,用户要先加入序列的档案。
一般序列的文本文件格式都可以加入程序中,而含有定序讯号的序列档案一般都为abi的文件格式,如果没有相关的程序是无法读取进行分析,而ContigExpress 可以读取和分析这种类型的档案。
要将序列档案或是定序的abi档案加载程序中的话,只要选择Project→Add Fragments就可以选择要加载程序的档案了(图12.5):图12.5 选择Project→Add Fragments加载程序的档案用户可以从窗口的左边的Fragments(图12.6)按下右键后选择Add Fragments 加入序列档案:图12.6 在Fragments 右键单击加入序列档案接下来由文件夹中选择欲加入的序列,可按住Ctrl键后用鼠标进行复选(图12.7):图12.7 可利用 Ctrl键,选取多个档案点选开启后,如果程序出现一连串的警告声并出现是否的寻问,那只是文件名一致性的修正询问,使用者可以忽略该状态并持续点选“是”的选项直到所有档案加载全程序为止(图12.8)。
NTI使用手册中文版

第一章安装和执行程序第一章安装和执行程序Vector NTI目前为网络版本,需要连到主机确认用户权力后才能运作。
厂商并没有针对授权时间进行限制,因此只要设定好License Server 的相关设定即可使用,不需要额外进行申请。
但用户IP 位置必须位于海洋大学校内,方可与授权服务器联机。
请下载下面这个档案进行安装.tw/Vector NTI Advance 10.exe 以下将说明如何设定的过程,变更为自服务器取得授权的方式: 1.选取开始→程序集→Invitrogen→Vector NTI Advance10→License Manager(图1.1)。
图1.1 授权设定2.在Applications 页面中(图1.2),点选下面的Dynamic 按钮,上面四个字段请填上使用者的姓名,系所单位,电话和电子邮件位置,最下面一栏URL of DLS 请入以下字符串:.tw:8080/vntidls.cgi。
DLS Server requires authentication 目前不需要勾选。
.tw/CMBB_REFIX/main/nti 1 Vector NTI教育训练手册图1.2 授权设定窗体3.在Internet Settings(图 1.3)里面建议选取Direct Connection,这样当IE 设定Proxy 之后,才不会导致Vector NTI因为IP 不在校内而不能联机。
图1.3 Vector NTI授权网络设定 4.设定之后可以点选Test Connection,程序会自动联机测试,如果在右边的联机结果显示Connection OK(图1.4),就表示设定成功;如果显示Connection error,则表示联机不成功,如果有开启防火墙/防病毒软件,请关闭或调整设定后再试一次。
图 1.4 Vector NTI授权网络设定,成功右下角会显示Connect OK .tw/CMBB_REFIX/main/nti 2 第一章安装和执行程序5.设定成功后,在Dynamic license 的页面记得按下Apply,最后在Applications (图1.5)把所有项目的Demo mode 改成Dynamic license。
vector nti 11 使用教程 附录A:如何取得序列

附录A 如何取得欲分析之序列透过网络抓取序列范例:首先,我们可以先进入NCBI 网站( /) (图A.1),在首页 “Search”下拉式选单及“for”文字盒中选择及输入适当选项内容,例如Search→Nucleotide;for →cyp1A,然后按下”Go ”按钮进行搜寻。
A.1 NCBI 网站此时 NCBI会传回如下之画面,目前 NCBI Nucleotide database 已经分成三个独立数据库,适当选择所需内容,点选所显示之数字进入适当数据库并进行下一步选择。
三个数据库分别为∙CoreNucleotide contains all Nucleotide records that are not in EST or GSS.They are of interest to most users.∙EST contains only Expressed Sequence Tag records.∙GSS contains only Genome Survey Sequence records.A.2 CoreNucleotide数据库例如点选图A.2中 “81 CoreNucleotide”之选项,可以进入更细部的选择,在CoreNucleotide数据库中总共含有81笔相关记录(图A.3),可依使用者所需进行点选,于此我们选择斑马鱼(zebrafish Danio rerio) 为范例进行说明,所以将点选第4笔数据NM_131879(Danio rerio cytochrome P450, family 1, subfamily A (cyp1a), mRNA)并得到如下图A.4:A.3 得到81笔斑马鱼的相关数据A.4 点选NM_131879得到的结果可以在左上角“Display”下拉式选单中选取“FASTA”,并得到以下仅含序列内容的画面信息(A.5)。
A.5 NM_131879FASTA格式的数据确定为所需之序列内容后,在“Display”下拉式选单右边 “Send to” 的下拉式选单选取“File”,此时系统将启动档案下载对话窗口,按下“储存”进行档案(FASTA格式)下载。
Vector NTI的使用-文档资料

功能模块主要包括:序列编辑、引 物设计、模拟翻译、模拟电泳、序列比 对、Contig、第三方附加模块等。
4
一、本机数据模块:
5
✓ 数据库分类
✓ 核酸分子 ✓ 蛋白质分子 ✓ 内切酶 ✓ 寡核苷酸 ✓ 电泳marker ✓ 引文 ✓ BLAST搜索结果 ✓ (PCR)分析结果
Vector NTI的使用
1
软件: ✓Vector NTI 8.0 / 10.3
此课件仅是提纲,具体使用 库的维护 ✓ 限制性内切酶 ✓ DNA→Protein ✓ Contig ✓ 序列比对及多重比对 ✓ DNA/质粒图谱绘制
3
软件包含两大模块: 数据库模块和功能模块。数据库用
✓酶切片段
✓ 完全酶切 ✓ 部分酶切
21
22
部分酶切
23
部分酶切
24
部分酶切
25
✓序列比对
26
按住“Ctrl”键后,用鼠标左键可以进行复杂的选择, 然后在选择的项目上点鼠标右键,关联菜单弹出……
27
这里先做选取
28
✓Contig
✓ *.abi或*.scf文件与序列文件 ✓ 构建一个Contig ✓Contig是“contiguous sequences group”的缩
✓ 每类数据库又分为基库(MAIN)和子库(subset)
✓ 真正的数据都在基库里,子库只是用户自己建立的数据条目(item)的子集,里面 是数据的联接不是数据本身,但是可以象操作数据一样进行直接操作;
✓ 对数据条目进行“拷贝”、“粘贴”操作,只会产生一个新的联接,不会产生新 的数据;要产生数据复制,需要用“Duplicate”(数据条目上点右键);
VectorNTI使用中文说明

Vector NTI它主要包括四个组件,分别对DNA、RNA和蛋白质进行各种分析和操作。
一、Vector NTI 作为Vector NTI Suite的核心组成部分,它可以在各种分子生物学研究项目的全过程中提供数据组织、编辑和分析支持。
(一)对分子序列的操作我们以一个DNA序列为例,进行一系列的常规分析;最后将此DNA序列翻译成氨基酸序列,并对此氨基酸序列进行各种分析. A,DNA序列为猪生长激素的cDNA序列,长为761bp。
首先使用Vector NTI的Create New命令将此序列导入到Vector NTI的数据库中: 1,第一种方法:如果只知道序列时,点击Molecule才菜单中的Create New—-Using Sequence Editor(DNA/RNA……); 2,在出现的“New DNA/RNA Molecule"对话框中,首先在General填入导入序列的名称——PGH; 3,在DNA/RNA Molecule活页中,选中Linear DNA, Animal/other Eukaryotes,Replicon Type中选Chromosome;4,Description中填入:S.Scrofa Growth hormone mRNA; 5,在Sequence and Maps中点击“Edit Sequence”按钮,将DNA 序列复制后,点“Paste”按钮-点“OK”-确认后就可以完成序列导入。
B,如果是一个从GenBank上下载的序列文件,则:点击“Molecule" 菜单-Open-Molecule files命令,找到序列文件,在File format中选中GenBank Files;点击OK。
(二)常规操作:当序列导入完成后,在桌面出现三个窗口,上左侧的窗口中显示的是该序列的常规信息,上右侧窗口则以图形的格式展示序列的特征区及酶切图谱等。
下面一个窗口显示的是序列:默认状态下以双链形式出现,也可以更改为单链显示。
vector nti

vector ntiVector NTI 是一种强大的分子生物学工具,广泛用于分析、设计和操作DNA、RNA和蛋白质序列。
本文将介绍Vector NTI 的主要功能、特点以及在生物科学研究中的应用。
一、介绍Vector NTI 是由Invitrogen公司开发的一款分子生物学软件,旨在为科学家们提供一套完整的工具,用于分析、设计和操作生物分子。
该软件集成了多种功能,包括序列分析、比对、克隆、构建基因工程实验方案等等。
二、主要功能1. 序列分析Vector NTI 提供了各种序列分析的功能,例如序列比对、绘制序列图谱、查找开放阅读框、预测转录起始点和终止点等等。
这些功能可以帮助科学家们更好地理解DNA、RNA和蛋白质序列的结构和功能。
2. 克隆工具Vector NTI 提供了丰富的克隆工具,可以帮助科学家们进行DNA 重组、构建基因工程实验方案等。
通过这些工具,科学家们可以轻松地进行基因克隆,并设计出符合实验需要的DNA序列。
3. DNA构建与设计Vector NTI 提供了功能强大的DNA构建和设计工具,可以帮助科学家们设计合适的引物、多聚酶链反应(PCR)方案以及合成DNA 序列等。
科学家们可以根据实验需求,通过这些工具快速设计出理想的DNA序列。
4. 蛋白质序列分析除了DNA和RNA序列分析外,Vector NTI 也可以进行蛋白质序列分析。
科学家们可以利用软件提供的功能,预测蛋白质二级结构、寻找蛋白质结构域、进行蛋白质序列比对等。
5. 实验方案管理Vector NTI 提供了实验方案管理功能,科学家们可以记录和管理自己设计的实验方案。
这方面的功能还包括实验标签、实验进度跟踪等,方便用户进行实验管理工作。
三、特点1. 用户友好的界面Vector NTI 提供了直观、易于使用的用户界面,使得科学家们能够快速上手并进行工作。
软件的菜单和工具栏都设计得非常直观,用户可以轻松找到自己需要的功能。
2. 数据库支持Vector NTI 可以与各种常见的生物信息数据库进行集成,例如GenBank、EMBL和SwissProt等。
vectorNTI中文使用说明书

vectorNTI中文使用说明书Vector NTI7.0 User's Manual软件包中文翻译者:宋厚辉(浙江大学),记住这个伟大的人物吧!前言(Introduction)1.程序附带的数据库(Vector NTI database)包括:DNA/RNA、蛋白质、内切酶、寡核苷酸、凝胶mark。
另外程序还提供数据库开发(Database Explorer)功能,用户能够自己修改、添加、拷贝感兴趣的各类数据库。
2.创立新分子(有四种方法)A.用GenBank/GenPept, EMBL/SWISS-PROT and FASTA、ASCII等格式输入DNA或氨基酸。
B.手工粘帖,然后保存到数据库中C.从其它分子、接头、载体中剪切、拼接构键D.从DNA或RNA分子的编码区翻译成蛋白质3.关于新分子的序列特征图谱:利用GenBank/GenPept,EMBL/SWISS-PROT or FASTA等格式输入的分子都能显示出序列和结构图,但自己手工粘帖的没有,需要自己编辑第一章Chapter 1Tutorial: Display Windows(显示窗口)目的:创立显示窗口,并对图、序列和文本进行操作• 1.登录Vector NTI安装后首次登录,系统将提示是否允许填充空库,点OK。
这样DNA molecules, proteins, enzymes, oligos, and gel markers将组成NTI的数据库。
并出现下列两个窗口。
• 2. 观察出现的 Vector NTI 工作窗口和 Database Explorer窗口上面的第一个窗口为工作窗口,由菜单栏和工具条两栏,移动鼠标到工具栏任意选项处,鼠标自动显示每个工具条的功能。
第二个窗口为exlporing——local vector NTI database,显示的是上次打开的DNA/RNA或蛋白分子。
3. Create a Display Window for pBR322激活exlporing——local vector NTI database窗口中的DNA/RNA Molecules (MAIN) 数据库,找到pBR322分子并双击打开。
04VectorNTI的使用

04VectorNTI的使用Vector NTI(简称NTI)是一款常用于分子生物学研究的软件,主要用于序列分析、蛋白质结构模拟和分子克隆实验的设计等方面。
本文将介绍NTI的安装和使用方法,并详细讲解其主要功能和特点。
一、安装NTI1. 打开浏览器,“Vector NTI”。
二、使用NTI1.启动NTI安装完成后,可以在开始菜单或桌面上找到NTI的快捷方式,点击打开软件。
2.序列分析NTI可以对DNA、RNA和蛋白质序列进行多方面的分析。
(1)打开序列文件点击菜单栏的“文件”>“打开”选项,选择要分析的序列文件,可以是FASTA、GenBank或其他格式的文件。
(3)序列比对点击菜单栏的“分析”>“序列比对”选项,选择要比对的序列文件,进行多序列比对分析。
(4)ORF预测点击菜单栏的“分析”>“ORF预测”选项,输入要预测的序列及参数设置,进行开放阅读框(ORF)的查找和分析。
(5)启动子预测点击菜单栏的“工具”>“启动子预测”选项,输入要预测的序列及参数设置,进行启动子的查找和分析。
3.蛋白质结构分析NTI可以对蛋白质的结构进行建模和分析。
(1)打开蛋白质结构文件点击菜单栏的“文件”>“打开”选项,选择要分析的蛋白质结构文件,可以是PDB、MOL或其他格式的文件。
(2)蛋白质结构模拟选中要模拟的蛋白质结构,点击右键选择“模拟结构”进行分子动力学模拟或力场优化。
(3)蛋白质结构可视化点击菜单栏的“显示”>“蛋白质结构”选项,可以调整蛋白质的显示方式,包括线框图、空间填充等。
4.分子克隆实验设计NTI可以辅助设计分子克隆实验。
(1)打开DNA序列文件点击菜单栏的“文件”>“打开”选项,选择要设计的DNA序列文件。
(2)引物设计点击菜单栏的“功能”>“引物设计”选项,输入目的序列及参数设置,进行引物的设计和注释。
(3)酶切位点分析点击菜单栏的“功能”>“酶切位点分析”选项,输入目的序列及参数设置,进行酶切位点的查找和分析。
VNTI9_Users_Manual中文版

版权声明:本站几乎所有资源均搜集于网络,仅供学习参考,不得进行任何商业用途,否则产生的一切后 果将由使用者本人承担! 本站仅仅提供一个观摩学习与交流的平台, 将不保证所提供资源的完 整性,也不对任何资源负法律责任。
所有资源请在下载后 24 小时内删除。
如果您觉得满意, 请购买正版,以便更好支持您所喜欢的软件或书籍!☆☆☆☆☆生物秀[]☆☆☆☆☆中国生物科学论坛[/bbs/]☆☆☆☆☆生物秀下载频道[/Soft/]生物秀——倾力打造最大最专业的生物资源下载平台!■■■ 选择生物秀,我秀我精彩!!■■■欢迎到生物秀论坛(中国生物科学论坛)的相关资源、软件版块参与讨论,共享您的资源,获 取更多资源或帮助。
Vector NTI User's Manual前言(INTRODUCTION) (3)第一章 DISPLAY WINDOWS (4)一. 顯示DNA序列及圖形 (5)二. 顯示蛋白質序列的方式 (10)第二章 MOLECULE OPERATIONS (13)一. 對P BR322’S 的常用資料(GENERAL DATA)進行編輯 (13)三. 插入新的序列片段 (15)四. 編輯TC(R) 圖示 (16)五. 刪除P2_P標示,並加入新的序列特徵標示 (17)六. 修改M Y P BR322的起始座標 (18)第三章 GRAPHICAL REPRESENTATION (19)一. 爲P BR322圖形建立一個展示視窗 (19)二. 修改圖形的自動排列設置 (19)三. 修改圖形的編碼區信號(CDS SIGNALS)設置 (19)四. 打開圖片編輯方式 (20)五. 修改TC(R)圖形 (20)六. 加入註解注釋 (22)七. 將P BR322 分子顯示結果保存到文檔文件中 (22)第四章 BLAST SEARCH AND BLAST VIEWER (24)一. 簡介 (24)二. BLAST 搜尋視窗 (24)三. BLAST S EARCH R ESULTS (28)四. S A VING BLAST S EARCH R ESULTS (29)第五章 ALIGNX (31)一. 開啟與執行: (31)二. 參數設定與調整: (35)三. 資料輸出與列印 (39)第六章 ALIGNX-BLOCKS (40)一. 開啟與執行: (40)二. 參數設定與調整: (42)三. 資料輸出與列印 (44)第七章 BIOPLOT (45)一. 開啟與執行: (45)二. 資料輸出與列印 (49)第八章 CONTIGEXPRESS (50)一. I NTRODUCTION (50)二. P ROJECT E XPLORER (50)三. W ORKING IN F RAGMENT W INDOW (54)四. W ORKING IN THE C ONTIG W INDOW (60)第九章 MISCELLANEOUS VECTOR NTI TOOLS (63)一. I NTRODUCTION (63)二. P UB M ED/E NTREZ S EARCH (63)三. C ITATION V IEWER (66)附記:如何執行反安裝 (69)- 2 -前言(Introduction)程式附帶的資料庫(Vector NTI database)包括:DNA/RNA序列、蛋白質序列、限制酶、寡核苷酸、電泳marker。
Vector NT使用教程之第一章安装和执行程式

5
第一章 安裝和執行程式
第一章 安裝和執行程式
Vector NTI 目前為網路版本,需要連到主機確認使用者權限後才能運作。廠商並 沒有針對授權時間進行限制,因此只要設定好 License Server 的相關設定即可使用, 不需要額外進行申請。但使用者 IP 位置必須位於海洋大學校內,方可與授權伺服器 連線。 請下載下面這個檔案進行安裝 .tw/Vector NTI Advance 10.exe 以下將說明如 何設定的過程,變更為自伺服器取得授權的方式: 1.選取開始→程式集 →Invitrogen→Vector NTI Advance 10→License Manager(圖 1.1)。
圖 1.7 左邊為文字窗口,右邊為圖譜窗口,下方為序列窗口
文字窗口
圖譜窗口
序列窗口
這三個窗口為文字窗口、圖譜窗口和序列窗口。文字窗口會將分析結果,作簡 單的文字敘述,並且可以自動做註記;圖譜窗口會將序列以一個卡通圖形進行說明,
4
第一章 安裝和執行程式
同時具有繪圖的功能;序列窗口會將分析結果,標定在序列上面,以序列呈現出來。 接下來就是將序列載入程式中,我們可以將實驗或是搜尋所獲得的序列資料放
圖 1.1 授權設定
2.在 Applications 頁面中(圖 1.2),點選下面的 Dynamic 按鈕,上面四個欄位請填 上使用者的姓名,系所單位,電話和電子郵件位置,最下面一欄 URL of DLS 請入以 下字串: .tw:8080/vntidls.cgi。 DLS Server requires authentication 目前不需要勾選。
vector nti 载体分析

载体(vector) ,指在基因工程重组DNA技术中将DNA片 段(目的基因)转移至受体细胞的一种能自我复制的DNA 分子。
载体必备条件 ①含有复制起始位点,能够独立复制,在宿主细胞中能保存下来 并能大量复制。 ②有多个限制酶切点,而且每种酶的切点最好只有一个。 ③有一定的标记基因,便于进行筛选。 三种最常用的载体是细菌质粒、噬菌体和动植物病毒。
①Ori的位置,了解质粒的 类型
③筛选标记
载体必备条件 ①含有复制起始位点, 能够独立复制,在宿主 细胞中能保存下来并能 大量复制。 ②有多个限制酶切点, 而且每种酶的切点最好 只有一个。 ③有一定的标记基因, 便于进行筛选。
①Ori的位置,了解质粒的 类型(原核/真核/穿梭质 粒)
②看多克隆位点(MCS)
③筛选标记 ④表达系统元件
谢谢
pBR322是一个人工 构建的重要质粒, 有万能质粒之称。 它是由pSF2124、 pMB1及pSC101三个 亲本质粒经复杂的 重组过程构建而成 的。
如何读懂质粒图谱
p BR 322
Plasmid 研究者Bolivar和Rogigerus
实验编号
载体必备条件 ①含有复制起始位点, 能够独立复制,在宿主 细胞中能保存下来并能 大量复制。 ②有多个限制酶切点, 而且每种酶的切点最好 只有一个。 ③有一定的标记基因, 便于进行筛选。
vector NTI 11 使用教程 第14 章文献搜寻
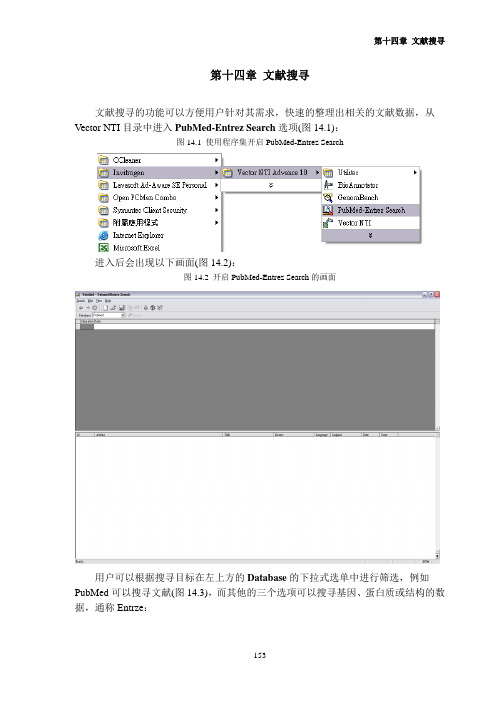
第十四章文献搜寻文献搜寻的功能可以方便用户针对其需求,快速的整理出相关的文献数据,从Vector NTI目录中进入PubMed-Entrez Search选项(图14.1):图14.1 使用程序集开启PubMed-Entrez Search进入后会出现以下画面(图14.2):图14.2 开启PubMed-Entrez Search的画面用户可以根据搜寻目标在左上方的Database的下拉式选单中进行筛选,例如PubMed可以搜寻文献(图14.3),而其他的三个选项可以搜寻基因、蛋白质或结构的数据,通称Entrze:图14.3 在下拉式选单中,选择要筛选的项目用户先在上方Term的项目中输入关键词(图14.4):图14.4 直接在Term项目输入关键词若要增加搜寻的条件可以在Term的字段中按下鼠标右键后点选Insert row (图14.5),也可以从主画面最上方Edit项目中选择Insert row的功能:图14.5在Term字段点选 Insert row进行增加字段的动作这时候窗体下方会出现另外一个新的字段(图14.6),使用者可以在新的字段中再增加条件:图14.6 点选Insert row后会增加一列使用者可以选择OR、AND、NOT来做为搜寻的条件(图14.7),还可以按下鼠标右键后点选Subcondition功能,使搜寻方式更加细腻:图14.7使用者可以选择OR、AND、NOT来搜寻若要消除搜寻条件只要使用鼠标把字段反白后,按下鼠标右键后选择Delete row 即可(图14.8):图14.8 删除搜索条件设定好搜寻的条件之后,接下来就是要设定显示的条件:从Edit的项目中选择Settings(图14.9):图14.9显示方式的设定进入以后可以调整单一页面所显示的篇数以及文献发表的时间(图14.10),设定好以后点选OK即可。
图14.10 Documents per page为每页显示的篇数,右边字段则是文献出版及发表时间当设定完成之后点选主画面最上方的Search,系统就会透过网络开始进行搜寻,搜寻的结果如图14.11所示:图14.11 搜寻完的结果会显示在窗口的下方用户可以看到程序下方会出现搜寻的数据,可以用鼠标点选开启(图14.12):图14.12 直接点击左方窗口的数据,开启数据内容在这个窗口的右上方有个PubMed ID的项目(图14.13)可以链接到NCBI的数据库,用户可透过此数据库下载文献:图14.13 点选PubMed ID里的项目连接到NCBI图14.14 利用NCBI查看较新的文献数据NOTE:有些文章需要付费才能下载全文,若无法下载该文献表示本校并没有购买该期刊的版权,除此之外有些非常新的发表也会暂时无法下载(图14.14)。
vector nti 11 使用教程 第十一章_AlignX Blocks

第十一章 AlignX BlocksAlignX Blocks可以用在蛋白质序列比对,和AlignX不同的地方在于比对的序列会以图形表示比对的结果。
启动AlignX Blocks的方式和AlignX是一样的,可直接开启或是从主程序上方开启(图11.1-2):图11.1 直接于程序集中开启AlignX Blocks图11.2 于Vector NTI程序内开启AlignX Blocks开启程序后会看见和AlignX很类似的操作界面(图11.3):图11.3 开启AlignX程序后的接口首先用户要把序列档案加载程序里面,可以点选或者从左上方的Project →Add Files把序列档案载入,序列只能分析蛋白质序列(图11.4):图11.4 利用Project→Add Files将序列加载到左上窗口进行比对前先将要比对的序列用鼠标反白(图11.5),操作和AlignX相同:图11.5 全选欲进行比对的序列比对前可以依各人需求调整序列比对的条件跟参数(图11.6)。
可点选或是从Blocks→AlignX Blocks Setup进行调整:图11.6 参数的设定会影响程序的运算,要适当的调整得到所要的结果参数的设定会影响到程序的运算,并造成不同的比对结果。
使用者可针对这三个参数进行调整,直到得到满意的结果为止。
Blocks’colors项目(图11.7)表示比对后各序列相似区域所显示的颜色,默认值为红色,可以依照喜好设定颜色。
图11.7 Blocks’ colors改变显示的颜色和AlignX相同,使用者也可以设定Score matrix的参数(图11.8),但是不建议一般使用者更改。
图11.8 更改Score matrix的参数,设定好了以后按下或者是选取Blocks→Search for blocks程序进行运算,经运算完成之后会出现下面的画面:图11.9 运算的结果:左下为相似的区域,右上为序列的图形显示,右下为比对的画面在左下的窗口(图11.9)中所显示的是程序依照蛋白质序列和设定的参数所比对出相似的区域,此区域称之为block;右上方的窗口是把比对的序列以图形显示,可以判别block之间的位置关系;右下方则是序列的比对画面。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
Vector NTI它主要包括四个组件,分别对DNA、RNA和蛋白质进行各种分析和操作。
一、Vector NTI 作为Vector NTI Suite的核心组成部分,它可以在各种分子生物学研究项目的全过程中提供数据组织、编辑和分析支持。
(一)对分子序列的操作我们以一个DNA序列为例,进行一系列的常规分析;最后将此DNA序列翻译成氨基酸序列,并对此氨基酸序列进行各种分析。
A,DNA序列为猪生长激素的cDNA序列,长为761bp。
首先使用Vector NTI的Create New命令将此序列导入到Vector NTI的数据库中:1,第一种方法:如果只知道序列时,点击Molecule才菜单中的Create New——Using Sequence Editor(DNA/RNA……);2,在出现的“New DNA/RNA Molecule”对话框中,首先在General填入导入序列的名称——PGH;3,在DNA/RNA Molecule活页中,选中Linear DNA,Animal/other Eukaryotes,Replicon Type中选Chromosome;4,Description中填入:S.Scrofa Growth hormone mRNA; 5,在Sequence and Maps中点击“Edit Sequence”按钮,将DNA序列复制后,点“Paste”按钮-点“OK”-确认后就可以完成序列导入。
B,如果是一个从GenBank上下载的序列文件,则:点击“Molecule”菜单-Open-Molecule files命令,找到序列文件,在File format中选中GenBank Files;点击OK。
(二)常规操作:当序列导入完成后,在桌面出现三个窗口,上左侧的窗口中显示的是该序列的常规信息,上右侧窗口则以图形的格式展示序列的特征区及酶切图谱等。
下面一个窗口显示的是序列:默认状态下以双链形式出现,也可以更改为单链显示。
1.选择序列区域:在图形区域或序列区域直接拖动鼠标左键,同时在最下端的状态栏中显示出所选区域的范围。
2.删除:选中后直接点击键盘上的Delete键,确认后即可删除。
3.选中序列片段后,点击Edit菜单,用其中的命令可以完成对此片段的剪切、复制、删除、定义为新的特征区和用其它序列来代替等。
4.当点在其一特定位置时,我们也可以在此位置插入新的序列:Edit –New –Insert Sequence as 5.当希望序列显示单链时,点击View –Show Both Strands (三)常规分析:1.设计PCR Sequence Primer, Hybridization, Probes:选中设计引物的模板区或点击Analyze中的相应命令即可。
需要注意的是,在设计前,首先得将序列存入数据库中,具体设计由于我们推荐使用Oligo,所以此处不详述。
2.序列基本信息分析:选中序列区段后,选Analyze –Oligo Analysis, 在Oligo Analysis对话框中,点击Analyze按钮,即可得到分子量、GC含量、Tm值、3‘端的自由能、回文结构及重复序列等基本信息。
3.酶切图谱分析点击Analyze菜单中Restriction Sites命令,出现“Restriction Map Setup”对话框,点击Add按钮,填入需要分析的位点,不需要的位点夜可以选中后点Remove按钮移除。
为了显示正确,我们可以设定超过一定位点数量的酶不显示,可以限定分析的区域等。
点击OK后程序自动完成酶切分析。
4.Motif查找点击Analyze菜单中的Motifs命令,在出现的Motifs Setup 对话框中我们可以添加新的Oligo或从Oligo Database和Oligo List中选取;选中后点击OK按钮,程序完成Motif的查找,同时给出相似性的百分比。
5.ORF查找点击Analyze菜单中的ORF命令,在出现的ORF Setup对话框中,填入ORF的最小长度(多少个密码子)以及其它一些设定后点击OK,程序自动完成ORF的查找。
6.翻译翻译前选中一个ORF或一个区域,这里我们希望把pGH cDNA基因完全翻译成蛋白质,因此选中最长的一个ORF(7-657bp)。
点击Analyze菜单中的Translate命令中的“Into New Protein”-“Direct Strand”,在出现的“New Protein Molecule”对话框中给出新蛋白质的名称后点击确定,程序完成翻译并打开一个新的窗口,显示氨基酸序列。
7.反翻译选中氨基酸序列片断,点击Analyze菜单中的BackTranslate命令,确认是“整个序列”还是“仅为选中的序列”后,即可设定简并度及组织特异性来完成反翻译。
(三)模拟电泳:模拟电泳是指对DNA片段进行酶切分析后,通过电脑模拟电泳过程,将酶切片段分离。
该功能有利于评估电泳时间,便于验证实际电泳结果的好坏。
1.点击Gel菜单–Create New命令:出现Gel Setup对话框,首先选择电泳介质的类型-“Agarose Gel”,以及胶的浓度、电压、胶长和Buffer种类。
2.点击OK后即打开一个电泳界面,左侧是对电泳情况的描述,右侧则为胶板。
3.首先配置一个Marker:点Gel菜单–Create Gel Marker, 出现New Gel Marker对话框, 首先输入Marker的名称:pGH-Marker,在Gel Marker活页中输入自己设定的片段,100,200,300,400,500,600,1000bp,点击确定。
4.样品制备:点Gel菜单–Create Gel Sample命令出现Create Gel Sample对话框,选择来源分子SSPGH,选中AvaI和SmaI两个酶,输入Sample的名称SSPGH- AS。
此时我们可以把酶切的片段直接加入到胶上,也可以加入到样品列表中或保存为Marker,我们点击Add to Gel,则在胶上出现1号样品。
5.点Marker到胶中:点击第二行工具栏中的Add Marker Lane图标,出现Choose Database Gel Marker对话框,选中pGH - Marker,点击OK,则Marker出现在2道6.电泳:用鼠标点击右侧窗口激活胶板,点击第二栏工具栏中的双箭头,开始电泳,同时在旁边显示电泳时间,再次点击双箭头,电泳停止。
7.计算两条带分开时间:选中没有分开的条带,点工具栏中的计算器Calculate Seperation Time,即可得到所需信息。
8.导出胶图:激活胶板,点击工具栏中的Camera工具,出现Camera对话框,选中需要导出的选项,结果可以到剪贴板,也可以保存成文件。
到剪贴板后就可以在Word等文档编辑器中粘贴。
(四)图形操作对于图形展示的序列信息,我们可以对图形进行各种修饰和改动,并最终导出需要的图形,如果序列是质粒,则可得到质粒图谱。
我们还是以SSPGH序列为例:1.激活图形栏,点击工具栏中的Edit Picture。
此时,点击任意一个需要改动的组件,则鼠标变为四方向箭头,按住左侧则可以任意拖动其位置。
2.点击左键,选中Properties命令,则在Properties对话框中可以改变文字,字体,连线的粗细和颜色。
3.对于特征序列,我们还可改变其填充方式,箭头方向等。
4.加入注释:点击工具栏中的回形针按钮,出现Annotation对话框,输入注释文字(支持中、英文),如:“这是一个测试Sequence”。
点击确定后,文字就出现在图示窗口中,同样可以改变其位置、字体、颜色等。
5.修改完成后,点工具栏中的命令,同样可以到剪贴板或文件。
二、组件AlignX运行AlignX后出现四个窗口,从上到下,从左到右分别为序列信息窗口,进化树窗口,同源比较图示窗口和序列比较窗口。
对于同源比较可以分为两类:一类是两个或几个序列(包括DNA/RNA和蛋白质序列)的比较,此时仅限于比较序列的相似性;另一类则是多个序列间的比较并得出系统进化树。
(一)导入外源序列的方法:点击Project菜单中Add Files命令,选择需要比较的序列文件,点击打开按钮,确认是DNA/RNA还是Protein Sequence后,点击Import按钮就可以完成序列的导入任务。
(二)我们以程序本身提供的演示Project来讲述AlignX的使用:1.选择Project菜单Open命令,找到Vector NTI的Demo Projects文件夹,打开DNA.apr文件;2.在文本窗口中,使用鼠标左键双击任一文件名,就可以得到该文件的所有基本信息;3.建立一个新的比较策略并进行比较:①在文本窗口中,按住Ctrl键选中四个序列:AF××××②点击Alignment菜单中Alignment Setup命令,出现Alignment Setup对话框,在此对话框中有30个选项来确定最终比较结果展示方式,这里我们使用默认值即可。
③点击Alignment菜单中的Align Selected Sequence命令,片刻后程序完成比较并给出结果和进化树;④在View菜单中,我们可以通过Edit Alignment命令来编辑比较结果,使用Display Setup命令来改变结果的展示方式和颜色等。
4.结果的导出:①进化树的导出:激活进化树窗口,点击工具栏中的Export Tree按钮即可将进化树保存为.Ph 文件,并可被其他树编辑软件所识别。
或者点击Edit菜单中的Camera命令,将图像保存到剪贴板后在Word 等文档编辑软件中粘贴;②序列比较结果的导出;点击Project菜单中的Export MSF Format命令,将结果保存为.msf文件后,用GeneDoc打开进一步进行编辑和修饰。
三、Contig Express 该组件让您能够直接从测序仪或其它文件格式(如GenBank和Fasta等)中导入序列,并将这些阿序列片段拼接成一个长片段。
在拼接的过程中可以显示测序图谱,可以自动去除载体序列及测序模糊序列。
同时能在拼接的完成序列碱基的改动。
1.点击Project菜单中的Open命令,找到Demo Project 文件夹中的DNA.cep文件。
当然我们也可以导入自己的序列,方法是点Project菜单中的Add Fragments命令,在此可以导入各种格式的文件。
2.建立拼接策略:点击Assemble菜单中的Assembly Setup命令,出现Assembly Setup对话框,在此我们使用程序的默认值,不作改动。
