circRNA基因敲除细胞株的经典方法及案例
circrna_沉默实验步骤

circRNA沉默实验步骤引言c i rc RN A(环状RN A)是一类形态和功能独特的非编码RN A分子,以环状结构存在于细胞质中。
近年来,c irc R NA的研究逐渐引起了科研人员的关注。
为了深入探究ci rc RN A的功能和作用机制,科学家们进行了大量的实验研究。
本文将介绍一种常用的c ir cR NA沉默实验步骤,帮助读者了解如何进行ci r cR NA的沉默实验。
实验材料1.细胞系:携带目标c ir cR NA的细胞系2.小干扰R NA(s iRN A):具有针对目标c ir cR NA的沉默效果的s i RN A3.载体:用于转染si R NA的合适载体4.转染试剂:用于将s iR NA转染入细胞的试剂,如脂质体试剂等5.细胞培养基:适合细胞系生长的培养基及其所需的补充物6.细胞裂解缓冲液:用于提取总RN A或总蛋白的缓冲液实验步骤1.细胞的预处理-将细胞培养在含有适当量的培养基的细胞培养皿中,使其达到合适的生长状态。
-确定细胞的适当密度,并将细胞均匀地分散在培养皿中。
-在培养箱中以37°C和5%CO2培养细胞,直至达到临界浓度。
2.转染si RN A-按照转染试剂的说明书,制备转染试剂的工作液。
-将合适浓度的s iR NA与转染试剂混合,并在室温下孵育一段时间,形成si RN A-转染试剂复合物。
-将复合物缓慢而均匀地滴加到细胞培养皿中,使其与细胞充分接触。
-将细胞培养皿放回培养箱中,继续在37°C的条件下培养一段时间,使复合物被细胞摄取。
3.提取总R NA或总蛋白-使用细胞裂解缓冲液裂解转染细胞,溶解细胞膜并释放内部物质。
-将细胞裂解液收集到离心管中,并进行离心以除去细胞碎片和细胞核等残留物。
-收集上清液中的总R N A或总蛋白,用于后续实验。
4.RT-q PC R检测c ir c RN A沉默效果-使用逆转录酶将总R N A逆转录为c DN A,并进行实时荧光定量P CR(R T-qP CR)检测ci r cR NA的表达水平。
CircRNA在结直肠癌中的研究进展

综述提供了 cricRNA进行抗癌治疗的原理。
关 键词:环状R N A ;结 直肠癌;增 殖 ;治疗
中 图 分 类 号 :R735.3
文 献 标 识 码 :A
文 章 编 号 :1009-881X(2021)01-0126-05
Research Development of CircRNA in Colorectal Cancer
Key words :circRNAs;colorectal cancer;proliferation;therapy
结直肠癌是世界范围内常见的消化系统恶性肿 瘤 。2021年发布的美国癌症统计数据显示, 目前结 直 肠 癌 发 病 率 在 消 化 系 统 癌 症 中 居 于 首 位 ,死亡人 数占全部恶性肿瘤死亡人数的8.7 % [1]。在 中 国 ,结 直 肠 癌 的 发 病 率 仅 次 于 肺 癌 和 胃 癌 ,位 居 所 有 恶 性 肿瘤的第3位 ,并且发病率还在不断上升[2]。近年来
1 CircRNA在结直肠癌中的角色
已 有 的 研 究 表 明 ,circR N A 在结直肠癌的发 生 和 发 展 过 程 中 发 挥 了 很 大 作 用 [181。一 项 研 究 通过使用A rraystar circRNA微 阵 列 分 析 ,确定了 1 8 1 7 个在结肠癌组织中具有明显不同表达谱的 circR N A , 其 中 1 2 3 6 个 上 调 ,5 8 1 个下调[19]。 CircRNA在 结 肠 癌 中 参 与 调 控 细 胞 增 殖 、细胞凋 亡 、肿 瘤 侵 袭 性 和 转 移 、细 胞 周 期 、癌症干细胞 以 及 上 皮 -间 充 质 转 化 (E M T ) 。这 一 部 分 将 具 体 阐 述 cricR N A 在 这 些 细 胞 过 程 中 的 作 用 并 分 析 circRNA在 结 直 肠 癌 中 的 机 制 。 1.1 CircRNA调 控 细 胞 增 殖 很 多 研 究 表 明 circRNA 在结直肠癌细胞增殖中起到重要作用。2以 !^等 [2(>] 通过实时荧光定量PCR实 验 、CCK8检测实验以及 细 胞 克 隆 形 成 实 验 研 究 发 现 ,circP P P lR 12A 的表 达加速了DLD-1、Caco-2、HCT-116和LoVo结肠癌
CRISPRCas9基因敲除细胞株详细构建流程

CRISPRCas9基因敲除细胞株详细构建流程Puro 抗性浓度摸索实验将细胞如下图1稀释。
给药7天后,弃培养液,用台盼蓝染色2 min,显微镜下观察细胞存活情况。
确定细胞多克隆细胞筛选和单克隆细胞筛选浓度。
图1 抗性浓度摸索单克隆形成验证实验细胞计数,将细胞悬液稀释混匀后加入96孔板,用封口胶将孔板封好,放于培养箱中培养。
静置培养48 h后每日观察并记录单克隆形成情况。
sgRNA 靶标设计根据基因序列信息,设计 sgRNA。
靶标位点序列信息确认PCR扩增(图2),测序验证基因序列,以确定sgRNA区域有无SNP。
图2 靶标序列扩增sgRNA克隆引物合成根据sgRNA设计sgRNA克隆引物。
lentiCRISPRv2-sgRNA载体构建退火,连接,转化,涂板(LB/Amp)培养。
lentiCRISPRv2-sgRNA载体验证每个实验组各挑取6个单克隆菌落,于LB/ Amp培养基中扩增,提取质粒,琼脂糖凝胶电泳检测质粒抽提效果(图3)。
图3 质粒抽提酶切验证:取3个单克隆进行酶切,琼脂糖凝胶电泳检测酶切效果(图4)。
选择2个样品送样测序。
图4 单克隆酶切验证病毒包装lentiCRISPRv2-sgRNA无内毒素质粒提取,病毒包装。
细胞转染配制梯度病毒稀释液,细胞于培养箱中静置培养48h。
阳性单克隆细胞株筛选细胞转染48h后,更换完全培养基,筛选至对照组大部分细胞死亡,实验组细胞扩大培养,进行单克隆筛选。
几天后挑选阳性单克隆进行扩增,并取样验证。
阳性单克隆细胞株验证测序验证阳性单克隆细胞株的基因序列,以确定是否敲除成功。
实验结果示例:该基因有两个单克隆细胞株,A1和A2。
单克隆细胞A1的目的基因在sgRNA2位置出现两种突变形式,分别缺失1个和19个碱基,在新序列的第337位和第355位碱基提前出现终止密码子。
单克隆细胞A2的目的基因在sgRNA2位置发生突变,插入1个碱基,在新序列的第340位碱基提前出现终止密码子。
细胞基因敲除策略

细胞基因敲除策略细胞基因敲除是一种常用的遗传学实验技术,可以通过改变细胞基因组的DNA序列,进而研究基因的功能和调控机制。
下面将介绍几种常见的细胞基因敲除策略。
1. CRISPR/Cas9系统CRISPR/Cas9系统是近年来最受关注的基因编辑技术之一。
它利用一种来自细菌的CRISPR RNA (crRNA)和转录后修饰RNA (tracrRNA)以及Cas9蛋白组成的复合物,通过靶向特定DNA序列并引导Cas9蛋白进行双链切割,实现基因敲除。
这种技术具有高效、简便、经济的特点,被广泛应用于细胞和动物模型中。
2. RNA干扰技术RNA干扰技术是通过使用小干扰RNA (siRNA) 或特殊的小分子RNA (shRNA) 来沉默目标基因的表达。
siRNA或shRNA与目标mRNA序列互补结合,形成RNA诱导沉默复合物(RISC),导致mRNA降解或翻译受阻,从而实现基因敲除。
这种技术适用于大多数细胞类型,可以有效地抑制基因的表达。
3. 基因敲除质粒转染基因敲除质粒转染是一种常见的基因敲除策略。
通过构建含有目标基因编码序列的质粒,并将其转染到细胞中,使细胞内产生反义RNA或RNA干扰,从而抑制目标基因的表达。
这种方法简单易行,适用于多种细胞类型,但效率相对较低。
4. 靶向核酸酶介导的基因敲除除了CRISPR/Cas9系统外,还有一些其他的核酸酶可以介导基因敲除。
例如,ZFN (锌指核酸酶) 和TALEN (转录激活样效应核酸酶) 等。
这些核酸酶通过特异性结合到目标DNA序列上,诱导DNA双链切割,并引发细胞内的DNA修复机制。
这种方法可以实现基因敲除,但需要设计和合成特异性的核酸酶,相对来说比较繁琐。
除了上述介绍的几种常见的细胞基因敲除策略外,还有许多其他的方法,如病毒介导的基因敲除、基因敲除动物模型等。
这些方法在研究基因功能和调控机制、治疗遗传疾病等方面发挥了重要的作用。
在进行细胞基因敲除实验时,需要注意以下几点。
circrna_沉默实验步骤 -回复

circrna_沉默实验步骤-回复沉默实验是一种用于研究环状RNA(circRNA)功能的实验方法。
circRNA是一类非编码RNA,其在细胞中广泛表达,并能够参与调控基因表达。
沉默实验可以通过抑制circRNA的表达,进一步研究其在细胞中的功能和调控机制。
本文将详细介绍circRNA沉默实验的步骤和操作方法。
第一步:确定circRNA的靶点在进行circRNA沉默实验之前,首先需要确定所研究的circRNA的靶点。
可以通过生物信息学分析,结合公共数据库和文献报道,确定某个circRNA与哪些基因存在靶向关系。
这一步是研究circRNA功能和调控机制的关键,因为确定了circRNA的靶点,才能有针对性地进行沉默实验。
第二步:选择沉默实验方法目前常用的circRNA沉默实验方法有RNA干扰(RNA interference,RNAi)和转座子介导的RNAi两种。
RNAi是通过引入特异性靶向circRNA的小分子干扰RNA,从而抑制circRNA的表达。
转座子介导的RNAi则是将干扰RNA的基因序列插入适当的载体中,通过转染的方式将其导入细胞,实现对circRNA的沉默。
选择具体的实验方法需要根据实验室的设备和技术优势进行权衡。
无论选择哪种方法,都需要将沉默载体构建好,并提前准备好所需的实验材料和试剂。
第三步:沉默载体构建沉默载体的构建是circRNA沉默实验的核心环节。
在RNAi实验中,通常选择合成特异性靶向circRNA的小干扰RNA(small interfering RNA,siRNA),并将其插入干扰载体中。
siRNA的设计需要根据circRNA的序列进行,确保其具有高度特异性和高效的沉默效果。
在转座子介导的RNAi实验中,需要将沉默RNA基因序列插入适当的载体中。
载体的选择需要考虑到转染效率和稳定性等因素。
常用的载体包括慢病毒、腺病毒和质粒等。
第四步:细胞培养和转染在进行circRNA沉默实验之前,需要将研究对象的细胞进行培养和扩增。
基因敲除的方法就这么些,2分48秒看清楚

基因敲除的方法就这么些,2分48秒看清楚现在的基因组编辑工具本身并不能改变基因序列的排布。
它们能做的只是在基因组序列上制造几个缺口。
而真正改变基因组序列,编辑基因组序列的是细胞本身。
而我们能做的,是最大限度地诱导细胞按照我们的意愿完成基因组编辑。
那么,到底是怎么个诱导方式呢?咱们从基因敲除说起~上视频~不愿意看视频的同学也可以看后面的文本哦~如果我们要进行基因敲除。
最简单但也最放任自流的方式是,在我们需要敲除的基因片段上制造一个切口,让细胞自行进行修补。
这个过程中,细胞有可能在缺口上缺失几个碱基,插入几个碱基。
如果缺失或插入的碱基不是3的倍数,这个基因就有可能因为移码突变而出现功能缺失,从而实现敲除。
我们从这些经过处理的细胞中随机挑选出一些,分别进行单个细胞的培养和扩增。
这些由单个细胞扩增形成的细胞群落被称作一个单克隆。
用western、PCR等方法分析这些单克隆的基因表达情况,就可以找到那些目的基因被敲除的细胞群落,也就是我们需要构造的基因敲除单克隆细胞系。
这些单克隆细胞系的目的基因功能被缺失掉了,但是编码基因的序列仍然被留在基因组里,只不过被破坏了,无法正常表达。
另外一个更可控的方式是我们在这个基因序列距离较远的两个位点上分别制造一个切口,两个切口间的大片段极有可能因此被删除。
我们可以筛选出这些发生大片段删除的单克隆。
它们的目的基因几乎可以从基因组中完全删除。
但是这种方法需要挑更多的单克隆进行验证,因为两个位点同时被切割的概率比单位点被切割的概率更低。
前面讲的这两种方法都需要随机挑取单克隆进行验证。
这个有点靠运气。
有一个办法可以让我们的单克隆挑选更有目的性,那就是给咱们被编辑细胞加标记。
比如gfp荧光蛋白?抗性基因等等,在它们的两边加上目的基因序列的同源臂做成标签序列。
把它们放进细胞的同时,也放进去一些基因组编辑工具,在目的序列上制造缺口,增加同源重组的概率,把这些标签给整合进去目的基因序列里。
动物中敲除某种细胞的方法

动物中敲除某种细胞的方法动物中敲除某种细胞是一种常见的研究方法,常用于生物医学研究、基因功能研究等领域。
它能够帮助科研人员深入了解特定细胞在生物体内的功能和影响,为人类健康和疾病治疗的相关研究提供重要的参考。
本文将介绍动物中敲除某种细胞的方法,包括CRISPR/Cas9技术、siRNA敲除、基因突变体筛选等等,希望对相关领域的科研人员有所帮助。
一、CRISPR/Cas9技术CRISPR/Cas9是一种革命性的基因编辑技术,可以用于敲除动物体内特定的基因。
通过设计合适的CRISPR引物和引导RNA,将Cas9蛋白靶向到目标基因上,发生双链断裂并引起基因敲除。
在实验动物中,通过注射CRISPR/Cas9蛋白或质粒构建的介导RNA来实现特定细胞的基因敲除,从而研究目标细胞的功能和影响。
二、siRNA敲除siRNA是一种特异性靶向特定基因的小分子RNA分子,可以在细胞内引起特定基因的沉默和敲除,从而研究目标细胞的功能和影响。
在动物实验中,可以通过注射siRNA分子或构建载体来实现特定细胞内基因的沉默和敲除,为相关研究提供重要的实验支持。
三、基因突变体筛选利用化学或物理诱发基因突变来筛选特定细胞的敲除变异体,是一种常见的方法。
通过暴露动物体内的细胞于化学诱变剂或放射线等物理因素,诱发基因突变,并筛选出特定细胞的敲除变异体,从而研究其功能和影响。
四、基因敲除动物模型基因敲除动物模型是一种通过基因敲除技术构建的动物模型,可用于研究特定细胞的功能和影响。
通过设计合适的基因敲除策略和动物繁殖技术,可以得到特定细胞敲除的动物模型,为相关研究提供可靠的实验数据。
动物中敲除某种细胞的方法包括CRISPR/Cas9技术、siRNA敲除、基因突变体筛选等多种技术手段。
这些方法在生物医学研究、基因功能研究等领域具有重要的应用价值,为相关研究提供了重要的技术支持和理论指导。
希望本文内容能够对相关领域的科研人员有所启发和帮助。
circrna筛选方法

circrna篩選方法标题:circRNA筛选方法:探索非编码RNA的研究策略在生物科学领域,环状RNA(circRNA)作为一种重要的非编码RNA分子,近年来引起了广泛关注。
circRNA具有稳定的环状结构,参与基因表达调控、疾病发生等多种生物过程。
为了深入研究circRNA的功能和机制,筛选高效、准确的circRNA成为科研工作的重要一环。
本文将详细介绍几种常见的circRNA筛选方法。
一、基于生物学信息的筛选方法1.生物信息学分析:通过高通量测序技术获得转录组数据,利用生物信息学软件进行circRNA的预测和筛选。
常用的软件有CIRCexplorer、find_circ 等。
此方法可快速发现大量候选circRNA,为进一步实验验证提供依据。
2.基因组浏览器:利用基因组浏览器(如UCSC Genome Browser)对已知circRNA的基因组位置进行检索,分析其与疾病相关的基因、miRNA等分子间的相互作用,从而筛选出具有潜在研究价值的circRNA。
二、基于实验技术的筛选方法1.Northern Blot:通过Northern Blot技术检测特定circRNA在组织或细胞中的表达水平,从而筛选出感兴趣的目标circRNA。
此方法具有较高的灵敏度和准确性,但操作复杂、成本较高。
2.实时荧光定量PCR(qPCR):利用qPCR技术对候选circRNA进行定量分析,筛选出表达量较高或具有显著差异的circRNA。
qPCR操作简便、成本低,适用于大量样本的筛选。
3.RNA干扰(RNAi):通过RNAi技术敲低候选circRNA的表达,观察细胞或生物体的表型变化,从而筛选出具有生物功能的circRNA。
三、基于生物功能的筛选方法1.功能缺失实验:通过基因敲除或基因编辑技术抑制circRNA的表达,观察细胞或生物体的生理、病理变化,筛选出具有潜在功能的circRNA。
2.功能获得实验:通过过表达circRNA,观察细胞或生物体的表型变化,筛选出具有生物功能的circRNA。
环状RNA在胰腺癌中的研究进展

环状RNA在胰腺癌中的研究进展纪道林;徐艺;冷开明;孙学英;崔云甫【摘要】胰腺癌的发病率和死亡率呈逐年上升趋势,其治疗主要为外科手术、放疗与化疗相结合的综合治疗方式。
由于胰腺癌早期缺乏特异性临床表现,多数患者确诊时已处于进展期,治疗效果欠佳,因此,寻求有效的早期诊断方法及治疗策略成为迫切需求。
随着对胰腺癌发生和发展的具体分子机制研究的不断深入,环状RNA(circular RNA,circRNA)成为胰腺癌早期诊断特异性肿瘤标志物及分子靶向治疗研究的热点,如利用高通量测序技术筛查circRNA在胰腺癌细胞株及病理组织中的表达谱,使得人类对circRNA与胰腺癌之间的关系有了更加深入的了解。
本文将对近些年发现的在胰腺癌中呈特异性表达的circRNA及其与胰腺癌发生和发展之间具体作用机制方面的研究作总结,并对其在胰腺癌诊断和治疗中的应用前景作展望。
【期刊名称】《中华胰腺病杂志》【年(卷),期】2019(34)4【总页数】7页(P302-308)【关键词】胰腺肿瘤/诊断;胰腺肿瘤/治疗;RNA;基因表达调控;微RNAs;生物标记,肿瘤【作者】纪道林;徐艺;冷开明;孙学英;崔云甫【作者单位】哈尔滨医科大学附属第二医院胆胰外科,黑龙江哈尔滨150086;哈尔滨医科大学附属第一医院肝脾外科中心,黑龙江哈尔滨150007【正文语种】中文【中图分类】R735.9;R730.4环状RNA(circularRNA,circRNA)是一类参与转录和转录后基因表达调节的非编码RNA(noncodingRNA,ncRNA)[1],与微小RNA(microRNA,miRNA)、长链非编码RNA(1ong non-coding RNA,lncRNA)一起构成了竞争性内源RNA(competing endogenous RNA,ceRNA)家族,是RNA研究领域的热点。
近年来多种circRNA被证实与胰腺癌的发生及发展有关,本文就circRNA与胰腺癌关系的研究进展作一综述。
thermo fisher scientific细胞质核分离 circrna具体步骤

Thermo Fisher Scientific 细胞质核分离circRNA 具体步骤一、实验准备1.实验器材:离心管、离心机、移液器、细胞培养皿/瓶、液氮罐。
2.试剂:细胞分离缓冲液、核染料、RNA酶抑制剂、无RNA酶水。
3.细胞系:选择适合的细胞系进行实验,circRNA 在不同细胞系中的表达可能存在差异。
二、实验步骤1.细胞培养:将细胞接种于适当大小的细胞培养皿或瓶中,根据细胞生长特性,选择适宜的培养条件进行培养。
2.细胞分离:当细胞生长至适宜密度时,使用细胞分离缓冲液将细胞分离为胞质和核两部分。
离心后,弃去胞质部分,保留核部分。
3.核染色:使用核染料对核部分进行染色,以便后续观察和分离。
4.circRNA 提取:使用RNA 酶抑制剂处理核染色后的样本,以防止circRNA 被降解。
随后,使用无RNA 酶水将circRNA 从核中洗脱。
5.circRNA 检测:通过qPCR、Northern blot 或其他相关技术对提取的circRNA 进行检测和定量。
circRNA 的检测通常涉及使用特定的引物或探针,以特异性和定量地检测circRNA 的表达。
6.数据分析和解释:对circRNA 的表达数据进行统计分析,比较不同处理或条件下的表达差异。
通过生物学功能分析,探讨circRNA 在细胞中的作用和潜在机制。
三、注意事项1.实验操作需在无菌条件下进行,以防止污染。
2.为确保实验结果的准确性,建议进行生物学重复和/或技术重复。
3.在提取circRNA 时,应确保使用RNA 酶抑制剂,以避免circRNA 被降解。
4.在数据分析过程中,应注意排除假阳性或假阴性结果,确保数据的可靠性。
5.对于涉及伦理和法律的问题,应遵循相关规定,确保实验的合法性和道德性。
通过以上步骤,可以成功地进行Thermo Fisher Scientific 细胞质核分离circRNA 的实验操作。
请注意,实验的具体步骤可能会因实验条件、细胞类型和circRNA 的特性而有所不同。
基因敲除技术的原理、方法和应用

基因敲除技术的原理、方法和应用2010-01-24 17:03:43 来源:易生物实验浏览次数:6302 网友评论 0 条1.基因敲除概述2.实现基因敲除的多种原理和方法:2.1.利用基因同源重组进行基因敲除 2.2利用随机插入突变进行基因敲除。
2.3.RNAi引起的基因敲除。
3.基因敲除技术的应用及前景4.基因敲除技术的缺陷关键词:基因敲除1.基因敲除概述:基因敲除是自80年代末以来发展起来的一种新型分子生物学技术,是通过一定的途径使机体特定的基因失活或缺失的技术。
通常意义上的基因敲除主要是应用DNA同源重组原理,用设计的同源片段替代靶基因片段,从而达到基因敲除的目的。
随着基因敲除技术的发展,除了同源重组外,新的原理和技术也逐渐被应用,比较成功的有基因的插入突变和iRNA,它们同样可以达到基因敲除的目的。
2.实现基因敲除的多种原理和方法:2.1.利用基因同源重组进行基因敲除基因敲除是80年代后半期应用DNA同源重组原理发展起来的。
80年代初,胚胎干细胞(ES细胞)分离和体外培养的成功奠定了基因敲除的技术基础。
1985 年,首次证实的哺乳动物细胞中同源重组的存在奠定了基因敲除的理论基础。
到1987年,Thompsson首次建立了完整的ES细胞基因敲除的小鼠模型 [1]。
直到现在,运用基因同源重组进行基因敲除依然是构建基因敲除动物模型中最普遍的使用方法。
2.1.1利用同源重组构建基因敲除动物模型的基本步骤(图1):a.基因载体的构建:把目的基因和与细胞内靶基因特异片段同源的DNA 分子都重组到带有标记基因(如neo 基因,TK 基因等)的载体上,成为重组载体。
基因敲除是为了使某一基因失去其生理功能,所以一般设计为替换型载体。
b.ES 细胞的获得:现在基因敲除一般采用是胚胎干细胞,最常用的是鼠,而兔,猪,鸡等的胚胎干细胞也有使用。
常用的鼠的种系是129及其杂合体,因为这类小鼠具有自发突变形成畸胎瘤和畸胎肉瘤的倾向,是基因敲除的理想实验动物。
如何用CRISPR技术敲低(敲除)内源circRNA-fwc
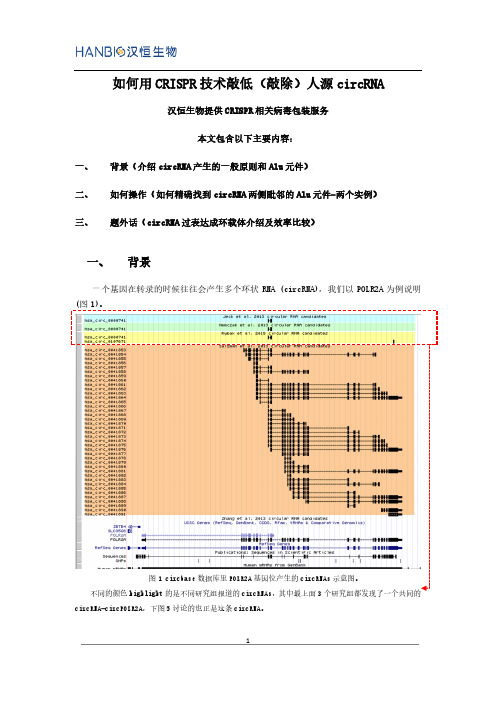
如何用CRISPR技术敲低(敲除)人源circRNA汉恒生物提供CRISPR相关病毒包装服务本文包含以下主要内容:一、背景(介绍circRNA产生的一般原则和Alu元件)二、如何操作(如何精确找到circRNA两侧毗邻的Alu元件-两个实例)三、题外话(circRNA过表达成环载体介绍及效率比较)一、背景一个基因在转录的时候往往会产生多个环状RNA (circRNA),我们以POLR2A为例说明(图1)。
circRNA-circPOLR2A,下图5讨论的也正是这条circRNA。
通常情况下circRNA的产生遵循以下原则(图1和2):图2环状RNA 的基因组特征.◆中间的外显子最容易成环,通常包含2~3个外显子;如果是一个外显子成环,则其长度会明显长于平均长度;◆成环外显子的两侧内含子较大(~5倍于随机内含子);◆通常,circRNA两侧毗邻内含子含有互补的序列,如短间隔重复序列(SINE)家族(图4)的Alu元件(the inverted repeated Alu elements, IRAlus)等,且距离成环外显子边界距离相当(图2和3)。
图3 POLR2A基因位产生的其中一个circRNA-circPOLR2A及其毗邻的IRAlus示意图。
Zhang, et al. (2014). "Complementary sequence-mediated exon circularization." Cell 159(1):134-147.Alu元件是灵长类基因组中一组散在分布的相关序列,每个长约300bp。
单个Alu元件成员的每个末端上有Alu限制酶的切割位点,并由此命名。
在灵长类的基因组中存在着大量不同种类的Alu元件。
事实上,Alu元件是人类基因组中丰度最高的转座元件。
特别地,Alu元件在人的基因组中占比11%,在影响基因表达等方面影响广泛。
Alu元件基本组成模式如下:Part A--AAAAATACAAAAAA--Part B-polyA,其中A和B序列往往类似。
基因敲除细胞系介绍及应用案例

基因敲除细胞系介绍及应用案例通过病毒法,化学转染法或电转法将gRNA和Cas9转入细胞中,根据转染方法不同进行不同时长的药筛,药筛完成后挑选单克隆培养。
选择不同的克隆分别进行靶位点扩增及测序验证,筛选出移码基因敲除的阳性克隆。
基因敲除细胞系应用案例:中国仓鼠卵巢(CHO)细胞被用作生物工厂,用于生产一系列重组治疗蛋白,包括单克隆抗体和Fc融合蛋白。
宿主细胞蛋白(HCP)是必须从治疗制剂中去除的杂质,因为它们具有潜在的免疫原性风险。
虽然在典型的下游净化过程中,大多数HCP杂质被有效去除,但清除少量存在的HCP仍然是一个挑战。
利用CRISPR/Cas9系统建立Anxa2和Ctsd基因敲除CHO细胞系,并证实了细胞裂解液中HCP完全消除。
在培养过程中,所有的基因敲除细胞系都显示出与野生型对照相似的生长和活力。
因此,敲除非必需基因可以减少重组治疗蛋白生产中HCP的污染。
(a)sgRNA打靶Anxa2基因的4号外显子。
对序列进行双向分析。
(b) sgRNA打靶Ctsd基因2号外显子。
对序列进行双向分析。
用SDS-PAGE和WB分析鉴定CHO基因敲除细胞株的蛋白表达。
(a) Anxa2基因敲除细胞系。
(b) Ctsd基因敲除细胞系。
细胞培养上清液(47μg蛋白)和细胞裂解液(20μg蛋白)经4-20%SDS-PAGE分析。
CBB染色检测总蛋白。
利用各自的捕获和检测抗体对每个蛋白质进行WB分析。
星号表示非特定波段。
双星号表示组织蛋白酶D的片段在蛋白质表达分析中,没有观察到CHO-K1(WT)细胞系中的细胞培养上清液中的膜联蛋白A2和组织蛋白酶D。
对贴壁的CHO-K1细胞进行静态培养,在培养过程中没有物理应力诱导细胞裂解,这表明CHO细胞分泌的这些蛋白质数量相当少。
此外,在对Anxa2和Ctsd基因敲除细胞系的蛋白质表达分析中,没有观察到任何截短的HCP。
看过这篇文章的人,做circRNA过表达都成功了(下)

看过这篇文章的人,做circRNA过表达都成功了(下)昨天湖人为大家支了五招,讲怎么构建circRNA过表达载体,今天就接着介绍怎么严谨地验证是否过表达成功。
构建好载体之后,自然是验证能否成功(正确)过表达了,常用的思路是瞬时转染细胞系,24-48h后取样做RT-qPCR检测,以2-△△Ct法计算表达差异。
而对于circRNA过表达来讲,因为其准确成环难度大,转录后还要经过剪切环化,仅仅做常规的RT-qPCR计算表达差异是不够的,而是一定要将PCR的产物进行电泳,检测是否单一条带,条带大小是否正确,并且进行sanger测序,比对PCR产物的序列是否和基因一致,确保环化位点处准确成环,没有错误添加进酶切位点或缺失序列。
此处还有一个难点,circRNA由于其反向剪切后环化位点(Splice Junction)在基因组上mapping是前后相反的,所以检测circRNA的特异引物一般设计为Divergent primers,这和用来检测线性基因的Convergent primers相对应。
多数文章里描述Divergent primers是相反方向设计,上下游引物分别在环化位点的两端,使扩增产物内包含环化位点。
但这样设计的引物具体位置和扩增特异性问题大都语焉不详,相信很多同学也发现这样设计引物总是有非特异条带或者扩增无条带的情况,稀里糊涂的做就不可取了。
这里的重点就是,针对检测circRNA过表达,需要同时设计两种Divergent primers,产物要经电泳确认大小,并且进行sanger测序。
首先来看一下引物设计,第一种普通的Divergent primers可参考大部分文章(如环状RNA引物设计链接),一般设计成背对背,使环化位点包含在产物中间,除了需要将序列首尾拼凑成环化后的形式外,和普通的引物设计基本一样的思路。
设计出来大概是这样的:对应在外显子上是这样的,此时扩增产物是绿色框选部分,中间包含一个环化位点(红色标记)。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
circRNA基因敲除细胞株的经典方法及案例
circRNA敲降(干扰)
在研究circRNA功能的方法中,最经典的抑制circRNA的方法是通过RNAi的方式(shRNA)进行敲降。
为了避免影响到mRNA,设计方案时需将干扰序列设计在反向剪接位点(BSS)处。
源井生物通过设计高效的shRNA,用慢病毒法将干扰载体转入细胞中,根据最佳药筛浓度对细胞进行药物筛选,直到对照组细胞全部死亡,获得circRNA敲降的稳定细胞株。
应用案例:
用siRNA进行敲降后,通过检测细胞增殖凋亡情况,说明circ-HIPK3敲除后抑制细胞增殖。
首先设计三组实验,分别针对HIPK3 mRNA线性转录本、
circ-HIPK3环状转录本和两种转录本共有部分设计siRNA,并在HEK-293 T细胞系上验证设计的siRNA只干扰相应的转录本。
利用增殖凋亡检测试剂盒:CCK-8和EdU进行细胞增殖凋亡检测,结果显示HIPK3 mRNA敲降后不明显影响细胞增殖,而circ-HIPK3敲降后,会明显抑制细胞增殖。
参考文献:
Zheng, Q., Bao, C., Guo, W., Li, S., Chen, J., Chen, B., ... & Liang, L. (2016). Circular RNA profiling reveals an abundant circHIPK3 that regulates cell growth by sponging multiple miRNAs. Nature communications, 7(1),
1-13.
circRNA过表达
circRNA过表达一直有成环效率低,容易错配成环等难点。
通过优化侧翼成环框架,如成环元件、QKI等RBP的结合位点,使circRNA准确高效环化。
过表达后仍需要检测是否成功成环,以及线性mRNA是否表达。
为了研究一种新环状RNA载体表达系统的成环效率,选择小鼠circRtn4环状基因在多种细胞系(包括Hela,N2a,HEK293)中进行表达验证。
根据不同细胞系中进行的RT-QPCR 实验数据显示,新载体系统pCircRNA-DMo-Rtn4成环效率在几种不同的细胞系中均比普通的载体系统(pCircRNA-BE-Rtn4)要高效得多。
circRNA的检测与鉴定
Northern Blotting是检测circRNA的金标准,探针通常跨反向剪接位点设计。
但由于Northern Blotting需要的circRNA量非常大,耗时间精力,而且探针一般是放射性标记,操作上比较困难。
常用的检测方案还是用RT-PCR或者是RT-QPCR,引物设计在反向剪接位点两端。
