微生物流程图
土壤微生物的分离纯化 实验:微生物的分离、培养和菌种保藏

思考题
❖ 什么叫无菌操作?分离放线菌和真菌为什么 需要分别加重铬酸钾和链霉素?
❖ 平板培养时为什么要把培养皿倒置? ❖ 如何确定平板上某单个菌落是否为纯培养?
Байду номын сангаас
实验七
结束
下周实验
理化因素对微生物的影响
(实验三十二、实验三十四、实验三十八)
每毫升样品中微生物细胞数=
每皿菌落平均数 × 稀释倍数 × 1/取样体积数
混匀 凝固
28~30℃ 培养
图7-4 混合法流程图
实验程序 Ⅳ——平板涂抹法(涂布法)
❖ 每人取无菌培养皿1付(每个同学做一只)。在皿底贴上标签,注明 分离菌名、稀释度、组别、班级。
❖ 取已熔化的高氏培养基,分别倒15-20ml入培养皿中,铺平,制成 平板。
养皿里。 ❖ 3.取已熔化的在水浴锅中保温500C左右的马铃薯培养基(PDA培养基),
分别倒约15-20ml入上述培养皿中,轻轻转动培养皿,使菌液、培养基 充分混匀铺平,放在平坦的桌面上,凝固后,倒置于28~300C恒温培养 5~6d。观察真菌菌落形态以及计数 菌落。并计算出每g土壤中真菌的数 量。即用某一培养皿内真菌的菌落数乘以该培养皿接种液的稀释倍数即 得。 (见图7-4)
挑取单个菌落接种于斜面培养基 上,如果不纯,再移植纯化,最 后得到纯培养。
实验程序Ⅵ——四大类微生物菌落形态的比较和
识别
❖ 区分和识别各大类微生物通常包括菌落形态(群体形态) 和细胞形态(个体形态)两方面的观察。
❖ 菌落形态: 菌落表面湿润:细菌——薄而小,酵母菌——厚而大。 菌落表面干燥:放线菌——密而小,霉菌——松而大。
淀粉琼脂培养基 马铃薯蔗糖培养基
❖ 无菌水:带有玻璃珠装有99mL无菌水三角瓶
高中生物选修一-第一章无菌操作技术实践-第1章 第2节

第二节分离特定的微生物并测定其数量1.认识各种微生物形成的菌落特征。
2.学会利用涂布平板法分离、培养微生物,并测定其数量。
(重难点)3.掌握利用选择培养基分离特定微生物的方法。
(重点)1.分离原理在实验室中利用选择培养基可以筛选和分离出某种微生物,然后在高度稀释的条件下,在固体培养基上培养该微生物形成的单个菌落,即为纯培养物。
2.分离方法(1)平板分离法:包括涂布平板法和混合平板法。
能将单个微生物分离和固定在固体培养基表面或里面,每个孤立的活微生物体经过生长、繁殖均可形成单个菌落。
(2)选择培养分离:科学家针对某种微生物的特性,设计一个特定的环境,使之特别适合这种微生物的生长,而抑制其他微生物的生长。
这种通过选择培养进行微生物纯培养分离的技术,称为选择培养分离。
(3)根据菌落特征初步分离:①菌落的含义:是指单个微生物在适宜的固体培养基表面或内部生长、繁殖到一定程度,形成肉眼可见的、有一定形态结构的子细胞生长群体。
②菌落特征:不同微生物在特定培养基上生长形成的菌落一般都具有稳定的特征,如菌落的大小、形状、边缘特征、隆起程度、颜色等,这些可以成为对微生物进行分类、鉴定的重要依据。
[合作探讨]探讨1:在琼脂固体培养基上长出的单个菌落含有多种细菌吗?提示:标准的单个菌落中只含有一种细菌。
探讨2:如果要分离厌氧菌,应在什么条件下培养?提示:在无氧条件下培养。
探讨3:在选择培养分离微生物的过程中,“选择培养”的培养基按物理性质划分属于什么培养基?为什么?提示:固体培养基。
便于选择、分离所需要的微生物。
[思维升华]1.涂布平板法与混合平板法的比较(1)原理:根据不同微生物的特殊营养要求或其对某化学、物理因素的抗性不同而制备培养基。
(2)方法:①在培养基中加入某种化学物质。
例如,加入青霉素可以分离出酵母菌和霉菌;加入高浓度的食盐可以得到金黄色葡萄球菌。
②改变培养基中的营养成分。
例如,缺乏氮源时可以分离固氮微生物;石油作为唯一碳源时,可以分离出能消除石油污染的微生物;用含无机碳源的培养基分离自养型微生物。
高中生物第1章微生物培养技术第3节测定微生物的数量学案中图版选修1

第三节测定微生物的数量一、测定微生物数量的方法测定微生物数量的方法可分为两类:直接计数法和间接计数法.前者常用的是显微镜直接计数法,这种方法是先将待测样品制成悬液,然后取一定量的悬液放在显微镜下进行计数。
该方法适用于纯培养悬浮液中各种单细胞菌体的计数。
后者最常用的是稀释平板计数法,需要将待测样品配制成均匀的系列稀释液并使其均匀分布于平板中的培养基内,经培养后统计培养基中出现的菌落数,从而推算出样品中的活菌数。
利用血球计数板测出的菌体数与平板计数法相比哪个多些?【提示】血球计数板测出的是全部菌体数,而平板计数法只能测出活菌数,而且比实际数偏少。
二、间接计数法的实验设计1.制备土壤稀释液取土壤表层5~10 cm处的土样.准确称取1 g土样,放入盛有99 mL无菌水的锥形瓶中,振荡20 min后制成102倍的稀释液.然后进行系列稀释,得到103、104、105、106倍的系列稀释菌液。
2.取样及倒平板3.培养4.观察记录(1)计数时对于细菌、放线菌以每个培养皿内有30~300个菌落为宜,霉菌以每个培养皿内有10~100个菌落为宜。
(2)计算每克样品菌数公式为:每克土壤样品菌数=错误!×稀释倍数.预习完成后,请把你认为难以解决的问题记录在下面的表格中问题1问题2问题3问题4一、微生物生长量的测定1.测重量法干重法:将单位体积待测的培养液离心后,用清水洗涤1~5次,放入干燥器中加热干燥,加热温度一般在100~105 ℃之间,再称重。
以细菌为例,一个细胞一般重约10-12~10-13g,也可在较低温度(如40 ℃)下进行真空干燥。
湿重法:将一定量的菌悬液离心或过滤,得到菌体,反复洗涤后称湿重,干重一般为湿重的20%~25%。
2.计数法(1)直接计数法这种方法是利用特定细菌计数板或血球计数板,在显微镜下计算一定容积里样品中微生物的数量。
此法的缺点是不能区分死菌与活菌。
计数板是一块特制的载玻片,上面有一个特定的面积1 mm2和高0。
沙门氏菌检验详细流程图

1 mL+SC10 mL 36 ℃±1 ℃,18 h~24 h
BS
XLD(或HE、科玛嘉显色培养基)
36 ℃±1 ℃,40 h~48 h
36 ℃±1 ℃,18 h~24 h
挑取可疑菌落
TSI,赖氨酸,NA, 靛基质, 尿素 (pH 7.2), KCN
商 品 化 生 化 H2S+靛基质- 尿素- H2S+靛基质+ 尿 H2S-靛基质-尿素 反应结果与左
为了最大可能的检出沙门氏菌,原则上必须使用 两种以上选择性分离培养基。
进口-SS 进口-SS 国产-SS 进口-HE 国产-HE 进口-XLD 国产-XLD 进口-BS 国产-BS CHRO显色 国产-DHL 国产-WS TSA(对照)
65.3 81.0 52.0 83.2 80.0 83.0 86.2 106.6 77.7 68.4 98.4 71.6 100.0
初筛微生 物
沙门氏菌
柠檬酸杆 菌属
变形杆菌
菌落颜色
紫色(直径约 1mm)
蓝色(直径约 1mm)
无色或被抑 制
特异性 灵敏度
89% 100%
---
---
(铜绿假单胞菌也呈紫红色, 可以通过前增菌排除。所以推 荐在检测中的前增菌用TTB。 粉红色、深红色、干燥菌落、 边缘锯齿状等 均非沙门氏菌菌 落。)
被分成2500多个血清型。
Vi抗原 O抗原 H抗原
沙门氏菌命名与书写方式
目前的命名方法规定:
肠道沙门菌肠道亚种(亚种Ⅰ)给予专用名,并采用 标本分离地址的地名。菌名的第一个字母需大写,且 亚种Ⅰ的所用菌名不能用斜体。例如:肠道沙门菌鼠 伤寒血清型应书写为Salmonella enterica subsp.enterica serotype Typhimurium,但在实际工作中,可简写为S. Typhimurium。
微生物工程实验报告

实验1 培养基的配制和灭菌一、目的要求1.了解培养基的配制原理和方法,掌握其配制过程。
2.了解几种灭菌方法,掌握高压蒸汽灭菌法的原理及其使用方法。
3.熟悉菌种分离筛选的前期准备工作及操作方法。
4.每2人一组,每人一份实验报告。
二、实验材料1.药品:酵母粉、蛋白胨、氯化钠、琼脂、葡萄糖、KNO3、MgSO4•7H2O、FeSO4•7H2O、1N HCl、1N NaOH2.设备:高压蒸汽灭菌锅、紫外线灭菌灯、75%酒精棉。
3.材料:天平、药匙、电炉、pH 试纸、烧杯、量筒、5mL、10mL 注射器、玻璃棒、试管、培养基、吸管、移液器(1mL 0.2mL)、牛皮纸、线绳、标签等。
三、实验内容(一) 培养基的配制1.培养基的配制方法和步骤1)称量:按照配方正确称取所需药品放于烧杯中。
2)溶化:在烧杯中加入所需水量,玻棒搅匀,加热溶解。
3)调pH 值:用1N NaOH 或1N HCl 调pH,用pH 试纸对照。
4)加琼脂溶化:加热过程要不断搅拌,可适当补水。
5)分装:注意不要污染试管塞或纱布。
6)包扎成捆:用记号笔注明何种培养基。
7)灭菌:在高压锅中, 115 °C 高温灭菌30min。
2.培养基的配制A.富集培养基(液体):海水2216E 液体培养基(g/L):蛋白胨5.0,酵母粉1.0,海水1L,pH 8.0。
取50mL 分装250mL 三角瓶后,8 层纱布封口,外包1层牛皮纸,121℃,高温灭菌20min。
B. 2216E 斜面(固体):海水2216E 液体培养基中加入2%的琼脂粉,加热溶化琼脂,每支试管内。
加入3mL,121℃高温灭菌20min,灭菌后摆斜面。
C. 无菌生理盐水和保种液:生理盐水: NaCl 0.85% 蒸馏水配制。
每支试管加入5mL,盖上试管帽、包扎、121°C 高温灭菌20min。
保种液:生理盐水,加25%甘油D. 碳源优化培养基:以海水2216E 液体培养基(蛋白胨0.5%,酵母粉0.1%,磷酸铁0.01%,人工海水,pH7.6)为基础,碳源分别是:葡萄糖、蔗糖、甘油、柠檬酸钠、乳糖,每种碳源的添加量为1%。
医院感染控制工作流程图

医院感染控制工作流程图一、多重耐药菌监测、报告、处置流程图二、中心静脉导管相关血液感染(CR-BSI)监测标准操作流程图三、呼吸机相关性肺炎监测标准操作流程图四、导尿管相关泌尿道感染(CA-UTI)监测标准操作流程图五、医院感染病例监测处理流程图六、手术部位感染监测规范及操作流程七、洗手流程图用水打湿双手取适量洗手液涂抹双手所有皮肤掌心对掌心揉搓手指交叉,掌心对手背揉搓手指交叉,掌心对掌心揉搓手指互握,一手手指背部放于另一只手手掌中,揉搓手指拇指在掌中揉搓指尖在掌心中揉搓用水冲洗用一次性毛巾彻底擦干用毛巾关水龙头你的手是安全的手八、手消毒流程图取适量产品于于掌心中并涂抹双手至所有皮肤掌心对掌心揉搓手指交叉,掌心对手背揉搓手指交叉,掌心对掌心揉搓手指互握,一手手指背部放于另一只手手掌中,揉搓手指拇指在掌中揉搓指尖在掌心中揉搓一旦干燥,你的手是安全的九、ICU呼吸机相关肺炎监测流程图十、ICU导尿管相关泌尿道感染监测流程图十一、暴露级别的评估流程图十二、职业暴露后的应急处理报告流程图十三、确定HIV 暴露源头严重程度流程图根据暴露级别和暴露源病毒载量水平预防性用药的推荐处理方案(表)表 职业暴露后预防性用药的处理方案*PEP:HIV/AIDS 的职业暴露后预防(Postexposure Prophylaxis ,PEP )应包括急救、对暴露级别的评估、暴露源严重程度的评估、预防性用药的推荐处理方案、报告与保密。
注:(1)基本用药程序:两种逆转录酶制剂,使用常规治疗剂量,AZT (每次200mg ,每日3次,或每次300mg ,每日2次)+拉米夫定(每次150mg 每日2次),连续使用28d 。
或双汰芝(AZT 与3TC 联合制剂)300mg/次,每日2次。
(2)强化用药程序:基本用药程序加一种蛋白酶抑制剂,茚地那韦(800mg ,tid ,饭前1h 及饭后2h 服用),均使用常规治疗剂量。
如病人无血源性传染病,则不需要进行任何处理,仅密切观察。
微生物菌种毒株和样本标准操作程序与流程 (2)

病原微生物菌(毒)种与样本的标准操作规程流程1.目的:对本实验室菌种、毒种的申购、保存、保管、领用、处理等各个环节实行有效的监督控制,防止意外事故发生,确保疾病预防与控制检验业务及科研教学工作。
2.适用范围及菌种、毒种专职管理人员适用于本科微生物实验室菌种、毒株的管理;菌种、毒种专职管理人员:于瑞芳、侯恩言3、职责①微生物实验室负责菌、毒种的出入库保管、保存及处理等日常管理。
②科室指定2名菌种、毒种库管理人员承担菌、毒种日常管理。
③科室负责人负责一、二类菌、毒种的出入库与向上级索取及对下级发送的审核。
④技术管理层批准本实验室一、二类菌、毒种的出入库与向上级索取及对下级发送的审批。
4.工作程序⑴报送及入库①当微生物实验室检出菌、毒株应及时报送市疾病预防控制中心。
②新发现的菌、毒种,要做好原始记录,逐级报送进行复核确认,报送时须2人参加。
③一、二类菌、毒种入库前,科室审核,技术管理层批准后入库④个人不得擅自保留菌、毒种,必须由科室进行统一编号、登记入库管理⑤菌、毒种入库时, 2名菌、毒种保管人员须认真做好菌毒种的编号、登记工作。
⑵日常管理①保管人员由于瑞芳、侯恩言2名检验人员组成。
②菌、毒种入库时,保管人员应及时验收,统一编号,填写《菌、毒种登记表》③严禁随意将菌、毒种置于非菌、毒种专用保存场所,应做到三专(专室、专柜、专锁)。
④菌、毒种库由2名保管人员双锁管理,铁门与锁必须牢固有效,发现损坏须及时报修。
未经各科室负责人同意,不得擅自将钥匙委托她人代管。
⑤菌、毒种保管人员应定期对库内温度、湿度、通风及冰箱、冰柜等菌、毒种保藏设备运转情况进行检查,并做好记录。
⑥菌、毒种保管人员根据菌、毒种的保存期限,及时通知分管病种的检验人员进行传代,定期鉴定,并详细记录在《菌、毒种登记表》⑦菌、毒种保管人员发现菌、毒种发生变异与死亡,应及时向科室负责人报告,并填写《菌、毒种登记表》。
科室须将变异及死亡的一、二类菌、毒种通报技术管理层。
微生物检验操作规程

微生物检验操作规程1. 目的建立微生物检验标准操作规程,保证检验操作规范化。
2. 范围适用于本公司相关的微生物检验活动的全过程。
3. 职责3.1微生物检验员负责微生物检验的全过程;3.2QC 主管负责监督检查本规程的有效实施。
4. 操作规程4.1微生物检验玻璃器皿吸管的洗涤4.1.1一般的玻璃器皿(例如培养皿、试管、三角瓶等),可用毛刷及洗涤剂洗去灰尘、油垢,然后用自来水、蒸馏水充分冲洗,直至玻璃器皿内壁均匀分布一层薄的水膜,即器壁既不挂水珠也无条纹,即洗涤达到标准。
4.1.2玻璃器皿内有污迹不能洗净时,可浸泡于洗液中,待器皿中有机物氧化分解后,取出用自来水、蒸馏水冲洗干净,备用。
4.1.3新购的玻璃器皿先用 2%盐酸浸泡,再用用自来水、蒸馏水充分冲洗。
4.2 器皿的包扎4.2.1试管:塞上胶塞,然后用牛皮纸或报纸把试管头包扎起来。
做发酵试验时,将试管头塞上专用的耐高温 PVC 试管帽。
4.2.2三角瓶、抽滤瓶:将三角瓶(抽滤瓶)口塞上胶塞,然后用牛皮纸把塞头部包起来。
4.2.3吸管:将耐高温的移液枪头装盒,用用牛皮纸或报纸包裹。
4.2.4平皿: 10 个为一组,用牛皮纸包裹。
4.3 消毒和灭菌:4.3.1化学灭菌:此法适用于微生物操作过程中不能加热、紫外线的地方。
如人手等。
( 1)用 75%的乙醇溶液或 0.1%新洁尔灭擦拭或浸泡。
( 2)一般用 1~ 2%的甲醛液浸泡软管和用具。
( 3)一般成品漂白粉的有效成分是25~ 32% 。
使用时配成有效成份的 2%溶液洒在地面或容器内,(对金属有腐蚀作用)放置 10~ 15min 后冲洗即可。
( 4)氯灭菌剂的能力以有效氯表示,将氯水配制成 50~ 100PPm 的有效氯溶液,可用于浸泡,冲洗和表面杀菌。
但氯对金属有腐蚀作用。
4.3.2物理灭菌:固体试液、液状石蜡等。
灭菌条件一般选用在 160℃干热空气灭菌 2h 。
(1)器具用牛皮纸或灭菌袋包(装)扎,放入金属容器内。
2.1 微生物的实验室培养(2)

B.在皿底上标记日期等方便
C.正着放臵容易破碎
D.防止皿盖上的冷凝水落入培养基造成污染
解析 平板倒臵主要是防止皿盖上的水珠落入培养基造成污染。
是平板划线法和稀释涂布
平板法。下列有关这两种方法的叙述错误的是( B )
1
2
3
4
A.均将大肠杆菌接种在固体培养基的表面
孢子繁殖形成,这种菌落才符合要求。
解析答案
1
2
3
4
4.右表是从土壤中分离纯化土壤细菌的培养基配方。请回答下面的问题。
(1) 培养基的类型很多,但培养基中一
般都含有水、碳源、 氮源 和无机盐。
上述培养基配方中能充当碳源的成分
试剂
用量
试剂
用量
牛肉膏/g
蛋白胨/g
5
10
琼脂/g —
20 —
是 牛肉膏和蛋白胨 。培养基配制完成
答案
应用示例
2.右图中甲是稀释涂布平板法中的部分操作,乙是平板划线法操作结果。 下列叙述中,错误的是( 后才能取菌液 )
A.甲中涂布前要将涂布器灼烧,冷却 B.对于浓度较高的菌液,如果甲中的
稀释倍数仅为102,则所得的菌落不
是由单一的细胞或孢子繁殖而成
C.乙中培养皿中所得的菌落都符合要求
D.乙中的连续划线的起点是上一次划线的末端
微生物感染的病原学实验室诊断分析前

清洁、快速、经济
自动化染色仪
手工染色
标准、清洁、安全
痰标本>10鳞状上皮细胞/每低倍视野=拒收
标本质量评估-痰涂片
Garbage in!!
有PMN的痰标本,且鳞状上皮细胞特别少=接受,进行培养
粘液丝
细胞内吞噬细菌
呼吸道标本的涂片评估模式
肺炎-可接受的
很少的鳞状上皮细胞 <10个每低倍视野 很多的中性粒细胞
标本质量
脓痰
粘液样痰
唾液or诱导痰 (?)
带血痰
革兰氏染色
——所有微生物检验的开端
Gram Staining
What Am I ?
Gram -
Gram +
革兰氏染色的意义
• 1.判断标本的合格性; • 2.标本菌群初始状态的确立与培养 条件的选择; • 3.致病菌的初步判断; • 4.与培养结果互相对照,提高检验准 确性; • 5.初步报告,为临床快速药物选择 和处理提供辅助意见;
采样时严格执行无菌操作,将污染情形降至最低!
护士采样的标本类型:
– 尿液 – 粪便 – 痰和拭子 – 伤口分泌物 – 血培养 –……
采样人员应接受专门培训,严格参考微生物室提供的标本采 集要求进行采样!
注意防止标本被污染或者造成患者机会感染!
标本采集的要求
• 1,在用药前采样;
• 2,按照采样规则要求,选择正确的采样部位、采样 容器、及采样量;
手工染色方法
• 影响因素:
– 1,脱色时间; – 2,涂片的质量; – 3,干燥和固定 – 4,细菌菌龄、用药后等形态变异 – 5,染液沉渣; – 6,交叉污染; – 7,接触感染性标本,危险性试剂
应用特点
微生物涂抹法菌落总数检测作业指导

微生物涂抹法菌落总数检测作业指导范围:适用于设备、成品面包装容器、塑料薄膜等的微生物检测、菌落总数的测定。
1 实验试剂及器材1.1 实验试剂1.1.1 灭菌生理盐水(0.85%)称取8.5g NaCl溶于1000mL蒸馏水,经121℃灭菌20min后,PH值应为7.0±0.1。
1.1.2 营养琼脂培养基(平板计数琼脂):按GB 4789.28中4.7规定。
1.1.3 75%乙醇1.2 实验仪器1.2.1 培养箱:36±1℃1.2.2 冰箱:0~4℃1.2.3 恒温水浴:46±1℃1.2.4 天平1.2.5 移液管:1mL,5mL,10mL;1.2.6 吸耳球1.2.7 广口瓶或三角瓶1.2.8 培养皿1.2.9 灭菌镊子1.2.10 称量纸1.2.11 硫酸纸、牛皮纸、线1.2.12 脱脂棉球1.2.13 高压灭菌锅1.2.14 放大镜1.2.15 酒精灯2 实验步骤2.1 准备实验2.1.1 将洗净的三角瓶、广口瓶、移液管、镊子、培养皿等一同经180℃,75min 灭菌后放入酒精棉球擦拭过的超净台上备用。
2.1.2 配制一定量0.85%的生理盐水,分装于广口瓶中,每个广口瓶装99mL,并给每个广口瓶装一个大约3×3×3cm3大小的无菌棉球。
另配制一定量的营养琼脂培养基(按照营养琼脂培养基外包装标示的浓度配制),与生理盐水一起经121℃,20min(干热121℃,30min)灭菌后,放置于超净台,待用。
2.1.3 打开超净台及室内紫外灯,灭菌30min,与上面生理盐水和培养基的30min 灭菌可以同步进行。
2.1.4 将灭菌后的生理盐水放置于酒精擦拭后的超净台备用,并将培养基放置于46℃水浴锅中保温。
2.2 样品采集进行实验前须用酒精棉球擦拭双手及无菌台的台面,然后以无菌镊子取蘸有少量灭菌生理盐水的无菌棉球,以无菌操作方式于无菌操作台内反复擦拭包装膜内表面,接触面积视卫生状况约100~200cm2,然后将擦拭后的棉球迅速装入盛有99mL灭菌生理盐水的瓶中,盖上瓶盖,摇匀待用。
微生物基因组denovo测序分析流程

#流程大放送#微生物基因组Denovo测序分析知因无限一介绍微生物基因组De novo测序分析也叫微生物基因组从头测序分析,指不依赖于任何参考序列信息就可对某个微生物进行分析的测序分析技术,用生物信息学的方法进行序列拼接获得该物种的基因组序列图谱,然后进行注释等后续一系列的分析。
微生物Denovo基因组测序及分析技术可以应用于医药卫生等领域。
二技术应用领域1、基因组图谱的系统性构建例子:过去几个月,肠病毒D68令数百名美国儿童患病。
华盛顿大学的研究人员测序和分析了肠病毒D68(EV-D68)的基因组,这一成果将发表在新一期的Emerging Infectious Diseases杂志上。
(Genome Sequence of Enterovirus D68 from St. Louis, Missouri, USA)肠病毒D68(EV-D68)能在儿童中引起严重的呼吸道疾病。
其基因组序列可以“帮助人们开发更好的诊断测试,”共同作者Gregory Storch说。
“有助于解释病毒感染为什么会造成严重的疾病,以及EV-D68为什么比过去传播得更广。
”(来自于生物通的报道)2、微生物致病性和耐药性位点检测及相关基因功能研究例子:根据分泌蛋白、毒力因子、致病岛、必需基因等结果去探讨所测物种致病性和耐药性。
3、微生物的比较基因组分析,确定各个近缘微生物中的系统发育关系二基本分析流程图三可能的结果展示图示例图1 微生物基因组的功能注释示例图2 微生物基因组的系统进化关系注:以上图片和文字来自参考文献21。
六参考文献[1] Hong-Bin Shen, and Kuo-Chen Chou, "Virus-mPLoc: a fusion classifier for viral protein subcellular location prediction by incorporating multiple sites", Journal of Biomolecular Structure & Dynamics, 2010, 28: 175-86.[2]Hong-Bin Shen and Kuo-Chen Chou, "Virus-PLoc: A fusion classifier for predicting the subcellular localization of viral proteins within host and virus-infected cells.", Biopolymers. 2007, 85, 233-240.[3] Ren Zhang and Yan Lin, (2009) DEG 5.0, a database of essential genes in both prokaryotes and eukaryotes. Nucleic Acids Research 37, D455-D458.[4] The CRISPRdb database and tools to display CRISPRs and to generate dictionaries of spacers and repeats. BMC Bioinformatics. 2007 May 23;8(1):172.[5] The Pfam protein families database: M. Punta, P.C. Coggill, R.Y. Eberhardt, J. Mistry, J. Tate,C. Boursnell, N. Pang, K. Forslund, G. Ceric, J. Clements, A. Heger, L. Holm, E.L.L. Sonnhammer, S.R. Eddy, A. Bateman, R.D. Finn Nucleic Acids Research (2014) Database Issue 42:D222-D230.[6] Clustal W and Clustal X version 2.0.(2007 Nov 01) Bioinformatics (Oxford, England) 23 (21) :2947-8.PMID: 17846036.[7] Felsenstein, J. 2004. PHYLIP (Phylogeny Inference Package) version 3.6. Distributed by the author. Department of Genome Sciences, University of Washington, Seattle.[8] Li et al (2010). De novo assembly of human genomes with massively parallel short readsequencing. Genome Res vol. 20 (2).[9] Li et al (2008). SOAP: short oligonucleotide alignment program. Bioinformatics Vol. 24 no.5 2008.[10] A.L. Delcher, D. Harmon, S. Kasif, O. White, and S.L. Salzberg (1999) Improved microbial gene identification with GLIMMER, Nucleic Acids Research 27:23 4636-4641.[11] S. Salzberg, A. Delcher, S. Kasif, and O. White (1998) Microbial gene identification using interpolated Markov models, Nucleic Acids Research 26:2, 544-548.[12] Delcher AL, Bratke KA Powe,rs EC,et al(2007). Identifying bacterial genes and endosymbiont DNA with Glimmer. Bioinformatics,23(6):673-679.[13]G. Benson(1999). Tandem repeats finder: a program to analyze DNA sequences. Nucleic Acids Research, Vol. 27, No. 2, pp. 573-580.[14] Kanehisa M, Goto S, Kawashima S, Okuno Y, Hattori M (2004). The KEGG resource for deciphering the genome. Nucleic Acids Res 32 (Database issue): D277–80.[15] Kanehisa M, Goto S, Hattori M, Aoki-Kinoshita KF, Itoh M, Kawashima S, et al. (2006). From genomics to chemical genomics: new developments in KEGG. Nucleic Acids Res 34(Database issue): D354–7.[16] Tatusov RL, Koonin EV, Lipman DJ(1997). A genomic perspective on protein families. Science. Oct 24;278(5338):631-7.[17] Tatusov RL, Fedorova ND et al.(2003). The COG database: an updated version includes eukaryotes. BMC Bioinformatics. Sep 11;4:41.[18] Magrane, M. and UniProt Consortium (2011) UniProt Knowledgebase: a hub of integrated protein data. Database (Oxford) , bar009.[19] Bard J, Winter R (2000). Gene Ontology:tool for the unification of biology. Nat Genet. 25:25-29.[20] ZODOBNOV.E.M,APWEILER.R.InterProScan—an intergration plaftorm forthe signature recognition methods in InterPro[J].Bioinform atics,2001,17(9):847-848.[21] Van den Bogert B1, Boekhorst J2, Herrmann R1, Smid EJ3, Zoetendal EG1, Kleerebezem M4. Comparative genomics analysis of Streptococcus isolates from the human small intestine reveals their adaptation to a highly dynamic ecosystem. PLoS One. 2013 Dec 30;8(12):e83418.。
实验室生物安全应急预案流程图
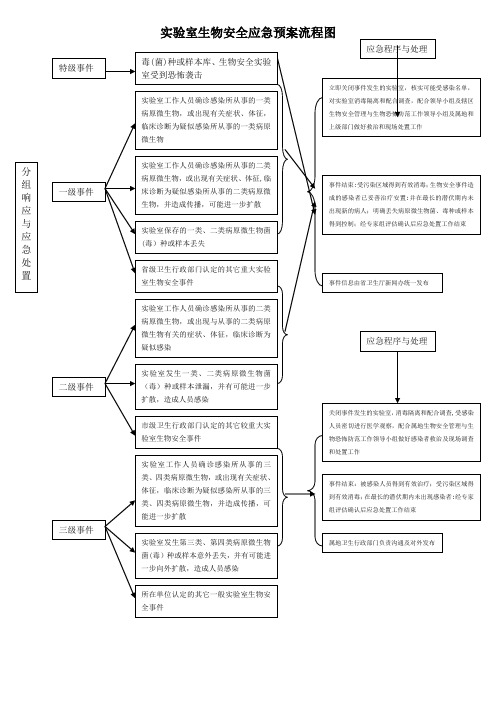
分
组
响
一级事件
应
与
应
急
处
置
二级事件
三级事件
实验室生物安全应急预案流程图
毒(菌)种或样本库、生物安全实验 室受到恐怖袭击
实验室工作人员确诊感染所从事的一类 病原微生物,或出现有关症状、体征, 临床诊断为疑似感染所从事的一类病原 微生物
应急程序与处理
立即关闭事件发生的实验室,核实可能受感染名单, 对实验室消毒隔离和配合调查,配合领导小组及辖区 生物安全管理与生物恐怖防范工作领导小组及属地和 上级部门做好救治和现场处置工作
省级卫生行政部门认定的其它重大实验 室生物安全事件
事件信息由省卫生厅新闻办统一发布
实验室工作人员确诊感染所从事的二类 病原微生物,或出现与从事的二类病原 微生物有关的症状、体征,临床诊断为 疑似感染
应急程序与处理
实验室发生一类、二类病原微生物菌 (毒)种或样本泄漏,并有可能进一步 扩散,造成人员感染
实验室工作人员确诊感染所从事的二类 病原微生物,或出现有关症状、体征,临 床诊断为疑似感染所从事的二类病原微 生物,并造成传播,可能进一步扩散
实验室保存的一类、二类病原微生物菌 (毒)种或样本丢失
事件结束:受污染区域得到有效消毒;生物安全事件造 成的感染者已妥善治疗安置;并在最长的潜伏期内未 出现新的病人;明确丢失病原微生物菌、毒种或样本 得到控制;经专家组评估确认后应急处置工作结束
市级卫生行政部门认定的其它较重大实 验室生物安全事件
实验室工作人员确诊感染所从事的三 类、四类病原微生物,或出现有关症状、 体征,临床诊断为疑似感染所从事的三 类、四类病原微生物,并造成传播,可 能进一步扩散
关闭事件发生的实验室,消毒隔离和配ห้องสมุดไป่ตู้调查,受感染 人员密切进行医学观察,配合属地生物安全管理与生 物恐怖防范工作领导小组做好感染者救治及现场调查 和处置工作
实验四-微生物的菌落形态观察、平板菌落计数及菌种保藏(张理珉)

十分多样
相同
一般不同
可见球状,卵圆状或假 丝状细胞
有时可见细丝状细胞
较快
慢
多带酒香味
常有泥腥味或抗生素气 味
不透明 较牢因结合
十分多样 一般不同 可见粗丝状细胞 一般较快 往往有霉味
二、细菌菌落形态的多样性(示意图)
延展性较强的一些霉菌菌丝(松散)
平板中菌落形态
Hale Waihona Puke 三、菌落特征描写方法说明:检测方法建立的基础——可 测、稳定、重现、可行、简便
浊度与微生物生物量测定
原理:
①细胞的存在会 造成光的散射, 降低光线的透过;
②在一定范围内 细胞浓度与吸光 度成正比
二、平板菌落计数的优缺点
▪ 优点:反映的是微生物活菌数量,可获 得活菌的信息,所以被广泛用于生物制 品检验(如活菌制剂),以及食品、饮 料和水(包括水源水)等的含菌指数或 污染程度的检测。
适用具有特殊生理功能的类群——存在但 不能人工培养形成单菌落,可以利用生化手段 检测出;利用统计学原理计算,结果粗放,只 能得到数量级(近似数)。
(3)菌体浊度的测定(只针对单细胞微 生物);
利用分光光度计在600nm测定吸光度 值。
(4)细胞干重和湿重的测定(有时湿重 以体积计量);
(5)各种细胞物质含量的测定(蛋白质、 核酸、 ATP、磷、叶绿素等定量测 定)——建立了多种快速检测方法;
4、若所有稀释度的平均菌落数均大于 300,则应按稀释度(倍数)最高的平均菌落数 乘以稀释倍数(见下表,例4)。
5、若所有稀释度的平均菌落数均小于30, 则应按稀释度(倍数)最低的平均菌落数乘以 稀释倍数(见下表,例5)。
6、若所有稀释度的平均菌落数均不在 30~300之间,则以最近300或30的平均菌落 数乘以稀释倍数(见下表,例6)。
霉菌毒素试剂盒操作流程图
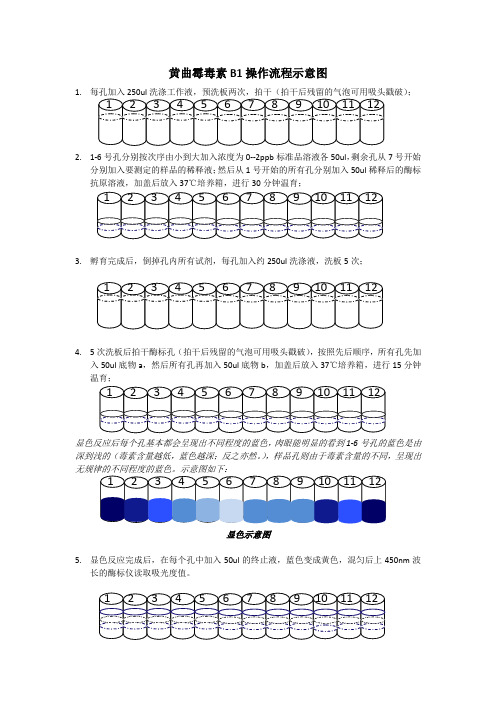
1.;2. 1-6号孔分别按次序由小到大加入浓度为0--2ppb 标准品溶液各50ul ,剩余孔从7号开始分别加入要测定的样品的稀释液;然后从1号开始的所有孔分别加入50ul 稀释后的酶标抗原溶液,加盖后放入37℃培养箱,进行30分钟温育;3. 孵育完成后,倒掉孔内所有试剂,每孔加入约250ul 洗涤液,洗板5次;4. 5次洗板后拍干酶标孔(拍干后残留的气泡可用吸头戳破),按照先后顺序,所有孔先加入50ul 底物a ,然后所有孔再加入50ul 底物b ,加盖后放入37℃培养箱,进行15分钟温育;显色反应后每个孔基本都会呈现出不同程度的蓝色,肉眼能明显的看到1-6号孔的蓝色是由深到浅的(毒素含量越低,蓝色越深;反之亦然。
),样品孔则由于毒素含量的不同,呈现出显色示意图5. 显色反应完成后,在每个孔中加入50ul 的终止液,蓝色变成黄色,混匀后上450nm 波长的酶标仪读取吸光度值。
1.;2. 1-6号孔分别按次序由小到大加入浓度为0--360ppb 标准品溶液各50ul ,剩余孔从7号开始分别加入稀释好的样品液;然后从1号开始的所有孔分别加入50ul 酶标记物溶液,最后在每孔加入50ul 抗试剂,加盖后放入37℃培养箱,进行20分钟温育;3. 孵育完成后,倒掉孔内所有试剂,每孔加入约250ul 洗涤液,洗板5次;4. 5次洗板后拍干酶标孔(拍干后残留的气泡可用吸头戳破),按照先后顺序,所有孔先加入50ul 底物液,然后所有孔再加入50ul 显色液,加盖后放入37℃培养箱,进行15分钟温育;显色反应后每个孔基本都会呈现出不同程度的蓝色,肉眼能明显的看到1-6号孔的蓝色是由深到浅的(毒素含量越低,蓝色越深;反之亦然。
),样品孔则由于毒素含量的不同,呈现出显色示意图5.显色反应完成后,在每个孔中加入50ul 的终止液,蓝色变成黄色,混匀后上450nm 波长的酶标仪读取吸光度值。
玉米赤霉烯酮毒素试剂盒操作流程示意图1.;2. 1-6号孔分别按次序由小到大加入浓度为0—2.4ppb 标准品溶液各50ul ,剩余孔从7号开始分别加入稀释好的样品液;然后从1号开始的所有孔分别加入50ul 酶标记物溶液,最后在每孔加入50ul 抗试剂,加盖后放入37℃培养箱,进行20分钟温育;3. 孵育完成后,倒掉孔内所有试剂,每孔加入约250ul 洗涤液,洗板5次;4. 5次洗板后拍干酶标孔(拍干后残留的气泡可用吸头戳破),按照先后顺序,所有孔先加入50ul 底物液,然后所有孔再加入50ul 显色液,加盖后放入37℃培养箱,进行15分钟温育;显色反应后每个孔基本都会呈现出不同程度的蓝色,肉眼能明显的看到1-6号孔的蓝色是由深到浅的(毒素含量越低,蓝色越深;反之亦然。
