实验四.重组质粒载体的构建
实验四.重组质粒载体的构建

5'…G↓AATTC…3' →5'… G AATTC…3' 3'…CTTAA↑G …5' →3'… CTTAA G…5'
DNA纯度、缓冲液、温度条件及限 制性内切酶本身都会影响限制性内切酶 的活性。大部分限制性内切酶不受RNA 或单链DNA的影响。当微量的污染物进 入限制性内切酶贮存液中时,会影响其进 一步使用,因此在吸取限制性内切酶时,每 次都要用新的吸管头。如果采用两种限 制性内切酶,必须要注意分别提供各自的 最适盐浓度。若两者可用同一缓冲液,则 可同时水解。若需要不同的盐浓度,则低 盐浓度的限制性内切酶必须首先使用,随 后调节盐浓度,再用高盐浓度的限制性内 切酶水解。
2、 混匀反应体系后,将eppendorf管置于适 当的支持物上(如插在泡沫塑料板 上),37℃水浴保温2-3小时,使酶切反应 完全。 3、 每管加入2μl 0.1mol/L EDTA(pH8.0), 混匀,以停止反应,置于冰箱中保存备用。
注意事项
1. 酶切时所加的DNA溶液体积不能太大,否则DNA溶液中其他成 分会干扰酶反应。 2、酶活力通常用酶单位(U)表示,酶单位的定义是:在最适反应 条件下,1小时完全降解1mg lDNA的酶量为一个单位,但是许多 实验制备的DNA不象lDNA那样易于降解,需适当增加酶的使用 量。反应液中加入过量的酶是不合适的,除考虑成本外,酶液中 的微量杂质可能干扰随后的反应。 3、市场销售的酶一般浓度很大,为节约起见,使用时可事先用酶 反应缓冲液(1×)进行稀释。另外,酶通常保存在50%的甘油 中,实验中,应将反应液中甘油浓度控制在1/10之下,否则,酶 活性将受影响。
构建DNA限制性内切酶图谱有许多 方法。通常结合使用多种限制性内切酶, 通过综合分析多种酶单切及不同组合的 多种酶同时切所得到的限制性片段大小 来确定各种酶的酶切位点及其相对位置。 酶切图谱的使用价值依赖于它的准确性 和精确程度。
同源重组实验步骤

同源重组实验步骤2.构建质粒载体:获得目标DNA片段后,需要准备一个质粒载体,由于同源重组实验通常使用的载体为质粒,因此需要选择合适的质粒载体。
通常选择的载体具有以下特点:小尺寸,方便操作和复制;至少具有一个选择性标记,如抗生素抗性基因;具有多个限制性内切酶切位点,方便插入DNA片段。
3.酶切质粒载体:为了在质粒中插入目标DNA片段,需要通过酶切将质粒切开。
选择适当的限制性内切酶,将质粒切割成线性的DNA分子。
切割后的质粒将具有相应的粘性末端。
4.酶切目标DNA:同样的,在切割质粒之前,需要通过酶切目标DNA片段来制备粘性末端。
选择适当的限制性内切酶,将目标DNA片段切割成线性的DNA分子,并留有与质粒末端相兼容的粘性末端。
5.进行连接:将酶切后的质粒和目标DNA片段进行连接。
将目标DNA片段与线性的质粒反应,使其两者的粘性末端互相连接。
由于粘性末端具有互补性,使得两者可以进行连接。
连接实验通常在适当的缓冲液中进行,并添加DNA连接酶。
6.转化宿主细胞:连接完成后,将混合物转化到宿主细胞中。
这通常是通过电穿孔、热激冷冻法、钙磷法等方法来实现的。
转化宿主细胞后,用培养基培养细胞,以便DNA在细胞中复制。
7.选择重组细胞:培养转化后的细胞后,需要选择具有重组质粒的细胞。
这通常通过添加对应抗生素或其他选择性培养基的方法来实现。
只有携带重组质粒的细胞才能生存并继续生长。
8.鉴定重组细胞:确定是否成功进行了同源重组,通常通过PCR检测、酶切鉴定或测序等方法来进行。
PCR检测、酶切鉴定和测序可以确定质粒中是否存在目标DNA片段,以及目标DNA片段是否插入到了正确的位置。
9.扩大培养重组细胞:验证重组细胞后,可以通过扩大培养的方式,获得更多的重组细胞或质粒。
10.应用:获得重组细胞或质粒后,可以根据实验需要进行其他研究。
比如,可以通过转录和翻译来获得重组蛋白,用于功能鉴定;也可以将重组质粒转入其他宿主细胞,用于产生大量的重组蛋白。
重组载体构建的方法和步骤

重组载体构建的方法和步骤全文共四篇示例,供读者参考第一篇示例:重组载体构建是基因工程领域中非常重要的一项技术,它可以用来将特定的基因插入到目标细胞中,实现基因的转移和表达。
在科学研究、医学诊断和治疗等领域中都有广泛的应用。
下面我们来详细介绍一下重组载体构建的方法和步骤。
一、选择载体首先我们需要选择一个适合的载体作为基础,常见的载体有质粒、病毒、原核生物等。
在选择载体时需要考虑载体的大小和特性,以及目标基因的大小和需要表达的水平。
同时还需要考虑载体的复制原点、抗生素抗性基因等相关元件。
二、线性化载体接下来我们需要将选择的载体进行线性化处理,以便将目标基因插入到载体中。
线性化可以通过受控的限制酶酶切处理来实现,将载体的环状DNA骨架切割成线性DNA片段。
三、插入目标基因将目标基因与线性化的载体进行连接。
目前常用的方法包括:内切酶切割连接法、PCR扩增连接法、接头连接法等。
这些方法可以有效地将目标基因插入到载体中,并确保插入的正确性和稳定性。
四、转化目标宿主将构建好的重组载体导入到目标宿主细胞中,使其稳定地存在和复制。
转化的方法多样,包括热激转化、电穿孔转化、化学法转化等。
转化效率和载体稳定性是评价转化效果的主要因素。
五、筛选重组子对转化后的细胞进行筛选,筛选出含有目标基因的重组子。
常用的筛选方法包括抗生素筛选、荧光筛选、酵素检测等。
筛选过程中需要注意筛选压力和筛选条件的优化,以提高筛选效率。
六、鉴定重组子对筛选出的重组子进行鉴定,确保其构建正确。
常用的鉴定方法包括PCR扩增、酶切鉴定、序列分析等。
通过这些方法可以验证重组子的结构和功能是否正确,确保后续实验的准确性和可靠性。
七、表达目标基因对鉴定合格的重组子进行表达。
通过选用适当的启动子和调控元件,可以实现目标基因的高效表达。
表达的方法有多种选择,包括转染法、感染法、转基因法等。
表达的效果可以通过荧光显微镜观察、酶活性测定、Western blot等方法进行检测和验证。
克隆载体构建实验报告(3篇)

第1篇一、实验目的1. 学习克隆载体的构建方法,掌握分子克隆的基本原理和操作步骤。
2. 掌握利用限制性内切酶和DNA连接酶进行DNA片段的插入和连接。
3. 熟悉重组质粒的鉴定和扩增方法。
二、实验原理克隆载体是分子生物学研究中常用的工具,它可以将目的基因插入其中,并在宿主细胞中进行扩增。
克隆载体的构建主要包括以下步骤:1. 设计引物:根据目的基因序列设计特异性引物,用于PCR扩增目的基因片段。
2. PCR扩增:利用引物扩增目的基因片段。
3. 载体线性化:利用限制性内切酶将载体线性化,使其具有末端粘性。
4. DNA片段连接:将目的基因片段与载体进行连接。
5. 转化宿主细胞:将连接后的重组质粒转化至宿主细胞。
6. 鉴定和扩增:通过PCR、酶切等方法对转化后的细胞进行鉴定和扩增。
三、实验材料1. 试剂:PCR引物、限制性内切酶、DNA连接酶、DNA分子量标准、Taq酶、pUC19载体、感受态细胞等。
2. 仪器:PCR仪、电泳仪、凝胶成像系统、移液器、DNA纯化柱等。
四、实验步骤1. 设计引物:根据目的基因序列设计特异性引物,引物长度一般为20-30个碱基,其中包含酶切位点。
2. PCR扩增:利用引物扩增目的基因片段,PCR反应体系如下:- 10×PCR缓冲液5μl- dNTPs(每种2.5μmol/L)4μl- 引物(上下游引物各1μmol/L)2μl- DNA模板1μl- Taq酶0.5μl- ddH2O补充至50μl3. 载体线性化:利用限制性内切酶将载体线性化,反应体系如下:- 载体DNA 5μl- 10×酶切缓冲液5μl- 限制性内切酶1μl- ddH2O补充至20μl4. DNA片段连接:将PCR产物与载体进行连接,反应体系如下:- 线性化载体DNA 5μl- PCR产物5μl- 10×连接缓冲液5μl- DNA连接酶1μl- ddH2O补充至20μl5. 转化宿主细胞:将连接后的重组质粒转化至感受态细胞,具体操作方法如下:- 将感受态细胞铺板于含有适当抗生素的培养基上,37℃培养过夜。
重组质粒的构建

重组质粒的构建重组质粒的构建是基因工程的核心步骤之一,其目的是将目的基因插入到质粒载体中,以实现目的基因的稳定表达和克隆化。
以下是重组质粒构建的主要步骤:1.目的基因获取首先需要获取目的基因。
目的基因可以从基因文库、PCR、基因组测序等方法中获取。
根据需要选择合适的方法,将目的基因克隆到质粒载体中。
2.载体质粒选择选择适合的质粒载体是重组质粒构建的关键步骤之一。
根据目的基因的特点和表达要求,选择适合的质粒载体。
常见的质粒载体有pET、pUC、pBluescript等。
3.限制性酶切限制性酶切是重组质粒构建的重要步骤之一。
通过限制性酶切,将目的基因和质粒载体分别切开,露出粘性末端,以便于连接反应。
4.连接反应将切好的目的基因和质粒载体的粘性末端连接在一起,形成重组质粒。
连接反应需要使用T4DNA连接酶或其它连接酶进行催化。
连接反应需要在适宜的温度和pH 条件下进行一定时间,以确保重组质粒的正确构建。
5.转化宿主细胞将连接反应得到的重组质粒转化到宿主细胞中。
常见的宿主细胞有细菌、酵母、昆虫等。
转化方法有多种,如电穿孔法、化学转化法等。
转化后需要在适宜的培养条件下进行培养,以获得大量的重组质粒。
6.克隆筛选克隆筛选是重组质粒构建的重要步骤之一。
通过克隆筛选,可以确定重组质粒是否正确构建。
常见的克隆筛选方法有蓝白斑筛选、酶切法等。
7.序列验证最后需要对重组质粒进行序列验证,以确保目的基因的正确插入和序列的准确性。
序列验证可以通过Sanger测序等方法进行。
重组表达质粒的构建——原核表达载体选择
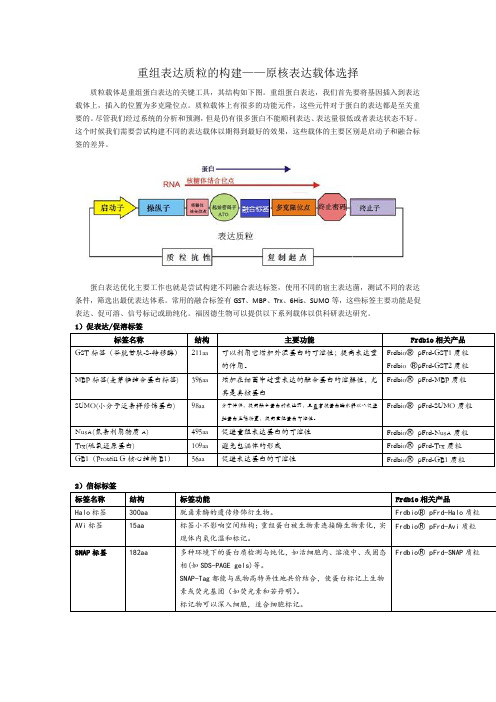
重组表达质粒的构建——原核表达载体选择质粒载体是重组蛋白表达的关键工具,其结构如下图。
重组蛋白表达,我们首先要将基因插入到表达载体上,插入的位置为多克隆位点。
质粒载体上有很多的功能元件,这些元件对于蛋白的表达都是至关重要的。
尽管我们经过系统的分析和预测,但是仍有很多蛋白不能顺利表达、表达量很低或者表达状态不好。
这个时候我们需要尝试构建不同的表达载体以期得到最好的效果,这些载体的主要区别是启动子和融合标签的差异。
蛋白表达优化主要工作也就是尝试构建不同融合表达标签,使用不同的宿主表达菌,测试不同的表达条件,筛选出最优表达体系。
常用的融合标签有GST、MBP、Trx、6His、SUMO等,这些标签主要功能是促表达、促可溶、信号标记或助纯化。
福因德生物可以提供以下系列载体以供科研表达研究。
1)促表达/促溶标签2)信标标签3)纯化标签我们选择表达载体的时候不但要考虑蛋白怎么表达成功,更要考虑蛋白怎么纯化出来,纯化的问题主要是考虑纯化标签和酶切位点的选择,下表我们列举了常见的纯化标签和酶切位点。
4)酶切位点以上为原核表达常用的标签和酶切位点,其性质也都作了简要的介绍,各专业网站或专业书籍已对此做详尽解释,科研工作者可根据具体实验设计方案,组合设计以上标签和酶切位点的使用。
特别值得注意的是,选用和设计蛋白酶切位点的时候首要考虑的是序列内部有没有蛋白酶位点,同时要考虑酶切的效率和蛋白酶试剂成本。
一般商业化载体,在标签蛋白与载体多克隆位点之间都设计有酶切位点。
标签可设计在N-端也可在C-端,设计在N-端的优势是,可通过标签高效翻译起始位点带动插入蛋白的表达,可溶性标签的高效表达更可促进蛋白的可溶性表达;同时,大部分的蛋白内切酶的切割位点在C-端,所以标签设计在N-端可将标签切割完全。
在设计标签序列与酶切位点的时候还要考虑N-端稳定性原则,也就是所谓宿主细胞的N-端规则(N-end rule),这个要避免;同时,还应该检查是否引入了可与别的蛋白相互作用的序列或者蛋白酶切位点。
4重组表达质粒的构建——基因的克隆

重组表达质粒的构建——基因的克隆长片段基因在大肠杆菌中表达往往比较困难,作为抗原使用的重组蛋白可以考虑选择抗原性好的区段原核表达,前文已作阐述。
对整个蛋白结构研究,必须全长表达该蛋白,此时最好考虑真核表达系统,特别是含有跨膜区的蛋白。
选定要克隆的区段,需先富集纯化之后才方便插入载体,常用的富集方法是PCR或者质粒繁殖复制。
为了防止在PCR扩增过程中引入碱基错误或者碱基缺失,PCR扩增基因时候必须使用高保真Taq酶。
为了满足科研工作者不同实验需求,福因德生物将高保真Taq酶优化为即用型Mix,使用时直接加引物和模板就可以扩增。
除此之外,福因德生物还开发出LA Taq、S-Taq Mix以及SYBR荧光定量PCR Mix(需要更高品质的可选用SYBR PCR SuperMix)。
原核重组表达常用克隆技术主要有以下几种:1)酶切连接这个是目前应用最为广泛的的克隆技术,主要优点是技术稳定;缺点是周期长、步骤多,任何一个环节产生的误差都会影响克隆构建的成败。
如用酶切连接的策略进行载体批量构建,不同载体和不同外源基因尽可能选用相同的上下游酶切位点,比如,批量克隆基因到某个载体上,可一次性大量双酶切将载体线性化后保存备用,每次构建载体只需酶切外源基因片段,载体可直接取用,不必每次都酶切,省时省力(此处需特别留意的是基因内部不能有与上述所用冲突的酶切位点)。
2)TA克隆TA克隆必须使用商业的线性化载体,线性载体3´末端有一个T碱基,与PCR扩增产物3´末端A正好匹配。
这种克隆策略最大的优点是载体使用方便,扩增产物可以直接克隆到载体上,不需要酶切位点等冗余序列;缺点是:必须依赖商业化载体,载体选择受限;扩增外源片段所使用的Taq酶也必须是可以在3´末端加A,这种Taq酶的保真度不高;外源片段插入之后还必须鉴定方向。
目前,这种构建表达载体的策略已经逐渐被淘汰。
3)TOPO克隆TOPO克隆载体利用DNA拓扑异构酶I识别序列中的CCCTT松弛双螺旋并重新连接,同时兼具限制性内切酶和连接酶的功能。
重组载体构建的方法和步骤

重组载体构建的方法和步骤
1. 选择载体,首先需要选择合适的载体,常用的载体包括质粒、病毒、人工染色体等。
选择载体时需要考虑载体大小、复制方式、
宿主范围等因素。
2. 线性化载体,将所选载体进行线性化处理,通常采用限制性
内切酶对载体进行切割,生成线性化的载体。
3. 外源基因插入,将待插入的外源基因与线性化载体进行连接。
这一步通常需要利用DNA连接酶或者DNA重组酶来实现。
4. 载体转化,将重组后的载体导入到宿主细胞中。
这一步通常
采用转化、转染或者电穿孔等方法。
5. 筛选正常重组子代,经过载体转化后,需要进行筛选以获得
正常重组的子代细胞。
通常采用抗生素筛选或者标记基因筛选的方式。
6. 鉴定重组子代,对筛选得到的细胞进行PCR、酶切、测序等
方法进行鉴定,确保重组载体的构建成功。
总的来说,重组载体构建的方法和步骤包括选择载体、线性化载体、外源基因插入、载体转化、筛选正常重组子代和鉴定重组子代。
这些步骤需要严格操作,并且需要根据具体实验情况进行调整和优化。
希望这些信息能够对你有所帮助。
载体构建连接实验报告(3篇)

第1篇一、实验目的本次实验旨在通过载体构建和连接技术,将目的基因与载体成功连接,构建一个含有目的基因的重组质粒。
此实验是基因工程中的重要步骤,对于后续的基因表达、蛋白质纯化等研究具有重要意义。
二、实验原理载体构建连接实验主要包括以下几个步骤:1. 目的基因的获取:通过PCR扩增或化学合成等方法获得目的基因。
2. 载体的选择与制备:选择合适的载体,并对其进行酶切处理,制备线性化载体。
3. 目的基因与载体的连接:利用DNA连接酶将目的基因与线性化载体连接,形成重组质粒。
4. 重组质粒的筛选与鉴定:通过PCR、酶切分析等方法筛选出含有目的基因的重组质粒。
三、实验材料1. 实验试剂:PCR引物、dNTPs、DNA聚合酶、限制性内切酶、DNA连接酶、T4 DNA聚合酶、Klenow片段、碱性磷酸酶(CIP)、质粒提取试剂盒、DNA纯化试剂盒等。
2. 实验仪器:PCR仪、电泳仪、凝胶成像系统、移液器、离心机、超净工作台等。
3. 实验材料:目的基因模板、载体质粒、感受态细胞等。
四、实验方法1. 目的基因的获取:根据目的基因的序列设计PCR引物,进行PCR扩增。
2. 载体的选择与制备:选择合适的载体,用限制性内切酶进行酶切处理,制备线性化载体。
3. 目的基因与载体的连接:a. 将目的基因和线性化载体进行PCR扩增,获得双链DNA。
b. 用Klenow片段或T4 DNA聚合酶将PCR产物进行末端修复,使末端具有3'-羟基。
c. 用CIP处理线性化载体,去除5'-磷酸基团。
d. 将目的基因和线性化载体进行连接反应,加入DNA连接酶。
e. 将连接产物进行PCR扩增,验证连接成功。
4. 重组质粒的筛选与鉴定:a. 将连接产物转化感受态细胞,涂布于含抗生素的琼脂平板上。
b. 在37℃恒温箱中培养过夜。
c. 挑取单菌落进行PCR扩增,验证重组质粒的存在。
d. 对重组质粒进行酶切分析,确认目的基因已插入载体。
重组质粒的构建、转化和重组子的筛选与鉴定

重组质粒的构建、转化和重组子的筛选与鉴定姓名:郑小煜学号:201100140069班级:11级生技班同组者:赵莉、高瑞【实验目的】1、学习在实现DNA的体外重组过程中,正确选择合适的载体和限制性内切酶,并利用限制性核酸内切酶对载体和目的DNA进行切割,产生利于连接的合适末端。
2、通过对DNA的酶切,学习设计构建重组DNA分子的基本方法,掌握载体和外源目的DNA酶切的操作技术。
3、学习利用T4 DNA连接酶把酶切后的载体片段和外源目的DNA片段连接起来,构建体外重组DNA 分子的技术,了解并掌握几种常用的连接方法。
4、掌握利用CaCl2制备感受态细胞的方法。
5、学习并掌握热击法转化E.coli的原理和方法。
6、在使用红白菌落法筛选获得重组子的基础上,本实验学习通过对重组子进行重组质粒DNA的抽提鉴定,以进一步确定重组质粒中含有外源目的DNA片段,验证重组子是期望的重组子的方法。
7、通过学习掌握重组DNA分子鉴定的基本方法。
8、掌握α互补筛选法筛选重组子的方法。
并鉴定体外导入目的DNA片段的大小。
9、学习用试剂盒提取重组质粒DNA的方法。
10、复习琼脂糖凝胶电泳的原理及方法。
【实验原理】外源DNA与载体分子的连接即为DNA重组技术,这样重新组合的DNA分子叫做重组子。
重组的DNA分子在DNA连接酶的作用下,有Mg2+、ATP存在的连接缓冲系统中,将分别经限制性内切酶酶切的载体分子和外源DNA分子连接起来。
将重组质粒导入感受态细胞中,将转化后的细胞在选择性培养基中培养,可以通过α互补筛选法筛选出重组子,并可通过酶切电泳及PCR检验的方法进行重组子的鉴定。
1、限制性核酸内切酶及酶切反应体外构建重组DNA分子,首先,要了解目的基因的酶切图谱,选用的限制性内切酶不能目的基因内部有专一的识别位点,即当用一种或两种限制性核酸内切酶切割外源供体DNA时,能得到完整的目的基因。
其次,要选择具有相应的单一酶切位点的质粒或者噬菌体等载体分子作为克隆的载体。
DNA重组-载体构建常见问题及解决方法

载体构建中常见问题及解决方法重组质粒构建是常用的分子生物学手段,是研究相关分子及蛋白功能的基础,是生化分子实验中最基本的方法,但是其中还是有些基本的技巧需要掌握。
下面我总结一下自己的经验,以期能为虫友们提供借鉴,希望多少给大家一下借鉴。
所涉及内容如下:1)克隆基因的酶切位点问题2)目的片段的扩增3) 载体酶切的问题4) 连接片段浓度比的问题5)菌液的提取6)酶切鉴定7)表达鉴定一、克隆基因的酶切位点问题1、克隆位点选择的问题。
首先要对目标基因进行酶切位点扫描分析,列出其所含酶切位点清单。
然后对照质粒多克隆位点,所选择的克隆位点必须是目标基因所不含的酶切位点。
双酶切时尽量选择酶切温度相同,buffer一致,双酶切效率高的两个内切酶,二者最好是您实验室常用的酶。
2、保护碱基数目的问题。
在设计PCR引物时,引入酶切位点后,常常要加入保护碱基,这是大家所熟知的。
但是保护碱基数量多少,可能被新手所忽视。
这种忽视可能会大大影响后续的实验进展。
参考常用的酶切位点保护碱基,选择酶切时间短且酶切效率高的碱基数。
注释:1.如果要加在序列的5‘端,就在酶切位点识别碱基序列(红色)的5’端加上相应的碱基(黑色),相同如果要在3‘端加保护碱基,就在酶切位点识别碱基序列(红色)的3’端加上相应的碱基(黑色)。
2.切割率:正确识别并酶切的效率3. 加保护碱基时最好选用切割率高时加的相应碱基二目的片段的扩增目的片段扩增失败的原因和可采取措施:1)无条带:引物设计错误,找人帮忙看看引物设计是否合理;退火温度不合适,设置梯度,选择最合适的退火温度;2)条带不单一:引物不特异,适当增长或引物序列长度;3)出现引物二聚体,适当提高退火温度或者重新设计引物。
三载体酶切的问题酶切效率直接影响后续实验的成功率,酶切过程的注意事项:1)酶切质粒浓度和纯度要好2)酶切温度和时间,如果两个酶的最适温度不同,建议单酶切,回收后在用另一个酶切,时间最好过夜切四连接片段浓度比的问题1)连接比例的确定:上一步酶切产物回收后,用琼脂糖凝胶电泳比较载体和目的片段的亮度,确定体积比,一般载体:片段=1:3~1:5;2)连接时间和温度:现在很多连接酶都比较高效了,室温2个小时就可以了,如果后续实验检测阳性克隆较少,可采用16℃过夜连接提高连接成功率;3)未避免后续菌落PCR的假阳性情况,建议连接转化时做对照组:酶切后的载体做自连接对照,如果对照组与实验组长出的克隆数相差不大则很可能存在载体自连或者没完全切开的情况,建议先不要做后续验证,重新从酶切开始,增加酶量及酶切时间、适当减少所切载体的量、改变连接比等;如果对照组没有克隆形成或鲜有克隆形成,则可继续进行后续验证。
目的基因T载体克隆实验步骤

目的基因T载体克隆实验步骤pcr产物的t载体克隆一.重组质粒的构建:重组的dna分子就是在dna连接酶的促进作用下,存有mg切的载体分子与外源dna分子进行连接。
dna连接酶存有两种:t4噬菌体dna连接酶和大肠杆菌dna连接酶。
两种dna连接酶都存有将两个具有相同粘性末端的dna分子连在一起的功能,而且t4噬菌体dna连接酶除了一种大肠杆菌dna连接酶没的特性,即为能够并使两个平末端的双链dna分子连接起来。
但这种相连接的效率比粘性末端的相连接率为高,通常可以通过提升t4噬菌体dna 连接酶浓度或减少dna浓度去提升平末端的相连接效率。
t4噬菌体dna连接酶催化剂dna 连接反应分成3步:首先,t4dna连接酶与辅因子atp构成酶-atp复合物;然后,酶-atp 复合物再融合至具备5’磷酸基和3’羟基切口的dna上,并使dna腺苷化;最后产生一个代莱磷酸二酯键,把切口封出来。
连接反应通常将两个相同大小的片断相连。
很多dna聚合酶在进行pcr扩增时会在pcr产物双链dna每条链的3’端加上一个突出的碱基a。
pucm-t载体是一种已经线性化的载体,载体每条链的3’端带有一个突出的t。
这样,pucm-t载体的两端就可以和pcr产物的两端进行正确的at配对,在连接酶的催化下,就可以把pcr产物连接到pucm-t载体中,形成含有目的片断的重组载体。
连接反应的温度在37℃时有助于连接酶的活性。
但是在这个温度下黏末端的氢键融合就是不稳定的。
因此实行折衷的温度,即12-16℃,相连接12-16h(过夜),这样既可以最大限度地充分发挥连接酶的活性,又兼具至较长时间接合结构的平衡。
2、atp存有的相连接缓冲器系统中,将分别经酶二.感受态制备原理细菌在0ccacl2低渗溶液中胀变成球形,遗失部分膜蛋白,沦为难稀释外源dna的状态。
三.β-半乳糖甘酶显色反应选择法(蓝白筛选)原理lacz基因是大肠杆菌乳糖操纵子中的一个基因,可以编码β—半乳糖核苷酶。
重组质粒的PCR实验

重组质粒的PCR实验李笃财 201200140048 生科一班一.实验目的1.学习利用PCR产物回收试剂盒回收PCR产物;2.掌握基因克隆的基本操作过程3.了解利用T载体进行PCR产物克隆的基本原理4.进一步巩固PCR扩增及电泳检测技术二.实验原理(一)重组质粒酶切鉴定将含有外源DNA的转化子的E.coliDH5α菌株进行培养,并用试剂盒提取其质粒DNA,将所提取的DNA用切pUC19质粒的同一种限制性内切酶进行切割以验证所插入的外源DNA的大小。
(二)PCRPCR(Polymerase Chain Reaction)即聚合酶链式反应是1986 年由Kallis Mullis 发现。
这项技术已广泛地应用于分子生物学各个领域,它不仅可用于基因分离克隆和核酸序列分析,还可用于突变体和重组体的构建,基因表达调控的研究,基因多态性的分析等方面。
本次实验旨在通过学习和掌握PCR反应的基本原理和实验技术,以验证重组质粒插入片段大小。
1.聚合酶链式反应原理PCR是一种利用两种与相反链杂交并附着于靶DNA两侧的寡核苷酸引物,经酶促合成特异的DNA 片段的体外方法。
反应过程由高温变性,低温退火和适温延伸等几步反应组成一个循环,然后反复进行,使目的的DNA 得以迅速扩增。
置待扩增DNA 于高温下解链成为单链DNA 模板,人工合成的两个寡核苷酸引物在低温条件下分别与目的片段两侧的两条链互补结合,DNA聚合酶在72℃将单核苷酸从引物3'端开始掺入,沿模板5'—3'方向延伸,合成DNA 新链。
由于每一循环所产生的DNA均能成为下一次循环的模板,所以PCR 产物以指数方式增加,经25—30次周期之后,理论上可增加109倍,实际上可增加107倍。
PCR 技术具有操作简便、省时、灵敏度高特异性强和对原始材料质量要求低等优点,但由于所用的TaqDNA 聚合酶缺乏5'—3'核酶外切酶活性,不能纠正反应中发生的错误核苷酸掺入,估计每9000个核苷酸会导致一个掺入错误,但是错误掺入的碱基有终止链延伸的作用倾向,使得错误不会扩大。
基因编辑技术实验报告(3篇)

第1篇一、实验目的基因编辑技术是近年来生物科学领域的一项重大突破,它能够精确地修改生物体的基因组,为生物学研究和应用提供了前所未有的可能性。
本实验旨在了解基因编辑技术的原理和操作流程,掌握CRISPR/Cas9技术的基本操作,并通过实验验证该技术的可靠性和有效性。
二、实验原理基因编辑技术是通过人工核酸酶对基因组进行精确修饰的一种基因工程技术。
CRISPR/Cas9技术是目前最常用的基因编辑技术之一,其原理是利用细菌内源性的CRISPR系统中的Cas9核酸酶,结合一段与目标基因序列互补的sgRNA(single-guide RNA),实现对目标基因的定点敲除、插入或替换。
三、实验材料1. 实验仪器:PCR仪、电泳仪、凝胶成像系统、移液器、培养箱、恒温振荡器等。
2. 实验试剂:CRISPR/Cas9试剂盒、dNTPs、Taq酶、限制性内切酶、连接酶、DNA 标记物、DNA提取试剂盒、质粒提取试剂盒等。
3. 实验菌株:大肠杆菌DH5α、CRISPR/Cas9菌株等。
四、实验方法1. 设计实验方案:根据实验目的,设计CRISPR/Cas9基因编辑实验方案,包括靶基因序列、sgRNA设计、靶位点验证等。
2. 构建CRISPR/Cas9表达载体:根据实验方案,设计sgRNA序列,并将其克隆到CRISPR/Cas9表达载体中。
3. 转化CRISPR/Cas9表达载体:将构建好的CRISPR/Cas9表达载体转化到大肠杆菌DH5α中。
4. 验证CRISPR/Cas9表达载体:通过PCR和测序验证CRISPR/Cas9表达载体的构建。
5. 转化CRISPR/Cas9表达载体到CRISPR/Cas9菌株:将验证好的CRISPR/Cas9表达载体转化到CRISPR/Cas9菌株中。
6. 实验操作:将CRISPR/Cas9菌株接种到含有抗生素的培养基中,培养至对数生长期。
取适量菌株接种到含有靶基因的细胞培养基中,进行基因编辑实验。
基因工程实验

基因工程实验详细步骤一、目的基因LK的扩增——PCR(含电泳)(一)目的基因LK的扩增——NE201(1)实验准备材料:rLK重组质粒(模板)试剂:PCR试剂盒用具:移液枪(2.5μl、10μl、50μl)以及相应枪头(盒)、EP管(100μl)、EP板(大、小);冰袋两只、手套、废液桶仪器:离心机(14000rpm)、PCR扩增仪(2)反应体系(50μl)试剂(按照加样顺序)用量dH2O23μL重组质粒1μL上游引物F 0.5μL下游引物R 0.5μLTaq酶25μL(3)反应条件预变性95℃,10min变性95℃,30s退火56.6℃,35s延伸72℃,40s最终延伸72℃,10minTIP:*吸取或加入液体时,枪头尽量少伸入,可避免气泡产生;若有气泡,需10000rcf离心1min 再进行下一步实验*勤换枪头,避免污染试剂或产物*任何移液枪用完后及时调回最大量程*仪器用完后及时关闭(二)LK PCR产物的电泳——NE217(1)实验准备材料:LK PCR产物试剂:去离子水、50×TAE缓冲液、电泳用琼脂糖、6×Loading Buffer、DL1000marker(D526A)、Ultrapower DNA Stain染液用具:钥匙、称量纸、橡皮筋、100ml锥形瓶、50ml量筒;制胶槽、制胶卡、制胶梳(18teeth);移液枪(10μl、1000μl)及相应枪头(盒),100μl EP管,EP板(大、小);冰袋两只、手套(包括塑料和麻布)、废液桶仪器:电子天平、微波炉、电泳仪(含电泳槽)(2)制胶(3%)1. 称取电泳用琼脂糖0.6g(即总体积20ml 的3%),小心倒入100ml锥形瓶中2. 取TAE缓冲液400μl于50ml量筒中,去离子水定容至20ml(实为电泳缓冲液)3. 将量筒中液体转入锥形瓶中,盖上吸量纸,用橡皮筋封好,再用步骤2中用剩的枪头扎一个小孔4. 将制胶梳(18 teeth)、制胶卡、制胶槽洗净并用吸水纸擦干,组装好5. 至于微波炉中,注意观察,煮沸后立即拿出(戴上麻布手套),轻轻晃匀,重复2-4次,至胶完全澄清均一为止6. 及时将胶转入准备好的制胶槽,凝固30min后,连着横条纹塑料板一起放入电泳槽中(胶孔靠近负极)(3)加样和电泳1. 在EP管内分别加入A. DL1000marker 5μl,染液1μl;B. PCR产物5μl,6×loading buffer1.2μl,染液1μl2. 将样品全部转入胶孔中。
同源重组构建质粒步骤

同源重组构建质粒步骤
同源重组构建质粒是一种常用的基因工程技术,用于将外源基因插入到质粒中。
以下是同源重组构建质粒的一般步骤:
1. 选择合适的质粒载体:根据实验需要选择适当的质粒载体,通常选择含有适当多个限制酶切位点的质粒。
2. 执行酶切反应:将选定的质粒载体进行限制酶切反应,使用限制酶切酶将质粒切割为线性片段。
3. 扩增外源基因:使用PCR等技术扩增所需的外源基因片段,同时设计引物使得其两端带有与质粒载体的酶切位点相同的序列。
4. 接头连接:将扩增的外源基因片段与酶切后的质粒片段进行连接,使用连接酶将二者进行连接。
连接酶可以是T4 DNA连接酶等。
5. 转化宿主细胞:将连接后的质粒导入到合适的宿主细胞中,常用的宿主细胞包括大肠杆菌等。
6. 选取转化子:利用筛选法选择成功转化质粒的宿主细胞,可以使用抗生素等抗性筛选方法。
7. 确认质粒插入:通过PCR、酶切等方法对转化细胞进行筛
选和验证,确认质粒中外源基因的插入情况。
8. 提取质粒:从筛选验证通过的细胞中提取质粒,通常使用质粒提取试剂盒等方法进行质粒提取。
9. 验证质粒:通过测序等方法验证质粒中外源基因的序列,确认构建成功。
以上步骤仅为一般的同源重组构建质粒步骤,具体实验操作可能因实验设计和需求的不同而有所差异。
重组载体构建的方法和步骤

重组载体构建的方法和步骤全文共四篇示例,供读者参考第一篇示例:重组载体是基因工程领域中的一个非常重要的技术手段,通过对DNA序列的重组,可以构建出具有特定功能的载体,进而实现对目标基因的操控和表达。
在生物学研究、新药开发、农业生产等领域都有着广泛的应用。
下面将介绍一下重组载体构建的方法和步骤。
一、选择载体和目标基因在进行重组载体构建之前,首先要选择适合的载体和目标基因。
一般来说,选择的载体应该具有良好的表达性能和较高的复制稳定性,常用的载体有质粒、病毒等。
而目标基因则应该是我们需要研究或者操控的基因,例如编码重要酶类、蛋白质、抗性蛋白等。
二、获取目标基因目标基因的获取可以通过多种方式,包括PCR扩增、基因合成、基因克隆等。
其中PCR扩增是最为常用的方法,通过设计引物,可以在DNA模板上扩增出目标基因的片段,然后进行纯化和检测,确保其纯度和完整性。
三、线性化载体将选择的载体进行线性化处理,通常是通过限制性内切酶将载体切割成线性片段,以便于后续的连接和转化。
线性化后的载体需要经过纯化和鉴定,确保其完整性和纯度。
四、连接目标基因和载体将目标基因和线性化载体连接起来,一般通过DNA连接酶催化反应实现。
连接时需要考虑目标基因和载体的互补性,以确保连接的正确性和稳定性。
连接后的重组载体需要经过转化操作,将其导入到宿主细胞中。
五、转化宿主细胞将构建好的重组载体导入到宿主细胞中,通常通过化学法、电击法、病毒介导等方式实现。
转化后的宿主细胞需要进行筛选、鉴定和培养,确保其稳定表达目标基因,并能够满足后续实验和研究的需要。
六、鉴定和验证对构建好的重组载体进行鉴定和验证是非常重要的一步,可以通过PCR、限制性酶切割、测序等方法进行验证。
同时也需要进行功能性和表达性的分析,确保重组载体的构建是成功的,能够稳定地表达目标基因。
七、应用和拓展构建好的重组载体可以应用于多个领域,如基因治疗、转基因作物、蛋白质表达等。
同时也可以通过改进和拓展构建方法,提高载体的稳定性和表达效率,开拓更广阔的应用领域。
分子生物学重组质粒的制备

实验一感受态细胞的制备一、实验材料大肠杆菌DH5a 、LB培养基、0.1M CaCl2,50ml大Ep管,冰,80%甘油。
二、操作步骤氯化钙法1.将单个菌落或液体菌种(1:50)接种于20mL LB培养液的试管中,37℃振荡(200r/min)培养过夜。
2.取过夜2ml菌体加入100mLLB培养液37℃振荡培养至光密度(OD600)值在0.4-0.5之间。
(约需1h-2h).3.预冷0.1M CaCl2,和2个50ml大Ep管。
4.将细菌培养物分别倒入预先冰浴的无菌50mL大Ep管中,冰浴30min.5.于4℃下离心10min,(4000r/min),弃去上清夜,放于冰浴中。
6.加10mL冰冷的0.1mol/L MgCl2溶液混匀,冰浴30min。
7.4℃下离心10min,(4000r/min),弃去上清夜(吸尽),放于冰浴中。
8.加入2ml 0.1 mol/L CaCl2轻混。
9.分装于1.5ml离心管中,(每支200uL),-80℃储存备用。
1)转化用:200ul/Ep2)保存:180ul+20ul甘油(80%)注意:1.为了获得高感受态细胞,应选用处于生长对数期的细胞,OD600值不应高于0.6,并且在整个试验过程中均需将细胞置于冰上。
2.细胞在冰冻后即获得了感受性,在-80℃几小时后感受性达到最高值,几个月不会改变。
实验二质粒DNA的小量提取一、试剂及溶液LB培养基、葡萄糖(1)用于碱法提取质粒DNA的溶液a.溶液Ⅰ(GET)缓冲液(50ml):葡萄糖0.49g,0.5M的EDTA(pH8.0)1ml,1M的Tris-HCl(pH8.0)1.25ml。
b. 溶液Ⅱ(变性液):(现用现配)c. (50ml)]:60mL的5M的KAc,11.5mL冰醋酸,28.5mL H2O. PH4.8。
(2)缓冲液TBE缓冲液(10Χ):称取Tris 108g,硼酸55g,5M EDTA(Ph8.0)40ml,用H2O定容到1000mL。
质粒构建小窍门

重组质粒构建是常用的分子生物学手段,其实只是最基本的方法,一般一个星期同时构建三二个组质粒是没有问题的。
在国内先进的实验中,也大都是由实验员搞定。
但是其中还是有些基本的技巧需要掌握。
在这里将我的心得分享于大家,这也是我本人几年来一线工作时的经验积累,以期能为谷友提供借鉴,让大家在实验中少走弯路。
所涉及内容如下:1) 克隆基因的酶切位点问题2) 载体酶切的问题3) 连接片段浓度比的问题在阐明上述问题同时,本人尽可能举些实验中的问题案例予以说明。
一、克隆基因的酶切位点问题1、克隆位点选择的问题。
首先要对目标基因进行酶切位点扫描分析,列出其所含酶切位点清单。
然后对照质粒多克隆位点,所选择的克隆位点必须是目标基因所不含的酶切位点。
这是常识,不赘述。
2、保护碱基数目的问题。
在设计PCR引物时,引入酶切位点后,常常要加入保护碱基,这是大家所熟知的。
但是保护碱基数量多少,可能被新手所忽视。
这种忽视碰可能会大大影响后续的实验进展。
一般情况下,普通的内切酶只加入两个保护碱基,其内切反应就可以正常进行;而有一类,仅仅只加入两个保护碱基,其内切反应就不能正常进行,这是因为内切酶不能正常结合DN段上。
如NdeI就属这类,需要加入至少6个保护碱基,常用的HindIII也要三个。
下面是我提供这类酶的列表及其所需最少的保护碱基数,相信下列将有助于大这家的实验设计。
NcoI 4NdeI 6NheI 3NotI 8PmeI 6SacI 3SalI 3SmaI 3HindIII 3BstI 8SphI 4XhoI 3XbaI 3SmaI 4案例分析一:本人最初曾选用NdeI克隆位点,未注意到保护碱基数目的问题,设计PCR引物时,引入NdeI酶切位点后,只加上两个保护碱基,一个月内没有进展,始终不能成功构建重组载体。
后查文献得知症结所在,在NdeI序列后加上六个保护碱基后,迎刃而解。
大家引以为戒啊。
现在普通酶我都引入三个保护碱基。
现在碱基合成价格也不贵了,为保证酶切充分,连接顺利,不用节约那点钱,再说若一次不成功,重复实验花费时间与金钱更多,孰利孰弊,不言自明。
实验报告构建重组质粒(3篇)

第1篇一、实验目的1. 学习DNA体外重组技术的原理和方法。
2. 掌握重组质粒的构建、转化、筛选和鉴定技术。
3. 熟悉实验操作步骤和注意事项。
二、实验原理DNA体外重组技术是指将外源DNA片段与载体DNA片段在体外进行连接,形成重组DNA分子的技术。
通过该技术,可以将外源基因导入宿主细胞,实现基因表达和功能研究。
三、实验材料1. 载体:pUC19质粒2. 目的基因:基因片段3. 限制性核酸内切酶:EcoRI、BamHI4. DNA连接酶:T4 DNA连接酶5. DNA聚合酶:Klenow片段6. 琼脂糖凝胶电泳试剂7. DNA标记物8. 转化试剂:CaCl2、SDS、乙醇9. 受体细胞:大肠杆菌DH5α四、实验步骤1. 目的基因的扩增(1)设计引物:根据目的基因序列设计一对引物,引物长度一般为20-25bp,5'端添加EcoRI和BamHI酶切位点。
(2)PCR扩增:以基因片段为模板,引物为引物,进行PCR扩增。
2. 载体的酶切(1)将pUC19质粒和PCR扩增的目的基因分别用EcoRI和BamHI进行酶切。
(2)回收酶切片段:琼脂糖凝胶电泳分离酶切片段,回收目的片段。
3. DNA连接(1)将回收的目的基因片段和载体片段混合,加入DNA连接酶,进行连接反应。
(2)将连接产物转化到大肠杆菌DH5α感受态细胞中。
4. 转化后细胞的培养与筛选(1)将转化后的细胞在含有氨苄青霉素的LB培养基中培养过夜。
(2)取适量菌液进行PCR检测,筛选出阳性克隆。
5. 阳性克隆的鉴定(1)提取阳性克隆的质粒DNA。
(2)用EcoRI和BamHI进行酶切,琼脂糖凝胶电泳检测酶切片段。
(3)将酶切片段与标记物进行对比,验证重组质粒的正确性。
五、实验结果与分析1. PCR扩增结果:在PCR产物中,可见一条与预期片段大小相符的条带。
2. 酶切结果:在琼脂糖凝胶电泳中,可见目的基因片段和载体片段的酶切片段。
3. 阳性克隆的鉴定:在琼脂糖凝胶电泳中,可见与预期片段大小相符的条带,证明重组质粒构建成功。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
5'…G↓AATTC…3' →5'… G AATTC…3' 3'…CTTAA↑G …5' →3'… CTTAA G…5'
DNA纯度、缓冲液、温度条件及限 制性内切酶本身都会影响限制性内切酶 的活性。大部分限制性内切酶不受RNA 或单链DNA的影响。当微量的污染物进 入限制性内切酶贮存液中时,会影响其进 一步使用,因此在吸取限制性内切酶时,每 次都要用新的吸管头。如果采用两种限 制性内切酶,必须要注意分别提供各自的 最适盐浓度。若两者可用同一缓冲液,则 可同时水解。若需要不同的盐浓度,则低 盐浓度的限制性内切酶必须首先使用,随 后调节盐浓度,再用高盐浓度的限制性内 切酶水解。
也可在第一个酶切反应完成后,用等体积酚/氯仿 抽提,加0.1倍体积3mol/L NaAc和2倍体积无水乙 醇,混匀后置-70℃低温冰箱30分钟,离心、干燥 并重新溶于缓冲液后进行第二个酶切反应。 DNA限制性内切酶酶切图谱又称DNA的物理 图谱,它由一系列位置确定的多种限制性内切酶酶 切位点组成,以直线或环状图式表示。在DNA序 列分析、基因组是不可缺少的环节,近年来发展起来的 RFLP(限制性片段长度多态性)技术更是建立在它 的基础上。
Ⅱ类中的限制性内切酶在分子克隆中 得到了广泛应用,它们是重组DNA的基础。 绝大多数Ⅱ类限制酶识别长度为4至6个核 苷酸的回文对称特异核苷酸序列(如 EcoRⅠ识别六个核苷酸序列:5'G↓AATTC-3'),有少数酶识别更长的序列 或简并序列。
Ⅱ类酶切割位点在识别序列中, 有的在对称轴处切割,产生平末端的 DNA片段(如SmaⅠ:5'-CCC↓GGG3');有的切割位点在对称轴一侧,产生 带有单链突出末端的DNA片段称粘 性未端, 如EcoRⅠ切割识别序列后 产生两个互补的粘性末端。
生物技术大实验—实验四
重组质粒载体的构建
• 限制性酶切概述 • DNA片段的回收 • 重组质粒的构建 • 实验仪器设备 • 实验试剂 • 实验步骤 • 注意事项
一、限制性酶切概述 限制性内切酶能特异地结合于一段 被称为限制性酶识别序列的DNA序列之 内或其附近的特异位点上,并切割双链 DNA。
它可分为三类:Ⅰ类和Ⅲ类酶在同一 蛋白质分子中兼有切割和修饰(甲基化)作 用且依赖于ATP的存在。Ⅰ类酶结合于 识别位点并随机的切割识别位点不远处 的DNA,而Ⅲ类酶在识别位点上切割 DNA分子,然后从底物上解离。Ⅱ类由两 种酶组成: 一种为限制性内切核酸酶(限 制酶),它切割某一特异的核苷酸序列; 另 一种为独立的甲基化酶,它修饰同一识别 序列。
4、 观察DNA离不开紫外透射仪,可是紫外光对 DNA分子有切割作用。从胶上回收DNA时,应 尽量缩短光照时间并采用长波长紫外灯(300360nm),以减少紫外光切割DNA。 5、 EB是强诱变剂并有中等毒性,配制和使用时 都应戴手套,并且不要把EB洒到桌面或地面上。 凡是沾污了EB的容器或物品必须经专门处理 后才能清洗或丢。 6、 当EB太多,胶染色过深,DNA带看不清时,可将 胶放入蒸馏水冲泡,30分钟后再观察。
二、DNA片段回收 质粒、噬菌体等经酶切、电泳; PCR产物经电泳后,常常需要对一 些DNA电泳片段进行回收和纯化, 用于亚克隆、探针标记等。DNA回 收和纯化常用方法有压碎法、低融 点琼脂糖法、冻融法等,也有现成 的试剂盒供应。
三、重组质粒的构建
将经过酶切、回收纯化后的 PCR产物与质粒载体用T4DNA连 接酶连接的过程称为重组质粒载 体的构建。
四、实验仪器设备
水平式电泳装置,电泳仪,台式高速 离心机, 恒温水浴锅, 微量移液枪, 微波炉或电炉,紫外透射仪, 照相支 架, 照相机及其附件。
五、实验试剂 1、 6×电泳载样缓冲液:0.25% 溴粉 蓝,40%(w/v) 蔗糖水溶液,贮存于 4℃。 2、 溴化乙锭(EB)溶液母液:将EB配制 成10mg/ml,用铝箔或黑纸包裹容器, 储于 室温即可。
六、实验步骤
1、 将清洁干燥并经灭菌的eppendorf管(最好0.5ml) 编号,用微量移液枪分别加入DNA 1μg和相应的 限制性内切酶反应10×缓冲液2μl,再加入重蒸水 使总体积为19μl,将管内溶液混匀后加入1μl酶 液,用手指轻弹管壁使溶液混匀,也可用微量离心 机甩一下,使溶液集中在管底。此步操作是整个 实验成败的关键,要防止错加,漏加。使用限制 性内切酶时应尽量减少其离开冰箱的时间,以免 活性降低。
构建DNA限制性内切酶图谱有许多 方法。通常结合使用多种限制性内切酶, 通过综合分析多种酶单切及不同组合的 多种酶同时切所得到的限制性片段大小 来确定各种酶的酶切位点及其相对位置。 酶切图谱的使用价值依赖于它的准确性 和精确程度。
在酶切图谱制作过程中,为了获得 条带清晰的电泳图谱,一般DNA用量 约为0.5-1μg。限制性内切酶的酶解反 应最适条件各不相同,各种酶有其相应 的酶切缓冲液和最适反应温度(大多数 为37℃)。对质粒DNA酶切反应而言, 限 制性内切酶用量可按标准体系1μg DNA加1单位酶,消化1-2小时。但要完 全酶解则必须增加酶的用量,一般增加23倍,甚至更多,反应时间也要适当延长。
2、 混匀反应体系后,将eppendorf管置于适 当的支持物上(如插在泡沫塑料板 上),37℃水浴保温2-3小时,使酶切反应 完全。 3、 每管加入2μl 0.1mol/L EDTA(pH8.0), 混匀,以停止反应,置于冰箱中保存备用。
注意事项
1. 酶切时所加的DNA溶液体积不能太大,否则DNA溶液中其他成 分会干扰酶反应。 2、酶活力通常用酶单位(U)表示,酶单位的定义是:在最适反应 条件下,1小时完全降解1mg lDNA的酶量为一个单位,但是许多 实验制备的DNA不象lDNA那样易于降解,需适当增加酶的使用 量。反应液中加入过量的酶是不合适的,除考虑成本外,酶液中 的微量杂质可能干扰随后的反应。 3、市场销售的酶一般浓度很大,为节约起见,使用时可事先用酶 反应缓冲液(1×)进行稀释。另外,酶通常保存在50%的甘油 中,实验中,应将反应液中甘油浓度控制在1/10之下,否则,酶 活性将受影响。
