EcoR I
基因工程简答题

(1)修复反应,制备平末端
可用Klenow酶修复限制性内切核酸酶或其他方法产生的5'或3'突出末端,制备平末端,这样可以使原来具有不相容的黏性末端的DNA片段通过平末端重组。如在反应系统中加入放射性同位素标记的脱氧核苷酸,用这种末端填补的方法可以制备3'末端标记的探针。
答:
基因型是lac-.原因是在该密码子中插入了两个碱基,造成了移码突变,所以Lac的基因是缺陷的
27.在基因工程中,为了在细菌细胞中表达真核生物的基因产物,为什么通常要用cDNA
而不用基因组DNA?为什么要在cDNA前加上细菌的启动子?
答:
这是因为细菌没有内含子剪接系统,并且不能识别真核生物的启动子之故。
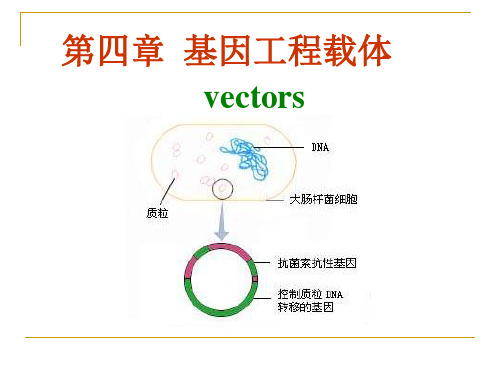
10. 列举质粒载体必须具备的4个基本特性。
.答:
(1)独立复制; (2)有选择标记; (3)有独特的酶切位点; (4)能转化但不扩散。
11. 什么叫穿梭载体?
答:
含有细菌质粒和克隆的真核生物DNA片段的杂种质粒,有两个复制起点和既能在细菌又能在真核细胞中进行选择的选择标记,所以,很容易从一宿主转到另一个宿主(来回穿梭)。
条件的改变会出现第二活性的酶的右上角加一个星号表示,因此第二活性又称为星
活性。
概括起来,诱发星活性的因素有如下几种:(1)高甘油含量(>5%, v/v);(2)限制性
内切核酸酶用量过高(>100U/ugDNA);(3)低离子强度(<25 mmol/L);(4)高pH(8.0
如果你的杂交探针是双链的,可能因为在加人杂交混合物之前忘记将探针变性而得到空白的放射自显影结果。
23.切口移位(nick translation)标记探针的主要步骤有哪些?
载脂蛋白B基因第29外显子EcoRI位点基因变异与冠心病、脑梗死易感性的关系

20 0 7年 第 1 0卷 第 1 期
. 。 。
广西 中医学 院学报
・5 ・
●
临 床 研 究
;
{
t _ ・ _ . . 州, ・¨・ . l ・ ・ ・ . , ◆¨ H ・ .
载 脂 蛋 白 B基 因第 2 9外 显 子 E o I 点 基 因 cR 位 变 异 与 冠 心病 、 梗 死 易 感 性 的关 系 脑
t e ae fn i d a muainw ss nfa t ieet ew e h nrl ru dteC ru P<0 0 ) [ o c s n i t o dv u tt a g icnl df r tentec t o pa HD go p( rr i i l o i i y f nb o og n h .5 . C nl i ] uo
W EIYu 1n ,Z . HONG e—a 1 HUANG n m ig ,Z NG a - u 2 U h n —u a W i n, g Ya - n 2 E Xio y n ,S S a g g i
( . a g i C Unvri ,Na nn a g i 5 0 0 ; 1Gu n x M ies y T t n i Gun x, 3 0 1 g
韦玉兰 钟 卫 干 黄 艳明 曾小 云 苏上贵 。 , , ,
(. 1 广西 中医学院, 广西 南宁 5 00 ; . 3 0 12 广西 医科大学, 广西 南宁 5 02 ) 3 02
摘 要 : 目 ] 讨载脂蛋 白 B基 因第 2 [ 的 探 9外显子 E 0 I 点基 因变 异与 冠心病 、 cR 位 脑梗 死 易感性 的关 系。[ 法] 用 方 采
r p c iey s e e t l .Th o iie r t fi dv d a t t n we e6 3 5 - 0% -1 . 7 v ep st a e o ii u l v s n mu a i r . 1 % 2 o 7 7 8% f rt e t re g o p ,r s  ̄t dy o h h e r u s ep i v .Th o ep d・
基因工程第四章载体
(4) 插入失活型质粒载体
载体的克隆位点位于其某一个选择性 标记基因内部。
如pDF41、pDF42、pBR329。
外源DNA
抗菌素抗性
无抗菌素抗性
(5)正选择的质粒载体 Direct selection vectors
直接选择转化后的细胞。
只有带有选择标记基因的转化菌细胞才 能在选择培养基上生长。
如pUR2、pTR262等。
目前通用的绝大部分质粒载体都是正 选择载体。
(6) 表达型质粒载体
主要用来使外源基因表达出蛋白质产物。
注意启动子的性质,终止子、起始 密码、终止密码的阅读正确。
如果在大肠杆菌里表达,必须把所克隆的 真核生物的基因置于大肠杆菌的转录—翻 译信号控制之下。
表达载体的结构
1)普通载体元件
b)细菌抗性原理 Ampr基因编码-内酰胺酶,特异地 切割氨苄青霉素的-内酰胺环。
ii)氯霉素(chloramphenicol,Cml)
a)抑菌原理 通过与50S核糖体亚基结合,干扰细胞 蛋白质的合成并阻止肽键的形成。杀死 生长的细菌。
b)细菌抗性原理
Cmlr 编码乙酰转移酶,特异地使氯霉 素乙酰化而失活。
(2)长度 6.3 kb。
(3)选择标记
大肠杆菌素(colicin)E1和对E1免疫 的基因(immE1)
① colicin E1基因的结构
cea 结构基因
imm
kil
免疫基因 溶菌基因
② 杀死不含有ColE1细菌的原因 cea + kil基因产物
③ 不被其他细菌的colicin E1所杀死的原因 imm基因
① 双抗菌素抗性选择标记 插入失活,分两次先后选择: 没有获得载体的寄主细胞 在Amp或Tet中都死亡。
酶切鉴定-BamHI and EcoRI

注意事项
1. 直接在抽提的质粒 直接在抽提的质粒DNA管中加入酶切体系。 管中加入酶切体系。 管中加入酶切体系 2. 为使微量操作更精确,可以4个人(8 tubes)一起做 为使微量操作更精确,可以 个人 个人( )一起做9 体系混合液,然后再分装10ul于各质粒管中。 于各质粒管中。 份酶切 体系混合液,然后再分装 于各质粒管中 3. 上样时要小心操作,防止样品溢出。 上样时要小心操作,防止样品溢出。 4. 接触凝胶时,因其中含有EB或荧光染料,要戴手套, 接触凝胶时,因其中含有 或荧光染料 要戴手套, 或荧光染料, 废物丢弃要在固定位置。不接触凝胶时不需戴手套! 废物丢弃要在固定位置。不接触凝胶时不需戴手套! 5. EB是极强致癌物!小心! 是极强致癌物!小心! 是极强致癌物
限制性内切 酶切割DNA 酶切割
A: Strategy of construction
f1 ori Ampr pGEX Vector (4900 bp)
BamH I EcoR I
B: The product of PCR
1 2 3
4900 bp
X-gene cDNA
Primer1
740 bp
Primer2
体积( ) 体积(µl) 10 2 1 1 1 5 20
酶切一小时后,每管加入 酶切一小时后,每管加入5 ul的6xloading 的 buffer,中止反应,混匀。 ,中止反应,混匀。 20~25 ul上样,在一个泳道中加入 ul DNA 上样, 上样 在一个泳道中加入10 Marker。 。
核酸电泳 根据核酸的解离性质, 根据核酸的解离性质,用中性或偏碱性的缓冲液使核 酸解离成阴离子,置于电场中便向阳极移动, 酸解离成阴离子,置于电场中便向阳极移动,这就是 电泳。 电泳。 凝胶电泳有许多优点:简单、快速、灵敏、成本低。 凝胶电泳有许多优点:简单、快速、灵敏、成本低。 常用的凝胶电泳有琼脂糖 琼脂糖(agarose)凝胶电泳和聚丙烯 凝胶电泳和 常用的凝胶电泳有琼脂糖 凝胶电泳 酰胺(poly-acrylamide)凝胶电泳。可以在水平或垂直 凝胶电泳。 酰胺 凝胶电泳 的电泳槽中进行。 的电泳槽中进行。凝胶电泳兼分子筛和电泳双重效 果,所以分离效率很高。 所以分离效率很高。
takara-EcoRI

Eco R IG A A T T C C T T A A GV2010.07TaKaRa Code: D1040A包 装 量: 4,000 Units附带试剂: 10×H Buffer 500 μl 10×Loading Buffer 500 μl纯 度:1) Overdigestion Test :≥12 Units2) Ligation-Recutting Test : Ligation Effi.:100%, Recutting Effi.:100% 3) pKF3 Cloning Test :<2%●酶贮存液:10 mM Tris-HCl, pH7.5100 mM KCl 0.1 mM EDTA 1 mM DTT 0.15 % TritonX-100 0.01 % BSA 50 % Glycerol●起 源:Escherichia coli RY13●一般反应体系:Eco R I 1 μl10×H Buffer 2 μlDNA ≤ 1 μg灭菌水 up to 20 μl●反应温度:37℃●活性确认:在50 μl 反应液中,37℃温度下反应1小时,将1 μg 的λ DNA 完全分解的酶量定义为1个活性单位(U)。
●纯度检测: 1) Overdigestion Test:在1 μg DNA 中加入过量的该限制酶,进行长时间(24小时)酶切反应,然后进行琼脂糖电泳,确认切出的DNA 片段的电泳谱带不发生变化。
2) Ligation-Recutting Test:在经过10倍量该酶切出的DNA 片段中,加入 T4 DNA Ligase,使其连接,然后再使用该酶进行切断反应,判断Ligation-Recutting 效率。
3) pKF3 Enforcement Cloning Test:使用10倍量的该酶,将Enforcement Cloning Vector pKF3 DNA 切开,然后再进行连接后,转化至TH2感受态细胞中,判断该酶切位点受到影响的重组体所占的比率。
NEB EcoRI

EcoRI 收藏货号规格价格#R0101L 50,000 units 1,909.00元#R0101M (高浓度5X)50,000 units 1,909.00元#R0101S 10,000 units 429.00元#R0101T (高浓度5X)10,000 units 429.00元#R0101V 5,000 units 219.00元识别位点∙isoschizomers |∙compatible ends |∙single letter code在不同反应缓冲液的活性NEBuffer 1: 100%NEBuffer 2: 100%NEBuffer 3: 100%NEBuffer 4: 100%酶的特性浓度:20,000 和100,000 units/ml(通过λ DNA鉴定)。
反应缓冲液:(随酶提供)NEBuffer EcoRI。
反应温度:37°C温育。
热失活:65°C 20 分钟。
概述来源:重组E. coli 菌株,包含有从E. coli RY 13(R.N. Yoshimori)克隆的EcoRI 基因。
连接和再切:经过EcoRI 100 倍过量消化,> 95%的DNA 片段能被连接和再切。
贮存条件:贮存于-20°C (含50%甘油)。
稀释兼容性:稀释液C。
甲基化敏感性:对哺乳动物CpG 甲基化敏感。
省时酶切:5 分钟能消化λDNA。
注意事项1.如下条件可能导致星号活性:低离子强度、高酶浓度、甘油浓度> 5% 或pH 值> 8. 0。
2.用EcoRI 做双酶切。
EcoRI限制性内切酶的

什么是黏性末端?为什么叫黏性末端? 被限制酶切开的DNA两条单链的切口,带
有几个伸出的核苷酸,他们之间正好互补配对, 这样的切口叫黏性末端。
DNA连接酶——“分子缝合针”
1. 常用种类 2. 作用:
E.coli DNA连接酶 T4 DNA连接酶
形成磷酸二酯键 E.coli DNA连接酶
E.coli DNA连接酶把黏性末端之间的缝隙 “缝合” 起来,即把梯子两边扶手的断口连接起来,这样一 个重组的DNA分子就形成了。
SmaI限制酶的作用特点是什么?
什么Байду номын сангаас平末端?
当限制酶从识别序列的中心轴线处切 开时,切开的DNA两条单链的切口,是 平整的,这样的切口叫平末端。
《基因工程与细胞工程》质粒DNA的限制性酶切及琼脂糖凝胶电泳鉴定酶切图谱实验

《基因工程与细胞工程》质粒DNA的限制性酶切及琼脂糖凝胶电泳鉴定酶切图谱实验【实验目的】1、掌握实用的分子生物学基本操作技术;2、提高处理DNA样品的操作技能;3、学会使用限制性内切酶对DNA样品进行酶切;4、学会配制琼脂糖凝胶;5、学会使用电泳技术分析和鉴定DNA分子。
【实验原理】1、质粒DNA的限制性酶切DNA的酶法操作是DNA重组技术中一项最常用的工具。
特别是一系列限制性内切核酸酶的使用,能够在特异性位点切割DNA,对从分子水平上认识基因的结构与功能和进行重组DNA技术研究是非常有用的。
限制性内切酶来源于细菌,能够在特异性的目标序列中(即限制性酶切位点)切割双链DNA,从而产生特定的DNA片段(即限制性酶切片段)。
内切酶是细菌限制与修饰体系中的一员,能够使细菌细胞免受外源性DNA的侵害,即通过切割噬菌体DNA中的特异性位点来限制细菌噬菌体的繁殖,从而抑制噬菌体对细菌细胞内的入侵。
细菌通过修饰限制酶的识别位点来防止限制酶破坏其自身的DNA,通常是利用对识别位点中1个碱基的甲基化修饰来实现的。
历年来,从细菌细胞内分离纯化的限制性内切酶的种类在不断增加,并越来越多的被分子生物学家应用到DNA的体外操作中。
每种限制酶都能识别1段特异的DNA序列,其中最常见的是长度为4-6 bp的回文序列(反向重复序列)。
同时,不同种类的限制酶在识别的切割位点是不同的,有些可能是在识别位点的中间切开,产生平末端(钝末端);而另一些限制酶可能是将识别位点错位切开,生成5’或3’突出末端(黏性末端)。
表2列举了本实验中所使用的2种限制酶的识别位点和切割位点。
表2 2种限制酶的识别位点和切割位点注:↓或↑:表示酶切位点。
2、DNA限制性内切酶酶切图谱(1)图谱简介DNA限制性内切酶酶切图谱,又称DNA的物理图谱,它由一系列位置确定的多种限制性内切酶酶切位点组成,以直线或环状图式表示。
在DNA序列分析、基因组的功能图谱绘制、DNA的无性繁殖、基因文库的构建等工作中,建立限制性内切酶图谱都是不可缺少的环节,近年来发展起来的RFLP(限制性片段长度多态性)技术更是建立在它的基础上。
质粒pBR322DNA与内切酶EcoRI相互作用的研究

EoI cR 的浓度为 5 nt L iu u -
DN 内切 酶 复 合 物 的 切 割 图 像 A一
E o I 浓 度 为 75 ntt。 cR 的 .u ir ・ L
\
\
丑
妞
丑
I Ⅱ I
D
E
F
切割后不同 D NA结构的统计图像( D、E、F分别与上面 的 A、B、C对应) 图 1 有 Mg 存在 的条件时。内切酶 E o I DN 的切割作用 图 cR 与 A
增加.
关键词 :D NA; 内切酶;切割 ;结合
中 图分 类 号 :Q6 3 3 文 献 标 志 码 :A 文 章 编 号 : 17 —5 32 1)50 3 .6 643 6 (0 10 —0 20 本文 的 P DF文件 可 以从 x ea .Z . uc 得 ub oW Ue . d a获
内切酶 浓度 增加 对切 割 率 的影 响不 是很大 ,当 内切 酶 的浓 度大 于 5 nt i u ・ 时 ,D NA 被切 割 的 比
3 4
例 明显增 加 .
温州大学学报 ・ 自然科学版 ( 1) 3 卷第 5 2 1第 2 0 期
A
B
C
Eo1 cR 的浓度 为 25u i . nt -
异性作用与非特异性作用 的热动力学系数 ,18年 ,Mc l i等人L 确定了E o I N 96 Ca n r l cR 与D A作用复
合物 的具体 结构 ,19 年 ,Wr h[1 又进 一步 提 出 了E o I 99 i t5 g 1等人 cR 与DN A结合与 分 离的动 力 学机制 , 这 些研 究结 果为进 一步 在 云母片 上 面研究 D NA与 内切 酶 的相互 作用 提供 了主要依 据 .
3.1重组DNA技术的基本工具——高二生物学人教版(2019)选择性必修三同步课时训练

3.1重组DNA技术的基本工具——2023-2024学年高二生物学人教版(2019)选择性必修三同步课时训练一、单选题1.同尾酶是指一组识别序列不同,但切出的黏性末端相同的限制酶。
下图为EcoRI、Bam-HI、BglI和MboI四种限制酶的识别序列及酶切位点。
下列说法正确的是( )A.BamHI、BgⅡ、MboI属于同族酶,他们的识别序列相同B.使用同尾酶构建基因表达载体时,切割位点的选择范围扩大C.选用两种不同的限制酶切割目的基因,都可以防止目的基因自身环化D.用DNA连接酶将BamHI和MboI形成的黏性末端连接后,可被二者重新切开2.红细胞生成素(EPO)是体内促进红细胞生成的一种激素物质,是当今最成功的基因工程药物,可用于治疗肾衰性贫血等疾病。
由于天然EPO来源极为有限,目前临床使用的红细胞生成素主要来自基因工程技术生产的重组人红细胞生成素(rhEPO)。
其简要生产流程如图,下列相关叙述正确的是( )A.过程①用到的工具酶有DNA聚合酶、限制酶和DNA连接酶B.构建的重组表达载体中终止子的作用是终止翻译过程C.检测重组细胞是否表达出rhEPO常用抗原—抗体杂交技术D.用乳腺生物反应器生产EPO可将人EPO基因导入哺乳动物体细胞获得3.甲、乙、丙三种限制酶均识别6个核苷酸序列,下列有关如图所示的黏性末端的说法,正确的是( )A.甲、乙、丙三种限制酶识别的核苷酸序列由核糖核苷酸组成B.限制酶切割DNA分子一次可断开2个磷酸二酯键,产生2个游离的磷酸基团C.甲、乙、丙是由不同的限制酶切割产生的相同黏性末端D.甲、乙黏性末端形成的重组DNA分子片段能被甲、乙限制酶切割4.甲型流感病毒的抗原性与感染性与其表面的R蛋白(血凝素蛋白)密切相关,现利用基因工程的方法生产相关疫苗。
图甲为构建R蛋白基因表达载体的过程,图乙为重组质粒被相关酶切后的电泳结果。
下列相关叙述错误的是( )A.电泳技术可以用于人类亲子鉴定、生物间亲缘关系的鉴定B.图甲过程中至少需要用到逆转录酶、DNA聚合酶、限制酶、DNA连接酶C.通过PCR技术从1个cDNA分子中特异性扩增出R蛋白基因时,在第四轮循环产物中开始出现两条脱氧核苷酸链等长的R蛋白基因D.据图乙可推测重组质粒长度最短为200bp,Bam HⅠ在重组质粒上最少有3个酶切位点5.基因编辑是一种基因工程技术,CRISPR/Cas9基因编辑技术,可以实现对DNA的定点切割。
载脂蛋白B基因EcoRⅠ位点多态性对儿童生长发育及营养状况的影响

E + / E 一 组 ;④ 等 位 基 因 E 一 组血清 T P和 A I B水 平 显 著 低 于 E 姐 ( P <0 . 0 5 、< 0 . 0 1 ) ,而 T C、H DL - C及 L D L — C水平 显 著高于 E + 组( P<0 . 0 5 ) ;⑤ 血 清 L DL — C分 别 与 T P及 A I B呈 高 度 负 相 关 ( P<0 . 0 5 ) ,而 H DL — C与 A I B呈 显 著 正 相
Ab s t r a c t : Ob j ct e i v e s T o e x p l o r e t h e e f e c t o f E c o R I l o c u s p o l y m o r p h i s m o f a p o l i p o p r o t e i n B( A p o B )g e n e o n
HU P e ,J I N G C h e n g - x u e ,Q I N Y u a n — h a n ,L I Mi n g - f a n g ,C HE N P £ ,P E I J u a n( D e p a r t m e n t o f P e d i a t r i c s ,T h e F i r s t A il f i a t e dH o s p i t a l o fG u a n g x i Me d w d U n i v e r s i t y ,N a n n i n g 5 3 0 0 2 1 ,G u a n g x i ,C h i n a )
EcoR I 说明书

pUC118 Eco R I/BAP使 用 说 明 书●TaKaRa Code :D3320●包 装 量:5 μg(0.1 OD)●浓 度:50~100 μg/ml●制品说明pUC118 Eco R I/BAP 载体是由pUC118 DNA 经其多克隆位点上的限制酶Eco R I 酶切后,用E.coli 来源的碱性磷酸酶(BAP)去磷酸化后得到的。
这种经BAP 处理过的载体可以防止自身环化,转化时可以降低含有空载体的转化细胞数量。
这种载体使用前不需要任何处理,可直接用于克隆。
●贮存溶液10 mM Tris-HCl(pH8.0),1 mM EDTA。
●保 存: -20℃●制 备使用CsCl-EtBr 密度梯度超离心法纯化的pUC118 DNA,经限制酶Eco R I 消化后,再使用E.coli 来源的碱性磷酸酶(BAP)进行了去磷酸化处理。
●链 长3,162 bp(Messing 等构建方法的计算值)。
●GenBank 登录号pUC118:Accession No. U07649●纯 度1. 用1% Agarose 电泳,显示单一条带。
2. 使用T4 DNA 连接酶对pUC118 Eco R I/BAP进行自身连接时的转化效率是未切断pUC118 转化效率的1%以下。
3. 使用T4 Polynucleotide Kinase将pUC118 Eco RI/BAP的5’-OH末端磷酸化后,再使用T4 DNA Ligase进行自身连接,然后转化至E.coli JM109 Competent Cell (转化效率为:1×107 Colonies/1 μg pUC118)时,其转化效率在1×106 Colonies/1 μg DNA以上,此时在含有IPTG、X-Gal的蓝白筛选平板培养基上的白色菌落数在1%以下。
●使 用 例pUC118 Eco R I/BAP 50 ng(25 fmol) DNA Fragment/Eco R I 25 fmol-250 fmol* DNA Ligation Kit Ver.2.0 (TaKaRa Code:D6022)●参考文献:Vieira, J.and Messing, J. (1987) Methods inEnzymolgy , 153, 3-11.pUC118多克隆位点图V2010.04。
DNA 的限制性内切酶酶切分析

观察酶切后与未酶切质粒的DNA带位置。
100bp DNA Marker 未酶切质粒
已酶切质 粒
点样孔
3000bp 1500bp 1000bp
500bp
2.96kp 1.9kp
注意事项
1. 第一步确保样品加到反应体系中,若有粘壁,用掌型离心机离 心到底部!
EcoR I
Bcl-2 (1.9kb)
操作步骤(改动)
1.酶切:
20ul反应体系
组分
加入体积
灭菌双蒸水
11ul
10×buffer H(含BSA) 2ul
质粒DNA
5ul
10U/ul EcoRI 2ul
掌型离心机混匀,37℃水浴反应1h
2.制胶(0.8%):称取0.48g琼脂糖倒入三角瓶中,加入1×TAE缓 冲液60ml,微波炉加热至沸腾,摇匀,至无颗粒。
3.倒胶:胶冷却至60℃左右(不烫手),缓缓倒入电泳槽 (先胶布 封口,放好梳子)。 4.点样:1 ul含SYBR Green I的载样buffer
+5ul酶切产物 或+5ul 未酶切的质粒, 混匀。 (点样孔置负极端)
5.电泳:稳压条件下90V电泳,待染料(指示过5V/cm胶长度)
6.电泳时电场强度不超过5V/cm胶长
思考题:
1. DNA的纯度会不会影响酶切产物的质量?如果会,请 说明原因。
2. 比较本实验酶切后与未酶切质粒的电泳图谱,综合前 两个实验,分析可能的原因
2. 当样品在37℃ 水浴时,要盖紧盖子,否则水汽进入管内,使 反应体积大大增加,造成酶切失败
3. RE一定要在低温(-20℃)下贮存(含50%甘油) ,每次吸取后 应放在冰盒,用完后立即放在-20℃,新购的大包装酶应先分装
ECoRI限制性内切酶的使用

5
0
1
1
1
1
1
Protocol for Fast Digestion of Different DNA
1. Combine the following reaction components at room temperature in the order indicated:
Water, nuclease-free (#R0581) 10X FastDigest® or 10X FastDigest® Green Buffer DNA FastDigest® enzyme Total volume: Plasmid DNA 15 µl 2 µl 2 µl (up to 1 µg) 1 µl 20 µl PCR product 17 µl 2 µl 10 µl (~0.2 µg) 1 µl 30 µl Genomic DNA 30 µl 5 µl 10 µl (5 µg) 5 µl 50 µl
Description
FastDigest® enzymes are an advanced line of restriction enzymes for rapid DNA digestion. All FastDigest® enzymes are 100% active in the universal FastDigest® and FastDigest® Green buffers and are able to digest DNA in 5-15 minutes. This enables any combination of restriction enzymes to work simultaneously in one reaction tube and eliminates the need for sequential digestions. FastDigest® enzymes can be used to digest plasmid, genomic and viral DNA as well as PCR products and do not show star activity even in prolonged incubations. Enzymes used in common downstream applications such as ligation, blunting and dephosphorylation reactions also have 100% activity in FastDigest® and FastDigest® Green Buffer. FastDigest® Green Buffer includes a density reagent along with blue and yellow tracking dyes that allow for direct loading of the reaction mixtures on a gel. The blue dye of the FastDigest® Green Buffer migrates with 3-5 kb DNA fragments in a 1% agarose gel and has an excitation peak at 424 nm. The yellow dye of the FastDigest® Green Buffer migrates faster than 10 bp DNA fragments in a 1% agarose gel and has an excitation peak at 615 nm. For applications that require analysis by fluorescence excitation FastDigest® Buffer is recommended, as the dyes of the FastDigest® Green Buffer may interfere with some fluorescence measurements.
AFLP分子标记技术的改进——内切酶EcoRⅠ/TruⅠ组合与EcoRⅠ/MseⅠ组合的比较

中 图分 类 号 : 73 1 Q 8 . 文 献 标 识 号 : A 文 章 编 号 :0 1 44 ( 08 0 0 o o 10 — 9 2 2 0 )9— 04一 4
I p o e e t0 m r v m n f AFLP o e u a a k r Te h i u M lc l r M r e c n q e
要: 通过对 E o /Tu1 E o /Ms 两个双酶切组合的 比较 , cRI r 与 cR I eI 证明在 A L F P标记体系中 , 完全
可 以用内切酶 TuI 代昂贵 的内切酶 M eI。E o T I可同时加入 , r 替 s cR I与 m 在不 同温度下分段 进行酶切 : 3 ̄ 7C酶切 4h 然后 6 c , 5c酶切 2h 可达到分步酶切的效果。 ,
Ab ta t I hss d h f c ftersr t n e zme c mbn t n E o I T u IfrAF P wa sr c n ti t y.teef to et ci n y o iai c R / r o L s u e h i o o
山东农 业科 学
20 ,: 69 0894~ ,
AL F P分子 标 记 技术 的改进
内切 酶 E o /TuI 合 与 E o /M eI cRI r 组 cR I s 组合 的 比较
徐 文玲 , 王淑芬 , 牟晋华 , 王翠花 , 贤娴 刘
( 山东省农业科学院蔬菜研究所 , 山东 济南 摘 20 0 ) 5 10
c mp rd w t h o iainEc R I/Ms o ae i tec mbn t o h o eI.I hsmeh d,Ec R Ia dT u 1w r d e g te t n ti to o n r eea d d t eh rwi o h
各种酶切体系

-
2
6
2.4
10×buffer D
2
1
1
1
3
4
BSA(10µg/µl)
0.2
0.1
0.1
0.2
0.6
0.6
DNA(1µg/µl)
8.3
4
7.9
6
18
30
Not I(10U/µl)
0.5
0.3
0.5
0.4
1.2
1.5
Sal I(10U/µl)
0.5
0.3
0.5
0.4
1.2
1.5
反应条件
37℃,1~3hr,电泳检测
NheI单酶切
依DNA量定体系
20µl
10µl
灭菌去离子水
-
-
10×buffer
2
1
DNA(1µg/µl)
17
8.5
Nhe I
1
0.5
反应条件
37℃,1~3hr,电泳检测
Xbal I和Sal I双酶切体系鉴定
依DNA量定体系
20µl
10µl
10µl
灭菌去离子水
8.5
4.3
-
10×buffer
2
1
1
BSA(10µg/µl)
0.2
0.1
0.1
DNA(1µg/µl)
8.3
4
7.9
Xba I(10U/µl)
0.5
0.3
0.5
Sal I(10U/µl)
0.5
0.3
0.5
反应条件
EcoR I酶切鉴定
组分
体系
质粒DNA
常用限制性内切酶酶切位点保护残基

酶切位点保护碱基-PCR引物设计用于限制性内切酶发布: 2010-05-24 20:19| 来源:生物吧| 编辑:刘浩| 查看: 161 次本文给出了分子克隆中常用限制性内切酶的保护碱基序列,如AccI,AflIII,AscI,AvaI,BamHI,BglII,BssHII,BstEII,BstXI,ClaI,EcoRI,HaeIII,HindIII,KpnI,MluI,NcoI,NdeI,NheI,NotI,NsiI,PacI,PmeI,PstI,PvuI,SacI,SacII,SalI,ScaI,SmaI,SpeI,SphI,StuI,XbaI,XhoI,XmaI,为什么要添加保护碱基?在分子克隆实验中,有时我们会在待扩增的目的基因片段两端加上特定的酶切位点,用于后续的酶切和连接反应。
由于直接暴露在末端的酶切位点不容易直接被限制性核酸内切酶切开,因此在设计PCR引物时,人为的在酶切位点序列的5‘端外侧添加额外的碱基序列,即保护碱基,用来提高将来酶切时的活性。
其次,在分子克隆实验中选择载体的酶切位点时,相临的两个酶切位点往往不能同时使用,因为一个位点切割后留下的碱基过少以至于影响旁边的酶切位点切割。
该如何添加保护碱基?添加保护碱基时,最关心的应该是保护碱基的数目,而不是种类。
什么样的酶切位点,添加几个保护碱基,是有数据可以参考的。
添加什么保护碱基,如果严格点,是根据两条引物的Tm值和各引物的碱基分布及GC含量。
如果某条引物Tm值偏小,GC%较低,添加时多加G或C,反之亦反。
为了解不同内切酶对识别位点以外最少保护碱基数目的要求,NEB采用了一系列含识别序列的短双链寡核苷酸作为酶切底物进行实验。
实验结果对于确定双酶切顺序将会有帮助(比如在多接头上切割位点很接近时),或者当切割位点靠近DNA末端时也很有用。
在本表中没有列出的酶,则通常需在识别位点两端至少加上6个保护碱基,以确保酶切反应的进行。
实验方法:用γ-[32P]ATP在T4多聚核苷酸激酶的作用下标记0.1A260单位的寡核苷酸。
常用同位酶问题回答

常用同位酶同位酶是一种可以切割或识别DNA的酶类。
在DNA分析、遗传学和进化研究中,同位酶有着广泛的应用。
常用的同位酶包括EcoRI、HaeIII、HindIII、BamHI等。
1. EcoRIEcoRI是最常用的同位酶之一,它的切割序列为GAATTC。
EcoRI可以切割DNA的双链,从而将其切割成两个长链。
由于EcoRI的切割序列相对较短,因此在DNA片段较小的实验中,其效果比较明显。
而在大片段的DNA分析中,则需要配合其他酶类。
2. HaeIIIHaeIII是另一种常用的同位酶,它的切割序列为GGCC。
HaeIII可以切割DNA的双链,从而将其切割成两个短链。
HaeIII的切割序列长度较短,因此在DNA片段较小的实验中其效果更好。
与EcoRI一样,在大片段的DNA分析中,HaeIII也需要配合其他酶类。
3. HindIIIHindIII也是一种常用的同位酶,它的切割序列为AAGCTT。
HindIII可以切割DNA的双链,从而将其切割成两个长链。
与EcoRI相比,HindIII的切割序列长度更长。
这意味着,HindIII在大片段的DNA分析中,其效果更好。
4. BamHIBamHI是一种从Bacillus amyloliquefaciens分离出来的同位酶,它的切割序列为GGATCC。
BamHI可以切割DNA的双链,从而将其切割成两个短链。
BamHI的切割序列长度较短,因此在DNA片段较小的实验中其效果更好。
与EcoRI、HaeIII和HindIII相比,BamHI使用的情况并不常见。
总体而言,常用的同位酶主要包括EcoRI、HaeIII、HindIII和BamHI。
每一种酶类都有其特定的切割序列和使用情况。
熟练掌握各种同位酶的使用方法和剪切条件,对于DNA分析和遗传学研究都具有重要意义,也是科学家们在实验中取得成功所必不可少的一步。
ecori名词解释

"ECORI"是一个生物学中常用的术语,它代表了一种特定的酶。
ECORI是一种限制性内切酶(restriction endonuclease),也被称为EcoRI酶。
限制性内切酶是一类能够识别DNA分子中特定的核酸序列,并在该序列上切割DNA链的酶。
ECORI 酶属于E. coli(大肠杆菌)中发现的一类限制性内切酶。
ECORI酶的作用是识别并切割DNA中的G/AATTC序列,切割后产生两个黏性末端(sticky ends),这些末端具有未配对的碱基,可以与其他DNA分子的相应末端互相结合。
ECORI酶的活性可用于DNA重组、DNA克隆等分子生物学技术中。
ECORI酶在分子生物学研究和实验室操作中被广泛使用,它是一种常见而重要的限制性内切酶,有助于DNA分析、基因工程和遗传学研究等领域的进展。
生物技术概论试题

生技28、查找阅读有关生物技术的英文文献并翻译成中文一、名词解释Plasmid目的基因单克隆抗体技术细胞融合基因库问答题:1.疾病基因治疗有哪四大策略,肿瘤的基因治疗主要有哪两种策略?2.从cDNA文库和基因文库中获得目的基因有什么不同?3.如何从一片嫩叶经组织培养培育出众多的完整植株?4.开展干细胞研究对人类有何积极的意义?5.为什么说干细胞的应用将具有广泛的前景?6.人类基因组计划任务是什么?将解决什么问题?它对医学的发展有什么影响?7.生物法处理污水或废水有哪几种常见方法?污水治理的意义何在?8.空气污染治理与水污染治理有什么关系,常见的方法有哪几种9.什么事生物修复技术,方法,举例说明其应用价值10. 生物技术对经济社会发展的影响,试举例说明。
第二部分综合练习一、单项选择题4. 下面哪个不是生物技术包括的基本内容?细胞工程基因工程遗传工程酶工程1、现代生物技术是一项高新技术,它具有高新技术的“六高”特征,下面哪个不属于“六高”?A、高效益B、高风险C、高势能D、高效率2、生物技术是以下面哪个为核心?A、基因工程B、细胞工程C、酶工程D、发酵工程3、分子克隆主要是指A、DNA的大量复制B、DNA的大量转录C、DNA的大量剪切D、RNA的大量反转录4、多数限制性核酸内切酶切割后的DNA末端为A、平头末端B、3突出末端C、5突出末端D、粘性末端5、设计聚合酶链反应的引物时,应考虑引物与模板的A、5‘端特定序列互补B、5’端任章序列互补C、3‘端特定序列互补D、3’端任意序列互补6、用于鉴定转化于细胞是否含重组DNA的最常用方法是A、抗药性选择B、分于杂交选择C、RNA反转录D、免疫学方法7、下列哪些是发酵技术独有的特点A、多个反应不能在发酵设备中一次完成B、条件温和、能耗少、设备简单C、不容易产生高分子化合物D、发酵过程中不需要防止杂菌污染8、不是工业生产上常用的微生物为A、担子菌B、细菌C、酵母菌D、霉菌9、机械搅拌发酵罐与通风搅拌发酵罐相比,下列不属于前者特点的是A、发酵罐内没有搅拌装置,结构简单B、发酵罐内有搅拌装置,混合速度快C、耗能少,利于生产D、发酵罐内没有搅拌装置,结构复杂10、从微生物细胞制备酶的流程一般包括破碎细胞、溶剂抽提、离心、过滤、干燥这几个步骤。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
Eco R I
G A A T T C
C T T A A G
TaKaRa Code:D1040A
包 装 量:4,000 Units
附带试剂: 10×H Buffer 500 μl
10×Loading Buffer 500 μl
纯 度:
1) Overdigestion Test:≥12 Units
2) Ligation-Recutting Test:
Ligation Effi.:100%,Recutting Effi.:100%
3) pKF3 Cloning Test:<2%
●酶贮存液:
10 mM Tris-HCl, pH7.5
100 mM KCl
0.1 mM EDTA
1 mM DTT
0.01 % BSA
0.15 % TritonX-100
50 % Glycerol
●起 源:Escherichia coli RY13
●一般反应体系:
Eco R I 1 μl
10×H Buffer 2 μl
DNA ≤1 μg
灭菌水 up to 20 μl
●反应温度:37℃
●反应时间:
在上述20 μl的反应体系中,37℃反应5分钟可以完全切断λDNA,满足各种实验需求。
针对特殊酶切底物DNA,如果得不到良好的酶切效果时,可以将反应时间延长至1小时。
●活性确认:
在50 μl反应液中,37℃温度下反应1小时,将1 μg的λDNA完全分解的酶量定义为1个活性单位(U)。
●纯度检测:
1) Overdigestion Test:在1 μg DNA中加入过量的该限制酶,进行长时间(24小时)酶切反应,然后进行琼脂糖电泳,确认切出的DNA片段的电泳谱带不发生变化。
2) Ligation-Recutting Test:在经过10倍量该酶切出的DNA片段中,加入 T4 DNA Ligase,使其连接,然后再使用该酶进行切断反应,判断Ligation-Recutting效率。
3) pKF3 Enforcement Cloning Test:使用10倍量的该酶,将Enforcement Cloning Vector pKF3 DNA切开,然后再进行连接后,转化至TH2感受态细胞中,判断该酶切位点受到影响的重组体所占的比率。
●在各种Universal Buffer中的相对活性:
L M H K T(+BSA)相对活性(%)(20)(100) 100 (120) (80)
●各种DNA的切断数:
●甲基化的影响:
根据识别序列后续碱基的不同,有时受CG methylase影响。
●Star活性:
高甘油浓度、Mn2+存在、低离子强度条件下,识别序列会发生变化。
如果在反应液中添加Spermine (0.2 mM左右),活性降低20~30%,但可以抑制30~50%的Star活性。
●Basal Buffer组成:
100 mM Tris-HCl, pH7.5
7 mM MgCl2
50 mM NaCl
7 mM 2-Mercaptoethanol
0.01 % BSA
●Universal Buffer组成(-20℃保存):
1.10 × L 100 mM Tris-HCl,pH7.5 4.10 × K 200 mM Tris-HCl,pH8.5
100 mM MgCl2100 mM MgCl2
10 mM Dithiothreitol 10 mM Dithiothreitol 2.10 × M 100 mM Tris-HCl,pH7.5 1,000 mM KCl
100 mM MgCl2 5.10 × T 330 mM Tris-Ac,pH7.9
10 mM Dithiothreitol (BSA 100 mM Mg-Ac
500 mM NaCl -Free) 5 mM Dithiothreitol 3.10 × H 500 mM Tris-HCl,pH7.5 660 mM K-Ac
100 mM MgCl2 6. 0.1% BSA
10 mM Dithiothreitol 7. 0.1% Triton X-100
1,000 mM NaCl
●10×Loading Buffer组成
(开封后室温保存)
0.9% SDS
50% Glycerol
0.05% Bromophenol Blue
使用时添加反应液量的1/10,即可停止反应,进行电泳。
-20℃保存时,会出现SDS沉淀,请于温水浴中溶解后使用。
在室温下保存时,SDS有时也会出现沉淀,此时同样请在温水浴中溶解后使用。
λ Ad2SVφX pBR pUC pUC M13Col
40174322 19 119 mp18E1
5 5 1 0 1 1 1 1 1
V2010.10。
