Genotyping protocol
Genotyping

Genotyping ProtocolCollect Tails1.Fill syringe in animal facility with isoflurane and attach to exposure box.2.Place folded up paper towel inside box.3.Inject 4-5cc isofluorane into container while pressing against gauze to ensure thatanesthetic evaporates into chamber.4.Immediately close the top so that the isoflurane can fill the chamber5.Set up razors, ear tags, tagging tool, recovery cage, and paper towels for cutting on.6.Retrieve first cage of animals.7.Place first animal into chamber, leaving it open as short of time as possible so thatisoflurane concentration stays high.8.Watch animal carefully. Animal will follow these stages of anesthetic:1.Loss of consciousness (test by rolling animal in chamber, LORR indicatesunconsciousness)2.Breathing will become very regular, deep and fast.3.Breathing will suddenly shift to a much lower rate4.Remove animal from chamber after 2-4 low rate breaths, much more could killthe animal.9.Tag animal with number facing rostral and internal to the pinnae (closer to the head).10.Cut a 1cm section of tail from the animal of interest.∙Be very careful not to contaminate tail samples with blood or tissue from the other animals as that DNA can confound the genotyping results.∙Make sure you use a different razor each tail and don’t cut in to previously bloodied areas of the paper towel.11.Place animal in recovery cage, then put tail sample into tube numbered for the eartag used.12.Place tub into dry ice and proceed to the next animal.13.Goto step 3 for each animal.14.Once animals are all awake and bleeding has subsided, return to home cage andretrieve next cage to be genotyped.DNA Extraction and PCR1.Turn on heat block(s) to 95°C, use both if you have enough samples.2.Take out the Sigma REDExtract-N-Amp Tissue PCR Kit (Cat: XNAT)3.Place REDExtract-N-Amp PCR Mix on ice and thaw the other solutions at roomtemperature.4.Take out ParpAPP primer mix from Ari’s box (recipe below), thaw, then place on ice.5.Syringe-filter 10mL of ddH2O into a falcon tube.6.Make a master mix of extraction buffer for your samples at n + 1 (one more than thenumber of samples you have).∙Mix Extraction Solution and Tissue Preparation Solution in a 4:1 ratio up to 2 hours before extraction. (If 15 tails, make enough for 16: 1.6mL ExtractionSolution and 400uL Tissue Prep)∙Vortex for 10s.7.Add 125uL of master extraction mix to each tail sample.8.Incubate at room temperature for 10 minutes.9.Incubate at 95°C for 3 minutes.10.Remove from heat and quickly add 100ul Neutralization Solution B to each sample.11.Vortex each sample for 5 sec to mix12.Shake tail down to bottom of tube∙Remove tails from extraction mixture and store at 4°C for up to 6 months.∙Make sure to discard tails into freezer bag to be taken back to animal facility.13.Take out enough PCR tubes for the number of tails you have, or a 96-well plate14.Put the tubes in tube rack and cover with strip caps15.Make a master PCR mix for n + 1 samples (preferably while step 8 is happening).∙10ul REDExtract-N-Amp PCR Reaction Mix per tail sample∙1ul ParpAPP primer mix per tail sample∙5ul filtered ddH2O per tail sample∙Vortex for 10 seconds, centrifuge at full speed for 15 sec∙Place on ice14.Place PCR tubes on ice and perform all PCR prep on ice as well.15.Place 16ul of PCR Master Mix into each tube that will be used for tails.ing only filter-tips, carefully pipette 4ul of each tail extraction into each tube17.Mix by slowly pipetting up and down 5x, make sure as much of the PCR reactionsolution stays at the bottom of each tube as much as possible18.Repeat for all samples, making sure that you do not contaminate any tail extractionwith another (the smallest amount of DNA can mess up the reaction).19.Cap the tubes (or 96-well plate), leave on ice.20.Turn on the PCR machine, log in as AEB.21.Go to the protocol library, scroll to the ParpGT folder and select the program.22.Select “Run Program”.23.Set “Hot Start” option to Yes and to 94°C.24.Set reaction volume to 20ul and temperature method to Algorithmic.25.Start the reaction∙The machine will raise the temperature of the heated lid to 100°C∙The block will then go to 94°C and hold26.Open the lid and place the PCR tubes quickly and carefully into the heated block.27.Close the lid as soon as possible to prevent PCR reaction mix evaporation.28.Select “Resume” on the PCR machine, which will start the PCR run.29.The PCR machine will tell you when the projected completion of the PCR reactionwill be, come back then or let go overnight. The machine will hold the samples at 4°C when the run is finished.ParpAPP Primer Mix (500ul):∙50ul Parp1F primer∙25ul JaxParp1 primer∙25ul NeoRB2r primer∙25ul Pd-APP-1F primer∙25ul Pd-APP-1R primer∙350uL filtered ddH2O∙Vortex for 10s, place at -20°CRun the gel* All measurements will be listed for the (large/small) gel rigs.1.Take out either the large or small agarose gel rig (or both)Gel rigs can handle (38/24) samples with two combs.2.Rinse the gel mold with water and make sure the gasket is very wet3.Place the mold into the running rig so that the gaskets are sealed against the sidewalls of the rig.4.Have 1-2 combs (green/blue) ready and cleaned next to the rig5.Make sure the rig is level!! (an uneven gel won’t run well)6.Make a 3% agarose gel7.Measure out (4.5g/2.25g) agarose and place into 250mL beaker.8.Add (150mL/75mL) 1x TAE to the beaker and swirl to mix.9.Place beaker in microwave for 1:30.10.Take beaker out and swirl (don’t burn yourself, it is hot!)11.Heat for another (35s/20s)12.Swirl again, repeat until no visible beads are in the solution.13.Add (15ul/8ul) SYBRSafe DNA dye to the molten gel and swirl gently in bothdirections to mix thoroughly.14.Pour the mixture into the mold, place the beaker into the sink and start running waterinto it.15.Quickly place the comb(s) into the comb sockets on the gel mold, 1.5mm side down(thicker wells).16.Cover the gel loosely with foil to protect from light.17.Let stand for 30 minutes or until gel is room temperature and solid to the touch.18.Take out 100bp DNA ladder and 6x loading dye to thaw on the bench top while gelis setting up.19.Pull out combs by pulling steadily straight up (it takes a little strength to get combsout of 3% gel).20.Remove gel mold and turn it so that if the leads of the rig are facing right, the wellsare pointed toward to the top of the rig.21.Fill the gel rig with 1x TAE until fluid level just covers the gel (about 2-3mm).22.Mix 6uL 100bp ladder and 1ul loading dye per comb (if using two combs, doublethese amounts) in a tube and mix by pipetting up and down several times.23.Pipette 7ul DNA ladder/dye mix into the far left well in each row of wells.24.Pipette 10uL of each PCR reaction straight from the PCR tube into each of the wells,taking care to keep track of which well is which and making sure that you don’tcontaminate the other wells or other PCR samples with the other reaction mixtures.25.Once the gel(s) is loaded, Place the top on the gel rig and attach to the power supply.26.Run the gel for 1hour at 120V.27.Recap the PCR tubes and place at 4°C in case you need to rerun the gel.28.Get the memory card from Ari’s desk drawer and place it into the digital camera inthe microscope room.29.Place the gel directly onto the UV transilluminator.30.Turn on the camera, activate the screen, turn off the flash, and turn on the macro lens.31.Make sure the UV filter is in front of the lens.32.Place a UV shield over your face (the shortwavelength UV emitted from the UV boxis extremely harmful to your eyes with very little exposure, and not good for your skin either).33.Zoom into your gel, center it, and take a picture or two.34.Turn off the UV box, turn off the camera, take the gel off of the box and the boxdown with a Kimwipe.35.Throw the gel away and give the card to Ari to analyze.。
genotyping原理

genotyping原理基因型(genotype)是指个体或生物体其中一种特定基因的种类或基因组合。
通过对一些生物的基因进行分析,可以确定其基因型,进而了解其遗传特征和相关性状。
基因型分析在遗传学研究、医学诊断和生物技术应用中具有重要的意义。
基因型分析的方法有很多种,其中最常见的是基于DNA序列的分子生物学方法。
基因型分析的原理主要包括DNA提取、基因放大、基因分型和数据分析等步骤。
首先,需要对样本中的DNA进行提取。
DNA提取是分子生物学实验中的第一步,常用的提取方法包括酚/氯仿法、离心法和商用DNA提取试剂盒等。
通过这些方法可以将DNA从细胞或组织中提取出来。
接下来,需要将提取得到的DNA进行放大。
常用的放大方法有PCR(聚合酶链式反应)和SNP(单核苷酸多态性)分析等。
PCR是一种体外的DNA复制技术,可以在短时间内扩增特定的DNA片段。
SNP分析则是通过扩增特定的基因片段来检测个体间的单核苷酸变异。
基因分型是基因型分析的核心步骤。
常见的基因分型技术有限制性片段长度多态性(RFLP)、序列特定引物扩增(SSP)和基因芯片等。
RFLP分析是基于特定限制性内切酶酶切位点的DNA片段长度变异来进行基因分型的技术。
SSP则是通过引物的特异性扩增决定基因分型。
基因芯片则是一种高通量的基因分型平台,可以同时检测数千个基因的单核苷酸多态性。
最后,需要对分型结果进行数据分析和解读。
根据不同的分型技术可以得到不同的分型结果,需要将这些结果与已知的基因型进行比对。
数据分析包括基因型预测、相关性状分析和基因关联研究等。
基因型分析在包括人类疾病遗传研究、种质资源鉴定、遗传育种和亲子鉴定等领域具有重要的应用价值。
通过基因型分析,可以确定个体或物种的基因型,进而了解其遗传特征和相关性状,对于人类健康和物种保护具有重要的意义。
总之,基因型分析是通过对生物体DNA序列进行分析,确定其基因型从而了解其遗传特征的一种方法。
它在分子生物学、遗传学和医学研究中具有重要的意义,可以为人类健康、物种保护和种质资源利用等领域提供有力的支持。
SNPs解析 (Genotyping)

基质辅助激光离子化飞行时间型质谱仪应用文集 No. 4SNPs解析 (Genotyping)1.前言2.原理3.分析实例之一┉┉同类结合体4.分析实例之二┉┉变异型结合体5.分析实例之三┉┉多引物的同时鉴定1.前言近年来,解读人类全部遗传信息的基因组碱基序列的基因组计划是以国家规模推进,到2000年30亿碱基序列(包含A-G-C-T四种碱基)几乎全部被解析和发表。
与详细的基因组碱基序列的分析一样,SNPs分析(Single Nucleotide Polymorphisms, 单核苷酸多态性)同样得到了广泛的重视。
SNPs是当碱基序列中只有一个碱基被其他碱基置换所造成的单个碱基所呈现出的变异·多样性的情况。
据推测在每个人所具有的遗传基因中约有30~1000万处SNPs。
在活体中遗传信息是由3个碱基为一组形成1个氨基酸的信息,由特定氨基酸决定的序列构成各种各样功能的蛋白质。
由于碱基序列决定组成蛋白质的氨基酸序列,当一个碱基被置换就可能产生功能不正常的蛋白质。
例如,有人善饮酒或不能饮酒,有些药起作用或不起作用,由于个人体质的差异容易患某些疾病等情况。
通过调查个体的SNPs希望探索某些疾病与个体等之间的相互关系,针对个体的差异,掌握正确的处方,开展个体差异性治疗是二十一世纪的新型医疗模式。
对于容易造成高血压或癌症等疾病的SNPs,其应用的场合就更加清楚,需要针对个体特定的SNPs中有哪些碱基是否发生置换的情况,进行快速的诊断和治疗。
这里利用AXIMA-CFR可对已知SNPs分析的genotyping (确定碱基)应用实例。
2.原理图1为实现SNPs的genotyping的样品准备过程,该方法称为单核苷酸延伸反应(SNuPE,Single Nucleotide Primer Extension)。
由Kuppuswamy在1991年发表。
图1:单核苷酸延伸反应(SNuPE)流程图①采用PCR方法扩增含有SNPs部位的DNA片段②针对第①步的PCR产物,作为SNPs部位延长一个核苷酸的引物③进行SNuPE单核苷酸延伸反应④在反应进行脱盐纯化⑤在AXIMA-CFR的样品板上,混合样品和基质后进行分析3.分析实例之一 --- hemozygotes (同类结合体)作为模型(标准)样品选用ras Mutant Set c-Ha-ras codon 12 (Takara),基质采用0.7M 3-hydroxypicllic acid (3-HPA)的50%乙腈0.07M ammonium citrate的饱和溶液,在分析之前采用ZipTip TM(纯化柱,Millipore)进行脱盐。
转录因子和基因互作的验证

转录因子和基因互作的验证转录因子是一类能够调控基因表达的蛋白质,它们通过与DNA结合,调节基因的转录过程。
在生物体内,转录因子与基因之间的互作关系非常复杂,需要通过一系列实验验证来加以证实。
一、基因表达谱分析基因表达谱分析是一种常用的验证转录因子和基因互作关系的方法。
通过对不同组织或细胞类型中基因表达谱的比较,可以发现一些共同的基因表达模式,这些模式往往与特定的转录因子有关。
例如,研究人类乳腺癌细胞中的基因表达谱,发现与转录因子FOXA1有关的基因表达模式,进一步证实了FOXA1与乳腺癌的发生和发展密切相关。
二、染色质免疫共沉淀实验染色质免疫共沉淀实验是一种验证转录因子和基因互作关系的重要方法。
该实验通过将转录因子与抗体结合,然后将其与细胞核中的染色质共同沉淀下来,最后通过PCR或测序等方法检测共沉淀下来的DNA 序列,从而确定转录因子与哪些基因发生了互作。
例如,研究人类肝癌细胞中的转录因子HNF4A,通过染色质免疫共沉淀实验,发现HNF4A与多个肝癌相关基因发生了互作,进一步证实了HNF4A在肝癌发生和发展中的重要作用。
三、基因敲除实验基因敲除实验是一种验证转录因子和基因互作关系的直接方法。
该实验通过CRISPR/Cas9等技术,将目标基因从细胞中完全删除,然后观察细胞的表型变化,从而确定该基因与哪些转录因子发生了互作。
例如,研究人类胰岛素分泌细胞中的转录因子PDX1,通过基因敲除实验,发现PDX1与多个胰岛素分泌相关基因发生了互作,进一步证实了PDX1在胰岛素分泌中的重要作用。
综上所述,转录因子和基因之间的互作关系非常复杂,需要通过一系列实验验证来加以证实。
基因表达谱分析、染色质免疫共沉淀实验和基因敲除实验是常用的验证方法,它们为我们深入了解转录因子和基因互作关系提供了重要的实验手段。
illumina芯片介绍

Whole Genome
• WG Resequencing • Candidate • WG Gene Expr. Resequencing • miRNA Discovery • Chromosome & Profiling sequencing • WG ChIP-Seq • HT Biomarker • Gene Expr. Validation • WG GT • CNV Screening• Custom GT • Focused Expr. • Biomarker • FFPE Discovery
Detected duplication
B allele frequency data (AAB genotype) A shift in the LogR value is detectible, but the integration of B allele data improves the signal to noise ratio
From discovery to single target Validation
Sequencing & Arrays Platform Synergy
Discovery
>109 108 107
Focused Research/Validation
106 105 104 103
Screening
660kmarkerspersample4samplesperchipcomprehensivegenomiccoverage5000commoncnvregionsinfiniumhdassaylowdnainput200ng410persample700kmarkerspersample12samplesperchiphapmapallphases1000genomesprojectinfiniumhdassaylowdnainput200ng260persample?1millionmarkerspersample4samplesperchiphapmapallphases1000genomesprojectcnvprobesinfiniumhdassaylowdnainput200ng468persample??????????????????software???developedwithandforthecytogeneticistautomatedcrossmatchingandreportingfunctionsadjustabledatabaseofknownregions???numerousanalysisoptionsforcopynumberanalysisgraphicaltableanddatadisplayslinksto3rdpartysoftwareofferingsforresearchuseonlyhumancytosnp12beadchipoptimizedforefficientcytogeneticanalysishigherdensityincytohighvalueregions40ofgenomeallpericentromeresandsubtelomeressexchromosomescommonregionsofinteresteg
小鼠基因型鉴定说明书Mouse Genotyping

1.2. 在灭菌1.5 ml EP管中,添加200 μl 1×裂解液至所需裂解组织中,涡旋震荡后在55oC水浴中孵育20 min。为保证DNA 释放效率,请务必将组织全部浸没至裂解液中。孵育结束后组织块可能并未消化完全,属正常情况,不影响使用。对于常 规大小目标片段,20 min孵育已足以释放足量的DNA模板。孵育时间也可根据实际情况进行调整,下表为55oC下推荐孵育 时间:
2.2. 推荐PCR反应条件设置:
94oC 94oC 55oC* 72oC 72oC
5 min(预变性)
30 sec
30 sec
}
30 sec/k需要根据引物Tm值进行调整,一般设置成低于引物Tm值1-2℃即可。 2.3. 扩增产物直接进行琼脂糖电泳检测,无需添加DNA Loading Buffer。
to 50 μl 25 μl 2~5μl 2 μl 2 μl 10 μl
*1 Taq Plus Master Mix (Dye Plus)中预混有1.5 mM的Mg2+。在实际使用过程中,可使用试剂盒提供的25 mM MgCl2进行 调整,调整间隔为每次增加0.5 mM。 *2 裂解产物加入量不应超过PCR反应总体积的1/10。 *3 5 × PCR Enhancer可显著提高GC-rich片段的扩增效率。在初次使用某引物对进行扩增时,可设置添加/不添加两平行 对照组。如添加后扩增产量或特异性无明显提升,则无需添加。
One Step Mouse Genotyping Kit
PD101-01
Vazyme biotech co., ltd.
产品简介
本试剂盒包含整套的DNA粗提取以及PCR扩增体系,可用于小鼠基因型快速鉴定(Rapid Genotyping)。该试剂盒可用于从 小鼠尾巴、耳朵以及脚趾等组织快速提取基因组DNA,提取产物可直接进行PCR扩增,无需匀浆、破碎、过夜消化、酚氯 仿抽提、DNA沉淀或柱式纯化等操作,极大缩短了实验耗时。使用时,将组织浸泡在预添加了Proteinase K的裂解液中, 55oC孵育20 min再95oC加热5 min灭活Proteinase K。裂解产物经离心后可直接用做PCR扩增模板,经反复测试广泛适用于 2 kb以内目标片段扩增,并适用于4对引物以内的多重PCR反应。 试剂盒中配有2 × Taq Plus Master Mix(Dye Plus)(P212),包含高性能的Taq Plus DNA Polymerase,dNTP以及优化的缓 冲体系。PCR反应时只需加入引物和模板即可进行扩增,减少了开管/移液等操作,显著降低了样品交叉污染并且提高了检 测通量和结果的重现性。独特的保护剂使得Taq Plus Master Mix经过反复冻融后仍可保持稳定的活性。体系中预混有电泳 缓冲液和染料,可在反应结束后直接进行电泳,使用方便快捷。PCR产物的3’端带A,可克隆至T载体,并适用于 ClonExpressTM快速克隆试剂盒(C112/C113)。
GeneXpert新型分子生物学诊断试剂数量及技术参数

GeneXpert新型分子生物学诊断试剂数量及技术参
数
一、试剂采购数量
400人份
二、主要技术参数
1、主要用途
1.1用于体外定性检测痰液样本
1.2用于痰沉淀样本中的结核分支杆菌复合群DNA和利福平耐药相关的rpoB基因突变
2、检测原理
2.1 GeneXpert系统使用实时PCR,整合并自动进行样品纯化、核酸扩增、单一或复杂样品中的目标序列测定。
2.2 XpertMTB/RIF用于确认目标细菌的充分处理和监测是否有PCR 反应抑制物的存在,探针检查质控PCC用于确认试剂再水合、检测匣中PCR管加样、探针完整性和染料稳定性。
2.3 XpertMTB/RIF针对rpoB基因81bp利福平耐药核心区(RRDR)设计引物、探针,检测其是否发生突变。
3、主要组成成分
3.1内含反应管的检测匣 10人份/盒
3.2 1号珠:内含引物、探针、氯化钾、氯化镁、4-羟乙基哌嗪乙磺酸、牛血清白蛋白 2个/检测匣
3.3 2号珠:聚合酶、探针、氯化钾、氯化镁、脱氧核糖核甘三磷酸、4-羟乙基哌嗪乙磺酸、牛血清白蛋白 2个/检测匣
3.4 3号珠:样本处理质控 1个/检测匣
3.5试剂1:三羟甲基氨基甲烷缓冲液、乙二胺四乙酸、表面活性剂4ml/检测匣
3.6试剂2:三羟甲基氨基甲烷缓冲液、乙二胺四乙酸、表面活性剂4ml/检测匣
3.7样本处理试剂:氢氧化钾、异丙醇 8ml*10瓶/试剂盒
3.8一次性移液管 12支。
小鼠胚胎-精子冷冻保种申请表-清华大学试验动物中心

小鼠胚胎/精子冷冻保种申请表需要填写的资料:说明:1.*表示必须填写;2.保种方式的选择:我们采用精子冻存和胚胎冻存的方法进行保种;假如为杂合子,雄性数量≥3只并且可以和B6交配做净化,我们推荐安乐死一只雄鼠使用精子冻存的方式保种,可以有效控制保种周期,不受雌鼠排卵多少的限制。
3.保种对提供小鼠的要求:精子冻存冷冻精子需要提供的小鼠信息:阳性纯合子雄鼠,2-3只,12-18W(最佳),生育能力良好,在实验前7d内进行过成功交配,而在3d内未交配过;冻存2只/品系,20个麦管。
费用:200元/麦管冷冻的流程和所需的时间:1)提前1周邮件预约申请,写明小鼠品系名称、数量、出生日期、所在位置。
2)通过申请后安排实验,精子冻存后至少1周进行复苏检测实验(1麦管)。
3)等待代孕小鼠生仔(孕期19.5天,小鼠出生后10-12天剪尾,通知取回鼠尾检测,(一周内反馈检测结果。
)冷冻后的保存:液氮中保存终生免费保存。
精子复苏(除复苏检测外)a、外来精子复苏提前1周邮件预约申请,写明小鼠冻精信息(品系,数量),送达时间。
从接收之日起,1周内安排复苏实验精子复苏采用体外受精的方式得到2-cell期胚胎,输卵管移植生仔。
(孕期19.5天,小鼠出生后10-12天剪尾,通知取回鼠尾检测,一周内反馈检测结果。
)费用:IVF 500元,IVF用B6(含激素)40元/只,移植用受体单侧100元/只,双侧180元/只,试剂和人工费100元。
如需其他品系雌鼠,需从外单位订购。
b、动物中心精子复苏提前1周邮件预约申请,写明复苏品系。
精子复苏采用体外受精的方式得到2-cell期胚胎,输卵管移植生仔。
(孕期19.5天,小鼠出生后10-12天剪尾,通知取回鼠尾检测,一周内反馈检测结果。
)费用:IVF 500元,IVF用B6(含激素)40元/只,移植用受体单侧100元/只,双侧180元/只,试剂和人工费100元。
注意事项:1. 运输过程中:请使用专业运输工具(航空罐)进行运输,运输过程中请避免磕碰,注意液氮充足。
小鼠基因型鉴定说明书Mouse Genotyping

One Step Mouse Genotyping Kit
PD101-01
Vazyme biotech co., ltd.
产品简介
本试剂盒包含整套的DNA粗提取以及PCR扩增体系,可用于小鼠基因型快速鉴定(Rapid Genotyping)。该试剂盒可用于从 小鼠尾巴、耳朵以及脚趾等组织快速提取基因组DNA,提取产物可直接进行PCR扩增,无需匀浆、破碎、过夜消化、酚氯 仿抽提、DNA沉淀或柱式纯化等操作,极大缩短了实验耗时。使用时,将组织浸泡在预添加了Proteinase K的裂解液中, 55oC孵育20 min再95oC加热5 min灭活Proteinase K。裂解产物经离心后可直接用做PCR扩增模板,经反复测试广泛适用于 2 kb以内目标片段扩增,并适用于4对引物以内的多重PCR反应。 试剂盒中配有2 × Taq Plus Master Mix(Dye Plus)(P212),包含高性能的Taq Plus DNA Polymerase,dNTP以及优化的缓 冲体系。PCR反应时只需加入引物和模板即可进行扩增,减少了开管/移液等操作,显著降低了样品交叉污染并且提高了检 测通量和结果的重现性。独特的保护剂使得Taq Plus Master Mix经过反复冻融后仍可保持稳定的活性。体系中预混有电泳 缓冲液和染料,可在反应结束后直接进行电泳,使用方便快捷。PCR产物的3’端带A,可克隆至T载体,并适用于 ClonExpressTM快速克隆试剂盒(C112/C113)。
应用实例
操作注意事项: - 用70%的乙醇(自备)预先清洗组织分离过程中所使用的所有工具; - Proteinase K灭活步骤(95oC,5min)必须进行,其残留活性会抑制后续PCR反应; - PCR反应体系配制过程应于冰水浴中进行,以提高扩增特异性(热启动)。
ch02

Difficulties for whole genome sequencing
Repeats
Tandem repeats Genome-wide repeats
Gaps
Genetic mapping
2.1 Approaches to genome sequencing 2.2 Distinguish between the terms ‘genetic map’ and ‘physical map’ 2.3 DNA markers for genetic mapping 2.4 Linkage analysis with different types of organism
Genomics
Chapter 2 Genetic mapping
Genome Mapping
人类的单倍体基因组分布在22条常染色体和X、Y 性染色体上,最大的1号染色体有263 Mb,最小 的21号染色体也有50 Mb,平均130Mb。人类基 因组计划的首要目标是测定全部DNA序列,但由 于人的染色体不能直接用于测序,因此人类基因 组计划的第一阶段是要将基因组这一巨大的研究 对象进行分解,将其分为容易操作的小的结构区 域,这个过程简称为基因组作图(Mapping)。
RFLP?
1) Allele Specific Hybridization
Also known as ASO (allele specific oligonucleotide hybridization), this protocol relies on distinguishing between two DNA molecules differing by one base by hybridization. Fluorescence labeled PCR fragments are applied to immobilized oligonucleotides representing SNP sequences. After stringent hybridization and washing conditions, fluorescence intensity is measured for each SNP oligonucleotide.
Golgi Staining Protocol
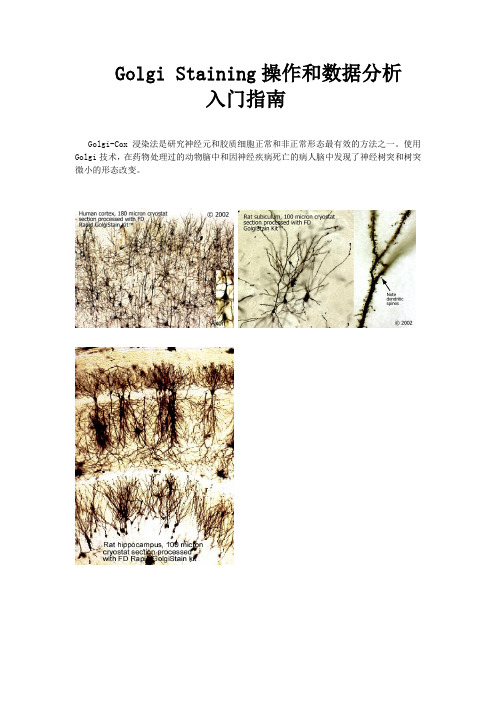
Golgi Staining操作和数据分析入门指南Golgi-Cox浸染法是研究神经元和胶质细胞正常和非正常形态最有效的方法之一。
使用Golgi技术,在药物处理过的动物脑中和因神经疾病死亡的病人脑中发现了神经树突和树突微小的形态改变。
Part A 高尔基染色原理:用重铬酸钾和铬酸钾,氯化汞作为初步固定剂浸润组织,铬盐和神经细胞中的蛋白质结合,氯化汞通过白色沉淀来标记单个细胞,进一步经过碱处理,使沉淀物变黑(硫化汞)。
该法最显著的特点是其反应终产物都局限于单个神经细胞的内部,而其周围去多类似的神经细胞不着色,从外观上看,就好像从固定到脱水的全过程在细胞膜水平都存在一个阻止可见产物扩散的屏障。
The beauty of the method lies in one of its shortcomings: the Golgi protocol stains only a few hundred neurons out of the millions present. Thus, it is possible to visualize neurons and trace their path and connections in the brain against a pale yellow background with unsurpassed clarity. Without this ability to stain the few select neurons, everything would have looked like a large black blob on the brain section.。
1.明胶玻片的制备高尔基染色的脑片较厚,如果使用普通玻片贴片,在染色过程中,脑片很容易从玻片上脱落下来,影响实验结果,对此需制备特殊明胶包被载玻片,以供高尔基染色使用。
方法:加热500 mL双蒸水,后加入10g明胶,搅拌溶解。
genotyping3种引物

genotyping3种引物1.引言1.1 概述概述引物在基因分型研究中起着重要的作用。
它们是一类短链的DNA或RNA序列,能够与待测DNA中的特定区域发生亲和性结合。
通过引物的选择和设计,我们可以准确地扩增和分离待测区域,从而实现基因分型的目的。
本文旨在介绍和探讨三种引物的基本信息和设计原理。
通过对这些引物的详细分析,我们可以了解它们在基因分型研究中的应用和优缺点。
在接下来的篇章中,我们将依次介绍引物1、引物2和引物3的基本信息和设计原理。
每种引物都有其特定的目的和优势,我们将分析其适用性和限制,以便更好地理解它们在基因分型研究中的应用。
最后,我们将对比这三种引物的优缺点,并探讨未来的发展方向。
通过对这些引物的研究和改进,我们有望提高基因分型的准确性和效率,为基因研究领域的发展做出更多的贡献。
本篇长文将详细介绍这三种引物的基本信息和设计原理,以期为基因分型研究提供有益的指导和启示。
希望读者通过阅读本文能够更好地理解和应用引物在基因分型研究中的作用,为科学研究和生物医学领域的进展做出贡献。
文章结构部分的内容是对整篇文章的组织架构进行介绍和概述。
以下是对文章结构部分的内容的编写示例:1.2 文章结构本文将分为引言、正文和结论三个部分,具体的章节安排如下:1. 引言部分将对基因分型的背景和意义进行概述,介绍引物的作用和重要性,并明确本文的目的。
2. 正文部分将详细介绍三种引物的设计原理和基本信息。
其中,2.1 将详细描述引物1的基本信息,包括引物序列、长度、GC含量等。
2.2 将解释引物1的设计原理,并讨论其在基因分型中的应用。
同样地,2.3 和2.4 将分别介绍引物2和引物3的基本信息和设计原理。
3. 结论部分将对比分析三种引物的优缺点,评估它们在基因分型中的适用性和限制,并探讨未来发展的方向。
通过以上的章节安排,本文将全面、系统地介绍三种引物的基本信息和设计原理,并评估它们在基因分型中的应用潜力,为读者提供一份详尽的参考。
新生小鼠genotyping protocol

从小鼠合笼到基因分型一,小鼠合笼:1,准备需要合笼的老鼠(要求为性成熟老鼠:8—10周龄),记录老鼠基本信息,包括编号,性别,出生日期,基因型。
2,挑选需要合笼的公鼠,每笼放一只,记录笼号和卡片流水号,并在卡片上标记该老鼠的基本信息(编号,基因型)。
3,挑选需要合笼的母鼠,每笼只,同样在卡片上标记母鼠的基本信息(编号,基因型),注意,同一笼中母鼠的耳号不能相同。
4,合笼时间的选择(具体情况具体分析)。
5,母鼠验栓。
合笼次日晨8:00验栓,有阴栓的母鼠更换到仅放有受孕母鼠笼中,在卡片上标记好母鼠信息(包括与该母鼠交配的公鼠的编号),在原鼠笼的卡片上划掉该母鼠信息。
6,重复步骤5连续三天,将仍未受孕的母鼠统一分装到不含有公鼠的笼中,标记相应信息卡片,至此合笼结束。
二,新生小鼠基因分型1,新生小鼠从出生时P0开始计数,到4周龄p28时需要对其进行基因分型且进行分笼。
2,对小鼠耳号的编号方式见下图:背部3,按照一窝新生老鼠8只(3只Male, 5只Female)为例,按上图耳号的位置打耳号,编号方式为(Male)1-1, 1-2, 1-3, (Female)2-1, 2-2, 2-3, 2-4, 2-5, 做好分笼,将打耳号时产生的ear punch biopsy放于对应编号的PCR管中,管上做对应编号。
4,Add 40μl Sol Ⅰ,Heat to 95℃15min,Add 40μl Sol Ⅱ5,2μl for PCR reaction.6,PCR结果凝胶电泳,电泳结果拍照并打印,清晰标记每个泳道的对应小鼠编号。
7,按照小鼠编号和电泳结果,在分笼后鼠笼卡片上标记对应小鼠编号的基因型。
相关试剂配方:。
小鼠GENOTYPE样本处理方法

superEase protocol to prep mouse tail DNA for genotyping Mouse tail: about 0.5CMReagents:Buffer A: 25mM NaOH +0.2mM EDTA, PH8.0To make 250ml of buffer,mix 625ul of 10N NaOH+100ul of 0.5M EDTA with water. Buffer B: Tris-HCl,40mM,PH3.0Produres:1.Add 100(50-100)ul Buffer A to a tube with mouse tail2.Cook the DNA at 90(90-95)℃ for 45-60 min to degrade mouse tissues and exposegenomic DNA3.Add 300ul(3time of bufferA)Buffer B to neutralize BufferA and mix well bygentle wortexing (5min to O/N,depending your “style”)4.Spin down in a bench-top centrifuge at top speed for 1mine 1ul of the supernatant for 25ul of PCR reactionPreparation of Genomic DNA from Mouse T ailMaterials:1.Lysis solution II(50mM Tris PH8.0,100mM EDTA,0.5%SDS)2.Proteinase K(10mg/ml)– dissolved in 10mM Tris HCl(PH7.5),20mM calcium chloride,50% glycerol3.5M NaCl solution4.Phenol: Chloroform:Isoamyl alcohol(25:24:1)5.Chloroform:Isoamyl alcohol(24:1)6.100% EtOH/70% EtOHProcedure:1.Cut 0.5-1cm mouse tail and put into 1.5ml microcentrifuge tube2.Cut it into smaller pieces and add 500ul of Lysis solutionII3.Add 10ul proteinase K(glycerol+)and mix well4.Incubate at 55℃ in water bath overnight.5.Cool the samples down to R.T.6.Add 0.25 vol(ie.131ul)5M NaCl and vigorously shake the tube7.Spin the tube at maximum speed for at least 15min at 4℃(longer spin will bebetter)8.Transer supernatant to a fresh tube.9.Add 1vol.(i.e. 500ul)of Phenol:Chloroform:Isoamyl alcohol mixture and gentlyshake it for 20min10.spin the tube at maximum speed for 10min11.Transfer the upper phase to a fresh tube12.Add 1vol. of Chloroform:Isoamyl alcohol mixture and gently shake it for 5min13.Spin it at maximum speed for 5min14.Transfer the upper phase to a fresh tube15.Add 2 or 2.5vol. of 100%EtOH and mix thoroughly.(You will see white precipitate,我看到的是浑浊的白色液体)16.Spin the tube briefly and discard the supernatant(我得到的沉淀是近于透明的粘稠固体)17.Wash the pellet with 70%EtOH twice18.Dry and add 100ul of D.W19.Warm the sample at 50℃ for 30min.Keep it at 4℃一般要扩比较长的片段或者引物不好的情况下才用此方法,一般的GENOTYPE只要用最简单的方法(BUFFER A BUFFER B的方法)就可以了。
鼠库全书丨NCG小鼠概述

⿏库全书⼁NCG⼩⿏概述NCG⼩⿏品系名称:NOD/ShiLtJ-Prkdc em26Cd52 Il2rg em26Cd22基因官⽅名:Prkdc;Il2rg基因全名:protein kinase DNA activated catalytic polypeptide蛋⽩激酶DNA活化催化多肽;interleukin 2 receptor gamma chain ⽩细胞介素2受体γ链基因别名:AI326420;AU019811;DNA-PKcs;DNAPDcs;DNAPK;DNPK1;DOXNPH;HYRC1;XRCC7;dxnph;p460;NCBI号:19090,16186品系背景:NOD/ShiLtJ修饰⽅式:Knockout (KO) CRISPR/Cas9保存⽅式:活体冷冻微⽣物级别:SPF级品系描述NCG (NOD/ShiltJ-Prkdc em26Cd52 Il2rg em26Cd22)是使⽤基因编辑技术敲除了NOD/ShiltJ⼩⿏的Prkdc (Protein kinase, DNA activated, catalytic polypeptide)及Il2rg(Common gamma chain receptor)基因⽽获得的重度免疫缺陷品系。
NOD/ShiltJ遗传背景使该品系具有天然免疫缺陷,如补体系统、巨噬细胞缺陷,同时,该背景Sirpa与⼈类CD47具有⾼亲和⼒,使NOD/ShiltJGpt⽐其它品系更适合⼈源移植物(如:肿瘤和⼈源细胞)的定植。
Prkdc基因功能缺失导致V(D)J重组不能正常发⽣,造成T细胞和B细胞不能发育成熟。
IL2RG是多种⽩细胞介素细胞因⼦受体的共同亚基,IL2RG失活则导致6种不同细胞因⼦信号通路缺失,造成NK细胞缺陷。
因此,NCG是迄今为⽌免疫系统缺陷最为彻底的⼩⿏模型之⼀,⾮常适合⼈源肿瘤细胞移植(CDX)、⼈源肿瘤组织移植(PDX)、⼈外周⾎单个核细胞(PBMC)及⼈源造⾎⼲细胞(CD34+ HSC)移植进⾏免疫重建。
高分辨率溶解曲线.pptx

Rosario Muleo,Marir Chiara Colao,Dario Miano at el. 2009 Genome52:252-260
第35页/共46页
第8页/共46页
Mutation Scanning
未知SNP扫描
第9页/共46页
等对北京油鸡CAPN1基因外显子进行扫描,筛查其潜在SNPs。
第10页/共46页
Bernhard等对Barley的eukaryotic translation
initiation factor 4E (eIF4E) gene基因外显子进行突变扫描结果
— 筛查突变 (mutation scanning) — 基因分型(Mutation Genotyping)
—小片段+内标法( Small Amplicon Genotyping ) —非标记探针法(Unlabeled Probe Genotyping,LunaProbe) —SSR分析 — 检测甲基化 • HRM仪器
内容提示
• 高分辨熔解曲线(High Resolution Melting) • 高分辨熔解曲线在遗传育种中的应用
— 筛查突变 (mutation scanning) — 基因分型(Mutation Genotyping)
—小片段+内标法( Small Amplicon Genotyping ) —非标记探针法(Unlabeled Probe Genotyping,LunaProbe) —SSR分析 — 检测甲基化 • HRM仪器
• 0.3oC/秒 升温速度= 67 pts/ ℃ • 0.05oC/秒 升温速度 = 400 pts/ ℃
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
A new protocol for genotyping
李广瑞( Li Guangrui )
University of Tokyo
E-mail: grli1235@
2012-5-25
1.Digest approximately 2 mm mouse tail (may be stored at –20°C) with 300 μL tail lysis
buffer in a 1.5-mL Eppendorf® tube at 55-60°C overnight.
2.Next morning, label one set of microfuge tubes with mouse ear tag numbers.
3.Centrifuge at full speed ( 20,000 × g ) for 5 min.
4.Transfer 100 uL supernatant to a clean tube with identical number.
5.Add 300 uL 100% ethanol into each tube, mix completely.
6.Centrifuge at full speed ( 20,000 × g ) for 10 min, then pour out the ethanol.
7.Wash the DNA pellets by adding 300 uL 70% ethanol into the tubes very gently ( do not
flush the DNA pellets) and centrifuge at full speed ( 20,000 × g ) for 5 min, then pour out the 70% ethanol.
8.Turn tubes upside down on paper towel to air –dry the DNA for 1-2 min (do not over-dry
or DNA may be difficult to resuspend)
9.Dissolve the DNA pellets with 20-50 uL TE buffer.
10. Polymerase Chain Reaction (PCR)
FORMULA
DDW appropriate amount
10×Buffer Ver. 2 12.5 uL
Primer-Forward 10-100pmol
Primer-Reverse 10-100pmol
DNA Polymerase 0.1 uL (12.5 units/uL)
DNA Template 0.1-2 ug —————————————————————————
25 uL
11.Electrophoresis
Gel
Check what % gel needed*:
Agarose effective Resol DNA (kb)
0.5% 30 to 1
0.7% 12 to 0.8
1% 10 to 0.5
1.2% 7 to 0.4
1.5% 3 to 0.2
*from current protocol in Neuroscience
Lysis Buffer
0.5M EDTA 50ml
5M NaCl 10ml
1M Tris pH7.4 5ml
10%SDS 50ml Proteinase K (10mg/ml) 10mM Tris pH7.3
20mM CaCl2
50% glycerol
TE buffer
10 mM Tris-HCl
0.2mM Na2EDTA pH 7.5。
