毕赤酵母直接基因敲除方法的研究
酵母基因敲除方法

酵母基因敲除方法
酵母是一类广泛应用于基因工程和生物技术领域的微生物。
在研究酵母中的基因功能时,敲除基因是一种常用的方法。
敲除基因可以揭示该基因在细胞生命周期中的作用,从而更深入地了解酵母细胞代谢、信号传导、生长和分裂等重要生物学过程。
敲除基因的方法有很多种,但是在酵母中最常用的方法是使用CRISPR/Cas9系统。
CRISPR/Cas9系统是一种先进的基因编辑技术,可以通过靶向DNA序列来精准地剪切和修复基因。
这使得科学家可以利用CRISPR/Cas9系统来快速、高效地敲除酵母中的目标基因。
使用CRISPR/Cas9系统进行酵母基因敲除的步骤如下:
1. 设计引物和gRNA:首先要选择与目标基因的特定序列匹配的引物和gRNA,用于引导CRISPR/Cas9系统到目标基因上。
2. 转化酵母:将CRISPR/Cas9系统和引物/gRNA导入酵母中,通常使用化学法或电转化法。
3. 筛选突变体:通过筛选来确定哪些酵母细胞中已经发生了目标基因的敲除。
通常使用PCR、Western blotting或功能鉴定等方法来验证敲除的效果。
4. 确认敲除的突变体:将酵母敲除体进行复制和纯化,以获得足够数量和纯度的敲除体。
通过CRISPR/Cas9系统敲除酵母基因的方法简单、高效、精准,并且可以应用于多种酵母品系和基因。
这使得酵母成为研究基因功能和生物过程的重要模型生物之一。
酵母基因敲除方法

酵母基因敲除方法
酵母基因敲除是一种常用的基因功能研究方法,其原理是通过DNA重组技术将目标基因的编码序列替换为可识别的选择标记物,进而利用选择标记物筛选敲除目标基因的突变菌株。
常用的选择标记物有抗生素抗性基因、荧光蛋白基因等。
酵母基因敲除方法的步骤主要包括:1.设计合适的引物,扩增选择标记物和目标基因的DNA片段;2.将选择标记物和目标基因的DNA 片段进行重组;3.将重组的DNA片段转化到酵母细胞中;4.筛选并鉴定目标基因敲除的突变菌株。
酵母基因敲除方法不仅可以用于研究基因功能,还可以用于制备工业酶、发掘新的代谢途径等方面。
但是该方法也存在着一些限制,例如敲除基因可能会导致生长变差或死亡等问题。
因此,在进行酵母基因敲除实验时需要根据具体情况进行优化和调整。
- 1 -。
毕赤酵母基因组编辑技术原理
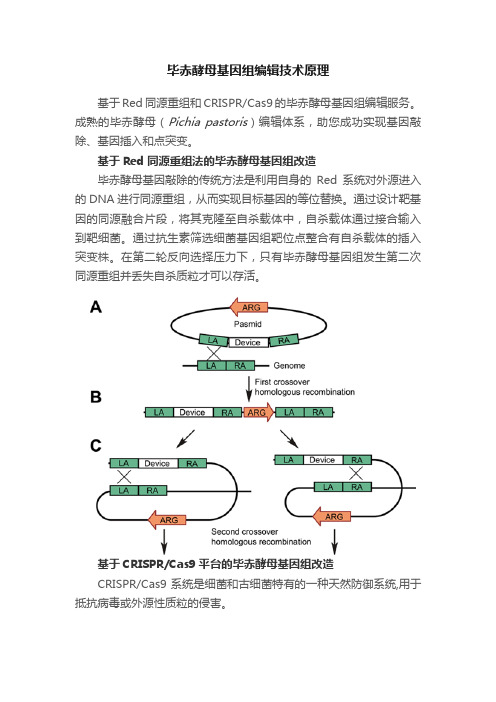
毕赤酵母基因组编辑技术原理基于Red同源重组和CRISPR/Cas9的毕赤酵母基因组编辑服务。
成熟的毕赤酵母(Pichia pastoris)编辑体系,助您成功实现基因敲除、基因插入和点突变。
基于Red同源重组法的毕赤酵母基因组改造毕赤酵母基因敲除的传统方法是利用自身的Red系统对外源进入的DNA进行同源重组,从而实现目标基因的等位替换。
通过设计靶基因的同源融合片段,将其克隆至自杀载体中,自杀载体通过接合输入到靶细菌。
通过抗生素筛选细菌基因组靶位点整合有自杀载体的插入突变株。
在第二轮反向选择压力下,只有毕赤酵母基因组发生第二次同源重组并丢失自杀质粒才可以存活。
基于CRISPR/Cas9平台的毕赤酵母基因组改造CRISPR/Cas9系统是细菌和古细菌特有的一种天然防御系统,用于抵抗病毒或外源性质粒的侵害。
研究内容•毕赤酵母基因敲除(Pichia pastoris gene knockout)•毕赤酵母基因敲入(Pichia pastoris gene knockin)•毕赤酵母基因点突变(Pichia pastoris gene point mutation)下游应用•抗生素及重要工业用酶•发现新的基因功能•优化代谢通路,提升代谢产物产量,实现工业化生产参考文献:•Selle K, Barrangou R. Harnessing CRISPR–Cas systems for bacterial genome editing[J]. Trends in microbiology, 2015, 23(4): 225-232.•Zerbini, F., Zanella, I., Fraccascia, D., König, E., Irene, C., & Frattini, L. F., et al. (2017). Large scale validation of an efficient CRISPR/Cas-based multi gene editing protocol in Escherichia coli.Microbial Cell Factories, 16(1), 68.。
巴斯德 [巴斯德毕赤酵母PEP4基因的剔除及对基因工程菌]
![巴斯德 [巴斯德毕赤酵母PEP4基因的剔除及对基因工程菌]](https://img.taocdn.com/s3/m/42946994dbef5ef7ba0d4a7302768e9951e76e3f.png)
3.2 毕赤酵母的高效转化及检测
验,按电泳条带亮度比较 PEP4 基因剔除前后毕赤酵母 MIP 的表达量,并
用 EcoRI 酶切质粒 pRS306/PEP4 L&R。由电泳结果可见,酶切后呈现
统计试验结果。
单一条带,酶切完全,见图 5。转化后在 SC-ura 平板上得到期望的转化
子。PCR 检测结果说明,置换前片段约为 1.7 kb,置换后片段约为 4.8 kb,
第3页共5页
本文格式为 Word 版,下载可任意编辑,页眉双击删除即可。
pRS306/PEP4L&R 进行转化,涂 SC-ura 平板,挑取单克隆在 YPD 培育基上
3 结果与商议
连CR 检测,根
据 DNA 片段大小判定毕赤酵母 PEP4 基因是否剔除〔置换前片段约为 1.7
激活液泡蛋白酶。在缺乏蛋白质水解酶 A 的状况下,有活性的蛋白质水解
巴斯德毕赤酵母〔Pichia pastoris〕是较理想的外源基因表达系统 酶 B 仍能在几个细胞生长周期内继续激活液泡蛋白酶,但蛋白质水解酶 B
[1]。在外源蛋白质的表达过程中,宿主菌毕赤酵母的胞内和胞外均有确 的自我激活能力并不是无限的,一旦其活性降低到某一临界值以下,细胞
3.1 构件质粒 pRS306/PEP4 L&R
kbp,置换后片段约为 4.8 kbp〕,并对 PCR 扩增得到的 DNA 片段测序。
3.1.1PCR 扩增 DNA 片段的鉴定 PCR 扩增 221 bp 毕赤酵母 PEP4 基因
将单菌落摇瓶培育后,依据羧肽酶 Y 平板检测法[10]鉴定 CPY 活性。 左臂和 225 bp 右臂 DNA 片段后进行琼脂糖凝胶电泳鉴定,说明其大小与
1.2 培育基
《毕赤酵母(Pichiapastoris)LNF013产纳豆激酶发酵优化及发酵动力学研究》

《毕赤酵母(Pichia pastoris)LNF013产纳豆激酶发酵优化及发酵动力学研究》一、引言随着生物技术的快速发展,酶类物质在工业、医药及生物工程等领域的应用日益广泛。
纳豆激酶作为一种天然存在的溶栓酶,因其具有良好的生物活性和稳定性而备受关注。
本文选取毕赤酵母(Pichia pastoris)LNF013作为纳豆激酶的生产菌株,通过对其发酵过程进行优化及发酵动力学研究,以期提高纳豆激酶的产量及发酵效率。
二、材料与方法2.1 材料本研究所用材料包括毕赤酵母LNF013菌株、发酵培养基、酶活性检测试剂等。
2.2 方法(1)发酵过程优化:通过调整培养基组成、发酵温度、pH 值、接种量等参数,对毕赤酵母LNF013产纳豆激酶的发酵过程进行优化。
(2)发酵动力学研究:采用动力学模型对发酵过程进行描述,分析各参数对纳豆激酶产量的影响,并建立相应的数学模型。
三、结果与分析3.1 发酵过程优化结果通过调整各参数,我们发现:(1)培养基组成:适当增加碳源、氮源浓度有助于提高纳豆激酶的产量。
(2)发酵温度:在30℃左右,毕赤酵母LNF013产纳豆激酶的活性较高。
(3)pH值:pH值为6.5左右时,菌株生长及纳豆激酶产量达到最佳状态。
(4)接种量:适宜的接种量有助于提高菌株的生长速度及纳豆激酶的产量。
3.2 发酵动力学研究结果通过建立动力学模型,我们发现:(1)菌体生长与纳豆激酶产量之间存在明显的相关性,可通过监测菌体生长情况来预测纳豆激酶的产量。
(2)各参数对纳豆激酶产量的影响程度不同,其中培养基组成、温度和pH值对纳豆激酶产量的影响最为显著。
四、讨论通过对毕赤酵母LNF013产纳豆激酶的发酵过程进行优化及发酵动力学研究,我们找到了提高纳豆激酶产量的关键因素。
在实际生产中,可根据实际情况调整这些参数,以实现更高的纳豆激酶产量及发酵效率。
此外,建立的动力学模型有助于预测和调控发酵过程,为工业化生产提供有力支持。
酵母基因敲除方法

酵母基因敲除方法
酵母基因敲除方法
酵母是基础研究中常用的模式生物之一,因其单细胞生长方式方便快速、易于操作以及基因遗传和代谢途径等知识已经得到很好的解析,
近年来尤其受到越来越广泛的关注。
对酵母中基因的敲除是进行基因
功能研究的重要手段之一,本文将讨论酵母基因敲除的一些方法。
一、全基因敲除法
该方法就是将酵母全部基因一一敲除,以期望发现哪些基因是细胞生长、分裂、代谢等生理过程所必不可少的基因,这些基因的缺失可能
导致细胞生长受到影响。
在这种方法中,酵母细胞需要被地基选择毒杀,仅有具备特殊生存能力的敲除株能够生长存活。
二、基因与载体插入融合法
该方法是在基因DNA序列的两端构建一对定向限制性内切酶切除位点,通过中间连接带卡宾磷酸基团的PCR产物和酵母选择载体的重组结合,将新构建的基因与选择载体融合。
将该构建好的载体转化到酵母中,
由于基因与选择载体已经融合在一起,所以密切相连,转化后选择带
有此载体的细胞株后,便得到含有新融合基因全部位点的酵母株。
三、基因敲除杂交法
该方法是在酵母中,将敲除目标基因的G418盐酸盐抗性基因与其他选
择基因杂交,将杂交产物导入到酵母中,筛选出具有双重辅助杂合基
因的酵母株。
接着,再将含有G418抗性基因的敲除载体转入酵母中,
经过选择,得到新的敲除株。
总结
以上是酵母基因敲除的三种方法,可以根据自己的需要选择实验方案。
酵母是广泛应用的系统生物,而基因敲除方法是酵母生物学研究中最
常用的工具之一,掌握好这些方法会使酵母生物学的研究更加得心应手。
《毕赤酵母(Pichiapastoris)LNF013产纳豆激酶发酵优化及发酵动力学研究》

《毕赤酵母(Pichia pastoris)LNF013产纳豆激酶发酵优化及发酵动力学研究》一、引言毕赤酵母(Pichia pastoris)作为一种重要的工业生产微生物,近年来在生产纳豆激酶方面展现出了良好的应用前景。
纳豆激酶是一种能够促进血液凝固和溶栓的酶,在医药和生物工程领域具有广泛的应用价值。
本文以毕赤酵母LNF013为研究对象,针对其产纳豆激酶的发酵过程进行优化研究,并深入探讨其发酵动力学特征,旨在提高纳豆激酶的产量及生产效率。
二、材料与方法2.1 材料实验所用毕赤酵母LNF013菌株、培养基及其他试剂均符合实验要求。
2.2 方法(1)发酵过程优化:通过调整发酵温度、pH值、接种量、培养时间等参数,探究各因素对纳豆激酶产量的影响。
(2)发酵动力学研究:通过实验数据,分析毕赤酵母LNF013在生长和产酶过程中的动态变化,建立发酵动力学模型。
三、结果与分析3.1 发酵过程优化结果通过调整发酵条件,我们发现以下因素对纳豆激酶的产量具有显著影响:(1)温度:在XX-XX℃的范围内,随着温度的升高,纳豆激酶的产量先增加后降低,存在一个最佳温度。
(2)pH值:pH值对纳豆激酶的产量也有较大影响,在pH 值为XX-XX的范围内,纳豆激酶产量较高。
(3)接种量:适当的接种量有利于提高纳豆激酶的产量,但过高的接种量可能导致竞争生长,降低产量。
(4)培养时间:随着培养时间的延长,纳豆激酶的产量逐渐增加,但达到一定时间后,产量增长速度减缓。
3.2 发酵动力学分析根据实验数据,我们绘制了毕赤酵母LNF013的生长曲线和纳豆激酶产量曲线。
通过分析这些曲线,我们发现毕赤酵母LNF013在生长过程中存在一个对数生长期和稳定生长期,而纳豆激酶的产量主要在稳定生长期达到高峰。
此外,我们还建立了发酵动力学模型,分析了各因素对毕赤酵母LNF013生长和产酶的影响。
四、讨论通过本文的研究,我们发现在一定的范围内调整发酵条件可以显著提高毕赤酵母LNF013产纳豆激酶的产量。
基因敲除酵母技术

技术简介
同源重组系统是微生物基因编辑经典的一种方法,可以实现对DNA分子的敲除、点突变、敲入等多种修饰。
该技术已被广泛地用于基因组DNA,如细菌人工染色体、大肠杆菌染色体等的遗传修饰研究以及基因工程药物的研究和开发中。
该生物研发团队在基于经典同源重组系统的基础上,通过自主研发优化构建载体和实验流程,开发了一项敲除效率和准确性均远高于传统方法的创新性的同源重组改良技术。
该技术可以广泛应用于酵母的基因编辑。
技术优势
独家创新技术,基因敲除效率提升10倍;
基因敲除效率和重组效率是传统技术的20-30倍以上;
轻松实现基因敲除,基因点突变和基因敲入;
技术流程
质控标准
1. 菌落PCR鉴定
2. 靶位点测序鉴定
南京瑞源生物技术有限公司坐落于南京市栖霞区生命科技园,专注于基因组高通量研究领域,致力于生物高科技研发,目前产品的技术水平已达到省级实验水平,瑞源生物立足于生命科学,为基础研究领域科学工作者提供生物学技术服务,自2019年创立以来,全体员工致力于利用酵母系统研发新药物靶点,专注于生物学研究,长期与全国各大农林/医药院校在大型科研技术研发上紧密合作。
毕赤酵母表达蛋白糖基化位点的方法

一、概述毕赤酵母是一种常见的真菌,它在生物技术和分子生物学领域有着广泛的应用。
在这些领域,研究人员经常需要对蛋白质进行糖基化修饰的研究,而毕赤酵母表达系统正是其中的一种重要工具。
本文将就毕赤酵母表达蛋白糖基化位点的方法进行介绍。
二、毕赤酵母表达系统简介1. 毕赤酵母表达系统的原理毕赤酵母表达系统是指利用毕赤酵母表达载体,将目标蛋白基因导入毕赤酵母中,使其在毕赤酵母中进行表达。
该系统具有高度的复制和表达效率,能够在较短的时间内高效地产生目标蛋白。
2. 毕赤酵母表达系统的优势和应用毕赤酵母表达系统具有许多优势,例如能够进行大规模的表达,提高了蛋白质的产量;同时也能够实现正常的翻译后修饰以及蛋白折叠功能。
在生物技术和分子生物学领域有着广泛的应用,如药物开发、生物能源等领域。
三、毕赤酵母表达蛋白糖基化位点的方法1. 利用质粒表达毕赤酵母表达载体中含有丰富的糖基化因子,对于糖基化位点的研究提供了便利。
研究人员可以将目标蛋白基因克隆至毕赤酵母表达载体中,通过大规模的表达筛选,筛选出糖基化位点进行研究。
2. 利用质粒诱导表达研究人员还可以通过对毕赤酵母进行质粒诱导,使其表达特定的糖基化酶,从而实现对特定蛋白质的糖基化位点的研究。
这种方法能够有效地降低研究成本,是当前常用的研究手段之一。
3. 基因敲除或过表达最近,基因敲除或过表达技术在毕赤酵母的研究中得到了广泛的应用。
研究人员可以通过敲除特定的糖基化酶基因或过表达其基因,从而实现对糖基化位点的研究。
这种方法能够帮助研究人员更深入地了解糖基化位点在蛋白质功能中的作用。
四、毕赤酵母表达蛋白糖基化位点研究的意义1. 为蛋白质功能研究提供重要依据研究糖基化位点能够帮助人们更深入地了解蛋白质的结构和功能。
糖基化位点通常与蛋白质的功能密切相关,通过研究糖基化位点,可以为蛋白质功能的研究提供重要的依据。
2. 为药物研发提供理论支持糖基化位点在药物研发中也有着重要的意义。
许多药物的研发过程中需要考虑蛋白质的糖基化修饰,因此对糖基化位点进行研究能够为药物研发提供理论支持。
毕赤酵母表达实验手册--生物秀
[ 生物部落—生物人网络家园 ]·[ 生物秀论坛—学术交流、资源共享与互助社区 ]·[ 生物秀知道—生命科学问题解决之道 ]·[ 生物百科—生命科学第一百科 ]
生物秀首页 生物部落 生物百科 新闻前沿 产业资讯 实验技术 招聘信息 会议会 展 生物秀知道 生物秀论坛 资源下载 生物人物 物秀商城 生物图片 仪器教程 生物考研 视频动 画 易生物商务
[1] [2] [3] [4] [5] [6] [7] [8] 下一页
◇ Alexander、5632285、he_furong 完成了有奖 任务 领取每日积分大礼包,领取了 5 个奖励积分 3分钟前
◇ he_furong 更新了自己的兴趣爱好等个人信息 3分钟前
◇ he_furong 更新了自己的教育情况 4分钟前
实验
虽然简化了产物的纯化,但不利于产物的活 生产PCR仪已有十年历史, 产品畅销国内 外多个城
电书 ——
性,为了得到有活性的蛋白,就需要进行变
市.欢迎来电咨询!
实验 视频
性溶解及复性等操作,这一过程比较繁琐,
——
同时增加了成本。
实验
大肠杆菌是用得最多、研究最成熟的基因工程表达系统,当前已商业化的基因工程产品大多是通过 下载
首页 | 基础 | PCR | DNA | RNA | 芯片 | 电泳 | 微生物 | 细胞 | 生化 | 生理 | 植物 | 免疫 | 遗传 | 神经 | 模式生物 | 实验室 | 仪器 | 工具 | 软件 | 教程 | 论坛
实验论坛>>>> [核酸基因技术] [细胞实验技术] [蛋白实验] [生化与免疫] [PCR交流] [模式生物] [实验室建设与安全] [综合讨论] 马上注册 ←生命科学实验交流阵地!
一种毕赤酵母高效基因敲除方法[发明专利]
![一种毕赤酵母高效基因敲除方法[发明专利]](https://img.taocdn.com/s3/m/3798bd579a6648d7c1c708a1284ac850ad02042d.png)
(19)中华人民共和国国家知识产权局(12)发明专利申请(10)申请公布号 (43)申请公布日 (21)申请号 201810853635.4(22)申请日 2018.07.30(71)申请人 惠州卫生职业技术学院地址 516000 广东省惠州市惠城区三栋镇福长岭惠南大道(72)发明人 李莉玲 (74)专利代理机构 惠州市超越知识产权代理事务所(普通合伙) 44349代理人 陈文福 陈惠珠(51)Int.Cl.C12N 15/90(2006.01)C12N 1/19(2006.01)C12R 1/84(2006.01)(54)发明名称一种毕赤酵母高效基因敲除方法(57)摘要本发明提供一种毕赤酵母高效基因敲除方法,其特征在于,包含以下步骤:S1.通过质粒pYES2构建敲除质粒pGE1203-ADE -URA3;S2.将敲除质粒分别敲入所述毕赤酵母组中目标基因och1的上游、下游;S3.具有敲除质粒活性的蛋白质识别、剪切所述核苷酸序列,敲除所述目标基因och 1,获得敲除所述目标基因och 1的毕赤酵母。
本发明有效改善毕赤酵母对蛋白的糖基化修饰,解决融合蛋白过度糖基化的问题,对毕赤酵母och1基因进行了高效敲除。
权利要求书1页 说明书4页序列表2页CN 109082444 A 2018.12.25C N 109082444A1.一种毕赤酵母高效基因敲除方法,其特征在于,包含以下步骤:S1. 通过质粒pYES2构建敲除质粒pGE1203-ADE -URA3;S2.将敲除质粒分别敲入所述毕赤酵母组中目标基因och 1的上游、下游;S3. 具有敲除质粒活性的蛋白质识别、剪切所述核苷酸序列,敲除所述目标基因och 1,获得敲除所述目标基因och 1的毕赤酵母。
2.根据权利要求1所述的一种毕赤酵母高效基因敲除方法,其特征在于,步骤S1中,所述敲除质粒pGE1203-ADE -URA3的构建方法为:以质粒pYES2为模板,通过扩增引物P1和P2进行PCR反应,生成pGE1201;通过已知毕赤酵母菌株为模板,设计目标基因och 1的同源臂序列P3和P4;通过毕赤酵母URA3基因设计寡核苷酸引物P5和P6;通过酿酒酵母ADE基因设计寡核苷酸引物P7和P8;将och 1 5’端同源臂以Bam H I/Not I酶切,克隆到用Bam H I/Not I酶切的pGE1201质粒上,形成pGE1202,再将3’端同源臂以Not I和KpnI酶切,克隆到用Not I和Kpn I酶切的pGE1202上,形成pGE1203;再将URA3基因以Pst I/Kpn I酶切,克隆到同样用Pst I/Kpn I 酶切的pGE1203上,形成pGE1203- URA3;将pGE1203- URA3以Not I酶切,再用CIAP去磷酸化,与同样用Not I酶切的ADE基因连接,形成敲除质粒pGE1203-ADE -URA3。
高质量毕赤酵母基因组DNA提取方法比较

Abstrac:t In orde r to find an optim ized and e ffective m ethod, the re w ere five m ethods o f yeast g enom ic DNA extrac tion to be compared, including sna ilase, u ltrasonic ex traction, liqu id nitrogen a ttr ition, ly ticase and yeast DNA k it m ethods T he genom ic DNA qua lity w as exam ined by e lectrophoresis and u ltrav iolet spectropho tom eter R esults dem onstrated that genom ic DNA of P ichia p astor is could be iso la ted by a ll the five me thods, in wh ich snailase m ethod w as the best one to get h igh qua lity DNA T here fore, the snailase m ethod show ed m any advantag es such as relatively low expense and h igh effic iency over the rest
g) 非常高质量的基因组 DNA 用于酶切消 化。本 试验通过几种提取酵母基因组 DNA 的不同方法, 探 索出适合大规模提取高质量的酵母基因组 DNA 的 方法, 为进一步鉴定提供保证。
收稿日期: 2009 10 09 基金项目: 国家 863 计划 ( 2007AA 02Z114) , 国家自然科学基金 项目 ( 30860368) , 重大新药 创制国家 科技重大 专项课题ห้องสมุดไป่ตู้项目 ( 2009ZX 09103
酵母基因敲除方法

酵母基因敲除方法
酵母基因敲除方法是一种常用的研究酵母遗传学的实验技术。
其主要思想是通过基因编辑技术将目标基因的DNA序列修改,使其无法继续编码功能性蛋白质,从而实现对基因功能的敲除。
在实验过程中,研究者首先需要设计并合成适当的sgRNA(单指引RNA),然后将其与Cas9核酸酶结合成复合物。
接下来,将该复合物导入到酵母细胞中,使其在靶向目标基因的DNA序列时发挥作用。
随后,由于Cas9核酸酶的活性,该酵母细胞的目标基因将被切割并失去功能性。
为了验证敲除效果,研究者通常会对敲除后的酵母细胞进行基因序列测序,以确认目标基因的DNA序列已被有效修改。
同时,研究者也会对敲除后的酵母细胞进行基因表达水平的分析,以确定目标基因敲除是否对酵母细胞的生长和代谢产生了影响。
总的来说,酵母基因敲除方法是一种非常有用的实验技术,它可以帮助研究者深入了解酵母的基因功能和生理特性,从而推动酵母遗传学领域的发展。
- 1 -。
酵母基因敲除方法

酵母基因敲除方法
酵母基因敲除方法是一种广泛应用于酵母遗传学研究中的技术。
该方法利用CRISPR/Cas9系统或者遗传转化的方式,将目标酵母基因的DNA序列进行改造或删除,从而实现对该基因的敲除。
这种方法可以用来研究酵母基因的功能、调控机制以及其在生物过程中的作用,对于探究生命科学领域的一系列问题,具有重要的意义。
在酵母基因敲除方法中,首先需要设计和合成sgRNA,这是一种由20个核苷酸组成的RNA序列,用于特异性识别和切割目标基因的DNA。
接着,将sgRNA和Cas9蛋白共同导入到酵母细胞中,通过与目标基因的DNA序列发生特异性配对,实现Cas9蛋白的定向切割。
切割后的DNA序列可以通过同源重组等方式进行修复,从而实现基因敲除。
此外,还可以利用遗传转化的方式,将外源的DNA序列导入到酵母细胞中,通过同源重组等机制,将外源DNA序列与目标基因的DNA 序列进行融合或替换,从而达到基因敲除的效果。
总之,酵母基因敲除方法是一种常用的酵母遗传学研究技术,可以用于研究酵母基因功能、调控机制以及其在生物过程中的作用。
该技术的应用,为生命科学领域的研究提供了新的手段和思路。
- 1 -。
采用基因敲除手段降低啤酒酵母蛋白酶A表达的研究

采用基因敲除手段降低啤酒酵母蛋白酶A表达的研究
周建中;王德良;王忠民;赵丽娟;葛邦国
【期刊名称】《酿酒》
【年(卷),期】2006(33)1
【摘要】采用醋酸锂转化法,将一段目的基因转入到酵母体内,破坏酵母PEP4基因的表达.通过对目的基因的扩增和羧肽酶Y酶活检测验证基因敲除情况,并通过传代抗性试验与传代发酵稳定性实验验证突变株遗传稳定性良好.
【总页数】3页(P62-64)
【作者】周建中;王德良;王忠民;赵丽娟;葛邦国
【作者单位】新疆农业大学食品学院,乌鲁木齐,830052;中国农业大学食品学院,北京,10009;中国食品发酵工业研究院酿酒工程部,北京,100027;新疆农业大学食品学院,乌鲁木齐,830052;沈阳农业大学食品学院,沈阳,110161;新疆农业大学食品学院,乌鲁木齐,830052
【正文语种】中文
【中图分类】TS261.11;TS262.5;Q789
【相关文献】
1.低蛋白酶A啤酒酵母突变株及其基因突变机理和中试、生产规模的试验研究 [J], 涂京霞;王德良;孔祥诚;宋绪磊;李惠萍
2.采用复合诱变选育低分泌蛋白酶A啤酒酵母菌株的研究 [J], 王德良;吴小映;李
惠萍;张五九
3.蛋白酶A检测技术在啤酒酵母选育中的应用 [J], 刘伟成;张五九;邓启华;贾凤超;
郑仰民;冯景章
4.甲基磺酸乙酯(EMS)诱变选育低产蛋白酶A啤酒酵母的研究 [J], 陈旭;王德良;俞雅琼;王晓娟;王志萍;杨海燕
5.溶酶体保护蛋白/组织蛋白酶A基因敲除小鼠听力和耳形态学初步研究 [J], 郭运凯;谢鼎华;杨新明
因版权原因,仅展示原文概要,查看原文内容请购买。
毕赤酵母PMR1的分子克隆及其无义突变株的构建的开题报告

毕赤酵母PMR1的分子克隆及其无义突变株的构建
的开题报告
一、研究背景和目的
细胞膜是细胞内外物质交换的通道和障碍物,对细胞功能的调节和
细胞生存至关重要。
钙离子是细胞内重要的信号分子,在细胞膜的钙离
子通道中发挥着重要功能。
毕赤酵母(Saccharomyces cerevisiae)作为
一种常见的真菌模式生物,其细胞膜的钙离子通道在生理学上具有研究
价值。
本研究旨在通过分子克隆和构建无义突变株,探究毕赤酵母PMR1基因在细胞膜钙离子通道中的作用机制。
二、研究内容和方法
1. PMR1基因的分子克隆
将含有PMR1基因的DNA片段扩增,并将其插入到载体上,经过蓝白斑筛选和PCR检测,最终得到PMR1基因的克隆片段。
将该片段进行
测序,验证其准确性。
2. 无义突变株的构建
构建PMR1基因敲除株和无义突变株,通过PCR检测、蓝白斑筛选
和测序验证。
3. 检测细胞膜钙离子通道的功能
通过荧光探针法检测细胞内钙离子水平及细胞膜钙离子通道的活性,比较PMR1基因敲除株和无义突变株与野生型毕赤酵母的差异。
三、研究意义和创新点
本研究通过PMR1基因敲除和无义突变的方式,探究毕赤酵母PMR1基因在细胞膜钙离子通道中的作用机制。
这将有助于深入了解细胞膜钙
离子通道的调控机制和信号转导途径,为相关疾病的研究提供新思路和
理论依据。
同时,本研究的创新点主要在于利用无义突变技术探究PMR1基因的功能,为细胞膜钙离子通道的研究提供一种新方法。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
—
竺
G S 1 l 5 H i r 染 色 体 上 发生插入替换后的 P R C1基因
性基 因重组 片段 , 整合 到宿主菌体 内 , 发生 同源重组 , 导致插入
的抗性 基因替换掉宿 主染色体上 的 目的基因 , 从而 实现 目的基
因的敲除。
图 1 基 因直 接插 入替 换 原 理 图
巴斯德毕赤酵 母( P i c h i a p a s t o r i s ) 是1 9 世纪 8 0 年代初期发展起来 的一种新型 的外源蛋 白表 达系统 , 在生物表 达 系统 中研 究最为广泛 。巴斯德毕赤酵母具 有易于高密度 发酵 、 培养基成本低 、 外源基 因整合稳定 、 外 源蛋 白以 分泌 蛋 白形 式存 在 、 内生蛋 白较 少 , 以及 蛋 白翻译后真核 加工等优点 。该 系统相 较其他表达系统 而言 , 弥补 了大 肠杆菌表达 系统不易表达复杂蛋 白的限制 以及植物 、 动物细胞表达系统周期长 、 操作繁琐 、 成本高 的缺点 , 完善 了 酿酒酵母 ( S a c c h a r o m y c e s c e r e v i s i a e ) 分 泌效 率低 、 表达菌株 不够稳定 、 表达质粒 易丢失等缺 陷 , 迅速成 为各 国科学 工作者 的研究热点 。 但是人们也发 现 了巴斯德毕赤 酵母外 源表达 系统 的不足。在表达外源蛋 白时 , 巴斯德毕赤 酵母 重组菌往往 存 在 比较严重 的蛋 白降解 现象 。酵母 表达外 源糖蛋 白时会对蛋 白进 行过度 N 一 糖基 化修饰 , 产生高甘 露糖型糖 链, 影 响蛋 白的活性 , 从而 限制 了巴斯德毕赤酵母表达人源型蛋 白表达在 医药方 面的应用 。随着 2 0 0 9 年 巴斯德毕 赤酵母 全基 凶组测序和 注释工作 的完成 , 人们对影响其转录调控 、 表达调控 及 翻译后修饰 等机 理有 了进一 步的 了解 , 可以通过相关 基因 的 敲除达到影响表 达蛋 白途径 , 从而实现改 良的毕赤酵母重 组菌
第3 4卷 第 6期 2 0 1 4年 1 2月
惠州学院学报 ( 自然科 学版)
J OURNAL OF HU I ZHOU UNI VE RS I TY
Vo 1 . 3 4 . N o . 6
De c , . 2 01 4
毕 赤 酵 母 直 接 基 因敲 除 方 法 的 研 究
收 稿 日期 : 2 0 1 4—0 7— 2 5
作者简介 : 李莉玲( 1 9 7 5一 ) , 女, 江西定南人 , 讲 师, 研究方 向为有机化学。
第 6期
李莉玲 : 毕赤酵母直接基 因敲除方 法的研 究
・3 3・
后 的重组 菌 。为 了提高筛选效率 , 常根据被敲 除的宿主菌 的性质 同时引入一个 正向筛选标记 , 进行正 反 向筛选 。
编码一个毒素 M a z F , 这是 一个 重要 的毒素一 抗 血清系统 , 介导 的细胞程序性死 亡 。M a z F 作为一种 m R N A干扰酶 ,
会优先切断在 A C A( 特定序列 ) 上 的单链 m R N A以阻止蛋 白合成 , 导致细胞生长阻遏 ] 。
m a z F 基 因被放置在诱 导 的表达 系统 里 , 同时 Z e o c i n 抗性基 因作为一个 正向的筛选标志 。但是一般 用于在功 能基 因片段 中插入一段序列 , 以破坏该基 因的正常转 录与翻译 , 从 而实现基因的功能缺 失。
的构建 。
巴斯德毕赤酵母 中常用 的直接基 因缺失 的方法主要分 为两
类: 有标记 的插入替换方法和无标记的 P o p — I n / P o p — O u t 方法…。
R扩 增
1 有标 记的插 入替 换方 法
有标记 的插入 替换方法 以抗 性基 因作 为筛选标记 , 在抗性
U R A 3 基 因与 T — u r f l 3 基 因是两个常用作 P o p — I n po / p — O u t 方法里 的反 向筛选标 记 , 但 由于它们分别给重 组毕赤酵
母带来 的生长缓慢 以及毒性导致 的低存 活率 都限制了这两种反向标记实现非标 记敲除基因方 法的应用 。 大肠 杆菌 的 m a z F 作 为一 种新 的反 向筛选 标志被 用于 巴斯德毕 赤酵母 的 P o p — I n / P o p — O u t 敲 除基 因 。m a z F
基 因片 段两边 通过 P C R扩增接上 被 敲除基 因在宿 主基 因两边
GS I 1 5 H i t染色 体 上 的 P R C1 基 因
● l
的同源片段 。将 两端 连有 同源片段 的抗性基 因通过双酶切后连 入载体质粒。以该 重组质粒 为模板得到两端连有 同源 片段 的抗
李莉玲
( 广东惠州 卫生职业技术学院 药学系 ,广 东 惠州 5 1 6 0 0 5 )
摘 要: 甲醇 营养 型巴斯德 毕赤酵母 已被 广泛应用于外 源蛋 白的表 达 , 但是 作为一个外 源蛋 白表 达 系统 , 其还
有待进一步 完善 。进一 步提 高外源蛋 白的表达量 以及形成折 叠正确的 目的蛋 白, 成为 了改造毕赤 酵母 菌株 的一个 热点 。基 因缺 失, 是一个常 用的改造和 构建新的毕赤酵母重组 菌株 常用的方法。 巴斯德毕赤 酵母 直接 基 因缺失方 法主要分 为两类 : 有标记 的插入 替换和无标记的 P o p — I n / P o p — O u t 。本文 系统地介绍 了这 两种方法 , 分别比较 了各 自 的优缺点 , 为它们在 巴斯德毕赤酵母 中进 行基因敲除提供 了一定参考 意义
关键 词 : 巴斯 德 毕 赤 酵 母 ; 基 因缺 失 ; 反 向 筛 选标 记 ; P o p — I n / P o p — O u t ; C r e — l o x P 中图 分 类 号 : Q 9 3 . 3 文献标识码 : A 文章编号 : 1 6 7 1 —5 9 3 4( 2 0 1 4 ) 0 6—0 0 2 8—0 8
