picbio 三代测序原理
三代基因组测序技术原理简介
三代基因组测序技术原理简介摘要:从1977年第一代DNA测序技术(Sanger法)1,发展至今三十多年时间,测序技术已取得了相当大的发展,从第一代到第三代乃至第四代,测序读长从长到短,再从短到长。
虽然就当前形势看来第二代短读长测序技术在全球测序市场上仍然占有着绝对的优势位置,但第三和第四代测序技术也已在这一两年的时间中快速发展着。
测序技术的每一次变革,也都对基因组研究,疾病医疗研究,药物研发,育种等领域产生巨大的推动作用。
在这里我主要对当前的测序技术以及它们的测序原理做一个简单的小结。
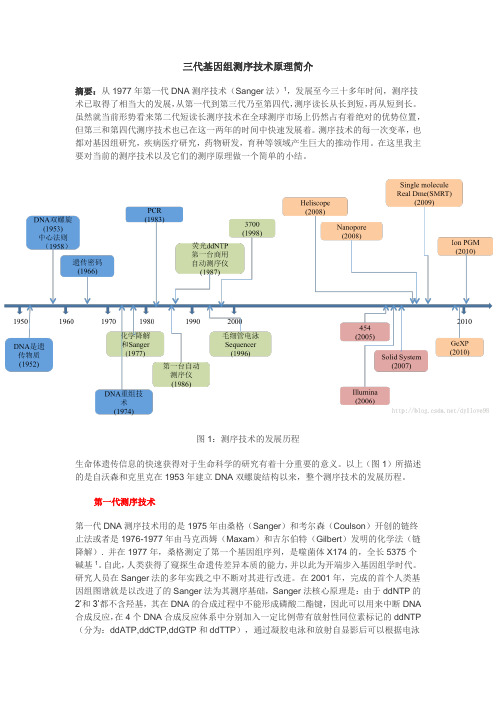
图1:测序技术的发展历程生命体遗传信息的快速获得对于生命科学的研究有着十分重要的意义。
以上(图1)所描述的是自沃森和克里克在1953年建立DNA双螺旋结构以来,整个测序技术的发展历程。
第一代测序技术第一代DNA测序技术用的是1975年由桑格(Sanger)和考尔森(Coulson)开创的链终止法或者是1976-1977年由马克西姆(Maxam)和吉尔伯特(Gilbert)发明的化学法(链降解). 并在1977年,桑格测定了第一个基因组序列,是噬菌体X174的,全长5375个碱基1。
自此,人类获得了窥探生命遗传差异本质的能力,并以此为开端步入基因组学时代。
研究人员在Sanger法的多年实践之中不断对其进行改进。
在2001年,完成的首个人类基因组图谱就是以改进了的Sanger法为其测序基础,Sanger法核心原理是:由于ddNTP的2’和3’都不含羟基,其在DNA的合成过程中不能形成磷酸二酯键,因此可以用来中断DNA 合成反应,在4个DNA合成反应体系中分别加入一定比例带有放射性同位素标记的ddNTP (分为:ddATP,ddCTP,ddGTP和ddTTP),通过凝胶电泳和放射自显影后可以根据电泳带的位置确定待测分子的DNA序列(图2)。
这个网址为sanger测序法制作了一个小短片,形象而生动。
值得注意的是,就在测序技术起步发展的这一时期中,除了Sanger法之外还出现了一些其他的测序技术,如焦磷酸测序法、链接酶法等。
一代,二代,三代测序原理
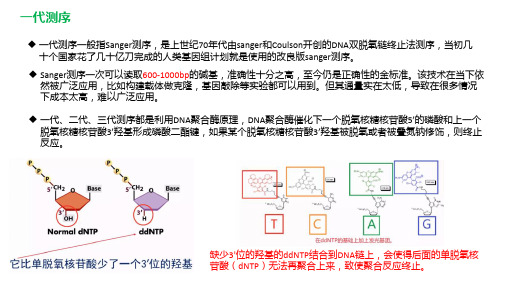
一代测序一般指Sanger测序,是上世纪70年代由sanger和Coulson开创的DNA双脱氧链终止法测序,当初几 十个国家花了几十亿刀完成的人类基因组计划就是使用的改良版sanger测序。
Sanger测序一次可以读取600-1000bp的碱基,准确性十分之高,至今仍是正确性的金标准。该技术在当下依 然被广泛应用,比如构建载体做克隆,基因敲除等实验都可以用到。但其通量实在太低,导致在很多情况 下成本太高,难以广泛应用。
二代测序
二代测序技术,又称为Next Generation Sequencing(NGS)技术,高通量测序技术, 是为了改进一代测序通量过低的问题而出现的。刚面世时主要包括Roche公司的454技 术、ABI公司的Solid技术和Illumina公司的Solexa技术。这三种技术都极大的提高了测 序的通量,大大降低了测序成本和周期。
➢ 二代测序和一代测序最大的不同点在于其边合成边测序技术。
二代测序
二代测序
测序流动槽(flowcell): 每个槽都有共价交联的两种oligo(P5和P7),分别与两 端的接头互补。DNA聚合酶
P5 P7
桥式PCR合成另一条链
NaOH解开双链
NaOH解开双链 后模板链被洗掉
二代测序
流动槽加入引物 Rd1 SP、DNA 聚合酶、荧光标 记的dNTP,对 第一条链测序
三代测序
SMRT Cell含有纳米级的零模波导孔,每个ZMW都能够包含一个DNA聚合酶及一条DNA样品链进行单分子测序, 并实时检测插入碱基的荧光信号。ZMW是一个直径只有10~50 nm的孔,当激光打在ZMW底部时,只能照亮很小 的区域,DNA聚合酶就被固定在这个区域。只有在这个区域内,碱基携带的荧光基团被激活从而被检测到,大幅 地降低了背景荧光干扰。
三代基因组测序技术简介及其原理整理

三代基因组测序技术简介及其原理整理第一代测序技术第一代DNA测序技术用的是1975年由桑格(Sanger)和考尔森(Coulson)开创的链终止法以及1976-1977年由马克西姆(Maxam)和吉尔伯特(Gilbert)发明的化学法(链降解)。
1977年,桑格测定了第一个基因组序列——噬菌体X174,全长5375个碱基。
自此,人类获得了窥探生命遗传差异本质的能力,并以此为开端步入基因组学时代。
研究人员在Sanger法的多年实践之中不断对其进行改进。
在2001年,完成的首个人类基因组图谱就是以改进了的Sanger法为其测序基础。
Sanger法原理:1)在模板指导下,DNA聚合酶不断将dNTP(N=A/G/T/ C)加到引物的3’- OH末端,合成出新的互补链。
在4个DNA合成反应体系中分别加入一定比例带有放射性同位素标记的ddNTP,在互补链在DNA聚合酶作用下延伸时,一旦连接上ddNTP,由于双脱氧核糖的2’和3’都不含羟基,故不能同后续的dNTP形成磷酸二酯键而终止反应,随即形成一系列不同长度的、以同样引物为起始、以同一碱基终止的短片段混合物。
2)双脱氧核苷酸在每个DNA分子中掺入的位置不同,采用聚丙烯酰胺凝胶电泳区分长度差一个核苷酸的单链DNA,从而读取DNA核苷酸序列。
化学裂解法原理:与Sanger法类似,将DNA模板分成4个反应。
在每个反应中,先在模板5’端进行放射性标记,再加入能特异性在其中一种碱基处切开DNA的化学试剂。
反应进行时,平均一个DNA分子只在随机位点产生一次裂解。
接着,通过凝胶电泳和放射自显影后可以根据电泳带的位置确定待测分子的DNA序列。
第二代测序技术第一代测序技术的主要特点是测序读长可达1000bp,准确性高达99.999%,但其测序成本高,通量低等方面的缺点,严重影响了其真正大规模的应用。
因而第一代测序技术并不是最理想的测序方法。
经过不断的技术开发和改进,以Roche公司的454技术、illumina公司的Solexa,Hiseq技术和ABI公司的Solid技术为标记的第二代测序技术诞生了。
三代测序原理

三代测序原理
三代测序原理是指第三代测序技术,又称为单分子测序技术。
与第一代(Sanger测序)和第二代(高通量测序)相比,第三代测序技术具有更高的速度、更低的成本和更长的测序读长等优点。
第三代测序技术的原理主要是基于测序模板的直接测序,而不需要PCR扩增。
这种直接测序的方法可以避免PCR扩增引入
的错误,并且能够在一个测序周期内得到完整的序列信息。
在第三代测序技术中,常用的方法是通过将DNA分子固定在
一个载体上,形成DNA聚集体。
然后,通过负电荷的方式将
这些DNA聚集体附着在固定的表面上,形成一个DNA分子
阵列。
接着,通过使用荧光染料将这些固定的DNA分子标记出来,
并且使用激光束在一个固定的区域内进行扫描。
这样,就可以得到每个DNA分子的位置和荧光信号强度信息。
在测序过程中,通常会使用一种特殊的酶来控制DNA链的合
成过程。
这种酶能够识别每个碱基的序列信息,并且在特定的条件下将其添加到适当的位置。
通过不断重复这个步骤,直到测序反应完成,就可以得到整个DNA分子的序列信息。
总结起来,第三代测序技术的原理是通过直接测序DNA模板,
不需要PCR扩增,通过固定DNA分子并使用荧光标记,通过酶的作用在特定条件下完成碱基的添加,最终得到完整的
DNA序列信息。
这种技术具有快速、低成本和长读长等优势,在各种生物学研究中得到了广泛的应用。
picbio三代测序原理

三代测序之PacBio SMRT技术全解析2017-05-11 11:29 来源:基因谷技术气温回升,天气渐暖,花儿开了一簇又一簇~在这美好的季节里,我们准备聊点新话题。
今天小编要来和你分享:PacBio SMRT测序那些事儿~测序技术在近几年中又有里程碑的发展,Pacific Biosciences公司成功推出商业化的第三代测序仪平台,让三代测序正式走入我们的视线。
与前两代相比,第三代测序有什么不同呢?今天小编带大家详细了解测序界新宠-PacBio SMRT测序平台。
PacBio SMRT测序原理Pacific Biosciences公司研发的单分子实时测序系统(Single Molecule Real Time,SMRT)应用了边合成边测序的原理,并以SMRT芯片为测序载体。
基本原理如下:聚合酶捕获文库DNA序列,锚定在零模波导孔底部4种不同荧光标记的dNTP随机进入零模波导孔底部荧光dNTP被激光照射,发出荧光,检测荧光荧光dNTP与DNA模板的碱基匹配,在酶的作用下合成一个碱基统计荧光信号存在时间长短,区分匹配碱基与游离碱基,获得DNA序列酶反应过程中,一方面使链延伸,另一方面使dNTP上的荧光基团脱落聚合反应持续进行,测序同时持续进行PacBio SMRT测序原理PacBio SMRT的单分子测序和超长读长是如何实现的?我们重点看一下该技术的两点关键创新:分别是零模波导孔(zero-mode waveguides, ZMWs)和荧光标记在核苷酸焦磷酸链上(Phospholinked nucleotides)。
SMRT Cell含有纳米级的零模波导孔,每个ZMW都能够包含一个DNA聚合酶及一条DNA样品链进行单分子测序,并实时检测插入碱基的荧光信号。
ZMW是一个直径只有10~50 nm的孔,当激光打在ZMW底部时,只能照亮很小的区域,DNA聚合酶就被固定在这个区域。
只有在这个区域内,碱基携带的荧光基团被激活从而被检测到,大幅地降低了背景荧光干扰。
第三代测序技术原理

第三代测序技术原理
第三代测序技术是一种新型的高通量DNA测序技术,相较于第二代测序技术,其具有更高的准确性和更高的读长,可以更好地解决一些基因组学研究领域中的难题。
第三代测序技术的原理是基于单分子测序,即将单个DNA分子进行直接测序,避免了PCR扩增等步骤对样本的影响。
该技术的主要方法包括单分子实时测序、纳米孔测序和光学显微镜测序等。
其中,单分子实时测序采用的是荧光标记的核苷酸,通过读取荧光信号来确定每个核苷酸的序列。
纳米孔测序则是将DNA分子通过纳米孔,测量通过纳米孔时所产生的电流变化,从而获得每个核苷酸的序列。
光学显微镜测序则是通过观察DNA分子的荧光信号,确定每个核苷酸的序列。
与第二代测序技术相比,第三代测序技术在读长、准确性和检测能力等方面都有明显提高。
它可以实现单分子、单细胞、全基因组和全转录组等领域的研究,有望在生物医学、农业、环境等领域产生广泛的应用。
- 1 -。
第三代PacBio测序技术的测序原理和读长

第三代PacBio测序技术的测序原理和读长针对PacBio单分⼦测序——第三代测序技术的测序原理和读长DNA基因测序技术从上世纪70年代起,历经三代技术后,⽬前已发展成为⼀项相对成熟的⽣物产业。
测序技术的应⽤也扩展到了⽣物、医学、制药、健康、农林、园艺、花卉、环保、法医等许多领域,并成为⼀项与我们⾐⾷住⾏密切相关的⾼技术产业。
据最新统计,2012年全球基因测序市场的产值已超过百亿,按最近⼏年增长速度,预计2017年市场产值将加倍。
因此可以说,基因测序在我国⽣物科技领域具有⾮常重要的战略意义。
“第三代测序技术”的研发已有近⼗年时间,商业化的第三代测序仪上市也有三年,⽬前,国内对Pacbio单分⼦测序研究也有了最新进展:⼀,中科院药植所采⽤PacBio单分⼦测序揭⽰丹参叶绿体DNA修饰之间复杂的相互作⽤:编码及⾮编码RNA的表达2014年6⽉10⽇,中科院药⽤植物研究所(IMPLAD)刘昶团队在《PLOS ONE》杂志上发表了利⽤PacBio测序技术揭⽰丹参(Salvia miltiorrhiza)叶绿体DNA修饰之间复杂相互作⽤的相关⽂章,该⽂章报道了丹参叶绿体中编码及⾮编码RNA的表达情况。
这也是国内PacBio第三代测序⽤户在国际性杂志发表的第⼀篇⽂章。
丹参是最⼴泛使⽤的药⽤植物之⼀。
作为基于叶绿体基因⼯程⼿段开发使丹参活性成分过表达⽅法的第⼀步,该研究团队从基因组,转录组,和碱基修饰三⽅⾯对丹参叶绿体进⾏了分析。
先从新鲜叶⽚中提取总基因组DNA和RNA,然后进⾏链特异性RNA测序和PacBio 公司的单分⼦实时(Single-Molecule Real-Time, SMRT)测序分析。
实验先是将RNA测序得到的reads mapping到基因组,使该研究⼩组确定了80个蛋⽩质编码基因的相对表达⽔平。
此外,还明确了19个多顺反⼦转录单元和136个假定反义和基因间⾮编码RNA(ncRNA)基因。
单细胞三代全长转录组原理

单细胞三代全长转录组原理概述在生物学研究中,了解细胞的转录组信息对于理解细胞的功能和特性至关重要。
传统的RNA测序方法通常只能获得平均细胞群体的总体信息,而单细胞转录组测序技术的出现可以帮助我们对个体细胞进行高分辨率的转录组测序。
单细胞转录组测序可以帮助我们揭示细胞的功能多样性及细胞类型的分化状态。
其中,单细胞三代全长转录组技术以其高精度、高效率的特点受到广泛关注。
三代测序技术概述三代测序技术是指第三代DNA测序技术,相比传统的第一代和第二代技术,第三代测序技术具有无需扩增、读长更长、错误率更低等特点。
常见的第三代测序技术包括PacBio和Oxford Nanopore技术。
基于第三代测序技术的单细胞转录组测序能够克服第二代测序技术中基因长度限制和错误率高的问题,为细胞内基因表达的全长信息提供了解决方案。
单细胞转录组测序工作流程单细胞三代全长转录组测序的工作流程可分为以下几个步骤:细胞分离与捕捉首先,需要将单个细胞从组织样本中分离出来,并将其分别封装到单个反应小室中,以避免测序过程中不同细胞之间的干扰。
目前常用的细胞分离与捕捉方法包括离心、流式细胞术和微流控芯片等。
全长转录组测序在单细胞转录组测序中,通过选择合适的方法,将细胞内的RNA转录本转录为cDNA,然后进行PCR扩增和文库制备。
与第二代测序技术不同的是,第三代测序技术可以获得更长的读长,从而可以捕获细胞中基因的全长信息。
数据分析和解读经过测序后,得到的数据需要进行分析和解读。
通常的分析包括数据质量控制、比对到参考基因组、转录本定量、细胞类型鉴定等步骤。
数据分析的结果可以帮助我们了解每个细胞的基因表达模式,进而探究细胞类型和功能的差异性。
单细胞三代全长转录组测序的优势和挑战单细胞三代全长转录组测序相较于传统的测序技术有许多优势,例如可以有效避免PCR扩增的引入的偏差,可以捕获更完整的转录本信息,还可以对每个细胞样本进行高通量的同步测序等。
picbio三代测序原理

p i c b i o三代测序原理The manuscript can be freely edited and modified三代测序之P a c B i o S M R T技术全解析2017-05-1111:29来源:气温回升;天气渐暖;花儿开了一簇又一簇~在这美好的季节里;我们准备聊点新话题..今天小编要来和你分享:PacBioSMRT测序那些事儿~测序技术在近几年中又有里程碑的发展;PacificBiosciences公司成功推出商业化的第三代测序仪平台;让三代测序正式走入我们的视线..与前两代相比;第三代测序有什么不同呢今天小编带大家详细了解测序界新宠-PacBioSMRT测序平台..PacBioSMRT测序原理PacificBiosciences公司研发的单分子实时测序系统SingleMoleculeRealTime;SMRT应用了边合成边测序的原理;并以SMRT芯片为测序载体..基本原理如下:聚合酶捕获文库DNA序列;锚定在零模波导孔底部4种不同荧光标记的dNTP随机进入零模波导孔底部荧光dNTP被激光照射;发出荧光;检测荧光荧光dNTP与DNA模板的碱基匹配;在酶的作用下合成一个碱基统计荧光信号存在时间长短;区分匹配碱基与游离碱基;获得DNA序列酶反应过程中;一方面使链延伸;另一方面使dNTP上的荧光基团脱落聚合反应持续进行;测序同时持续进行PacBioSMRT测序原理PacBioSMRT的单分子测序和超长读长是如何实现的我们重点看一下该技术的两点关键创新:分别是零模波导孔zero-modewaveguides;ZMWs和荧光标记在核苷酸焦磷酸链上Phospholinkednucleotides..SMRTCell含有纳米级的零模波导孔;每个ZMW都能够包含一个DNA聚合酶及一条DNA样品链进行单分子测序;并实时检测插入碱基的荧光信号..ZMW是一个直径只有10~50nm的孔;当激光打在ZMW底部时;只能照亮很小的区域;DNA聚合酶就被固定在这个区域..只有在这个区域内;碱基携带的荧光基团被激活从而被检测到;大幅地降低了背景荧光干扰.. SMRTCell和ZMWs将荧光染料标记在核苷酸的磷酸链而不是碱基上;当核苷酸掺入到新生的链中;标记基团就会自动脱落;减少了DNA合成的空间位阻;维持DNA链连续合成;延长了测序读长..SMRT测序最大限度地保持了聚合酶的活性;是最接近天然状态的聚合酶反应体系..荧光标记在焦磷酸链上的核苷酸PacBioSMRT测序送样要求PacBioSMRT测序建库流程DNA打断之后;经过末端修复、接头连接、片段筛选、杂交测序引物和聚合酶绑定;即可出库准备上机测序;建库过程无PCR反应..PacBioSMRT建库流程PacBioSMRT技术特点PacBioSMRT技术有两种测序平台;RSⅡ和Sequel;应该如何选择小编已将二者的对比表准备好啦~表3RSⅡ和Sequel测序平台比较Sequel平台与RSII平台相比具有很大的优势;Sequel平台测序通量高、单Gb数据成本低、周期短..1.第三代基因测序技术又被为"SingleMoleculeRealTimeSMRT DNASequencing"单分子实时DNA测序技术;该方法基于纳米孔的单分子读取技术;不需要扩增即可快速读取序列..目前;PacificBiosciences公司已经成功推出了商业化的第三代测序仪PacB ioRS平台;使得第三代测序正式走入人们的视角..PacBioRSII是PacificBiosciences公司研发的单分子实时测序系统SingleMolecu leRealTime;SMRT;其专利的SMRTCell含有15;000个纳米级的零模波导孔zero-mod ewaveguides;ZMWs;每个ZMW都能够包含一个DNA聚合酶及一条DNA样品链进行单分子测序;并实时检测插入碱基的荧光信号..2.2技术特点ü 利用测序过程聚合酶反应的动力学变化;首次实现对碱基修饰进行直接测序..ü 超长的读长:平均测序读长能达到7;000-8;000bp;最长读长能达到3;0000bp;ü 准确率高:对基因组组装和基因组变异检测;可以最多达到99.999%的准确率;ü 敏感性强:可以检测频率在0.1%的minorvariants;ü 无PCR扩增偏好性:样本不需要进行PCR扩增;避免了覆盖度不均一和PCRartif acts;ü 最小的GC偏好性GCbias:在极端高GC和极端低GC区域;可以轻松测定;从而保证序列的均匀覆盖度..3.3数据产出试剂盒平均读长数据量/SMRTCellP4-C2Chemistry 5.5-6Kb ~200MbP5-C3Chemistry 8-10Kb ~250Mb4.4应用领域1全基因组denovo测序与二代测序最高不超过1kb的读长相比;PacBioRSII的长读长将有效解决短序列数据的拼接难题..同时;与二代测序的模板样品需要扩增相比;PacBioRSII无需扩增可直接对单个分子进行测序;有效避免了PCR扩增偏好性和GC偏好性;PacBioRSII可轻松跨越GC含量异常过高或过低及高度序列重复的区域;实现序列覆盖的完整性和均一性..2基因组草图的优化或基因组完成图绘制对前期已开展测序的动植物、微生物基因组结合三代长读长测序数据进行完善和提升..针对前期已经开展全基因组测序;获得基因组草图的动植物、微生物等;可以结合PacBioRSII平台长读长reads进行补充;从而快速获得前期没有测得的信息及提升基因组的完整度..另外还可以针对前期没有检测获得的结构变异信息structural -variationevents、串联重复序列信息tandemduplication、易位信息Inversion 等..尤其在微生物基因组完成图的绘制中;可在一天之内、成本低于$1;000即可将基因组完善获得0gapcontig..3全长转录本测序PacBioRSII的长读长可实现全长转录本测序;并使基因可变剪接形式的识别成为可能;因此可以对新基因及其isoform进行更全面的研究..同时;长读长不再需要对RN A-Seq的reads进行组装;因此可以更完整的对基因模型和转录的基因进行更全面的注释;用以改进参考基因组中的基因注释信息..4宏基因组测序长读长reads能够更为精准地鉴定水体、土壤、肠道等生境中微生物的种类的鉴定;能够更加快捷地获得更多微生物种的全基因组序列..516SrDNA全长测序PacBioRSII的平均读长为8-10Kb;而16SrDNA长度大约为1540bp;因此结合该平台测序可以成功测序获得16S的全长序列..6细胞器基因组测序叶绿体基因组和线粒体基因组序列都包含重复序列、反向重复序列等复杂结构;Pac BioRSII的长读长可直接跨越这些区域获得这些细胞器的全基因组序列信息等;基因组组装不依赖于是否有近缘物种的线粒体和叶绿体基因组信息等、重测序能够检测到全面的SNPs及indel信息..7全基因组重测序&稀有变异鉴定PacBioRSII长读长测序reads无GC偏号;能够全面获得基因组的遗传变异;包括SN Ps的鉴定到CNV、SV结构变异等;可运用至人类癌症基因组重测序等..PacBioRSII 平台测序周期在10hours小时即可完成测序;可应用至需要快速反馈的临床检测中;如细菌感染疾病中细菌的鉴定、病毒的鉴定等;取样开展重测序和目前已有的细菌、病毒基因组数据库进行比对鉴定即可..8表观遗传学PacBioRSII利用测序过程聚合酶反应的动力学变化;首次实现对碱基修饰进行直接测序..当碱基有额外修饰时;DNA聚合酶的合成速度会减慢;对应的信号会被检测出来..每种碱基修饰事件都会使聚合酶的“停顿模式”PacBioRSII产生微小差异;最终反映到荧光脉冲信号的间隔上..除了甲基化修饰;还可以检测5-hC、5-hmU、5-h U、1-mA、6-mA、8-oxoA、BPDE、6-mT、6-mG等碱基修饰;甚至可以鉴别传统亚硫酸氢盐测序法无法区分的甲基化修饰和羟甲基化修饰..PacBio平台可以在测序的同时即可检测表观遗传学修饰信息;只需对测序数据选择合适的软件即可分析碱基修饰信息..5.5。
三代基因组测序技术原理简介
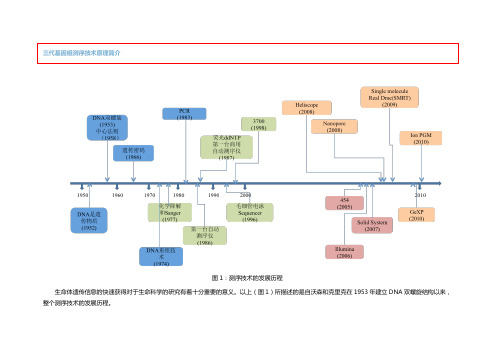
图1:测序技术的发展历程生命体遗传信息的快速获得对于生命科学的研究有着十分重要的意义。
以上(图1)所描述的是自沃森和克里克在1953年建立DNA双螺旋结构以来,整个测序技术的发展历程。
第一代测序技术第一代DNA测序技术用的是1975年由桑格(Sanger)和考尔森(Coulson)开创的链终止法或者是1976-1977年由马克西姆(Maxam)和吉尔伯特(Gilbert)发明的化学法(链降解). 并在1977年,桑格测定了第一个基因组序列,是噬菌体X174的,全长5375个碱基1。
自此,人类获得了窥探生命遗传差异本质的能力,并以此为开端步入基因组学时代。
研究人员在Sanger法的多年实践之中不断对其进行改进。
在2001年,完成的首个人类基因组图谱就是以改进了的Sanger法为其测序基础,Sanger法核心原理是:由于ddNTP的2’和3’都不含羟基,其在DNA的合成过程中不能形成磷酸二酯键,因此可以用来中断DNA合成反应,在4个DNA合成反应体系中分别加入一定比例带有放射性同位素标记的ddNTP(分为:ddATP,ddCTP,ddGTP和ddTTP),通过凝胶电泳和放射自显影后可以根据电泳带的位置确定待测分子的DNA序列(图2)。
这个网址为sanger测序法制作了一个小短片,形象而生动。
值得注意的是,就在测序技术起步发展的这一时期中,除了Sanger法之外还出现了一些其他的测序技术,如焦磷酸测序法、链接酶法等。
其中,焦磷酸测序法是后来Roche公司454技术所使用的测序方法2–4,而连接酶测序法是后来ABI公司SOLID技术使用的测序方法2,4,但他们的共同核心手段都是利用了Sanger1中的可中断DNA合成反应的dNTP。
图2:Sanger法测序原理第二代测序技术总的说来,第一代测序技术的主要特点是测序读长可达1000bp,准确性高达99.999%,但其测序成本高,通量低等方面的缺点,严重影响了其真正大规模的应用。
第三代基因测序原理及应用

发展前景展望
提高准确性
随着技术的不断发展和优化,未来第三代测序技术的准确性将得到 进一步提高,有望接近甚至超过传统测序技术。
降低成本
随着技术的普及和规模化应用,第三代测序技术的成本有望进一步 降低,使得更多人能够享受到高精度基因组测序服务。
拓展应用领域
随着第三代测序技术的不断发展和完善,其应用领域也将不断拓展 ,包括复杂疾病研究、精准医疗、生物多样性保护等。
缺点
测序准确度相对较低,且对DNA样 品的质量要求较高。
链接测序技术
原理
通过连接反应将DNA片段连接成 更长的序列,然后对连接产物进 行测序。连接反应具有高度的特 异性,可以准确识别碱基序列。
优点
测序准确度高、读长较长、适用 于复杂样本的测序。
缺点
测序速度相对较慢,且连接反应 可能受到多种因素的影响,如温 度、pH值等。
挑战与问题
高错误率
第三代测序技术的错误率相 对较高,尤其是在连续测序 过程中,错误率可能会逐渐 累积,影响测序结果的准确
性。
数据处理难度
由于第三代测序技术产生的 数据量巨大,对数据处理和 分析的要求也相应提高,需 要更强大的计算能力和更高
效的算法支持。
成本问题
目前第三代测序技术的成本 仍然较高,限制了其在大规 模基因组测序等领域的应用 。
多维度数据挖掘
利用多组学数据,挖掘基因、环境、生活方式等多因素对人体健康和疾病的影响,为个性化医疗和精准预防提供 科学依据。
智能化和自动化发展方向
自动化样本处理
通和准确性。
智能数据分析
利用人工智能和机器学习技术,对测序数据进行自动分析和 解读,提取有价值的信息和模式,为科研和临床应用提供智 能决策支持。
第三代测序技术原理及应用

Pacific Bioscience SMRT 技术
• 优势:长读长,耗时短。可以进行DNA甲基化,高GC含量区域、 RNA等测序。
• 缺陷:会出现插入和缺失错误。 缺失错误源于有时候碱基掺入速度过快, 超过了相机的拍摄帧数; 插
入错误源于有时候酶随机的选择一些碱基,但并未真的将这些碱基掺入 合成链中。但这些错误是随机的,并不会随着读长的增加而提高, 随着 测序覆盖深度的增加会逐渐被消除。
Oxford Nanopore Technologies 纳米孔单分子测序技术
• 优势:仪器构造简单使用成本低廉,因为它不需要对核苷酸进行标记, 也不需要复杂的光学探测系统 。能直接对 RNA 分子进行测序。同时 由于它是直接检测每一个碱基的特征性电流, 因而能对修饰过的碱基 进行测序, 这一点对于表观遗传学研究具有极高的价值。
Pacific Bioscience SMRT 技术
第一代、二代、三代测序仪比较
Rhoads A, Au KF. PacBio Sequencing and Its Applications. Genomics, Proteomics & Bioinformatics. 2015;13(5):278-289. doi:10.1016/j.gpb.2015.08.002.
• 缺点:测序的平均读长相对较短, 只有 35 bp, 准确率较低, 约为 ≤97% 。
Oxford Nanopore Technologies 纳米孔单分子测序技术
α-溶血素纳米孔 外:核酸外切酶 内:环糊精传感器
核酸外切酶 消化单链DNA
单碱基与环糊 精作用影响电 流
依据电信号大小和 停留时间识别碱基
测序准确度较高,测序碱基错误率 为1%
第三代测序技术原理

第三代测序技术原理
第三代测序技术是指最新一代的高通量测序技术,它与第一代和第二代测序技
术相比,具有更高的测序速度、更低的成本和更高的准确性。
第三代测序技术的原理主要包括单分子测序、纳米孔测序和合成孔测序等多种技术。
本文将重点介绍第三代测序技术的原理及其应用。
首先,单分子测序是第三代测序技术的核心原理之一。
它通过直接测序单个DNA分子,避免了PCR扩增和文库构建等步骤,大大简化了测序流程。
单分子测
序技术主要包括荧光基团标记、逐个核苷酸加入和荧光检测等步骤。
这种原理使得第三代测序技术在测序速度和准确性上有了质的飞跃。
其次,纳米孔测序是第三代测序技术的另一重要原理。
它利用纳米孔将DNA
分子拉伸成单链,然后通过电压驱动DNA分子逐个通过纳米孔,通过测量电流信
号来识别不同的核苷酸。
这种原理使得第三代测序技术可以实现长读长,大大提高了测序的准确性和覆盖度。
最后,合成孔测序是第三代测序技术的又一重要原理。
它利用合成纳米结构来
实现单分子测序,通过控制合成孔的尺寸和形状,可以实现对不同长度的DNA分
子进行测序。
这种原理使得第三代测序技术可以实现更高的测序速度和更低的成本,为基因组学研究提供了强大的工具。
总的来说,第三代测序技术的原理主要包括单分子测序、纳米孔测序和合成孔
测序等多种技术,它们共同推动了测序技术的发展,为基因组学研究提供了强大的工具。
随着第三代测序技术的不断进步,相信它将在生命科学研究和临床诊断中发挥越来越重要的作用。
一代、二代、三代测序技术原理与比较

一代、二代、三代测序技术原理与比较【写在前面的话】:首先,这一篇博文中的内容并非原创,而是对多篇文献中内容的直接摘录,有些图片和资料还来自身边的同事(在此深表谢意!),再夹杂自己的零星想法,写在这里分享与大家,同时也是为了方便自己日后若有需要能够方便获得,文章比较长。
摘要:从1977年第一代DNA测序技术(Sanger法)1,发展至今三十多年时间,测序技术已取得了相当大的发展,从第一代到第三代乃至第四代,测序读长从长到短,再从短到长。
虽然就当前形势看来第二代短读长测序技术在全球测序市场上仍然占有着绝对的优势位置,但第三和第四代测序技术也已在这一两年的时间中快速发展着。
测序技术的每一次变革,也都对基因组研究,疾病医疗研究,药物研发,育种等领域产生巨大的推动作用。
在这里我主要对当前的测序技术以及它们的测序原理做一个简单的小结。
图1:测序技术的发展历程生命体遗传信息的快速获得对于生命科学的研究有着十分重要的意义。
以上(图1)所描述的是自沃森和克里克在1953年建立DNA 双螺旋结构以来,整个测序技术的发展历程。
第一代测序技术第一代DNA测序技术用的是1975年由桑格(Sanger)和考尔森(Coulson)开创的链终止法或者是1976-1977年由马克西姆(Maxam)和吉尔伯特(Gilbert)发明的化学法(链降解). 并在1977年,桑格测定了第一个基因组序列,是噬菌体X174的,全长5375个碱基1。
自此,人类获得了窥探生命遗传差异本质的能力,并以此为开端步入基因组学时代。
研究人员在Sanger法的多年实践之中不断对其进行改进。
在2001年,完成的首个人类基因组图谱就是以改进了的Sanger法为其测序基础,Sanger法核心原理是:由于ddNTP 的2’和3’都不含羟基,其在DNA的合成过程中不能形成磷酸二酯键,因此可以用来中断DNA合成反应,在4个DNA合成反应体系中分别加入一定比例带有放射性同位素标记的ddNTP(分为:ddATP,ddCTP,ddGTP和ddTTP),通过凝胶电泳和放射自显影后可以根据电泳带的位置确定待测分子的DNA序列(图2)。
第三代测序技术原理及应用

SMRT cell
Sequel (2015年)
1,000,000 ZMWs /SMRT cell SMRT cell 的通量提高 7 倍 1/3体积
Oxford Nanopore Technologies 纳米孔单分子测序技术
• 优势:仪器构造简单使用成本低廉,因为它不需要对核苷酸进行标记, 也不需要复杂的光学探测系统 。能直接对 RNA 分子进行测序。同时 由于它是直接检测每一个碱基的特征性电流, 因而能对修饰过的碱基 进行测序, 这一点对于表观遗传学研究具有极高的价值。
第三代测序技术的基本原理及应用
baby诺安
目录
1. 简介 2. 基本原理 3. 应用
简介
第三代测序技术是指单分子测序技术。不同于二代测序依赖片段化 DNA的克隆性扩增,三代测序技术不需要经过PCR扩增,直接对模板中 的每条DNA分子单独测序,避免了潜在的PCR扩增错误和偏好性。同时 超长读长使得一条read可以横跨基因组上的复杂区段或者重复序列,为 基因组组装及准确挖掘与疾病、进化相关的重复原件、拷贝数变异、结 构性变异提供了技术支持。
成像定位 模板位置
洗涤
合成 检测
全内反射显微镜(TIRM)单色成像
Helico BioScience SMS技术
• 测序仪:HeliScope(2008年上市,$1,350,000)
• 优势:样本通量非常高,2 个流动槽可同时运行,每个流动槽有 25 个独立通道,每个通道又可以运行最多 96 个标记分子条形码的样本, 这样每次运行的样本数可高达 4 800 个。把 DNA 聚合酶用逆转录酶 代替还可以进行 RNA 直接测序。
picbio三代测序原理

p i c b i o三代测序原理集团标准化工作小组 #Q8QGGQT-GX8G08Q8-GNQGJ8-MHHGN#三代测序之PacBio SMRT技术全解析2017-05-11 11:29 来源:气温回升,天气渐暖,花儿开了一簇又一簇~在这美好的季节里,我们准备聊点新话题。
今天小编要来和你分享:PacBio SMRT测序那些事儿~测序技术在近几年中又有里程碑的发展,Pacific Biosciences公司成功推出商业化的第三代测序仪平台,让三代测序正式走入我们的视线。
与前两代相比,第三代测序有什么不同呢今天小编带大家详细了解测序界新宠-PacBio SMRT 测序平台。
PacBio SMRT测序原理Pacific Biosciences公司研发的单分子实时测序系统(Single Molecule Real Time,SMRT)应用了边合成边测序的原理,并以SMRT芯片为测序载体。
基本原理如下:聚合酶捕获文库DNA序列,锚定在零模波导孔底部4种不同荧光标记的dNTP随机进入零模波导孔底部荧光dNTP被激光照射,发出荧光,检测荧光荧光dNTP与DNA模板的碱基匹配,在酶的作用下合成一个碱基统计荧光信号存在时间长短,区分匹配碱基与游离碱基,获得DNA序列酶反应过程中,一方面使链延伸,另一方面使dNTP上的荧光基团脱落聚合反应持续进行,测序同时持续进行PacBio SMRT测序原理PacBio SMRT的单分子测序和超长读长是如何实现的我们重点看一下该技术的两点关键创新:分别是零模波导孔(zero-mode waveguides, ZMWs)和荧光标记在核苷酸焦磷酸链上(Phospholinked nucleotides)。
SMRT Cell含有纳米级的零模波导孔,每个ZMW都能够包含一个DNA聚合酶及一条DNA样品链进行单分子测序,并实时检测插入碱基的荧光信号。
ZMW是一个直径只有10~50 nm的孔,当激光打在ZMW底部时,只能照亮很小的区域,DNA聚合酶就被固定在这个区域。
一代-二代-三代测序原理
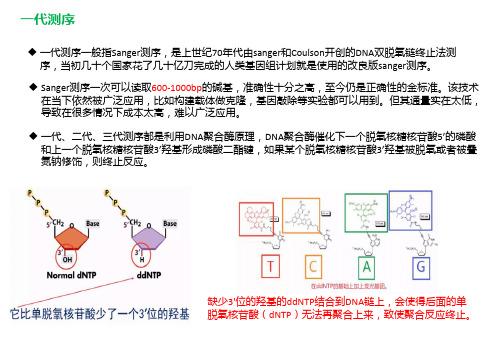
缺少3'位的羟基的ddNTP结合到DNA链上,会使得后面的单 脱氧核苷酸(dNTP)无法再聚合上来,致使聚合反应终止。
二代测序
二代测序技术,又称为Next Generation Sequencing(NGS)技术,高通量测 序技术,是为了改进一代测序通量过低的问题而出现的。刚面世时主要包括 Roche公司的454技术、ABI公司的Solid技术和Illumina公司的Solexa技术。这 三种技术都极大的提高了测序的通量,大大降低了测序成本和周期。
序引物;
I7:
第一条测序完,用I7引物测序第一条链上的index 1
序列;
P5:
再用P5为引物测序第一条链上的index 2;
Rd1 SP:第二条链测序引物;
➢ 因不同的样品可以同时在一个流动槽里进行测序反应,index 1和index 2 序列可以通 过加或者不加来区分不同的样本来源;
其中Illumina公司凭借超低的测序成本和可以接受的读长,成为了目前最主流的 二代测序公司,其测序成本近五年来从几千元1G(1G即10亿碱基)降到了到今 天的40多块钱。
• 化学试剂三羧基乙基膦(TCEP)淬灭荧光信号;有时荧光基团切割不完全给簇形成荧光背景,导致 测序够长。
• 叠氮保护基团遇到巯基试剂(如二巯基丙醇)会发生断裂,并在原来的位置形成羟基,供下一个碱基合 上。
P5与流动槽共价 连接的单链被切 断后洗掉
化学方法切断
一轮反应只能 加上一个碱基, 一个簇只能测 150-300个反应。
三代测序的原理

三代测序的原理三代测序是一种新型的高通量测序技术,它可以在较短时间内获得大量的 DNA 测序数据,为基因组研究、单细胞基因组学等领域提供了更为高效的方法和手段。
三代测序技术相比于传统的二代测序技术,在测序长度、测序速度等方面都具有明显的优势,因此备受关注。
那么,什么是三代测序?它的原理是什么?三代测序技术包括单分子测序技术、纳米孔测序技术等,这些技术都是基于不同的原理而发展的。
下面我们就一些常见的三代测序技术原理进行详细介绍。
一、单分子测序技术单分子测序技术,顾名思义,就是在一个分子的层面上进行 DNA 测序,这种技术可以突破传统二代测序技术中 DNA 放大和分离纯化等阶段的限制,避免了因 PCR 反应带来的测序误差,并可以直接测序双链 DNA 分子。
单分子测序技术主要有实时荧光测序技术和化学测序技术两种。
1. 实时荧光测序技术实时荧光测序技术是通过 DNA 上特异序列受体,与荧光信号的转导机制相结合来实现的测序技术。
这种技术可以检测位于 DNA 测序反应过程中的碱基,并通过光学探测来连续监测测序结果。
实时荧光测序技术的基本原理是在每一轮的 DNA 合成中加入荧光标记基团,然后通过不断检测从而读出 DNA 序列信息。
2. 化学测序技术化学测序技术是将 DNA 去除荧光标记基团后,采用荧光探针进行信号监测,从而得出碱基序列信息的一种测序技术。
先在反应过程中加入一个被称为“保护基覆盖”的基团,然后进行荧光信号检测,得出结果后再将前面添加的保护基覆盖去掉,继续进行下一轮检测,直到完成整个 DNA 序列的测定。
单分子测序技术擅长于检测低复杂度序列(如 GC/AT 重复序列)、基因组结构变异等重要问题,可以应用在很多领域,如生物医学、生物环境以及生物信息学等领域。
二、纳米孔测序技术纳米孔测序技术是利用纳米孔对单个 DNA 分子进行测序的技术,它是一种界面控制技术,主要包括固体纳米孔、液态纳米孔和气态纳米孔等。
三代基因组测序技术原理简介

三代基因组测序技术原理简介摘要:从1977年第一代DNA测序技术(Sanger法)1,发展至今三十多年时间,测序技术已取得了相当大的发展,从第一代到第三代乃至第四代,测序读长从长到短,再从短到长。
虽然就当前形势看来第二代短读长测序技术在全球测序市场上仍然占有着绝对的优势位置,但第三和第四代测序技术也已在这一两年的时间中快速发展着。
测序技术的每一次变革,也都对基因组研究,疾病医疗研究,药物研发,育种等领域产生巨大的推动作用。
在这里我主要对当前的测序技术以及它们的测序原理做一个简单的小结。
图1:测序技术的发展历程生命体遗传信息的快速获得对于生命科学的研究有着十分重要的意义。
以上(图1)所描述的是自沃森和克里克在1953年建立DNA双螺旋结构以来,整个测序技术的发展历程。
第一代测序技术第一代DNA测序技术用的是1975年由桑格(Sanger)和考尔森(Coulson)开创的链终止法或者是1976-1977年由马克西姆(Maxam)和吉尔伯特(Gilbert)发明的化学法(链降解). 并在1977年,桑格测定了第一个基因组序列,是噬菌体X174的,全长5375个碱基1。
自此,人类获得了窥探生命遗传差异本质的能力,并以此为开端步入基因组学时代。
研究人员在Sanger法的多年实践之中不断对其进行改进。
在2001年,完成的首个人类基因组图谱就是以改进了的Sanger法为其测序基础,Sanger法核心原理是:由于ddNTP的2’和3’都不含羟基,其在DNA的合成过程中不能形成磷酸二酯键,因此可以用来中断DNA 合成反应,在4个DNA合成反应体系中分别加入一定比例带有放射性同位素标记的ddNTP (分为:ddATP,ddCTP,ddGTP和ddTTP),通过凝胶电泳和放射自显影后可以根据电泳带的位置确定待测分子的DNA序列(图2)。
这个网址为sanger测序法制作了一个小短片,形象而生动。
值得注意的是,就在测序技术起步发展的这一时期中,除了Sanger法之外还出现了一些其他的测序技术,如焦磷酸测序法、链接酶法等。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
三代测序之PacBio SMRT技术全解析2017-05-11 11:29 来源:基因谷技术
气温回升,天气渐暖,
花儿开了一簇又一簇~
在这美好的季节里,
我们准备聊点新话题。
今天小编要来和你分享:
PacBio SMRT测序那些事儿~
测序技术在近几年中又有里程碑的发展,Pacific Biosciences公司成功推出商业化的第三代测序仪平台,让三代测序正式走入我们的视线。
与前两代相比,第三代测序有什么不同呢?今天小编带大家详细了解测序界新宠-PacBio SMRT测序平台。
PacBio SMRT测序原理
Pacific Biosciences公司研发的单分子实时测序系统(Single Molecule Real Time,SMRT)应用了边合成边测序的原理,并以SMRT芯片为测序载体。
基本原理如下:
聚合酶捕获文库DNA序列,锚定在零模波导孔底部
4种不同荧光标记的dNTP随机进入零模波导孔底部
荧光dNTP被激光照射,发出荧光,检测荧光
荧光dNTP与DNA模板的碱基匹配,在酶的作用下合成一个碱基
统计荧光信号存在时间长短,区分匹配碱基与游离碱基,获得DNA序列
酶反应过程中,一方面使链延伸,另一方面使dNTP上的荧光基团脱落
聚合反应持续进行,测序同时持续进行
PacBio SMRT测序原理
PacBio SMRT的单分子测序和超长读长是如何实现的?我们重点看一下该技术的两点关键创新:分别是零模波导孔(zero-mode waveguides, ZMWs)和荧光标记在核苷酸焦磷酸链上(Phospholinked nucleotides)。
SMRT Cell含有纳米级的零模波导孔,每个ZMW都能够包含一个DNA聚合酶及一条DNA样品链进行单分子测序,并实时检测插入碱基的荧光信号。
ZMW是一个直径只有10~50 nm的孔,当激光打在ZMW底部时,只能照亮很小的区域,DNA聚合酶就被固定在这个区域。
只有在这个区域内,碱基携带的荧光基团被激活从而被检测到,大幅地降低了背景荧光干扰。
SMRT Cell和ZMWs
将荧光染料标记在核苷酸的磷酸链而不是碱基上,当核苷酸掺入到新生的链中,标记基团就会自动脱落,减少了DNA合成的空间位阻,维持DNA链连续合成,延长了测序读长。
SMRT测序最大限度地保持了聚合酶的活性,是最接近天然状态的聚合酶反应体系。
荧光标记在焦磷酸链上的核苷酸
PacBio SMRT测序送样要求
PacBio SMRT测序建库流程
DNA打断之后,经过末端修复、接头连接、片段筛选、杂交测序引物和聚合酶绑定,即可出库准备上机测序,建库过程无PCR反应。
PacBio SMRT建库流程
PacBio SMRT技术特点
PacBio SMRT技术有两种测序平台,RSⅡ和Sequel,应该如何选择?小编已将二者的对比表准备好啦~ 表3 RSⅡ和Sequel测序平台比较
Sequel平台与RSII平台相比具有很大的优势,Sequel平台测序通量高、单Gb数据成本低、周期短。
1.第三代基因测序技术又被为"Single Molecule Real Time (SMRT™) DNA
Sequencing"(单分子实时DNA测序技术),该方法基于纳米孔的单分子读取技术,不需要扩增即可快速读取序列。
目前,Pacific Biosciences 公司已经成功推出了商业化的第三代测序仪PacBio RS平台,使得第三代测序正式走入人们的视角。
PacBio RS II是Pacific Biosciences公司研发的单分子实时测序系统(Single Molecule Real Time, SMRT),其专利的SMRT Cell含有15,0 00个纳米级的零模波导孔(zero-mode waveguides, ZMWs),每个ZMW 都能够包含一个DNA聚合酶及一条DNA样品链进行单分子测序,并实时检测插入碱基的荧光信号。
2. 2
技术特点
ü利用测序过程聚合酶反应的动力学变化,首次实现对碱基修饰进行直接测序。
ü超长的读长:平均测序读长能达到7,000-8,000bp,最长读长能达到3,0000bp;
ü准确率高:对基因组组装和基因组变异检测,可以最多达到99.999%的准确率;
ü敏感性强:可以检测频率在0.1%的minor variants;
ü无PCR扩增偏好性:样本不需要进行PCR扩增,避免了覆盖度不均一和PCR artifacts;
ü最小的GC偏好性(GC bias):在极端高GC和极端低GC区域,可以轻松测定,从而保证序列的均匀覆盖度。
3. 3
数据产出
试剂盒平均读长数据量/SM RT Cell
P4-C2 Chemistry 5.5-6Kb ~200Mb
P5-C3 Chemistry 8-10Kb ~250Mb
4. 4
应用领域
(1)全基因组de novo测序
与二代测序最高不超过1kb的读长相比,PacBio RS II的长读长将有效解决短序列数据的拼接难题。
同时,与二代测序的模板样品需要扩增相比,PacBio RS II无需扩增可直接对单个分子进行测序,有效避免了PCR扩
增偏好性和GC偏好性,PacBio RS II可轻松跨越GC含量异常(过高或过低)及高度序列重复的区域,实现序列覆盖的完整性和均一性。
(2)基因组草图的优化或基因组完成图绘制
对前期已开展测序的动植物、微生物基因组结合三代长读长测序数据进行完善和提升。
针对前期已经开展全基因组测序,获得基因组草图的动植物、微生物等,可以结合PacBio RS II平台长读长reads进行补充,从而快速获得前期没有测得的信息及提升基因组的完整度。
另外还可以针对前期没有检测获得的结构变异信息(structural-variation events)、串联重复序列信息(tandem duplication)、易位信息(Inversion)等。
尤其在微生物基因组完成图的绘制中,可在一天之内、成本低于$1,000即可将基因组完善获得0 gap contig。
(3)全长转录本测序
PacBio RS II的长读长可实现全长转录本测序,并使基因可变剪接形式的识别成为可能,因此可以对新基因及其isoform进行更全面的研究。
同时,长读长不再需要对RNA-Seq的reads进行组装,因此可以更完整的对基因模型和转录的基因进行更全面的注释,用以改进参考基因组中的基因注释信息。
(4)宏基因组测序
长读长reads能够更为精准地鉴定水体、土壤、肠道等生境中微生物的种类的鉴定,能够更加快捷地获得更多微生物种的全基因组序列。
(5)16S rDNA全长测序
PacBio RS II的平均读长为8-10Kb,而16S rDNA长度大约为1540bp,因此结合该平台测序可以成功测序获得16S的全长序列。
(6)细胞器基因组测序
叶绿体基因组和线粒体基因组序列都包含重复序列、反向重复序列等复杂结构,PacBio RS II的长读长可直接跨越这些区域获得这些细胞器的全
基因组序列信息等,基因组组装不依赖于是否有近缘物种的线粒体和叶绿体基因组信息等、重测序能够检测到全面的SNPs及indel信息。
(7)全基因组重测序&稀有变异鉴定
PacBio RS II长读长测序reads无GC偏号,能够全面获得基因组的遗传变异,包括SNPs的鉴定到CNV、SV结构变异等,可运用至人类癌症基因组重测序等。
PacBio RS II平台测序周期在10 hours小时即可完成测序,可应用至需要快速反馈的临床检测中,如细菌感染疾病中细菌的鉴定、病毒的鉴定等,取样开展重测序和目前已有的细菌、病毒基因组数据库进行比对鉴定即可。
(8)表观遗传学
PacBio RS II利用测序过程聚合酶反应的动力学变化,首次实现对碱基修饰进行直接测序。
当碱基有额外修饰时,DNA聚合酶的合成速度会减慢,对应的信号会被检测出来。
每种碱基修饰事件都会使聚合酶的“停顿模式”PacBio RS II产生微小差异,最终反映到荧光脉冲信号的间隔上。
除了甲基化修饰,还可以检测5-hC、5-hmU、5-hU、1-mA、6-mA、8-oxoA、BP DE、6-mT、6-mG等碱基修饰,甚至可以鉴别传统亚硫酸氢盐测序法无法区分的甲基化修饰和羟甲基化修饰。
PacBio平台可以在测序的同时即可检测表观遗传学修饰信息,只需对测序数据选择合适的软件即可分析碱基修饰信息。
5. 5。
