寻找基因外显子、内含子的几种方法
外显子查找方法范文

外显子查找方法范文外显子(exon)是基因组中编码蛋白质的片段,它们是基因转录后的成果。
外显子查找是基因组学领域的一个重要任务,它可以帮助我们了解基因的功能和结构,以及鉴定和研究基因突变和遗传疾病。
在过去的几十年里,外显子查找方法经历了多次技术革新,从最早的Sanger测序到现在的高通量测序技术。
下面将介绍几种常用的外显子查找方法。
1. Sanger测序:Sanger测序是一种经典的测序技术,通过反复合成DNA链并在每个碱基上加入一种特殊的标记物来测定DNA序列。
借助Sanger测序,我们可以逐个测定DNA的碱基顺序,并通过比对已知外显子序列来确定外显子的位置。
2. 基于EST序列的外显子查找:EST(Expressed Sequence Tag)是从cDNA文库中得到的短序列片段,它们通常来自于外显子区域。
利用EST序列可以通过比对已知外显子序列来推断新的外显子。
3. 基于数据库的外显子查找:利用已知的外显子序列建立外显子数据库,如Ensembl、NCBI等,可以快速比对新的DNA序列来鉴定外显子。
4. 基于高通量测序的外显子查找:高通量测序技术的发展使得我们可以快速测定大量的DNA序列,从而推断编码蛋白质的外显子序列。
常用的高通量测序技术包括二代测序技术(如 Illumina、Iontorrent)和三代测序技术(如 PacBio、Nanopore),它们通过将DNA序列拆解成短片段并进行平行测序来提高测序速度。
5. 基于RNA-Seq的外显子查找:RNA-Seq是一种利用高通量测序技术直接测定RNA序列的方法。
由于RNA是从基因组DNA转录而来的,因此RNA-Seq可以直接测定外显子序列。
此外,由于RNA-Seq还可以检测到转录后修饰和剪接等信息,因此它成为目前外显子查找的主要方法。
总的来说,外显子查找是基因组学研究中的一项重要任务。
不同的外显子查找方法有不同的优缺点,在实际应用中需要根据研究的目的、样本的可得性和测序平台的要求来选择合适的方法。
应用UCSC_Ensembl查找基因启动子(promoter)、内含子、外显子序列-表观遗传学论坛-生物秀论坛
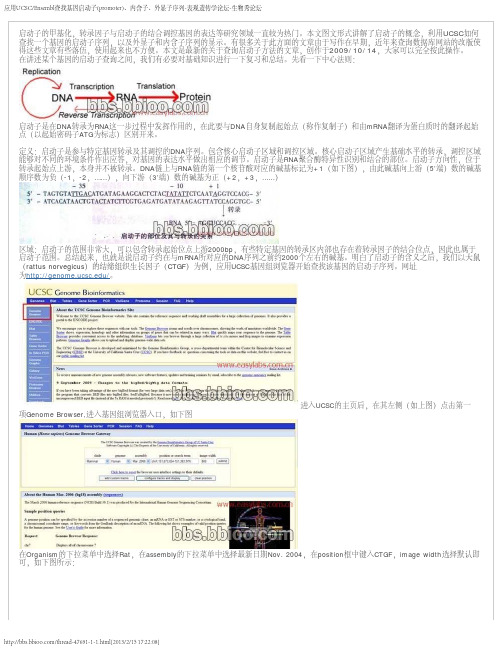
在讲述某个基因的启动子查询之间,我们有必要对基础知识进行一下复习和总结。
先看一下中心法则:启动子是在DNA转录为RNA这一步过程中发挥作用的,在此要与顺序数为负(-1,-2,……),向下游(3’端)数的碱基为正(+2,+3,……)区域:启动子的范围非常大,可以包含转录起始位点上游2000bp,有些特定基因的转录区内部也存在着转录因子的结合位点,因此也属于为/。
进入项Genome Browser,进入基因组浏览器入口,如下图在Organism的下拉菜单中选择Rat,在assembly的下拉菜单中选择最新日期Nov. 2004可,如下图所示:然后点击Submit,返回的页面如下:结果显示该基因的已知序列和相关mRNA序列,点击Known Gene中的第一个序列,出现包含这序列的图解概要。
为了获得这个区域更清晰的图像,可以点击紧靠zoom out的1.5X按钮,如下图:对于Known Genes(已知基因)和预测的基因路径来说,一般的惯例是以一个高的垂直线或块状表示每个编码外显子,以短的垂直线或块状表示5′端和3′端非翻译区。
起连接作用的内含子以非常细的线条表示。
翻译的方向由沿着细线的箭头指示。
本例的搜寻目的来说,默认设置不是理想的设置。
按照视图利用页面底部的Track Controls按钮,将一些路径设置为hide模式(即不显示),其他设置为dense模式(所有资料密集在一条直线上);另一些路径设置为full模式(每个特征有一个分开的线条,最多达300)。
在考虑这些路径内究竟存在那些资料之前,对这些路径的内容和表现做一个简要的讨论是必要的,许多这些讨论是由外界提供给UCSC的。
Ensembl Gene Predictions路径由Ensembl提供。
Ensembl基因通过许多方法来预测,包括与已知mRNA和蛋白质进行同源性比较。
若查询启动子区域,我们需要将Ensembl Genes选择为dense 或full模式,点击Refresh,即刷新,出现下图:图中多出了Ensembl Genes的预测路径,我们在红框中圈出。
使用UCSC查找基因启动子区域方法

应用UCSC查找基因启动子(promoter)、内含子、外显子序列(2015)本文原文来源于网络,因网站相关网页有所变化,特改写原文,望对同同仁有所帮助。
启动子的甲基化,转录因子与启动子的结合调控基因的表达等研究领域一直较为热门。
本文图文形式讲解了启动子的概念,利用UCSC如何查找一个基因的启动子序列,以及外显子和内含子序列的显示。
在讲述某个基因的启动子查询之间,我们有必要对基础知识进行一下复习和总结。
先看一下中心法则:启动子是在DNA转录为RNA这一步过程中发挥作用的,在此要与DNA自身复制起始点(称作复制子)和由mRNA翻译为蛋白质时的翻译起始点(以起始密码子ATG为标志)区别开来。
定义:启动子是参与特定基因转录及其调控的DNA序列。
包含核心启动子区域和调控区域。
核心启动子区域产生基础水平的转录,调控区域能够对不同的环境条件作出应答,对基因的表达水平做出相应的调节。
启动子是RNA聚合酶特异性识别和结合的部位。
启动子方向性,位于转录起始点上游,本身并不被转录。
DNA链上与RNA链的第一个核苷酸对应的碱基标记为+1(如下图),由此碱基向上游(5’端)数的碱基顺序数为负(-1,-2,……),向下游(3’端)数的碱基为正(+2,+3,……)区域:启动子的范围非常大,可以包含转录起始位点上游2000bp,有些特定基因的转录区内部也存在着转录因子的结合位点,因此也属于启动子范围。
总结起来,也就是说启动子约在与mRNA所对应的DNA序列之前约2000个左右的碱基。
明白了启动子的含义之后,我们以人(Human)的NDRG4(N-MycDownstream- Regulated Gene 4)为例,应用UCSC基因组浏览器开始查找该基因的启动子序列。
网址/index.html。
进入UCSC的主页后,在其左侧(如上图)点击第一项GenomeBrowser,进入基因组浏览器入口,如下图按照上图中的下拉选项,选择Mammal下的Human,assembly选择最新,search term 处填写需要查找的基因名称。
常用生物数据库 外显子、内含子、mRNA、CDS

常用生物数据库核酸序列数据库主要有GenBank, EMBL, DDBJ等.蛋白质序列数据库有SWISS-PROT, PIR, OWL, NRL3D, TrEMBL等,蛋白质片段数据库有PROSITE, BLOCKS, PRINTS等,三维结构数据库有PDB, NDB, BioMagResBank, CCSD等,蛋白质结构有关的数据库还有SCOP, CATH, FSSP, 3D-ALI, DSSP等,与基因组有关的数据库还有ESTdb, OMIM, GDB, GSDB等,文献数据库有Medline, Uncover等。
外显子、内含子、mRNA、CDS1.DNA复制:以DNA为模板,在DNA聚合酶的催化作用下,将四种游离的dNTP按照碱基互补配对原则合成新链DNA转录:以DNA为模版,在DNA指导的RNA聚合酶的作用下,将四种游离的NTP按照碱基互补配对的原则合成RNA翻译:以mRNA为模板,在核糖体内合成蛋白质的过程特点:模板特点原料引物DNA复制双链DNA 合成的新链与模板链一模四种dNTP 半保留复制需要一样四种NTP 半不连续转录不需要转录双链DNA 合成的新链除了把DNA上的T改为U外,其他一样翻译mRNA 3个碱基决定一个氨基酸20种游离的氨基酸2.mRNA(messenger RNA,信使RNA)信使RNA是由DNA经hnRNA剪接而成,携带遗传信息的能指导蛋白合成的一类单链核糖核酸。
3. 基因DNA分为编码区和非编码区,编码区包含外显子和内含子,一般非编码区具有基因表达的调控功能,如启动子在非编码区。
编码区则转录为mRNA并最终翻译成蛋白质。
外显子和内含子都被转录到mRNA前体hnRNA中,当hnRNA进行剪接变为成熟的mRNA 时,内含子被切除,而外显子保留。
实际上真正编码蛋白质的是外显子,而内含子则无编码功能,内含子存在于DNA中,在转录的过程中,DNA上的内含子也会被转录到前体RNA中,但前体RNA上的内含子会在RNA离开细胞核进行翻译前被切除。
《2024年若干模式生物基因组中ORF、Intron和Exon的识别与特征研究》范文

《若干模式生物基因组中ORF、Intron和Exon的识别与特征研究》篇一一、引言随着生物信息学和基因组学的快速发展,对模式生物基因组的研究已经成为揭示生命奥秘的重要手段。
在基因组中,开放阅读框(ORF)、内含子(Intron)和外显子(Exon)是基因结构的重要组成部分。
本文旨在探讨若干模式生物基因组中ORF、Intron和Exon的识别方法及其特征研究。
二、方法1. 数据来源:本文选取了若干种模式生物的基因组数据,包括人类、小鼠、果蝇等。
2. ORF识别:通过生物信息学软件和算法,对基因组序列进行扫描,识别出潜在的ORF区域。
3. Intron和Exon识别:利用已知的基因注释信息,结合生物信息学工具,对基因组中的Intron和Exon进行识别。
三、ORF的识别与特征1. 识别方法:通过比对已知的蛋白质序列,结合密码子使用偏好性分析,以及跨膜区域预测等方法,对基因组中的ORF进行识别。
2. 特征分析:ORF具有编码蛋白质的功能,其长度、位置、序列特性等均对基因的表达和功能具有重要影响。
本研究发现,不同模式生物的基因组中ORF的长度、数量及分布存在差异,这可能与物种的进化历程和基因表达调控机制有关。
四、Intron的识别与特征1. 识别方法:Intron是基因中的非编码区域,通过与Exon的对比分析,结合生物信息学软件进行识别。
2. 特征分析:Intron在基因组中具有调节基因表达、增强mRNA稳定性等功能。
本研究发现,不同模式生物的基因组中Intron的长度、数量及位置存在差异,这可能与物种的基因表达调控机制和进化历程有关。
此外,Intron的剪接方式也对基因的表达和功能具有重要影响。
五、Exon的识别与特征1. 识别方法:Exon是基因中的编码区域,通过与Intron的对比分析,结合生物信息学软件进行识别。
2. 特征分析:Exon是蛋白质编码的关键区域,其序列特性、长度、位置等均对蛋白质的结构和功能具有重要影响。
寻找基因外显子、内含子的几种方法
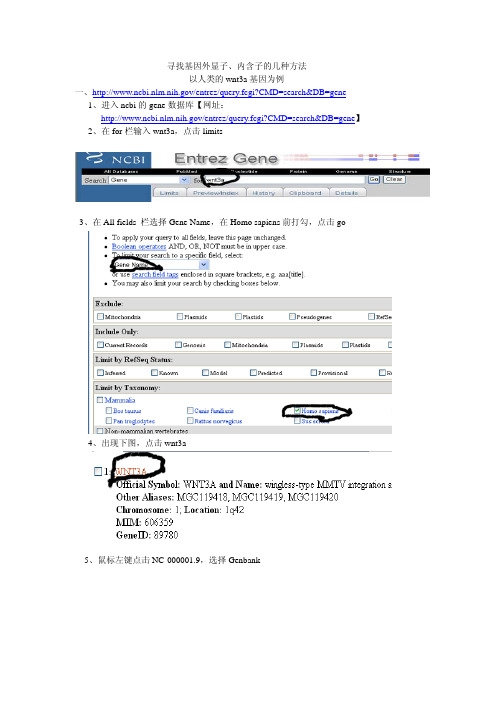
寻找基因外显子、内含子的几种方法以人类的wnt3a基因为例一、/entrez/query.fcgi?CMD=search&DB=gene1、进入ncbi的gene数据库【网址:/entrez/query.fcgi?CMD=search&DB=gene】2、在for栏输入wnt3a,点击limits3、在All fields 栏选择Gene Name,在Homo sapiens前打勾,点击go4、出现下图,点击wnt3a5、鼠标左键点击NC-000001.9,选择Genbank或在Genomic栏下点击Genbank【图中圈出的部分】6、出现下图,图中画线部分就是外显子的位点【注意不是图中圆圈的部分】外显子分别为1~14915617~1585843606~4387151936~54210二、/1、进入/的网页2、点击Gene Sorter3、在genome栏输入human,在search栏输入wnt3a,点击go4、出现下图,点击图中圈出来的部分:5、出现下图,点击sequence6、出现下图,点击Genomic7、出现下图,点击submit8、出现序列,其中外显子用大写字母,内含字用小写字母。
9、将其拷贝到word中,鼠标定位到大小写接头的位点,进行定位统计。
疑问:奇怪的是得出的结果与方法一、方法三不符,不知是什么原因?请大家指点,看看是哪里错了。
这种方法的介绍见:/bbs/actions/archive/post/6145797_1.html三、/1、进入/网页2、在search栏中选择homo sapiens,在for 栏输入wnt3a,点击go3、出现下图,点击图中圈出的部分。
4、出现下图,点击exon information5、出现下图。
从图中我们得知wnt3a基因位于1号染色体上,从226261375~226315584结束。
共有4个外显子,三个内含子。
外显子1 长149内含子1-2 长15467外显子2 长242内含子2-3 长27747外显子3 长266内含子3-4 长8064外显子4 长2275从外显子、内含子的长度,我们可以推出各外显子的序列位置,即1~14915617~1585843606~4387151936~54210。
基因外显子与内含子的查找方法

基因外显子与内含子的查找方法一、外显子/内含子的概念1. 外显子(exon) sequence of a gene's DNA that transcribes into protein structures外显子(expressed region) 是真核生物基因的一部分,它在剪接(Splicing)后仍会被保存下来,并可在蛋白质生物合成过程中被表达为蛋白质。
外显子是最后出现在成熟RNA中的基因序列, 又称表达序列。
既存在于最初的转录产物中,也存在于成熟的RNA分子中的核苷酸序列。
术语外显子也指编码相应RNA外显子的DNA中的区域。
*简言之,外显子就是指真核细胞的基因在表达过程中能编码蛋白质的核苷酸序列。
☆重要特点:①比较不同物种的相关基因,我们发现相应的外显子序列通常是保守的,而内含子序列则很少保守。
②编码蛋白质的序列通常处于选择压力之下,内含子由于没有选择压力,因此比外显子的进化快得多。
③通过确定在多种生物中出现的片段来鉴定编码区域,而外显子的保守性可以作为这种鉴定的基础2. 内含子(introns)内含子是基因内的间隔序列,不出现在成熟的RNA分子中,在转录后通过加工被切除。
大多数真核生物的基因都有内含子。
需注意的是,在古细菌中也有内含子。
在转录后的加工中,从最初的转录产物除去的内部的核苷酸序列。
术语内含子也指编码相应RNA内含子的DNA中的区域。
大多数真核结构基因中的间插序列(intervening sequence)或不编码序列。
它们可以转录,但在基因转录后,由这些间插序列转录的部分(也可用内含子这个术语表示)经加工被从初级转录本中准确除去,才产生有功能的RNA。
基因的编码部分称外显子。
内含子常比外显子长,且占基因的更大比例。
真核基因所含内含子的数目、位置和长度不尽相同,如鸡卵清蛋白基因的外显子被7个内含子隔开,鸡卵伴清蛋白基因有17个内含子,α-珠蛋白基因有2个内含子,卵粘蛋白基因有6个内含子等。
外显子、内含子、mRNA、CDS、ORF区

1、DNA复制:以DNA为模板,在DNA聚合酶的催化作用下,将四种游离的dNTP按照碱基互补配对原则合成新链DNA转录:以DNA为模版,在DNA指导的RNA聚合酶的作用下,将四种游离的NTP按照碱基互补配对的原则合成RNA翻译:以mRNA为模板,在核糖体内合成蛋白质的过程特点:DNA复制:模板为双链DNA,合成的新链与模板链一模一样,原料为四种dNTP,为半保留复制,需要引物转录:模板为双链DNA,为半不连续转录需要引物,原料为四种NTP,合成的新链除了把DNA上的T改为U外,其他一样翻译:模板为mRNA,原料为20中游离的氨基酸,3个碱基决定一个氨基酸2、mRNAmRNA(messenger RNA,信使RNA)信使RNA是由DNA经hnRNA剪接而成,携带遗传信息的能指导蛋白合成的一类单链核糖核酸。
3、基因DNA分为编码区和非编码区,编码区包含外显子和内含子,一般非编码区具有基因表达的调控功能,如启动子在非编码区。
编码区则转录为mRNA并最终翻译成蛋白质。
外显子和内含子都被转录到mRNA前体hnRNA中,当hnRNA进行剪接变为成熟的mRNA时,内含子被切除,而外显子保留。
实际上真正编码蛋白质的是外显子,而内含子则无编码功能,内含子存在于DNA中,在转录的过程中,DNA上的内含子也会被转录到前体RNA中,但前体RNA上的内含子会在RNA离开细胞核进行翻译前被切除。
4、CDSSequence coding for amino acids in protein蛋白质编码区CDS是Codingsequence的缩写,是编码一段蛋白产物的序列,是结构基因组学术语。
与开放读码框ORF的区别开放读码框是从一个起始密码子开始到一个终止密码子结束的一段序列;不是所有读码框都能被表达出蛋白产物,或者能表达出占有优势或者能产生生物学功能的蛋白。
CDS,是编码一段蛋白产物的序列。
cds必定是一个orf。
但也可能包括很多orf。
外显子、内含子、mRNA、CDS、ORF区别与联系

外显子、内含子、mRNA、CDS、ORF区别与联系1、DNA复制:以DNA为模板,在DNA聚合酶的催化作用下,将四种游离的dNTP依照碱基互补配对原则合成新链DNA转录:以DNA为模版,在DNA指导的RNA聚合酶的作用下,将四种游离的NTP依照碱基互补配对的原则合成RNA翻译:以mRNA为模板,在核糖体内合成蛋白质的过程特点:DNA复制:模板为双链DNA,合成的新链与模板链一模一样,原料为四种dNTP,为半保存复制,需要引物转录:模板为双链DNA,为半不连续转录需要引物,原料为四种NTP,合成的新链除了把DNA上的T改为U外,其他一样翻译:模板为mRNA,原料为20中游离的氨基酸,3个碱基决定一个氨基酸2、mRNAmRNA (messenger RNA,信使RNA)信使RNA是由DNA经hnRNA剪接而成,携带遗传信息的能指导蛋白合成的一类单链核糖核酸。
3、基因DNA分为编码区和非编码区,编码区包含外显子和内含子,一般非编码区具有基因表达的调控功能,如启动子在非编码区。
编码区则转录为mRNA并最终翻译成蛋白质。
外显子和内含子都被转录到mRNA前体hnRNA中,当hnRNA进行剪接变成成熟的mRNA时,内含子被切除,而外显子保存。
实际上真正编码蛋白质的是外显子,而内含子则无编码功能,内含子存在于DNA中,在转录的过程中,DNA上的内含子也会被转录到前体RNA中,但前体RNA上的内含子会在RNA离开细胞核进行翻译前被切除。
4、CDS Sequence coding for amino acids in protein 蛋白质编码区 CDS是Coding sequence的缩写,是编码一段蛋白产品的序列,是结构基因组学术语。
与开放读码框ORF的区别开放读码框是从一个起始密码子开始到一个终止密码子结束的一段序列;不是所有读码框都能被表达出蛋白产品,或者能表达出占有优势或者能发生生物学功能的蛋白。
CDS,是编码一段蛋白产品的序列。
RT-PCR引物设计与是否跨内含子的检测方法(图)

问题一:如何在pubmed上找到目标基因的DNA序列以及其内含子和外显子情况。
步骤:打开NCBI主页面,选GENE,输入基因名称-》显示出很多不同种但名字相同的基因(fig.1)-》找到你要的种属,打开-》显示基因信息,找到”Genomic regions, transcripts, and products”,点击基因名称,links到geneback(fig.2)-》显示了该基因DNA的信息(fig.3),可以看到这是基因组DNA,从mRNA选项中可以看到JION后面的是外显子的位置,该基因一个共有3个内含子,4个外显子,以及外显子的起至位置,为了以后的操作,可以把这个DNA序列和外显子的起至位置记下来。
问题二:如何在pubmed上找到目标基因的mRNA序列。
步骤:打开NCBI主页面,选GENE,输入基因名称-》显示出很多不同种但名字相同的基因-》找到你要的种属,打开-》显示基因信息,找到“mRNA and protein”前面那个NM***********,号码(fig.4),把它记下来(后面的blast要用)并点开-》显示这个基因的mRNA信息(Fig.5),最后的ORIGIN是CDNA序列,把它记下来,一会PRIMER的时候要用。
问题三:如何设计跨内含子的引物(利用primer-blast)1)打开primer-blast网站:(/tools/primer-blast/)2)将mRNA编号(NM***********)输入序列框中,real time PCR 一般产物长度为80~150bp也可以超一点,但是会影响扩增效率,这样就不能用ddCT法计算了,酌情考虑。
温度在55~63和GC含量在40%~60%,两条链不要差太远。
3)会得到若干条引物(fig。
6),红条中的黄圈标记的小白色缺口是内含子所在的位置,跨过小缺口就是跨了内含子。
根据下面列出的引物具体的温度GC含量之类的再进行选择。
4)将初步选择的引物用PRIMER 5验证引物二聚体,发卡结构等,方法见下个问题“问题四:用PRIMER 5查找已知引物的产物位置”。
外显子、内含子、mRNA、CDS、ORF区别与联系

外显子、内含子、mRNA、CDS、ORF区别与联系1、DNA复制:以DNA为模板,在DNA聚合酶的催化作用下,将四种游离的dNTP按照碱基互补配对原则合成新链DNA转录:以DNA为模版,在DNA指导的RNA聚合酶的作用下,将四种游离的NTP按照碱基互补配对的原则合成RNA翻译:以mRNA为模板,在核糖体内合成蛋白质的过程特点:DNA复制?:? 模板为双链DNA,合成的新链与模板链一模一样,原料为四种dNTP,为半保留复制,需要引物转录:模板为双链DNA,为半不连续转录需要引物,原料为四种NTP,合成的新链除了把DNA上的T改为U外,其他一样?翻译?:模板为mRNA,原料为20中游离的氨基酸,3个碱基决定一个氨基酸2、mRNAmRNA (messenger RNA,信使RNA)信使RNA是由DNA经hnRNA剪接而成,携带遗传信息的能指导蛋白合成的一类单链核糖核酸。
3、基因DNA分为编码区和非编码区,编码区包含外显子和内含子,一般非编码区具有基因表达的调控功能,如启动子在非编码区。
编码区则转录为mRNA 并最终翻译成蛋白质。
外显子和内含子都被转录到mRNA前体hnRNA中,当hnRNA进行剪接变为成熟的mRNA时,内含子被切除,而外显子保留。
实际上真正编码蛋白质的是外显子,而内含子则无编码功能,内含子存在于DNA中,在转录的过程中,DNA上的内含子也会被转录到前体RNA中,但前体RNA上的内含子会在RNA离开细胞核进行翻译前被切除。
4、CDS? Sequence coding for amino acids in protein 蛋白质编码区?? ? CDS是Coding sequence的缩写,是编码一段蛋白产物的序列,是结构基因组学术语。
与开放读码框ORF的区别开放读码框是从一个起始密码子开始到一个终止密码子结束的一段序列;不是所有读码框都能被表达出蛋白产物,或者能表达出占有优势或者能产生生物学功能的蛋白。
基因外显子与内含子的查找方法

基因外显子与内含子的
查找方法
本页仅作为文档封面,使用时可以删除
This document is for reference only-rar21year.March
基因外显子与内含子的查找方法
基因的内含子和外显子的序列
在NCBI找到基因序列之后,要确定其内含子和外显子的序列是比较简单的。
如果是RefSeq序列的话,那就更加容易了。
简单的思路就是跟基因组序列比对就行了。
%用NCBI的Splign工具,详见:图解:如何在NCBI上找到HNF-4基因第4个外显子的序列。
%但由于猪KIT(FJ938289)基因不是Refseq序列。
这就比较难了,由该序列的注释可知(/chromosome="8),该序列是属于第8号染色体。
%所以先确定猪(Pig,Sus scrofa)的第8号染色体是否已经测完序了。
先在Taxonomy数据库搜索,得到Taxonomy ID: 9823。
直接用关键词
txid9823[Organism:noexp]搜索Genome数据库。
猪有19条染色体,结果为11。
看图:
%但其中并没有第8号染色体的。
所以要确定猪KIT(FJ938289)基因的内含子和外显子的序列,好像就不大可能了。
反正我是没办法了。
基因结构图
1,NCBI提供的Graphics工具
在NCBI搜索FJ938289之后,接下来用NCBI提供的Graphics工具来查看,就能知道基因的内含子和外显子的序列了(同时也是一个基因结构图哦)。
看图:。
寻找基因外显子内含子的几种方法

寻找基因外显子内含子的几种方法1.基于基因组序列的预测方法:基因组预测是寻找基因外显子和内含子的常用方法。
这种方法基于一系列计算模型和算法,分析基因组序列的物理特性和编码信息,通过寻找开放阅读框(ORFs)和间隔序列来预测基因的位置。
常用的基因组预测软件包括Glimmer、GeneMark、FgeneSH等。
2.基于转录本信息的实验方法:转录本是基因表达时的中间产物,包含了编码区域的外显子以及非编码区域的内含子。
转录组学技术如全长转录组测序(full-length transcriptome sequencing)和转录组组装(transcriptome assembly)可以利用高通量测序技术直接检测和组装转录本,从而获得基因的外显子和内含子的信息。
3.基于剪接的分析方法:剪接是在基因表达过程中原始mRNA分子中排除内含子,并将外显子连接在一起的过程。
通过利用剪接位点上的保守序列和转录组学测序数据,可以推测基因的外显子和内含子的位置和组成。
常用的剪接分析软件包括Tophat、Cufflinks等。
4.基于保守序列的比较基因组学方法:比较基因组学方法是通过比较不同物种的基因组序列,找出其保守区域以预测基因的外显子和内含子。
保守序列通常指具有功能约束的序列区域,如编码区域和剪接位点。
这种方法可以通过多序列比对和保守区域预测软件来实现。
5.基于基因组特征的机器学习方法:机器学习方法是一种基于已知标注数据训练模型,然后利用该模型来预测新的数据或未标注数据。
在基因组学中,可以利用已知的基因和非基因区域的数据来训练模型,然后使用这些模型来预测新的区域是否为基因的外显子或内含子。
这种方法通常结合了基因组序列特征、保守序列和转录组学数据。
综上所述,寻找基因外显子和内含子的方法可以基于基因组序列的预测、转录本信息的实验、剪接的分析、比较基因组学和机器学习等方法。
这些方法可以相互结合,共同提供对基因组序列的准确和全面的理解,进而推动对基因功能以及相关疾病的研究。
基因外显子与内含子的查找方法

基因外显子与内含子的查找方法基因是遗传信息的基本单位,它们含有编码蛋白质所需的信息。
基因由外显子(exons)和内含子(introns)组成。
外显子是具有转录和翻译功能的区域,它们编码蛋白质序列;而内含子则是非编码区域,它们在转录过程中被剪切掉。
查找基因的外显子和内含子是基因组研究的重要一步,它能够帮助我们了解基因的结构和功能。
现代分子生物学技术的进步,尤其是基因组测序技术的发展,使得基因外显子和内含子的查找变得更加准确和高效。
下面将介绍一些常见的查找基因外显子和内含子的方法。
1.基于实验的方法:-准确分离基因组DNA:通过DNA提取实验,我们可以从细胞中准确分离出基因组DNA。
-cDNA合成:将基因组DNA反转录为互补DNA(cDNA),这可以消除内含子的干扰,使得外显子更容易被检测。
-基于PCR的方法:利用多聚酶链反应(PCR),我们可以特异性扩增外显子和内含子的DNA序列,并通过电泳进行分离和检测。
- 基于Southern印迹方法:Southern印迹是一种从DNA样品中分离具有特定DNA序列的方法,它能够定量测量基因外显子的存在和数量。
- 基于Northern印迹方法:Northern印迹是一种从RNA样品中分离具有特定RNA序列的方法,它能够帮助我们检测基因的转录情况,进而确定外显子的位置。
2.基于计算和生物信息学的方法:-基因组注释:通过分析已知基因组的注释信息,如转录组、蛋白质结构和功能等,可以预测基因的外显子和内含子的位置。
-基于比对的方法:将已知蛋白质或cDNA序列与未知基因组序列进行比对,通过寻找序列相似性和保守性来预测外显子的位置。
- 基于开放源代码软件的分析工具:如NCBI的BLAST、Ensembl、UCSC Genome Browser等在线工具,提供了基因组一站式注释和查找外显子和内含子的功能。
无论是基于实验还是基于计算和生物信息学的方法,都允许我们准确地查找基因的外显子和内含子。
内含子-外显子边界预测方法__解释说明以及概述

内含子-外显子边界预测方法解释说明以及概述1. 引言1.1 概述本文将介绍内含子-外显子边界预测方法的定义、原理以及应用。
内含子和外显子是基因进行转录和剪接过程中的关键元素,准确预测其边界位置对于理解基因结构和功能具有重要意义。
内含子-外显子边界预测方法是通过分析DNA或RNA序列和结构特征来推断内含子和外显子之间的连接点。
1.2 文章结构本文主要包括以下几个部分:- 引言:介绍文章的背景、目的和结构;- 内含子-外显子边界预测方法:阐述定义、背景以及两种常见的预测方法;- 解释说明内含子-外显子边界预测方法的原理和步骤:详细说明基于序列标记和RNA结构的预测方法;- 相关研究进展与应用案例分析:概述最新研究成果,并通过应用案例分析说明内含子-外显子边界在遗传疾病中的重要性;- 结论与展望:总结文章内容,回顾贡献点,并展望未来该领域的发展方向。
1.3 目的本文旨在介绍内含子-外显子边界预测方法的原理和步骤,并探讨该方法在遗传疾病研究中的应用。
通过深入了解这些预测方法,读者可以更好地理解基因结构和功能,为相关领域的进一步研究提供参考依据。
同时,本文也将总结目前的研究进展,探讨未来该领域的发展趋势和挑战。
2. 内含子-外显子边界预测方法2.1 定义和背景内含子-外显子边界预测方法是一种用于预测基因组中内含子和外显子之间的边界位置的技术。
在基因表达调控中,内含子和外显子的区别对于正确识别基因以及产生不同转录本具有重要意义。
准确预测内含子-外显子边界位置可以帮助我们了解基因的结构与功能,并在遗传疾病诊断和治疗等领域具有潜在应用价值。
2.2 方法一:基于序列标记的预测方法基于序列标记的预测方法是通过分析DNA或RNA序列中相对保守的模式、特征或信号来推断内含子-外显子边界位置。
这些标记可能涉及剪接位点、蛋白质结合位点、启动子区域等。
该方法通常包括以下步骤:(1) 收集训练数据集:从已知基因组中提取已注释的内含子-外显子边界序列,作为训练样本。
内含子和外显子的位置

偶最近也在做RT-PCR。
也正在设计跨内含子的引物。
在园子里看了些文献,也请教了一些人。
个人感觉主要是你找到这个基因的具体的内含子和外显子的位置后再设计引物一点也不难。
只要在跨越的引物中选取条件好的就行了。
其中对于如何找到内含子和外显子,偶是用以下的方法。
一、首先登录Pubmed找到你的基因序列,这个应该很简单。
偶不多说。
二、如下图进入Blastl界面,选取genomes中的相应物种。
三将你的序列拷贝到查询框中,进行查询(Begin search)。
然后点击format,一路点下去,出现下图样的对话框。
点击genome view然后点击其相应的染色体号,我查的是HIF基因,在14号染色体上。
相应的染色体上会出现红色的标记。
好了,如下图。
你可以看到右边的这些方框最后给的序列,就是外显子的序列。
你可以对着此来设计引物,找到哪些跨过内含子的引物。
首先要明白:RT-PCR的引物之所以要跨内含子设计,主要原因就是为了避免再PCR过程中基因组DNA的影响。
那么其实只要在提RNA时除去DNA也就没什么关系了。
RNA沉淀之后,在100ul反应体系中用RNAse free的DNAseI37C处理0.5h,然后直接用100ul 氯仿抽提,异丙醇(别忘了加1/10体积4M NaAc)再次沉淀后用适量DEPC水溶解即可。
NCBI>GENE>基因名称搜索(Click the picture to see the source)louischenPosts:518Score:432004-07-30 06:57点击基因名称进入左键击NC_000019>GRAPHICSintron,extron的排列,可以点击进入粗划线为外显子,并有对应的protein编码还在/也可以,在主页上输入基因的名字,找出该物种的基因序列,在transview和exonview上可以看到外显子和内含子,此网页简单,得到不同颜色标记的外显子。
《外显子连接序列与连接处内含子序列的相互作用》范文

《外显子连接序列与连接处内含子序列的相互作用》篇一一、引言在生物学和遗传学的研究中,基因的转录和翻译过程一直是研究的热点。
基因序列中包含着丰富的信息,包括外显子和内含子等结构。
外显子连接序列与连接处内含子序列的相互作用是基因表达过程中至关重要的环节。
本文将就这一主题展开讨论,分析其相互作用机制及其在生物学中的意义。
二、外显子与内含子的基本概念1. 外显子:外显子是基因序列中编码蛋白质的部分,也称为编码序列。
在基因转录过程中,外显子被保留并参与蛋白质的合成。
2. 内含子:内含子是基因序列中非编码部分,位于两个外显子之间。
在转录过程中,内含子被剪切掉,只留下外显子部分用于蛋白质的合成。
三、外显子连接序列与内含子序列的相互作用1. 连接序列的作用:外显子连接序列位于外显子之间的交界处,起着连接相邻外显子的作用。
连接序列的正确拼接对于基因表达至关重要。
2. 相互作用机制:在基因转录过程中,RNA聚合酶将DNA 上的遗传信息转录为RNA。
在这个过程中,内含子和外显子被剪切和拼接,形成成熟的mRNA。
外显子连接序列与内含子序列之间的相互作用是这一过程的关键环节。
四、相互作用的具体过程1. 剪切过程:在转录过程中,内含子被剪切掉,只留下外显子部分。
这一过程需要剪切酶的参与,如RNA剪切酶等。
2. 拼接过程:剪切后的外显子通过连接序列进行拼接,形成成熟的mRNA。
这一过程需要拼接酶的参与,如RNA拼接酶等。
连接序列与内含子序列的相互作用确保了拼接过程的准确性。
3. 调控作用:外显子连接序列与内含子序列的相互作用还受到多种调控因子的影响,如转录因子、剪切因子等。
这些调控因子通过与连接序列和内含子序列的结合,影响基因的表达水平和表达模式。
五、相互作用的生物学意义1. 维持基因结构的稳定性:外显子连接序列与内含子序列的相互作用确保了基因结构的稳定性。
在基因转录和翻译过程中,这一相互作用保证了基因信息的准确传递和表达。
寻找真核生物基因有无内含子及cDNA序列
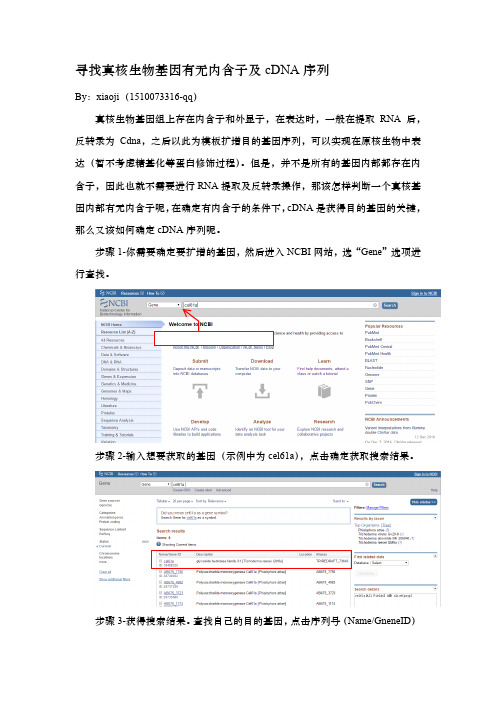
寻找真核生物基因有无内含子及cDNA 序列By :xiaoji (1510073316-qq )真核生物基因组上存在内含子和外显子,在表达时,一般在提取RNA 后,反转录为Cdna ,之后以此为模板扩增目的基因序列,可以实现在原核生物中表达(暂不考虑糖基化等蛋白修饰过程)。
但是,并不是所有的基因内部都存在内含子,因此也就不需要进行RNA 提取及反转录操作,那该怎样判断一个真核基因内部有无内含子呢,在确定有内含子的条件下,cDNA 是获得目的基因的关键,那么又该如何确定cDNA 序列呢。
步骤1-你需要确定要扩增的基因,然后进入NCBI 网站,选“Gene ”选项进行查找。
步骤2-输入想要获取的基因(示例中为cel61a ),点击确定获取搜索结果。
步骤3-获得搜索结果。
查找自己的目的基因,点击序列号(Name/GneneID)“Gene”模式下搜索基因进入详细目录。
步骤4-获得目的基因详细信息。
进入“Genomic regions, transcripts, and products”初步查看在基因组上该基因是否有内含子;外显子个数内含子在基因组上的位置步骤5-初步确定是否存在内含子。
在左上角的文字说明里可以看到“Exon count 2”可知有两段外显子,则确定存在一个内含子;Graphics中可以看到,基因中存在间隔,中间一部分没有加粗显示,说明是内含子,之后要确定内含子的确定位置及找到cDNA序列,点击Genebank进入基因序列界面;基因在基因组上的位置步骤6-基因详细信息界面;mRNA序列位置(间隔序列,说明存在内含子)进入蛋白氨基酸序列界面步骤7-将页面向下浏览,在mRNA序列部分会显示内含子位置,mRNA序列位置1-218bp,273-1351bp,则中间间隔部分为内含子。
下方CDS序列显示cDNA在基因组上的位置,比mRNA位置略短。
之后点击CDS区protein_id链接,进入氨基酸序列界面;步骤8-氨基酸序列界面,点击CDS右下角会出现提示信息;步骤9-点击CDS后出现的提示信息(右下角),点击下方“Display”部分的GenBank进入cDNA序列界面;cDNA序列步骤10-获得cDNA序列;cDNA序列位置(从ATG开始)点击可以进入引物设计界面步骤11-在右边操作界面,可以选择要显示的序列信息,也可以在此设计引物用于目的基因扩增。
外显子、内含子、mRNA、CDS、ORF区别与联系

外显子、内含子、mRNA、CDS、ORF区别与联系1、DNA复制:以DNA为模板,在DNA聚合酶的催化作用下,将四种游离的dNTP按照碱基互补配对原则合成新链DNAﻫ转录:以DNA为模版,在DNA指导的RNA聚合酶的作用下,将四种游离的NTP按照碱基互补配对的原则合成RNAﻫ翻译:以mRNA为模板,在核糖体内合成蛋白质的过程特点:DNA复制:模板为双链DNA,合成的新链与模板链一模一样,原料为四种dNTP,为半保留复制,需要引物转录:模板为双链DNA, 为半不连续转录需要引物,原料为四种NTP,合成的新链除了把DNA上的T改为U外,其他一样ﻫ翻译 :模板为mRNA,原料为20中游离的氨基酸,3个碱基决定一个氨基酸2、mRNAmRNA (messengerRNA,信使RNA)信使RNA是由DNA经hnRNA剪接而成,携带遗传信息的能指导蛋白合成的一类单链核糖核酸。
3、基因DNA分为编码区和非编码区,编码区包含外显子和内含子,一般非编码区具有基因表达的调控功能,如启动子在非编码区。
编码区则转录为mRNA并最终翻译成蛋白质。
外显子和内含子都被转录到mRNA前体hnRNA中,当hnRNA进行剪接变为成熟的mRNA时,内含子被切除,而外显子保留。
实际上真正编码蛋白质的是外显子,而内含子则无编码功能,内含子存在于DNA中,在转录的过程中,DNA上的内含子也会被转录到前体RNA中,但前体RNA上的内含子会在RNA离开细胞核进行翻译前被切除。
4、CDSSequence coding for aminoacids in protein蛋白质编码区CDS是Coding sequence的缩写,是编码一段蛋白产物的序列,是结构基因组学术语。
与开放读码框ORF的区别开放读码框是从一个起始密码子开始到一个终止密码子结束的一段序列;不是所有读码框都能被表达出蛋白产物,或者能表达出占有优势或者能产生生物学功能的蛋白。
CDS,是编码一段蛋白产物的序列。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
寻找基因外显子、内含子的几种方法
以人类的wnt3a基因为例
一、/entrez/query.fcgi?CMD=search&DB=gene
1、进入ncbi的gene数据库【网址:
/entrez/query.fcgi?CMD=search&DB=gene】
2、在for栏输入wnt3a,点击limits
3、在All fields 栏选择Gene Name,在Homo sapiens前打勾,点击go
4、出现下图,点击wnt3a
5、鼠标左键点击NC-000001.9,选择Genbank
或在Genomic栏下点击Genbank【图中圈出的部分】
6、出现下图,图中画线部分就是外显子的位点【注意不是图中圆圈的部分】外显子分别为1~149
15617~15858
43606~43871
51936~54210
二、/
1、进入/的网页
2、点击Gene Sorter
3、在genome栏输入human,在search栏输入wnt3a,点击go
4、出现下图,点击图中圈出来的部分:
5、出现下图,点击sequence
6、出现下图,点击Genomic
7、出现下图,点击submit
8、出现序列,其中外显子用大写字母,内含字用小写字母。
9、将其拷贝到word中,鼠标定位到大小写接头的位点,进行定位统计。
疑问:奇怪的是得出的结果与方法一、方法三不符,不知是什么原因?请大家指点,看看是哪里错了。
这种方法的介绍见:/bbs/actions/archive/post/6145797_1.html
三、/
1、进入/网页
2、在search栏中选择homo sapiens,在for 栏输入wnt3a,点击go
3、出现下图,点击图中圈出的部分。
4、出现下图,点击exon information
5、出现下图。
从图中我们得知wnt3a基因位于1号染色体上,从226261375~226315584结束。
共有4个外显子,三个内含子。
外显子1 长149
内含子1-2 长15467
外显子2 长242
内含子2-3 长27747
外显子3 长266
内含子3-4 长8064
外显子4 长2275
从外显子、内含子的长度,我们可以推出各外显子的序列位置,即
1~149
15617~15858
43606~43871
51936~54210。
