原位杂交技术步骤
荧光原位杂交 操作

荧光原位杂交操作荧光原位杂交(FISH)是一种基因组学的技术,它使用荧光探针将特定的DNA序列定位在染色体上。
这种技术被广泛用于分析染色体的结构和功能,以及人类基因组变异和疾病的研究。
本文将介绍荧光原位杂交操作的步骤。
步骤1:样品制备FISH技术可以应用于不同种类的样本,如人类细胞、动植物组织等。
对于人类细胞的样本,常用的方法是将细胞分离,制作成染色体悬液。
首先要通过荧光染色法把两种不同的染色体和特定的基因序列染成不同的颜色。
这需要用到已知基因序列的探针,可以从已有的文献中获取。
如果没有,可以通过设计和合成新的探针。
步骤2:制作探针FISH探针是由一段DNA片段制成的。
这个片段可用PCR方法从DNA中扩增出来,或者可以通过化学合成的方法制造。
制造探针时需要注意,探针的长度和序列应该与目标序列相匹配,可以通过BLAST或其他基因数据库检索确认。
通常使用荧光染料标记探针,以便在镜头下观察到探针的定位。
常用的标记分子有荧光素(FITC),荧光素同路胺(rhodamine)和锔(Cy3和Cy5)。
这些分子的选择取决于探针和荧光显微镜系统的兼容性。
在制作FISH探针时,需要注意探针的特异性和灵敏度。
为了达到这一目的,需要经过一系列的标记、纯化和探针肢解操作。
探针一般都是双链DNA,通过加入单链转化酶(S1)或DNA酶I,使双链DNA变成单链DNA,并标记在其5'-末端上。
标记完成后,进行染色体裂解、脱氧核糖核苷酸(dNTP)和DNA聚合酶的反应来进行回收和标记探针,以免对染色体质量产生影响。
对于高复杂度的基因组,可以使用克隆探针阵列,在单个染色体上定位多个序列。
步骤4:染色体固定和前处理将样本固定在载玻片上,这可以是用乙醛和甲醛等固定剂进行固定。
将载玻片上的样本通过逐步浸入水,逐步替换固定样本中的有机溶剂来除去样本中的RNA,然后用类碱/类酸(如乙酸/氯化锂,0.1M Na丙二酸缓冲pH 9.0)进行前处理,以增加荧光探针的穿透率,使其更好地结合DNA的目标序列。
原位杂交操作流程

原位杂交操作流程1、使用地高辛标记的核酸探针进行石蜡切片的RNA原位杂交第一天1)二甲苯于37℃脱蜡2次,每次15分钟;2)无水乙醇浸泡2次,每次3分钟;3) 95%乙醇浸泡2次,每次3分钟;4) PBS清洗3分钟;5) 2%焦碳酸二乙酯室温下浸泡10分钟;6) PBS清洗10分钟;7)加入胃蛋白酶25ul/ml,37℃孵育15分钟;8) PBS清洗2次,每次3分钟;9) 0.2N的HCl孵育30分钟;10)PBS清洗2次,每次3分钟;11)0.25%无水乙酸和0.1M三乙醇胺孵育10分钟;12)PBS清洗2次,每次5分钟;13)预杂交缓冲液孵育30分钟;14)准备核酸探针混合物:使用预杂交缓冲液稀释探针,85℃加热5分钟,置于冰块中10分钟;15)杂交;第二天16)将玻片置于SSC中2次,每次5分钟以去除封片;17)PBS清洗3分钟;18)RNA酶A溶液中(或0.1-1ng/mlPBS中),37℃孵育30分钟;19)PBS清洗5分钟;20)室温,2×SSC清洗10分钟;21)37℃,1×SSC清洗10分钟;22)37℃,0.5×SSC清洗10分钟;23)缓冲液A孵育10分钟;24)缓冲液A(1%正常绵羊血清和0.03%三重氢核X-100)孵育30分钟;25)加入抗地高辛抗体(1/200的上述缓冲液,来自Boehringer Mannheim),37℃孵育3 小时;26)缓冲液A清洗2次,每次10分钟;27)缓冲液B清洗2次,每次5分钟;28)制成NBT/BCIP暗处保存30-60分钟,显微镜下进行观察,如果背景尚佳,显色时间可延长到16小时;29)停止缓冲液B的反应,用水进行简单的清洗;30)固红,脱水以及封片进行核的复染。
2、使用地高辛标记的寡核苷酸探针进行石蜡切片的原位DNA杂交第一天1)二甲苯于37℃脱蜡2次,每次15分钟;2)无水乙醇浸泡2次,每次5分钟;3) 95%乙醇浸泡2次,每次5分钟;4) PBS清洗5分钟;5) 2%焦碳酸二乙酯室温下浸泡10分钟;6) PBS清洗5分钟;7)加入胃蛋白酶25ul/ml,37℃孵育10分钟;8) PBS清洗2次,每次5分钟;9) 0.2N的HCl孵育30分钟;10)PBS清洗2次,每次5分钟;11)0.25%无水乙酸和0.1M三乙醇胺孵育10分钟;12)PBS清洗5分钟;13)预杂交缓冲液孵育30分钟;14)准备寡核苷酸探针混合物:使用预杂交缓冲液稀释探针;15)杂交;第二天16)将玻片置于SSC中以去除封片;17)室温,2×SSC清洗10分钟;18)37℃,1×SSC清洗10分钟;19)37℃,0.5×SSC清洗10分钟;20)缓冲液A孵育10分钟;21)缓冲液A孵育30分钟;22)加入抗地高辛抗体37℃孵育3小时;23)缓冲液A清洗2次,每次5分钟;24)缓冲液B清洗2次,每次5分钟;25)制成NBT/BCIP暗处保存30-60分钟,显微镜下进行观察,如果背景尚佳,显色时间可长到16小时;26)停止缓冲液B的反应,用水进行简单的清洗;27)固红,脱水以及封片进行核的复染。
原位杂交实验步骤

原位杂交实验步骤原位杂交实验呀,就像是一场精心编排的舞蹈。
首先呢,得准备好舞台,也就是标本的处理。
这就好比给舞者们准备一个合适的场地,要把标本固定好,让它稳稳地待在那儿,可不能在中途出啥岔子。
接下来,就是探针的准备啦。
这探针就像是舞蹈的主角,得精心挑选和设计。
它要能准确地找到自己的目标,和标本上特定的序列结合。
然后,杂交这一步可太关键啦!就像舞者们在舞台上的精彩互动。
让探针和标本在合适的条件下相遇、结合,产生奇妙的反应。
之后呢,要进行信号的检测。
这就好比要看到舞者们的精彩表现呈现出来的效果。
通过各种方法,让杂交后的信号能够清晰地被我们看到。
再说说洗片这一步吧,就像是给舞台打扫干净。
把多余的杂质、干扰都去掉,只留下最精彩的部分。
整个过程中,每一步都得小心翼翼,就跟跳舞时每个动作都要精准到位一样。
要是哪一步出了差错,那可就像舞蹈中出现了失误,会影响整个演出的效果呀!做原位杂交实验,还得有耐心。
不能着急忙慌的,得一步一步慢慢来。
就像跳舞,不能一下子就跳到结尾,得按照节奏,一个动作一个动作地来。
而且呀,实验环境也很重要呢。
温度啦、湿度啦,都得控制好,这就好比舞蹈的灯光、音响,得配合得恰到好处,才能让整个表演更加完美。
想象一下,如果标本处理得不好,那后面的步骤不就都白费啦?或者探针没准备好,那还怎么去找到目标呢?所以啊,每一步都不能马虎,都得认真对待。
这原位杂交实验,可不就是一场科学与技术的精彩舞蹈嘛!咱可得把这场舞跳好,跳出精彩,跳出成果来!这就是原位杂交实验的步骤啦,大家可都得记住咯!。
原位杂交操作流程

原位杂交操作流程1、使用地高辛标记的核酸探针进行石蜡切片的RNA原位杂交第一天1)二甲苯于37°C脱蜡2次,每次15分钟;2)无水乙醇浸泡2次,每次3分钟;3)95%乙醇浸泡2次,每次3分钟;4)PBS清洗3分钟;5)2%焦碳酸二乙酯室温下浸泡10分钟;6)PBS清洗10分钟;7)加入胃蛋白酶25ul/ml,37°C孵育15分钟;8)PBS清洗2次,每次3分钟;9)0.2N的HCl孵育30分钟;10)PBS清洗2次,每次3分钟;11)0.25%无水乙酸和0.1M三乙醇胺孵育10分钟;12)PBS清洗2次,每次5分钟;13)预杂交缓冲液孵育30分钟;14)准备核酸探针混合物:使用预杂交缓冲液稀释探针,85°C加热5分钟,置于冰块中10分钟;15)杂交;第二天16)将玻片置于SSC中2次,每次5分钟以去除封片;17)PBS清洗3分钟;18)RNA酶A溶液中(或0.1-1ng/mlPBS中),37°C孵育30分钟;19)PBS清洗5分钟;20)室温,2XSSC清洗10分钟;21)37°C,1XSSC清洗10分钟;22)37°C,0.5XSSC清洗10分钟;23)缓冲液A孵育10分钟;24)缓冲液A(1%正常绵羊血清和0.03%三重氢核X-100)孵育30分钟;25)加入抗地高辛抗体(1/200的上述缓冲液,来自BoehringerMannheim),37°C孵育3小时;26)缓冲液A清洗2次,每次10分钟;27)缓冲液B清洗2次,每次5分钟;28)制成NBT/BCIP暗处保存30-60分钟,显微镜下进行观察,如果背景尚佳,显色时间可延长到16小时;29)停止缓冲液B的反应,用水进行简单的清洗;30)固红,脱水以及封片进行核的复染。
2、使用地高辛标记的寡核苷酸探针进行石蜡切片的原位DNA杂交第一天1)二甲苯于37C脱蜡2次,每次15分钟;2)无水乙醇浸泡2次,每次5分钟;3)95%乙醇浸泡2次,每次5分钟;4)PBS清洗5分钟;5)2%焦碳酸二乙酯室温下浸泡10分钟;6)PBS清洗5分钟;7)加入胃蛋白酶25ul/ml,37C孵育10分钟;8)PBS清洗2次,每次5分钟;9)0.2N的HCl孵育30分钟;10)PBS清洗2次,每次5分钟;11)0.25%无水乙酸和0.1M三乙醇胺孵育10分钟;12)PBS清洗5分钟;13)预杂交缓冲液孵育30分钟;14)准备寡核苷酸探针混合物:使用预杂交缓冲液稀释探针;15)杂交;第二天16)将玻片置于SSC中以去除封片;17)室温,2XSSC清洗10分钟;18)37°C,1XSSC清洗10分钟;19)37°C,0.5XSSC清洗10分钟;20)缓冲液A孵育10分钟;21)缓冲液A孵育30分钟;22)加入抗地高辛抗体37C孵育3小时;23)缓冲液A清洗2次,每次5分钟;24)缓冲液B清洗2次,每次5分钟;25)制成NBT/BCIP暗处保存30-60分钟,显微镜下进行观察,如果背景尚佳,显色时间可长到16小时;26)停止缓冲液B的反应,用水进行简单的清洗;27)固红,脱水以及封片进行核的复染。
原位杂交实验操作步骤

原位杂交实验操作步骤撰写人:范为民一、实验原理原位杂交是指借助于核酸分子杂交的方法,在显微镜水平检测和定位特异的核苷酸片段。
现在原位杂交已经成为在分子水平研究肿瘤和遗传性疾病的发生,发展和调控等根本性问题的有力工具。
二、试剂盒本实验室常用的原位杂交试剂盒是博士德公司生产的敏感加强型原位杂交检测试剂盒,此试剂盒分为两种,一种为过氧化物酶(POD)检测(MK1030型),一种为碱性磷酸酶(AP)检测系统(MK1032型),用于mRNA的杂交。
过氧化物酶(POD)检测的最终信号为棕黄色,而碱性磷酸酶(AP)检测的信号为紫色,因后者信号比较突出,所以一般采用后一种检测方法。
两种检测方法的实验步骤相差不多,所用洗脱缓冲液也大同小异。
用于杂交的探针也可以分为两种,一种是DNA探针,即是用DNA与组织中的mRNA杂交,另一种是RNA 探针,即用RNA与组织中的mRNA杂交。
DNA探针处理操作简单,但杂交信号一般不如RNA探针强烈,所以条件允许的话一般采用RNA探针。
下面先介绍碱性磷酸酶(AP)检测试剂盒,采用RNA作为探针的操作步骤。
三、实验步骤原位杂交实验主要包括三大部分,即组织冰冻切片、RNA探针标记、原位杂交三部分。
(一)组织冰冻切片1. 实验准备(1)原位杂交专用载玻片:用多聚赖氨酸处理后的载玻片,使切片紧密粘附在玻片上,可以用于后面的洗脱。
一般一张载玻片上可以贴至多十张切片(可以是不同组织的切片),所以需要玻片的数目需要根据实验的要求而定。
这种专用载玻片可以从中杉金桥公司购买,目前价格是每片2.2元,玻片有一面的一端是毛玻璃,用于标记组织名称等,切片应该贴在此面,切勿贴到反面。
(2)缓冲液配备1.1器具准备剪刀、镊子各三把,开壳钳一把,100ml量筒一个,磁力搅拌子一个;100ml试剂瓶一个,250ml试剂瓶三个。
以上器具均洗净后置于180摄氏度以上烘烤6小时以上。
铅笔、显微镜、冰、吸水纸、一次性塑料手套等。
原位杂交操作流程3
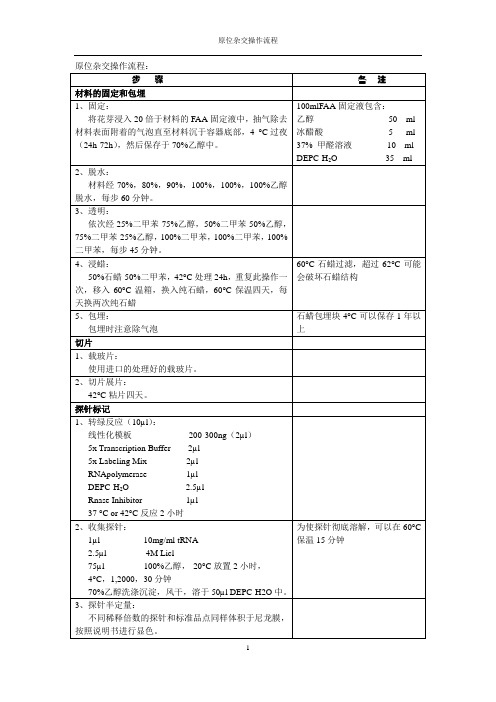
原位杂交操作流程:步骤备注材料的固定和包埋1、固定:将花芽浸入20倍于材料的FAA固定液中,抽气除去材料表面附着的气泡直至材料沉于容器底部,4 °C过夜(24h-72h),然后保存于70%乙醇中。
100mlFAA固定液包含:乙醇50 ml 冰醋酸 5 ml 37% 甲醛溶液10 ml DEPC-H2O 35 ml2、脱水:材料经70%,80%,90%,100%,100%,100%乙醇脱水,每步60分钟。
3、透明:依次经25%二甲苯-75%乙醇,50%二甲苯-50%乙醇,75%二甲苯-25%乙醇,100%二甲苯,100%二甲苯,100%二甲苯,每步45分钟。
4、浸蜡:50%石蜡-50%二甲苯,42°C处理24h,重复此操作一次,移入60°C温箱,换入纯石蜡,60°C保温四天,每天换两次纯石蜡60°C石蜡过滤,超过62°C可能会破坏石蜡结构5、包埋:包埋时注意除气泡石蜡包埋块4°C可以保存1年以上切片1、载玻片:使用进口的处理好的载玻片。
2、切片展片:42°C粘片四天。
探针标记1、转绿反应(10µl):线性化模板200-300ng(2µl)5x Transcription Buffer 2µl5x Labeling Mix 2µlRNApolymerase 1µlDEPC-H2O 2.5µlRnase Inhibitor 1µl37 °C or 42°C反应2小时2、收集探针:1µl 10mg/ml tRNA2.5µl 4M Licl75µl 100%乙醇,-20°C放置2小时,4°C,1,2000,30分钟70%乙醇洗涤沉淀,风干,溶于50µl DEPC-H2O中。
为使探针彻底溶解,可以在60°C 保温15分钟3、探针半定量:不同稀释倍数的探针和标准品点同样体积于尼龙膜,按照说明书进行显色。
原位杂交步骤整理

1.石蜡切片:每组一个蜡块,切片厚度5-6μm
2.脱蜡入水
3.DEPC水洗5min*2(1500ul/1500ml)
4.蛋白酶K消化,37℃,10-15min,DEPC水洗5min。
5.2×SSC平衡20min*1,DEPC 5min*2
6.42℃预杂交2h,切片上每个组织50μl,湿盒内(至少30min)(冰柜里面放的白色的液体就是预杂交液,标的H的)
7.沸水中变性探针,5min,立即冰浴(防止探针复性),放冰盒内(用预杂交液稀释)
8.稀释探针浓度为1-8ng/μl,(3ng/ul左右)切片上每个组织30μl,42℃过夜,湿盒内A:56ng/ul
9. 2×SSC洗两次,每次 5min
10.0.5×SSC洗两次,每次15min
11.马来酸洗一次,5min。
12.封闭液(由TBS溶解粉剂至1%)孵育60min,不洗。
(用马来酸稀释封闭液1:9)100/900(冰柜黄色液体)
13.用封闭液稀释生物素化抗地高辛抗体:混匀后50μL/片加至标本片上。
置样品于湿盒中,37℃反应120分钟。
0.5/500(上面稀释好的)
14.马来酸洗10分钟×3次。
15.detection buffer 5min*1
16.nbip 显色1ml加20μl(用detection buffer 稀释)时间3个小时以上。
(避光)
17.苏木素轻度复染。
蒸馏水洗。
脱水,透明,封片。
原位杂交介绍与步骤

原位杂交介绍与步骤原位杂交组织(或细胞)化学(In situ Hybridization Histochemistry,ISHH)简称原位杂交(In Situ Hybridization),属于固相分子杂交的范畴,它是用标记的DNA或RNA为探针,在原位检测组织细胞内特定核酸序列的方法。
根据所用探针和靶核酸的不同,原位杂交可分为DNA-DNA 杂交,DNA-RNA杂交和RNA-RNA杂交三类。
根据探针的标记物是否直接被检测,原位杂交又可分为直接法和间接法两类。
直接法主要用放射性同位素、荧光及某些酶标记的探针与靶核酸进行杂交,杂交后分别通过放射自显影、荧光显微镜术或成色酶促反应直接显示。
间接法一般用半抗原标记探针,最后通过免疫组织化学法对半抗原定位,间接地显示探针与靶核酸形成的杂交体。
原位杂交最初是以同位素标记探针进行的。
尽管同位素标记(如35S,3H,32P等)仍然广泛使用,但非同位素标记探针的迅速发展(尤其是生物素标记探针和地高辛标记探针),更引起科技工作者的极大兴趣。
一、基本要求1. 组织取材:组织取材应尽可能新鲜。
由于组织RNA降解较快,所以新鲜组织和培养细胞最好在30 min 内固定。
2. 固定目的是:(1)保持细胞结构;(2)最大限度地保持细胞内DNA或RNA的水平;(3)使探针易于进入细胞或组织。
最常用的固定剂是多聚甲醛,与其它醛类固定剂(如戊二醛)不同,多聚甲醛不会与蛋白质产生广泛的交叉连接,因而不会影响探针穿透入细胞或组织。
3. 增强组织的通透性和核酸探针的穿透性:(1)稀酸处理和酸酐处理:为防止探针与组织中碱性蛋白之间的静电结合,以降低背景,杂交前标本可用0.25%乙酸酐处理10 min,经乙酸酐处理后,组织蛋白中的碱性基团通过乙酰化而被阻断。
组织和细胞标本亦可用0.2 M HCl处理10 min,稀酸能使碱性蛋白变性,结合蛋白酶消化,容易将碱性蛋白移除。
(2)去污剂处理:去污剂处理的目的是增加组织的通透性,利于杂交探针进入组织细胞,最常应用的去污剂是Triton X-100。
原位杂交技术(单独一章)

第十三章原位杂交技术原位杂交(in situ hybridization,ISH)是分子生物学、组织化学和细胞学相结合的产物。
该技术是根据核酸碱基互补配对原则,将放射性或非放射性标记的外源核酸(探针)与染色体经过变性的单链DNA互补配对,结合成专一的核酸杂交分子。
经过一定的检测手段,可利用显微镜直接观察到所研究的目的序列在细胞或染色体上的位置和分布。
为宏观的细胞学与微观的分子生物学研究架起了一座桥梁,形成了一门新的交叉学科――分子细胞遗传学(molecular cytogenetics)。
原位杂交技术是由Gall 和Pardue利用同位素标记的rDNA探针与非洲爪蟾细胞核杂交,于60年代末首次获得成功。
第一节原位杂交基本程序原位杂交的基本程序包括5个基本步骤:第一步是制作用来进行原位杂交的染色体制片,方法与普通染色体制片完全相同。
材料可以是体细胞(如根尖细胞),也可以是花粉母细胞。
以3:1甲醇/乙醇:冰醋酸固定材料,而后做直接压片法或酶解火焰干燥法制片,选择具有理想的细胞分裂时期的片子以备后用。
第二步是对染色体DNA进行变性(denaturation)处理。
先把盖玻片揭开,用RNA酶处理制片,消化掉染色体上的内源RNA,而后用碱、酸或甲酰胺或高温处理,使DNA变性,成为单链状态。
第三步进行杂交。
用以进行杂交的DNA或RNA分子探针通常利用切刻平移法或随机引物标记法标记上放射性的3H、125I或非放射性的生物素(biotin)、地高辛(digoxingenin)。
目前常用的是,把非放射性探针、甲酰胺、硫酸葡聚糖、鲑鱼精DNA(ssDNA)、磷酸缓冲液(SSC)配成杂交液,置于制片上,盖上盖玻片,在37℃右的温箱中保温过夜,使探针同与其碱基序列具有互补性的单链DNA分子进行充分杂交,形成双链。
而后打开盖片,冲洗掉多余未杂交的探针。
第四步信号检出和对染色体进行染色。
对于使用放射性标记探针的,先将制片浸入一定浓度的感光乳剂溶液,并缓慢取出,使制片表面涂上一层均匀的感光乳剂,而后把制片放入暗箱,进行2-3天的感光,接着进行显影和定影。
荧光原位杂交实验具体步骤及详细说明

荧光原位杂交实验具体步骤及详细说明用已知的标记单链核酸为探针,按照碱基互补的原则,与待检材料中未知的单链核酸进行异性结合,形成可被检测的杂交双链核酸。
由于DNA分子在染色体上是沿着染色体纵轴呈线性排列,因而可以探针直接与染色体进行杂交从而将特定的基因在染色体上定位。
1. 探针变性将探针在75℃恒温水浴中温育5 min,立即置0℃,5~10 min,使双链DNA探针变性。
2. 标本变性(1)将制备好的染色体玻片标本于50℃培养箱中烤片2~3 h。
(经Giemsa 染色的标本需预先在固定液中退色后再烤片)。
(2)取出玻片标本,将其浸在70~75℃的体积分数70% 甲酰胺/2x SSC的变性液中变性2~3 min。
(3)立即按顺序将标本经体积分数70%、体积分数90%和体积分数100%冰乙醇系列脱水,每次5 min,然后空气干燥。
3. 杂交将已变性或预退火的DNA探针10 uL 滴于已变性并脱水的玻片标本上,盖上18x18盖玻片,用Parafilm封片,置于潮湿暗盒中37℃杂交过夜(约15~17 h)。
由于杂交液较少,而且杂交温度较高,持续时间又长,因此为了保持标本的湿润状态,此过程在湿盒中进行。
4. 洗脱此步骤有助于除去非特异性结合的探针,从而降低本底。
(1)杂交次日,将标本从37℃温箱中取出,用刀片轻轻将盖玻片揭掉。
(2)将已杂交的玻片标本放置于已预热42~50℃的体积分数50%甲酰胺/2xSSC中洗涤3次,每次5 min。
(3)在已预热42~50℃的1xSSC中洗涤3次,每次5 min。
(4)在室温下,将玻片标本于2xSSC中轻洗一下。
(5)取出玻片,自然干燥。
(6)取200 uL复染溶液(PI/antifade或DAPI/antifade染液)滴加在玻片标本上,盖上盖玻片。
5. 杂交信号的放大(适用于使用生物素标记的探针)(1)在玻片的杂交部位加150 uL封闭液I,用保鲜膜覆盖,37℃温育20min。
原位杂交步骤自己整理的精讲

原位杂交步骤自己整理的精讲荧光原位杂交试剂及其配制方法(以下试剂均需用RNase-free水配制):1×PBS:NaCl 8g∕L、KCl g∕L、Na2HPO4 g∕L、KH2PO4 g∕L,溶于DEPC处理的去离子水中,用pH计测其pH,再用NaOH调其pH为;PBST:PBS加%吐温-20;LiCI:配4M LiCI 100ml需要LiCI.H20 ,配好后加入千分之一的DEPC,37℃,12h放置,高温灭菌。
4%多聚甲醛(4%PFA):准确称取40g多聚甲醛,溶于1000ml的1×PBS中,避光于摇床(37℃,160r∕m)上,待其全部溶解后,用棕色瓶分装成小体积包装,保存于-20℃。
(注:本方法与其他一些文献用NaOH和HCl先后调节pH而促其溶解的方法略有不同,因是考虑到pH的稳定性;另外之所以采用上述方法使多聚甲醛缓慢溶解而没有采用文献中提及的用NaOH调节其pH至10促其溶解后再用HCl调节pH的原因,除了用一般的pH试纸测量配置好的4%多聚甲醛的pH不够准确外,也因为不能用pH计对其酸碱度进行测量,因为它会损坏pH 计)。
取以上各成分,溶于DEPC处理过一定量的纯水(或双蒸水)中,测定其pH(放置一段时间待其pH稳定后),据此以HCl或NaOH来调节其pH值至,定容至所要求的体积。
(注:根据实际情况,因DEPC处理过的水其pH会下降,故本实验中实际上是用1mol∕L的NaOH来调节其pH)。
75%甲醇/PBST:将无水甲醇按75%(V/V)比例溶于PBST;50%甲醇/PBST:将无水甲醇按50%(V/V)比例溶于PBST;25%甲醇/PBST:将无水甲醇按25%(V/V)比例溶于PBST;【HYB-溶液:50%甲酰胺(V/V)、5×SSC(终浓度)、%吐温-20(终浓度)、RNase-free水(-20℃)HYB+溶液:HYB-溶液、500ug/ml酵母tRNA、50ug/ml (终浓度)肝素,(-20℃);50%甲酰胺/2×SSCT:50%甲酰胺(V/V),2×SSCT(终浓度);2×SSCT:2×SSCT(终浓度),%吐温-20;MAB马来酸缓冲液(Maleic acid buffer):100mM maleic acid,150mM NaCl,pH (20℃),溶于双蒸水,用浓缩或固体的NaOH调节pH为,并过滤除菌(sterile)MABT:MAB,使用前加入%吐温-20;10×Blocking reagent(阻断剂):将阻断试剂(Blocking reagent)溶于马来酸缓冲液中,摇晃并在加热器或微波炉上加热,使其终浓度为10%(w/v);此储存液(原液)需高温除菌(is autoclaved)并分装后保存于-20℃。
荧光原位杂交操作步骤

荧光原位杂交操作步骤荧光原位杂交听起来很复杂呢,其实操作步骤也有章可循啦。
一、样本准备。
要做这个实验,先得把样本处理好。
如果是细胞样本的话,就得把细胞乖乖地固定在载玻片上,就像把小娃娃放在小床上一样安稳。
这一步可不能马虎,固定不好后面就不好办啦。
要是组织样本呢,得把组织切成薄片,薄到像纸片一样精致才行。
然后同样把它固定好,这就为后面的杂交打下了好基础。
二、探针准备。
接下来就是探针啦。
探针就像是专门去找目标的小侦探。
得按照实验要求把探针配好,浓度要刚刚好哦,太浓了可能会乱了阵脚,太稀了又可能找不到目标。
把探针在合适的缓冲液里调配好,就像给小侦探准备好装备一样。
三、杂交反应。
然后就到了杂交这一步啦。
把准备好的探针滴到有样本的载玻片上,再盖上盖玻片。
这时候就像把小侦探放到了有目标的地方,让它们去寻找匹配的东西。
然后把载玻片放到一个合适温度的环境里,这个温度很关键呢,就像给小侦探们创造一个舒适的工作环境。
在这个环境里让探针和样本中的目标DNA或者RNA去结合,这个过程需要一点时间,就像小侦探寻找目标也不能太着急呀。
四、洗涤。
杂交完了之后呢,就要把那些没有结合的多余探针洗掉。
这就像是把那些凑热闹的家伙赶走,只留下真正找到目标的小侦探。
用合适的洗涤液轻轻地洗,可不能太粗暴,不然把已经结合好的也冲走了就糟糕啦。
五、检测。
最后就是检测啦。
这时候要用专门的荧光显微镜去看。
打开显微镜,就像打开一个神秘的宝藏盒子。
如果杂交成功了,就能看到漂亮的荧光信号啦,就像在黑暗中看到了闪闪发光的小星星。
那些荧光信号所在的地方就是探针找到目标的地方呢。
荧光原位杂交虽然有点复杂,但是按照这些步骤一步一步来,就像照顾小宠物一样细心,也能顺利完成这个有趣的实验啦。
一种在g-显带的玻片上进行分子荧光原位杂交处理方法

一种在g-显带的玻片上进行分子荧光原位杂
交处理方法
分子荧光原位杂交技术(FISH)是一种重要的生物学技术,它能够检测和定位细胞核中的特定DNA序列。
在g-显带玻片上进行分子荧光原位杂交处理,可以用于研究染色体结构和基因组变异。
以下是在g-显带玻片上进行分子荧光原位杂交处理的基本步骤:
1.预处理玻片:在开始实验前,需要将玻片进行预处理,包括清洗、脱蜡、消化等步骤,以去除玻片表面的杂质和固定细胞。
2.制备探针:选择与目标DNA序列互补的DNA或RNA片段作为探针,将其标记上荧光染料或其他可检测的标记物。
3.杂交反应:将制备好的探针与玻片上的染色体进行杂交,使探针与目标DNA序列结合。
这一步需要控制杂交温度和时间,以确保探针与目标DNA的有效结合。
4.洗涤:杂交反应后,需要将未结合的探针洗涤掉,以避免背景干扰。
这一步需要严格控制洗涤条件,如洗涤液的成分、温度和时间等。
5.检测与分析:最后,使用荧光显微镜或其他检测设备对染色体的荧光信号进行检测和分析。
通过观察荧光信号的位置和强度,可以推断出染色体结构和基因组变异的情况。
总之,在g-显带玻片上进行分子荧光原位杂交处理需要严格控制实验条件和处理步骤,以确保结果的准确性和可靠性。
原位杂交实验操作步骤

原位杂交实验操作步骤一质粒制备1质粒的转化和扩增1.1制备XL1-Blue感受态细菌1.取400uLXL1-Blue菌种加入到含200mlLB培养基的锥形瓶中,37℃、100rpm 培养4h,离心,倒置,以冰冷的0.1mol/LCaCl_2重悬细菌,冰浴30min,离心,弃上清,倒置,再加4ml(含15%甘油)冰冷的CaCl2重悬细菌,分装(200μ/tube),-80℃保存。
2.转化:在冰浴中将1管XL1-Blue感受态菌解冻,将浓度为2ng/μ1的质粒DNA4μ1加入到8Oμ1感受态菌中。
3.轻轻摇匀,冰浴30min。
4.42℃热激9O秒,然后迅速冰浴2min。
5.加入LB培养液(无氨苄青霉素)0.8ml,在37℃,100转/min水浴孵育60min。
6.取200μl菌液铺于琼脂板上(涂有X-Gal(20mg/ml)-IPTG(200mg/ml)的LB-氨苄青霉素50mg/ml,1μl/ml培养基),待菌液全部被吸收后,倒置平板于37℃培养12-16h。
1.2鉴定和挑选含重组质粒的菌落1.用无菌牙签挑取单菌落,接种到10ml含氨苄青霉素的LB培养液的离心管中,于37℃,200转/分培养2h,取1ml之一Eppendorf离心管,加50μl10mmol/L EDTA(pH8.0)。
2.加入50μl新配置的0.2mol/LNaOH、0.5%SDS、20%蔗糖溶液后,振荡30秒。
3.在70℃温育5min,然后冷却到室温。
4.加1.5μ14mol/LKCl和0.5μ1含0.4%溴酚兰染液,振荡3O秒后,冰浴5min。
5.12000g,4℃离以3min,以除去细菌碎片。
6.制备1%的琼脂糖凝胶(含EB0.5μg/ml),取50μl上清液加入到样品孔中,其中一孔加入中等分子量DNAMarker。
恒压50V,进行电泳。
7.当溴酚兰迁移到凝胶全长的2/3-3/4时,停止电泳,在紫外灯下检查质粒DNA 分于量的大小是否与转入质粒相符。
原位荧光杂交操作规程(3篇)

第1篇一、目的原位荧光杂交技术是一种在细胞或组织切片上进行DNA或RNA定位的方法,通过对特定序列的DNA或RNA进行标记,从而实现对特定基因或染色体的定位和定量分析。
本规程旨在规范原位荧光杂交操作,确保实验结果的准确性和可靠性。
二、适用范围本规程适用于所有原位荧光杂交实验,包括细胞原位杂交、组织切片原位杂交等。
三、材料与设备1. 材料:- 标本:细胞或组织切片- 探针:标记有荧光染料的DNA或RNA探针- 酶标抗体:针对探针标记的荧光染料- 洗涤液:如磷酸盐缓冲盐溶液(PBS)- 抗荧光抗体:用于检测酶标抗体- 封片剂:如甘油封片剂2. 设备:- 荧光显微镜- 原位杂交仪- 热循环仪- 高速离心机- 显微镜载物台- 离心管- 移液器四、操作步骤1. 标本制备:- 将细胞或组织切片固定在载玻片上,进行脱水和透明处理。
- 将载玻片放入烘箱中,进行烘烤,使切片紧密贴合在载玻片上。
2. 探针制备:- 将探针进行变性,使其单链化。
- 将变性后的探针与标记有荧光染料的探针混合,进行杂交反应。
3. 杂交反应:- 将杂交混合物滴加在载玻片上的标本上,用封片剂封口。
- 将载玻片放入原位杂交仪中,进行杂交反应。
4. 洗涤:- 将杂交后的载玻片放入洗涤液中,进行洗涤。
- 洗涤过程中,注意轻柔操作,避免破坏杂交结构。
5. 酶标抗体反应:- 将酶标抗体滴加在载玻片上的杂交结构上,进行抗体结合反应。
- 将载玻片放入原位杂交仪中,进行抗体结合反应。
6. 洗涤:- 将抗体结合后的载玻片放入洗涤液中,进行洗涤。
7. 抗荧光抗体反应:- 将抗荧光抗体滴加在载玻片上的杂交结构上,进行荧光抗体结合反应。
- 将载玻片放入原位杂交仪中,进行荧光抗体结合反应。
8. 洗涤:- 将荧光抗体结合后的载玻片放入洗涤液中,进行洗涤。
9. 封片:- 将封片剂滴加在载玻片上的杂交结构上,进行封片。
10. 观察与分析:- 将载玻片放入荧光显微镜中,进行观察和分析。
原位杂交技术的原理及应用

原位杂交技术的原理及应用1. 原位杂交技术的概述原位杂交技术是一种用于研究基因组结构和功能的重要工具。
它基于两个主要原理:互补配对和探针标记。
通过互补配对,可以使DNA探针与目标DNA的特定序列发生结合。
探针通常被标记为荧光染料或放射性同位素,以便检测和定位。
2. 原位杂交技术的工作原理原位杂交技术的工作原理可以分为以下几个步骤:2.1 产生探针首先,需要产生特定序列的DNA探针。
这可以通过PCR扩增、限制性内切酶消化和合成等方法来实现。
探针的选择应根据研究需求和目标DNA的序列来确定。
2.2 标记探针为了使探针可视化和定位,需要对其进行标记。
常用的标记方法包括荧光标记和放射性同位素标记。
荧光标记通过使用特定的荧光染料,使探针在显微镜下可见。
放射性同位素标记则通过使用放射性同位素来标记探针,然后通过放射性计数器来检测和定位。
2.3 杂交反应将探针与目标DNA进行杂交,使它们发生互补配对。
杂交条件的选择取决于目标DNA的性质和探针的序列。
通常,在一定温度和盐浓度下,探针与目标DNA可以形成稳定的双链结构。
2.4 洗涤和检测完成杂交后,需要将非特异性的探针从样品中洗涤掉,以减少干扰和背景信号。
然后,使用显微镜观察或放射性计数器检测探针的信号。
荧光标记的探针可以通过荧光显微镜观察到特定位置的信号强度和分布情况,而放射性同位素标记的探针可以通过放射性计数器测量到辐射信号的强度。
3. 原位杂交技术的应用原位杂交技术在生物学研究中有广泛的应用,主要包括以下几个方面:3.1 基因定位和染色体映射原位杂交技术可以用于确定基因在染色体上的位置并进行染色体映射。
通过将特定序列的探针与目标DNA进行杂交,可以确定基因在染色体上的位置和分布。
这对于基因组研究、疾病基因的定位以及基因组结构和功能的理解都具有重要意义。
3.2 突变检测和基因表达分析原位杂交技术可以用于检测基因突变并分析基因的表达模式。
通过使用特定的突变探针或转录探针,可以检测到特定基因的突变或表达模式。
荧光原位杂交实验方法及步骤

荧光原位杂交实验⽅法及步骤2.38.1 实验⽅法及步骤(1) 探针变性将探针在75℃恒温⽔浴中温育5min,⽴即置0℃,5~10min,使双链DNA探针变性。
(2) 标本变性a.将制备好的染⾊体玻⽚标本于50℃培养箱中烤⽚2~3h。
(经Giemsa染⾊的标本需预先在固定液中退⾊后再烤⽚)。
b.取出玻⽚标本,将其浸在70~75℃的体积分数70%甲酰胺/2×SSC的变性液中变性2~3min。
c.⽴即按顺序将标本经体积分数70%、体积分数90%和体积分数100%冰⼄醇系列脱⽔,每次5min,然后空⽓⼲燥。
(3) 杂交将已变性或预退⽕的DNA探针10µL 滴于已变性并脱⽔的玻⽚标本上,盖上18×18盖玻⽚,⽤Parafilm封⽚,置于潮湿暗盒中37℃杂交过夜(约15~17h)。
由于杂交液较少,⽽且杂交温度较⾼,持续时间⼜长,因此为了保持标本的湿润状态,此过程在湿盒中进⾏。
(4) 洗脱此步骤有助于除去⾮特异性结合的探针,从⽽降低本底。
a.杂交次⽇,将标本从37℃温箱中取出,⽤⼑⽚轻轻将盖玻⽚揭掉。
b.将已杂交的玻⽚标本放置于已预热42~50℃的体积分数50%甲酰胺/2×SSC中洗涤3次,每次5min。
c.在已预热42~50℃的1×SSC中洗涤3次,每次5min。
d.在室温下,将玻⽚标本于2×SSC中轻洗⼀下。
e.取出玻⽚,⾃然⼲燥。
f.取200µL复染溶液(PI/antifade或DAPI/antifade染液)滴加在玻⽚标本上,盖上盖玻⽚。
(5) 杂交信号的放⼤(适⽤于使⽤⽣物素标记的探针)a.在玻⽚的杂交部位加150µL封闭液I,⽤保鲜膜覆盖,37℃温育20min。
b.去掉保鲜膜,再加150µL avidin-FITC于标本上,⽤保鲜膜覆盖,37℃继续温育40min。
c.取出标本,将其放⼊已预热42~50℃的洗脱液中洗涤3次,每次5min。
原位杂交操作过程

(一)、仪器设备医用微波炉;水浴锅。
(二)、试剂1)95%乙醇,DEPC水配制2)70%乙醇,DEPC水配制3)0.1M甘氨酸glycine(用0.1M PBS配制)4)0.3%TritonX-100(用0.1M PBS配制)5)TE缓冲液配制:0.1M Tria-HCl,0.05M EDTA,pH8.0,DEPC水配制6)蛋白酶K (5-20 ug/ml),(用PBS缓冲液配制)7)4% Paraformaldehyde(PFA)(用0.1M PBS配制)8)0.1mol/L Triethanolamine TEA (pH8.0): Triethanolamine 5.33ml,DEPC水定容0.4L。
9)醋酸酐溶液: 0.1mol/L Triethanolamine中醋酸酐(用前加至浓度0.25M)。
10)杂交缓冲液:50% deionized formamide, sterile 4X SSC(DEPC 水配制)11)杂交液:50% deionized formamide, 100 μg/ml heparin, 5X SSC, 0.1% Tween20, 5 mM EDTA, 1X Denhardt's, 1 mg/ml total RNA; stored frozen in 50 ml aliquots.12)20×SSC(pH7.0):20X SSC [175 g NaCl, 88.5 g trisodium citrate, DEPC-H2O and 200 ul 10M HCl to 1L, pH 7.4 (DEPC treated and autoclaved)]. Store frozen in 40 ml aliquots. All reagents are purchased from Sigma.13)10mg/ml RNA: 让RNase溶于NTE缓冲液。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
1.For each probe (control and experimental), set up a separate 100-ml PCR in a 0.5-ml sterile tube, as tabulated below. Either.cDNA inserted in plasmids or genomic DNA can be used as templates for the PCR (see REAGENT SETUP for details on primer design).每个探针(实验组和对照组),在0.5 -ml无菌管设立一个独立的100毫升PCR,正如下面的表。
另外。
互补脱氧核糖核酸插入到基因组DNA质体或可用作模板PCR(见试剂设置有关底漆设计)。
**注意关键:1.很多版本的实验反义核酸探针可以作为一种控制背景染色(见试剂设置)。
然而,我们相信最好的方法来演示特异性是获取相同的空间限制表达模式使用不同的非重叠探测器相同的基因。
2.小心不要污染pcr.使用无菌试管和过滤器的技巧和戴手套3.另外,PCR扩增,cDNAs质粒中可以使用约束线性化酶,独特的站点位于5¢(反义核酸探针)或3¢(对感官探测)来插入。
净化的线性DNA可以通过苯酚/氯仿萃取乙醇沉淀紧随其后。
2| Run the PCR using the conditions tabulated below.使用下面列出的条件运行PCR**暂停点:把扩增好的pcr产品放4℃降温和在-20℃贮藏几个星期。
3| Add the 100-ml PCR to a Microcon YM-50 column and add 400 ml of sterile water. Centrifuge for 15–20 min at 1,000 g at room temperature.加入100毫升PCR到Microcon YM-50列并加入400毫升的无菌水。
在室温下1000g离心15 - 20分钟。
**注意关键:膜应该是干的。
如果没有再离心4|Place the Microcon column into a new microfuge tube (provided in the kit), add 20ml of sterile water, vortex briefly and then turn the Microcon column upside down. Spin for 1 min at 1,000 g at room temperature to recover the DNA.把小层析柱放在一个新的离心管(在这个工具包中提供),增加20毫升的无菌水、短暂离心,然后颠倒层析柱。
自旋1分钟1000 g在室温下恢复了DNA**注意关键:离心的步骤应该快速。
离心机1分钟只是为了避免样本太干。
5|Check the quality, quantity and size of the PCR amplification product by loading 1/20 of the preparation on a 1% (wt/vol) agarose gel in 1 TBE buffer. DNA should appear as a band and not as a smear. The 1/20 of the preparation should contain at least 40 ng of DNA.通过装载1/20的稀释液在1*的TBE buffer缓冲液中的1% (wt/vol)的琼脂糖凝胶检查PCR的扩增产物的质量数量和大小。
DNA产物应该以一个条带而不是以一块污点表现出来。
这份1/20的稀释液至少含有40 ng 的DNA。
6| For each probe, add the components tabulated below to a microfuge tube. Mix and incubate for 2 h at 37 1C.按下面表格给每一份在离心管中的谈增加成分,混合均匀后放在37℃下保存2h.7| Add 2 ml of RNase-free DNase I and 18 ml of sterile water. Mix and incubate for 30 min at 37 1C.添加2毫升的RNase-free DNase I和18毫升的无菌水。
在37℃下混合和孵化30分钟。
8| Stop the reaction by adding 1 ul of sterile 0.5 M EDTA and 9 ul of sterile water.停止反应,通过添加1微升的无菌0.5 M EDTA和9微升无菌水。
9| Place a Sigmaspin post-reaction purification column on top of a microfuge tube. Centrifuge for 15 s at 750g.放一个Sigmaspin反应纯化柱到小离心管,750g离心15 s。
10| Break the base of the column and discard the lid. Spin for 2 min at 750g.破坏柱子的基柱,丢弃盖子,750g 离心旋转2 min11| Place the column on a new microfuge tube. Add the RNA template on top of the resin. Centrifuge for 4 min at 750 g. Discard the column.把柱子放在一个新的离心管上,在树脂上加上RNA模板。
750 g 离心4 min,丢掉柱子。
12| Add 1 ml of sterile EDTA 0.5 M and 9 ml of RNAlater to the sample; this protects the RNA from degradation.加1 ml 无菌的EDTA 0.5 M 和9 ml RNAlater到体系里;这样可以防止RNA降解**注意:由地高辛标记的反义(或正义)的RNA 能在-20℃环境下保存数月。
这个探针合成提供了足够的反义(或正义)RNA执行至少40个左右的不同原杂反应。
13| Visualize 1/20 of the synthesized RNA on 1% (wt/vol)agarose gel in 1 TBE buffer after 30 min of electrophoresis at 230 V. A good probe should appear as one or two discretebands on the gel, not as a smear—which would indicate degradation.设想1/20的合成RNA稀释液在1*的TBE buffer缓冲液中的1% (wt/vol)的琼脂糖凝胶在230 V下经过30 min 的电泳后,好的探针应该以一到两条条带在胶上,而不是一块污点,用来显示降解。
14| Set up pairs of male and female fish in breeding tanks the night before eggs are to be collected. Separate the male and female with a diagonal plastic divider.在鱼卵收集前的晚上建立一个饲养雄鱼和雌鱼的缸,用一个塑料对角分隔器将雄鱼与雌鱼分开饲养。
15| The next morning, when the light goes on, place the upper part of the breeding tank in a clean lower part filled with fresh water. Remove the divider of the breeding tank and let the fish mate for 10–20 min. The eggs laid will sink to the bottom of the breeding tank.第二天天亮后,把饲养缸上半部分移到一个新鲜的淡水体里。
移动分离器,让鱼交配10–20 min。
鱼卵就会沉到水底。
**注意:成人斑马鱼在野外交配时通常在广下。
在养鱼设施中,绝大多数的鱼产卵5 min 到1 h后把灯打开。
可以在上午晚些时候可以获得额外的蛋,但它们通常质量低劣。
16| Collect the eggs from the bottom tank by pouring them into Petri dishes.从水缸里收集沉在水底的鱼卵,放至培养皿里。
**注意:避免一个培养皿中放太多的鱼卵17|Clean the clutch under a stereomicroscope with a diascopic stand; discard dirt and unfertilized eggs using a Pasteur capillary pipette (with a large opening) and pipette pump filler.清理收集鱼卵的培养皿,用消毒过的移液器(有大口)吸掉坏的鱼卵或未受精的鱼卵。
18| Place the embryos in a 100-ml beaker, covered with a minimal amount of water (10 ml).把有用的胚胎放到一个100-ml大烧杯,烧杯里加合适的最少量水10-ml。
19| Pour 3 ml of pronase (1/100 wt/vol, warmed to 28.5 1C) into the beaker and incubate for 1 min. Gentle pronase treatment progressively softens the chorion without damaging the embryos. Alternatively, embryos can be dechorionated by hand using sharpened forceps, but this is a slow process, which can be used only for a small quantity of embryos.倒3毫升链霉蛋白酶(1/100 wt/vol, warmed to 28.5 1C)放入烧杯,孵化1分钟。
