pEASY-Blunt Cloning Kit使用说明书
巴西橡胶树HbPSKR2_基因克隆及互作蛋白鉴定

热带作物学报2024, 45(4): 653 662Chinese Journal of Tropical Crops巴西橡胶树HbPSKR2基因克隆及互作蛋白鉴定杜晓愚1,2,赵一杰3,张世鑫2,田维敏2,晁金泉2*1. 浙江农林大学现代农学院,浙江杭州 311300;2. 中国热带农业科学院橡胶研究所/热带作物生物育种全国重点实验室/农业农村部橡胶树生物学与遗传资源利用重点实验室/省部共建国家重点实验室培育基地-海南省热带作物栽培生理学重点实验室,海南海口 571101;3. 海南大学热带作物学院,海南海口 570228摘要:磺肽素是一种高等植物特有的小肽类激素,广泛参与植物的生长发育、分裂分化、生物或非生物胁迫等生物学过程。
磺肽素受体蛋白(phytosulfokine receptor, PSKR)是磺肽素的直接受体,对于磺肽素信号传导至关重要。
本研究采用RT-PCR(reverse transcription-polymerase chain reaction)技术克隆了橡胶树的HbPSKR2基因,并对其进行生物信息学、基因表达模式、互作蛋白筛选及鉴定分析。
结果显示HbPSKR2基因的开放阅读框全长3159 bp,编码1052个氨基酸,理论分子量为114.84 kDa,理论等电点为6.34。
结构域分析显示HbPSKR2属于典型的跨膜蛋白,前640个氨基酸是由亮氨酸重复组成的天线结构,第692~714个氨基酸是跨膜结构域,第765~1052个氨基酸是膜内激酶结构域。
对HbPSKR2膜内激酶结构域以及拟南芥和水稻的PSKR同源序列进行多重比对,结果显示均存在ATP binding site、CaM binding site、Activation segment、GC Centre等保守性位点。
表达模式分析显示,HbPSKR2基因在橡胶树形成层区高丰度表达,其表达量在冠菌素处理前期显著上升。
通过酵母双杂交技术筛选到12个与HbPSKR2互作的候选蛋白,并对其中的2个蛋白激酶(HbPBL8和HbPIX13)与HbPSKR2的互作关系进行荧光素酶互补成像验证。
M5 pBLUE-T Simple Cloning Kit 使用说明书
M5pBLUE-T Simple Cloning Kit使用说明书产品名称单位货号M5pBLUE-T Simple Cloning Kit20T MF134-01M5pBLUE-T Simple Cloning Kit4×20T MF134-04【储存条件】长期保存,请置于-20˚C,有效期6个月。
使用后请及时放入-20˚C保存以保证酶的活性。
【产品简介】pBLUE-T Simple Vector是一种高效克隆PCR产物(TA Cloning)的专用载体。
这种载体是由pBlueScript II SK(+)质粒改建而成,和传统T载体ECOR V切开后加T方法不同,pSURE-T Simple通过改造pBlueScript II SK(+),在原pBlueScript II SK(+)多克隆位点引入Xcm I酶切后使其原多克隆位点两侧的3’末端直接产生未配对的T碱基,因此有更高的重组效率。
同时它消除了pBlueScript II SK(+)载体上的多克隆酶切位点,需要在PCR扩增引物上导入合适的酶切位点。
此时如果使用PCR扩增引物导入的酶切位点进行DNA酶切时,酶切反应将不会受到T载体上其它多克隆酶切位点上的限制酶影响,可以大大提高酶切效率,增加亚克隆成功率。
此外,本公司载体连接体系还有背景低(蓝斑小于10%),重组率高(白斑中超过90%有插入片段),可快速连接等特点。
本说明书末列出了pBLUE-T Simple载体相关的技术资料,其全序列可参照pBlueScript II SK(+)序列,只是其多克隆酶切位点处序列稍有不同。
测序推荐采用M13通用测序引物和T3启动子引物(见后面图谱)。
【产品组份】20T4×20TpBLUE-T Simple Vector(30ng/µl)20µl80µl1000bp Control(30ng/µl)5µl5µl10x PEG Enhancer50µl200µl10x Ligation Buffer40µl160µl2x Quick Ligation Buffer100µl400µlT4DNA Ligase,5U/ul20µl80µl【操作步骤】1.连接反应的准备:PCR产物是否要进行纯化取决于扩增产物的质量。
实验总结-3’race

应用3'Full-RACE技术克隆PCDHB1基因3'非翻译区廖柔霞:汕头大学医学院2008级本科一班指导教师:许丽艳,方王楷,黄俏,吴健谊,吴炳礼,李恩民【摘要】目的:克隆PCDHB1基因3'非翻译区,为深入研究该基因在食管癌等肿瘤或内分泌性疾病中的功能提供基本实验材料。
方法:3'Full-RACE技术。
结果:未能成功获得PCDHB1基因3'非翻译区克隆。
结论:1)提示PCDHB1基因的结构组织形式可能十分复杂;2)今后,拟在具体方法学上改进,争取获得该基因3'非翻译区克隆。
PCDHB1(别名PCDH-BETA1),位于5q31。
该基因共含有2534个碱基。
这个基因是原钙黏连蛋白基因簇的一员,是5号染色体上的三个串连连接的成员之一。
该基因组显示了与B的细胞和T细胞受体基因簇的一个不寻常的基因相似。
其具体职能是未知的,但它很有可能在神经细胞间的连接和建立中发挥关键的作用。
β钙黏连蛋白在特定的细胞粘连的事件扮演一个重要的角色。
见诸于人类恶性肿瘤中的表达和调控功能,在很多情况下,导致肿瘤细胞浸润和转移的加剧。
由于该基因的3'端还未知,所以必须用3'Full RACE的技术将PCDHB1的3'端测出来。
对此实验设计如下:应用3’RACE试剂盒及设计好的基因特异性引物扩增得到的基因片段,并将其连接到pEASYTM-Blunt Simple coloning vector 并测序。
该基因片段由于出现“双峰”现象导致测序失败。
关键词:基因克隆,PCDHB1基因,3'非翻译区,3'Full-RACEThe clone of 3' untranslational region of PCDHB1 geneusing 3'Full-RACERX Liao: 2008 Undergraduates of Medical College of Shantou UniversitySupervisor: LY Xu, WK Fang, Q Huang, JY Wu, BL Wu, EM LiAbtract:Objective MethodsResults ConclusionPCDHB1(PCDHB-BETA1),is located in 5q31. This gene is a member of the protocadherin beta gene cluster, one of three related gene clusters tandemly linked on chromosome five. The gene clusters demonstrate an unusual genomic or ganization similar to that of B-cell and T-cell receptor gene clusters. Their specific functions are unknown but they most likely play a critical role in theestablishment and function of specific cell-cell neural connections.It can be seen in many human’scancer and mostly, it makes the stuation more serious as a result.Fu rthermore, Becase this gene’s3’ UTR is unknown until now,so we should make it out whit 3’ Full RACE technique. Therefore,the next tests were designed:we used the 3’ Full RACE Kit and the specical primer we foundbefore to get a gene fragment and pEASYTM-Blunt Simple coloning vector inserted into andsequenced.Alought this gene failed in sequence,it also tell us something important.Key Words: gene cloning, PCDHB1gene, 3'-nontranslational region, 3'Full-RACE人类的钙粘蛋白PCDHB1(protocadh erin β 1)主要在细胞间的相互识别连接中发挥功能,其编码基因定位于5q31。
pEASYT1载体

pEASY®-T1 Cloning Kit目录号:CT101试剂盒组成1.pEASY- T1 Cloning Vector (10 ng/μl)2.Control Template (5ng/μl), Control Primers (10 μM),3.M13F (10 μM),M13R (10 μM)4.Trans1-T1 Phage Resistant Chemically Competent Cell.保存Trans1-T1 Phage Resistant Chemically Competent cell 可于–70℃保存至少六个月, 其他组分-20℃保存至少六个月。
产品说明pEASY- T1 Cloning Kits适用于TA克隆.-快速克隆基因,仅需要5分钟反应时间。
-提供氨苄青霉素和卡那霉素两种筛选标记。
-对照插入片段克隆效率可达95% 以上。
-包含LacZ 基因,在含有IPTG,X-gal 的平板培养基上,可进行蓝白斑筛选。
工作原理pEASY- T1 Cloning Kits 以线性的形式提供:-3′端悬垂胸腺嘧啶(T)可用于TA克隆-拓扑异构酶与载体相互偶联(活化载体)Taq DNA聚合酶具有类似脱氧核糖核酸末端转移酶(TdT)的功能,可在新合成双链产物的3’端加上一个碱基。
尽管4种碱基均可被聚合到3’端,但Taq酶对dATP的聚合能力远高于其他3种dNTP.所以,在标准PCR条件下,PCR产物3’端这一非模板依赖的聚合碱基几乎总是A。
pEASY- T1 Cloning Kits在3’ 端提供一个悬垂的T 碱基,这样的结构使PCR片段可以高效的和载体进行连接。
牛痘拓扑异构酶I型可以与DNA双链的特定位置结合并切割特异位点5’-(C/T)CCTT-3’, 与3‘T的磷酸基团性成共价键,并切断其中一条DNA链,使DNA解链。
磷酸二酯键断裂释放的能量储存于3’磷酸与拓扑异构酶氨基酸残基(Tyr-274)形成的共价键中。
基于CRISPR

作物学报 ACTA AGRONOMICA SINICA 2024, 50(1): 138 148 / ISSN 0496-3490; CN 11-1809/S; CODEN TSHPA9 E-mail:***************本研究由山西省基础研究计划项目(20210302123409)和山西农业大学博士人才引进科研启动项目(2022BQ08)资助。
This study was supported by the Basic Research Program of Shanxi Province (20210302123409) and the Doctoral Talent Introduction Scientific Research Initiation Project of Shanxi Agricultural University (2022BQ08).*通信作者(Corresponding author): 上官小霞,E-mail:**************Received (收稿日期): 2023-03-01; Accepted (接受日期): 2023-05-24; Published online (网络出版日期): 2023-06-16. URL: https:///kcms2/detail/11.1809.S.20230615.1534.006.htmlThis is an open access article under the CC BY-NC-ND license (/licenses/by-nc-nd/4.0/).DOI: 10.3724/SP.J.1006.2024.34041基于CRISPR/Cas9的棉花GhbHLH71基因编辑突变体的分析上官小霞* 杨琴莉 李换丽山西农业大学棉花研究所, 山西运城 044000摘 要: 碱性/螺旋-环-螺旋(basic/helix-loop-helix, bHLH)转录因子在植物的生长发育、次生代谢、信号转导、逆境胁迫等方面起重要的调控作用。
植物基因转化与转基因植株鉴定
3. 目标基因 PCR 扩增 采用 PrimeSTARTM HS DNA Polymerase 高保真酶从玉米 cDNA 中扩增 ZmMKK4 全长编码序列(GenBank 序列号:NM_001158890.1)。 ZmMKK4 扩增引物:F-ZmMKK4,R-ZmMKK4。 25μL 反应体系为:5 μL 5× PrimeSTAR Buffer (Mg2+ Plus),2 μL dNTP Mixture (各 2.5 mM),1μL 上游引物(5μM),1μL 下游引物(5μM),1 μL cDNA,0.25 μL PrimeSTARTM HS DNA Polymerase,加 ddH20 至 25μL。PCR 扩增程序为 98C 98C 58C 72C 72C 1~3 min 10 s 15 s 1.5 min 10 min
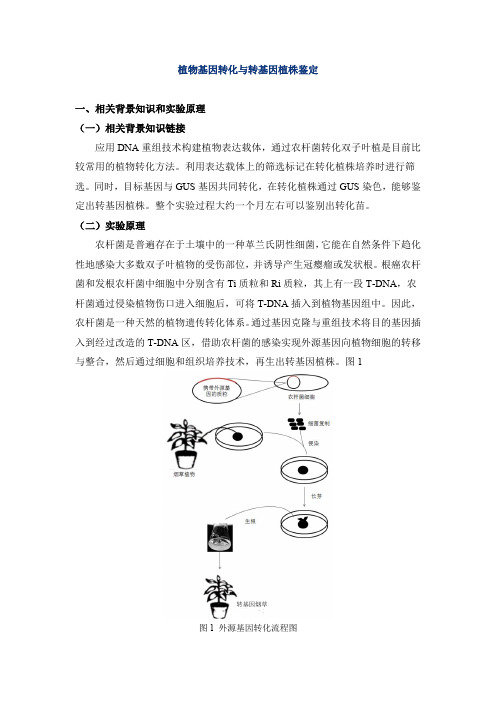
图 1 外源基因转化流程图
三、实验目的和实验要求 目的: 学习和掌握从基因分离克隆到植株转化及转基因植株的筛选整个实验 体系。 要求: 综合利用前面的单个实验环节所掌握的知识, 获得完整的实验理念, 并在实验过程中学会设计、构建适合的载体并将外源基因转化植株。 四、实验材料和方法 (一)实验仪器 PCR 仪, 凝胶成像系统, 台式高速离心机, 台式高速冷冻离心机, DU 640 紫 外分光光度计,UV-2450 分光光度计,精密 pH 计,微量移液器,控温摇床,电 子天平,制冰机,-80C 超低温冰箱,电热恒温水浴锅,自动电热压力蒸汽灭菌 器,超净工作台,光照培养箱等 (二)试剂及配方 Trizol试剂,RevertAid™ First Strand cDNA Synthesis Kit,DNase I,2× PCR Master Mix,FastDigest 限制性内切酶,T4 DNA Ligase,PrimeSTARTM HS DNA Polymerase 高保真酶, pEASY-Blunt C, 质粒提取试剂盒 (E.Z.N.A. Gel Extraction Kit) , 凝胶回收试剂盒 (E.Z.N.A. Plasmid Mini Kit I) , DNA提取试剂盒, Kanamycin (Kan) ,Ampicillin(Amp) ,Carbenicillin(Cb) ,电泳缓冲液,LB培养基、YEB 培养基,烟草培养基如下表1.
M5HiPerpTOPO-BluntCloningKit使用说明书

M5 HiPer pTOPO-BluntCloning Kit使用说明书产品名称单位货号M5 HiPer pTOPO-Blunt Cloning Kit 20T MF021-01M5 HiPer pTOPO-Blunt Cloning Kit 4x20T MF021-04【储存条件】长期保存,请置于-20˚C,有效期12个月。
使用后请及时放入-20˚C保存以保证酶的活性。
【产品简介】本制品和传统的T4连接酶原理不同,它利用了Topoisomerase可以在瞬间(几秒钟-几分钟)、高效(接近100%)连接DNA片段的原理采用本公司独创的工艺制成。
【产品特点】1. 可以在瞬间(几秒钟-几分钟)完成连接。
2. 无需冰浴和热休克,室温5分钟内完成转化;无需1小时复苏,只需37℃10分钟复苏便可以涂板。
从连接到涂板只需15-20分钟。
3. 无自连、零背景,无需繁琐蓝白斑筛选,见到长出的克隆便是有插入的(接近100%)。
4. 可以连接长达10kb片段。
5. 测序可以采用M13F/M13R通用引物测序(见后面图谱)【产品组份】20T 4x20TM5 HiPer pTOPO-Blunt Vector(30ng/µl) 20µl 80µl1kb Control(40ng/µl)5µl 5µl10x Enhancer 20µl 80µl【操作步骤】1. 连接反应的准备:<注意:PCR引物不能磷酸化>。
使用PCR扩增产物是平末端的高保真聚合酶扩增(如X5高保真酶,KOD酶,Phusion酶,Q5酶,Fastpfu酶,pfu酶)。
PCR产物(仅有目的条带、无非特异条带和引物二聚体)可直接进行连接反应,无需纯化;如果有非特异扩增,建议胶回收纯化(货号:MF029)。
如果是以质粒为模板的PCR产物,也必须进行切胶纯化,因为模板质粒也可能长出菌落(但不是想构建的目的载体)。
使用peasy-blunt simple cloning vector连接克隆载体原理

使用peasy-blunt simple cloning vector连接克隆载体原理1. 引言1.1 概述克隆载体是在分子生物学中常用的工具,用于将外源DNA序列插入到宿主细胞中进行研究。
peasy-blunt simple cloning vector是一种具有简易操作和高效性的克隆载体。
本文将介绍使用peasy-blunt simple cloning vector进行DNA 克隆的原理和方法步骤。
1.2 文章结构本文共分为五个部分。
首先,对peasy-blunt simple cloning vector进行详细介绍,包括其定义、功能、特点和优势以及应用领域。
接下来,在第三部分中,将概述DNA重组技术的基础知识,并解释克隆载体的作用和构建原理,以及peasy-blunt simple cloning vector在克隆过程中的应用。
第四部分将着重讲解使用peasy-blunt simple cloning vector进行克隆的具体方法步骤,包括实验前准备工作与材料准备、DNA放入克隆载体的方法与步骤详解,以及转化、筛选与验证克隆结果的步骤说明。
最后,在结论与展望部分,将总结实验结果并进行讨论,并提出存在问题和改进方向的展望。
1.3 目的本文旨在详细介绍使用peasy-blunt simple cloning vector进行克隆的原理和方法,以帮助读者全面了解这一克隆技术并能够正确应用于实验中。
通过本文的阅读,读者将能够掌握克隆载体原理、peasy-blunt simple cloning vector的特点和应用,并能够熟练操作克隆过程中的各项步骤。
2. peasy-blunt simple cloning vector简介:2.1 定义和功能:peasy-blunt simple cloning vector是一种常用的克隆载体,它被广泛应用于分子生物学研究中。
克隆载体是用来携带外源DNA片段并将其复制到宿主细胞中的DNA分子。
pEASY-Blunt clonning vector 载体说明书WORD版

pEASY-BluntComplete sequence AGCGCCCAATACGCAAACCGCCTCTCCCCGCGCGTTGGCCGA TTCATTAATGCAGCTGGCACGA CAGGTTTCCCGACTGGAAAGCGGGCAGTGAGCGCAACGCAATTAATGTGAGTTAGCTCACTCA TTAGGCACCCCAGGCTTTACACTTTATGCTTCCGGCTCGTATGTTGTGTGGAATTGTGAGCGGA T AACAATTTCACACAGGAAACAGCTATGACCATGA TTACGCCAAGCTTGGTACCGAGCTCGGATC CACTAGTAACGGCCGCCAGTGTGCTGGAATT GCCCTTAAGGGCAA TTCTGCAGATA TCCA TC ACACTGGCGGCCGCTCGAGCATGCA TCTAGAGGGCCCAA TTCGCCCTATAGTGAGTCGTA TTAC AA TTCACTGGCCGTCGTTTTACAACGTCGTGACTGGGAAAACCCTGGCGTTACCCAACTTAATC GCCTTGCAGCACATCCCCCTTTCGCCAGCTGGCGTAATAGCGAAGAGGCCCGCACCGATCGCCC TTCCCAACAGTTGCGCAGCCTGAATGGCGAATGGACGCGCCCTGTAGCGGCGCATTAAGCGCG GCGGGTGTGGTGGTTACGCGCAGCGTGACCGCTACACTTGCCAGCGCCCTAGCGCCCGCTCCT TTCGCTTTCTTCCCTTCCTTTCTCGCCACGTTCGCCGGCTTTCCCCGTCAAGCTCTAAA TCGGGG GCTCCCTTTAGGGTTCCGA TTTAGTGCTTTACGGCACCTCGACCCCAAAAAACTTGATTAGGGT GA TGGTTCACGTAGTGGGCCATCGCCCTGA TAGACGGTTTTTCGCCCTTTGACGTTGGAGTCCA CGTTCTTTAATAGTGGACTCTTGTTCCAAACTGGAACAACACTCAACCCTA TCTCGGTCTATTCT TTTGATTTA TAAGGGA TTTTGCCGA TTTCGGCCTATTGGTTAAAAAATGAGCTGATTTAACAAAA ATTTAACGCGAATTTTAACAAAATTCAGGGCGCAAGGGCTGCTAAAGGAAGCGGAACACGTAG AAAGCCAGTCCGCAGAAACGGTGCTGACCCCGGATGAA TGTCAGCTACTGGGCTA TCTGGACA AGGGAAAACGCAAGCGCAAAGAGAAAGCAGGTAGCTTGCAGTGGGCTTACA TGGCGA TAGCT AGACTGGGCGGTTTTA TGGACAGCAAGCGAACCGGAATTGCCAGCTGGGGCGCCCTCTGGTAA GGTTGGGAAGCCCTGCAAAGTAAACTGGA TGGCTTTCTTGCCGCCAAGGA TCTGATGGCGCAG GGGATCAAGATCTGA TCAAGAGACAGGA TGAGGATCGTTTCGCATGATTGAACAAGA TGGATTG CACGCAGGTTCTCCGGCCGCTTGGGTGGAGAGGCTATTCGGCTATGACTGGGCACAACAGACA ATCGGCTGCTCTGATGCCGCCGTGTTCCGGCTGTCAGCGCAGGGGCGCCCGGTTCTTTTTGTCA AGACCGACCTGTCCGGTGCCCTGAA TGAACTGCAGGACGAGGCAGCGCGGCTATCGTGGCTGG CCACGACGGGCGTTCCTTGCGCAGCTGTGCTCGACGTTGTCACTGAAGCGGGAAGGGACTGGC TGCTATTGGGCGAAGTGCCGGGGCAGGATCTCCTGTCA TCCCACCTTGCTCCTGCCGAGAAAGT ATCCATCATGGCTGA TGCAATGCGGCGGCTGCATACGCTTGATCCGGCTACCTGCCCATTCGACC ACCAAGCGAAACATCGCA TCGAGCGAGCACGTACTCGGATGGAAGCCGGTCTTGTCGATCAGG ATGA TCTGGACGAAGAGCATCAGGGGCTCGCGCCAGCCGAACTGTTCGCCAGGCTCAAGGCGC GCATGCCCGACGGCGAGGA TCTCGTCGTGACCCATGGCGATGCCTGCTTGCCGAA TATCA TGGT GGAAAATGGCCGCTTTTCTGGA TTCATCGACTGTGGCCGGCTGGGTGTGGCGGACCGCTATCAG GACATAGCGTTGGCTACCCGTGATATTGCTGAAGAGCTTGGCGGCGAATGGGCTGACCGCTTCC TCGTGCTTTACGGTATCGCCGCTCCCGA TTCGCAGCGCA TCGCCTTCTA TCGCCTTCTTGACGAG TTCTTCTGAATTGAAAAAGGAAGAGTATGAGTATTCAACATTTCCGTGTCGCCCTTATTCCCTTT TTTGCGGCATTTTGCCTTCCTGTTTTTGCTCACCCAGAAACGCTGGTGAAAGTAAAAGATGCTG AAGATCAGTTGGGTGCACGAGTGGGTTACATCGAACTGGATCTCAACAGCGGTAAGA TCCTTG AGAGTTTTCGCCCCGAAGAACGTTTTCCAATGATGAGCACTTTTAAAGTTCTGCTATGTGGCGC GGTATTATCCCGTATTGACGCCGGGCAAGAGCAACTCGGTCGCCGCATACACTATTCTCAGAAT GACTTGGTTGAGTACTCACCAGTCACAGAAAAGCA TCTTACGGATGGCA TGACAGTAAGAGAA TTATGCAGTGCTGCCA TAACCATGAGTGA TAACACTGCGGCCAACTTACTTCTGACAACGA TCG GAGGACCGAAGGAGCTAACCGCTTTTTTGCACAACATGGGGGATCATGTAACTCGCCTTGA TCG TTGGGAACCGGAGCTGAA TGAAGCCATACCAAACGACGAGCGTGACACCACGATGCCTGTAGC AA TGGCAACAACGTTGCGCAAACTATTAACTGGCGAACTACTTACTCTAGCTTCCCGGCAACAA TTAATAGACTGGATGGAGGCGGATAAAGTTGCAGGACCACTTCTGCGCTCGGCCCTTCCGGCTGGCTGGTTTATTGCTGA TAAA TCTGGAGCCGGTGAGCGTGGGTCTCGCGGTATCATTGCAGCACT GGGGCCAGATGGTAAGCCCTCCCGTA TCGTAGTTATCTACACGACGGGGAGTCAGGCAACTATG GA TGAACGAAATAGACAGA TCGCTGAGA TAGGTGCCTCACTGATTAAGCA TTGGTAACTGTCAG ACCAAGTTTACTCATA TATACTTTAGATTGA TTTAAAACTTCATTTTTAATTTAAAAGGATCTAGG TGAAGA TCCTTTTTGA TAA TCTCA TGACCAAAATCCCTTAACGTGAGTTTTCGTTCCACTGAGCG TCAGACCCCGTAGAAAAGA TCAAAGGATCTTCTTGAGATCCTTTTTTTCTGCGCGTAATCTGCTG CTTGCAAACAAAAAAACCACCGCTACCAGCGGTGGTTTGTTTGCCGGATCAAGAGCTACCAAC TCTTTTTCCGAAGGTAACTGGCTTCAGCAGAGCGCAGATACCAAATACTGTTCTTCTAGTGTAG CCGTAGTTAGGCCACCACTTCAAGAACTCTGTAGCACCGCCTACA TACCTCGCTCTGCTAATCCT GTTACCAGTGGCTGCTGCCAGTGGCGA TAAGTCGTGTCTTACCGGGTTGGACTCAAGACGATAG TTACCGGATAAGGCGCAGCGGTCGGGCTGAACGGGGGGTTCGTGCACACAGCCCAGCTTGGA GCGAACGACCTACACCGAACTGAGATACCTACAGCGTGAGCTATGAGAAAGCGCCACGCTTCC CGAAGGGAGAAAGGCGGACAGGTATCCGGTAAGCGGCAGGGTCGGAACAGGAGAGCGCACG AGGGAGCTTCCAGGGGGAAACGCCTGGTATCTTTATAGTCCTGTCGGGTTTCGCCACCTCTGAC TTGAGCGTCGATTTTTGTGATGCTCGTCAGGGGGGCGGAGCCTA TGGAAAAACGCCAGCAACG CGGCCTTTTTACGGTTCCTGGCCTTTTGCTGGCCTTTTGCTCACATGTTCTTTCCTGCGTTATCCC CTGA TTCTGTGGATAACCGTA TTACCGCCTTTGAGTGAGCTGATACCGCTCGCCGCAGCCGAAC GACCGAGCGCAGCGAGTCAGTGAGCGAGGAAGCGGAAGLac Z fragment: bases 1-548M13 reverse priming site: bases 205-221Multiple cloning site: bases 234-358T7 promoter/priming site: bases 364-381M13 Forward (-20) priming site: bases 389-404f1 origin: bases 546-983Kanamycin resistance ORF: bases 1317-2111Ampicillin resistance ORF: bases 2129-2989pUC origin: bases 3134-3807pEASY-BluntRestriction MapEnzyme # of cuts positionsAcc65I (1) ggtacc 246AceIII (3) cagctc 949, 2630, 3870AciI (47) ccgc 88, 191, 329, 561, 575,1014, 1159, 1515,1579, 1680AclI (2) aacgtt 2319, 2692AcuI (4) ctgaag 3283, 1576, 2008,2235AflII (1) cttaag 297AflIII (2) acrygt 1021, 3810AhdI (1) gacnnnnngtc 2922AloI (1) gaacnnnnnntcc 835AluI (22) agct 58, 122, 217, 239, 251,477, 691, 948, 1069,1122AlwI (16) ggatc 258, 1700, 2292, 3072,3169, 257, 1263, 1280,1310, 1621AlwNI (2) cagnnnctg 1073, 3399ApaBI (1) gcannnnntgc 1002ApaI (1) gggccc 356ApaLI (2) gtgcac 2250, 3496ApoI (3) raatty 964, 975, 986AseI (3) attaat 52, 111, 2746AvaI (1) cycgrg 338AvaII (3) ggwcc 1966, 2557, 2779BamHI (1) ggatcc 258BanI (7) ggyrcc 137, 246, 740, 1202,1450, 1485, 2969 BanII (4) grgcyc 252, 356, 706, 1812BbeI (2) ggcgcc 1202, 1450BbvI (18) gcagc 1002, 1410, 1594,1687, 2500, 3194,3400, 57, 457, 530 BccI (11) ccatc 771, 1243, 1269, 1334,1762, 2470, 2757,2881, 319, 789 BceAI (6) acggc 397, 1424, 3324, 270,737, 1860BcefI (6) acggc 397, 1424, 3324, 270,737, 1860BcgI (1) cgannnnnntgc 2401BciVI (2) gtatcc 1659, 3601BclI (1) tgatca 1292BfaI (7) ctag 263, 349, 626, 1150,2727, 3062, 3315 BfrBI (1) atgcat 345BglI (2) gccnnnnnggc 539, 2803BglII (1) agatct 1287Bme1580I (5) gkgcmc 356, 1393, 1486, 2250,3496BmrI (5) actggg 420, 1075, 1156, 1390,2872BpmI (1) ctggag 2832BpuEI (5) cttgag 1842, 3442, 2297,3165, 3704BsaAI (3) yacgtr 779, 1022, 1753 BsaBI (3) gatnnnnatc 319, 1286, 1310 BsaHI (3) grcgyc 1202, 1450, 2381 BsaI (1) ggtctc 2850BsaJI (7) ccnngg 142, 430, 1056, 1260,1614, 1883, 3650 BsaWI (5) wccggw 1184, 1482, 2626,3457, 3604BsaXI (2) acnnnnnctcc 833, 28BscAI (11) gcatc 1418, 1673, 1889,2230, 2670, 3722, 346,1740, 1804, 2079 BseMII (4) ctcag 2960, 3126, 3535,2420BseYI (3) cccagc 1197, 1955, 3506 BsiEI (8) cgrycg 272, 332, 508, 1357,2401, 2550, 3473,3897BsiHKAI (6) gwgcwc 252, 1560, 1750, 2250,2335, 3496BslI (9) ccnnnnnnngg 39, 561, 887, 1431,1975, 3331, 3610,3776, 3794BsmAI (2) gtctc 1298, 2850BsmFI (1) gggac 1587Bsp1286I (11) gdgchc 252, 356, 706, 1393,1486, 1560, 1750,1812, 2250, 2335Bsp24I (1) gacnnnnnntgg 3130BspCNI (4) ctcag 2960, 3126, 3535,2420BspHI (1) tcatga 3090BspMI (3) acctgc 1118, 1347, 1710 BsrBI (5) ccgctc 191, 335, 635, 2062,3879BsrDI (3) gcaatg 2863, 1677, 2681 BsrFI (4) rccggy 676, 1770, 1951, 2837 BsrI (20) actgg 277, 1032, 2446, 3394,3407, 81, 326, 393,419, 864BssHII (1) gcgcgc 1848BssSI (3) cacgag 2043, 2253, 3637 BstAPI (1) gcannnnntgc 1002BstF5I (10) ggatg 463, 1632, 1059, 1242,1304, 1761, 1786,2469, 2756, 2937 BstKTI (25) gatc 257, 507, 1263, 1280,1286, 1291, 1310,1621, 1699, 1780 BstNI (6) ccwgg 142, 430, 1836, 3649,3662, 3783BstUI (12) cgcg 33, 550, 574, 594, 970,1514, 1815, 1847,2360, 2853BstXI (2) ccannnnnntgg 284, 326BstYI (11) rgatcy 258, 1264, 1287, 1622,1868, 2275, 2292,3060, 3072, 3158BtgI (1) ccrygg 1883BthCI (34) gcngc 57, 273, 330, 457, 530,562, 576, 598, 1002,1255BtsI (4) gcagtg 2524, 94, 1129, 2496 Cac8I (24) gcnngc 61, 90, 343, 476, 500,619, 633, 676, 1101,1125CdiI (7) catcg 1888, 2669, 790, 1736,1939, 2080, 2265 ChaI (25) gatc 257, 507, 1263, 1280,1286, 1291, 1310,1621, 1699, 1780CjeI (7) ccannnnnngt 3228, 3783, 243, 482,3129, 3229, 3307 CjePI (4) ccannnnnnntc 3277, 2538, 3010,3130Csp6I (3) gtac 245, 1754, 2438 CviAII (14) catg 226, 342, 1139, 1320,1665, 1851, 1882,1908, 2474, 2510 CviJI (78) rgcy 41, 58, 122, 144, 165,217, 239, 251, 271,331DdeI (4) ctnag 2420, 2960, 3126,3535DpnI (25) gatc 257, 507, 1263, 1280,1286, 1291, 1310,1621, 1699, 1780 DraI (3) tttaaa 2342, 3034, 3053 DraIII (1) cacnnngtg 782DrdI (3) gacnnnnnngtc 828, 1478, 3708EaeI (9) yggccr 42, 272, 332, 396,1357, 1531, 1922,1949, 2529EagI (3) cggccg 272, 332, 1357EarI (4) ctcttc 494, 1800, 2010, 2128 EciI (4) ggcgga 1964, 2764, 3592,3738Eco57MI (5) ctgrag 3283, 1576, 2008,2235, 2832EcoHI (6) ccsgg 1056, 1453, 1613,2384, 2735, 3431 EcoICRI (1) gagctc 252EcoO109I (1) rggnccy 356EcoRV (1) gatatc 315EsaBC3I (9) tcga 337, 744, 1562, 1718,1742, 1778, 1940,2266, 3710FalI (1) aagnnnnnctt 1773FatI (14) catg 226, 342, 1139, 1320,1665, 1851, 1882,1908, 2474, 2510 FauI (6) cccgc 89, 579, 1580, 32, 500,633FmuI (8) ggncc 355, 498, 786, 1966,2557, 2779, 2796,2875Fnu4HI (34) gcngc 57, 273, 330, 457, 530,562, 576, 598, 1002,1255FokI (10) ggatg 463, 1632, 1059, 1242,1304, 1761, 1786,2469, 2756, 2937 FspI (3) tgcgca 528, 1551, 2697GdiII (11) cggccr 42, 272, 332, 396,1357, 1922, 1949, 272,332, 1357HaeI (4) wggccw 1531, 3334, 3786,3797HaeII (6) rgcgcy 7, 622, 630, 1202,1450, 3566HaeIII (19) ggcc 41, 271, 331, 355, 395,497, 786, 928, 1356,1530HaeIV (2) gaynnnnnrtc 1628, 2922HgaI (4) gacgc 3130, 3708, 549, 2380 HhaI (30) gcgc 6, 34, 99, 527, 551,564, 573, 595, 621,629Hin4I (8) gaynnnnnvtc 257, 346, 1628, 1658,2848, 2922, 1628,2922HindIII (1) aagctt 240HinfI (10) gantc 46, 376, 828, 850,1935, 2069, 2922,3439, 3835, 3910 HinP1I (30) gcgc 6, 34, 99, 527, 551,564, 573, 595, 621,629HpaII (21) ccgg 163, 675, 1056, 1183,1354, 1431, 1453,1481, 1612, 1702 HphI (6) ggtga 2202, 2443, 768, 2217,2839, 3066Hpy188I (15) tcnga 255, 721, 1266, 1289,1415, 1759, 2112,2421, 2541, 2552Hpy188III (21) tcnnga 77, 350, 415, 1084,1284, 1295, 1468,1784, 1793, 1876Hpy8I (7) gtnnac 776, 831, 848, 1236,2250, 3008, 3496Hpy99I (9) cgwcg 399, 412, 1536, 1564,1858, 1873, 2652,2915, 3709HpyAV (10) ccttc 1584, 2562, 3270,3580, 514, 654, 2085,2095, 2187, 2799 HpyCH4I (14) catg 226, 342, 1139, 1320,1665, 1851, 1882,1908, 2474, 2510 HpyCH4III (11) acngt 522, 804, 1045, 1828,1944, 2052, 2479,2994, 3307, 3777 HpyCH4IV (11) acgt 410, 668, 778, 821,833, 1021, 1565, 1752,2318, 2691HpyCH4V (17) tgca 55, 309, 344, 455,1126, 1228, 1340,1499, 1674, 1688 HpyF10VI (25) gcnnnnnnngc 15, 99, 143, 290, 339,363, 509, 539, 571,615KasI (2) ggcgcc 1202, 1450KpnI (1) ggtacc 246LpnI (6) rgcgcy 7, 622, 630, 1202,1450, 3566M.AacDam (25) gatc 257, 507, 1263, 1280,1286, 1291, 1310,1621, 1699, 1780M.AclI (2) aacgtt 2319, 2692M.AflIII (2) acrygt 1021, 3810M.AlaK2I (25) gatc 257, 507, 1263, 1280,1286, 1291, 1310,1621, 1699, 1780M.AloI (1) gaacnnnnnntcc 835M.AluI (22) agct 58, 122, 217, 239, 251,477, 691, 948, 1069,1122M.Alw26I (2) gtctc 1298, 2850M.AlwI (16) ggatc 258, 1700, 2292, 3072,3169, 257, 1263, 1280,1310, 1621M.ApaI (1) gggccc 356M.AquI (1) cycgrg 338M.AvaIX (4) rccggy 676, 1770, 1951, 2837 M.AvaV (25) gatc 257, 507, 1263, 1280,1286, 1291, 1310,1621, 1699, 1780M.AvaVI (25) gatc 257, 507, 1263, 1280,1286, 1291, 1310,1621, 1699, 1780M.AvaVII (19) ggcc 41, 271, 331, 355, 395,497, 786, 928, 1356,1530M.BabI (10) gantc 46, 376, 828, 850,1935, 2069, 2922,3439, 3835, 3910M.BanII (4) grgcyc 252, 356, 706, 1812 M.BbvI (18) gcagc 1002, 1410, 1594,1687, 2500, 3194,3400, 57, 457, 530 M.BbvSI (18) gcwgc 57, 457, 530, 598,1002, 1410, 1511,1553, 1594, 1687M.BcgI (1) cgannnnnntgc 2401M.BclI (1) tgatca 1292M.BcnIA (6) ccsgg 1056, 1453, 1613,2384, 2735, 3431M.BcnIB (6) ccsgg 1056, 1453, 1613,2384, 2735, 3431M.BepI (12) cgcg 33, 550, 574, 594, 970,1514, 1815, 1847,2360, 2853M.BfiIA (5) actggg 420, 1075, 1156, 1390,2872M.BfiIB (5) actggg 420, 1075, 1156, 1390,2872M.BflBF4I (18) gcsgc 273, 330, 562, 576,1255, 1358, 1421,1516, 1681, 1923M.BglII (1) agatct 1287M.BmeTI (1) tgatca 1292M.BnaI (1) ggatcc 258M.BsaAI (3) yacgtr 779, 1022, 1753M.BspKT6I (25) gatc 257, 507, 1263, 1280,1286, 1291, 1310,1621, 1699, 1780M.BspLU11IIIA (1) gggac 1587M.BspLU11IIIC (1) gggac 1587M.BspRI (19) ggcc 41, 271, 331, 355, 395,497, 786, 928, 1356,1530M.BspST5I (11) gcatc 1418, 1673, 1889,2230, 2670, 3722, 346,1740, 1804, 2079M.BstF5IA (10) ggatg 463, 1632, 1059, 1242,1304, 1761, 1786,2469, 2756, 2937M.BstF5ID (10) ggatg 463, 1632, 1059, 1242,1304, 1761, 1786,2469, 2756, 2937M.BstI (1) ggatcc 258M.BstNI (6) ccwgg 142, 430, 1836, 3649,3662, 3783M.BstVI (1) ctcgag 338M.BstYI (11) rgatcy 258, 1264, 1287, 1622,1868, 2275, 2292,3060, 3072, 3158M.BsuBI (2) ctgcag 310, 1500M.BsuEII (12) cgcg 33, 550, 574, 594, 970,1514, 1815, 1847,2360, 2853M.BsuFI (21) ccgg 163, 675, 1056, 1183,1354, 1431, 1453,1481, 1612, 1702M.BsuRI (19) ggcc 41, 271, 331, 355, 395,497, 786, 928, 1356,1530M.CboI (21) ccgg 163, 675, 1056, 1183,1354, 1431, 1453,1481, 1612, 1702rMI (10) gantc 46, 376, 828, 850,1935, 2069, 2922,3439, 3835, 3910M.Cfr10I (4) rccggy 676, 1770, 1951, 2837 M.Cfr13I (8) ggncc 355, 498, 786, 1966,2557, 2779, 2796,2875M.Cfr6I (4) cagctg 59, 478, 1195, 1555 M.CfrBI (2) ccwwgg 1260, 1883M.CfrI (9) yggccr 42, 272, 332, 396,1357, 1531, 1922,1949, 2529M.CglASI (18) gcsgc 273, 330, 562, 576,1255, 1358, 1421,1516, 1681, 1923M.CviAI (25) gatc 257, 507, 1263, 1280,1286, 1291, 1310,1621, 1699, 1780M.CviAII (14) catg 226, 342, 1139, 1320,1665, 1851, 1882,1908, 2474, 2510M.CviAIV (260) rgcb 7, 23, 41, 58, 122, 144,165, 217, 234, 239 M.CviBI (10) gantc 46, 376, 828, 850,1935, 2069, 2922,3439, 3835, 3910M.CviBIII (9) tcga 337, 744, 1562, 1718,1742, 1778, 1940,2266, 3710M.CviJI (260) rgcb 7, 23, 41, 58, 122, 144,165, 217, 234, 239 M.CviPI (309) gc 4, 15, 22, 32, 40, 54,57, 61, 86, 90M.CviQI (3) gtac 245, 1754, 2438M.CviQIII (9) tcga 337, 744, 1562, 1718,1742, 1778, 1940,2266, 3710M.CviQVI (10) gantc 46, 376, 828, 850,1935, 2069, 2922,3439, 3835, 3910M.CviQVII (14) catg 226, 342, 1139, 1320,1665, 1851, 1882,1908, 2474, 2510M.CviRI (17) tgca 55, 309, 344, 455,1126, 1228, 1340,1499, 1674, 1688M.CviRII (3) gtac 245, 1754, 2438M.CviSI (17) tgca 55, 309, 344, 455,1126, 1228, 1340,1499, 1674, 1688M.CviSII (14) catg 226, 342, 1139, 1320,1665, 1851, 1882,1908, 2474, 2510M.CviSIII (9) tcga 337, 744, 1562, 1718,1742, 1778, 1940,2266, 3710M.CviTI (78) rgcy 41, 58, 122, 144, 165,217, 239, 251, 271,331M.DdeI (4) ctnag 2420, 2960, 3126,3535M.DpnIIA (25) gatc 257, 507, 1263, 1280,1286, 1291, 1310,1621, 1699, 17801621, 1699, 1780M.DraI (3) tttaaa 2342, 3034, 3053M.DraIII (1) cacnnngtg 782M.DsaV (12) ccngg 142, 430, 1056, 1453,1613, 1836, 2384,2735, 3431, 3649M.EaeI (9) yggccr 42, 272, 332, 396,1357, 1531, 1922,1949, 2529M.Ecl18kI (12) ccngg 142, 430, 1056, 1453,1613, 1836, 2384,2735, 3431, 3649M.Eco1831I (6) ccsgg 1056, 1453, 1613,2384, 2735, 3431M.Eco57IA (4) ctgaag 3283, 1576, 2008,2235M.Eco57IB (4) ctgaag 3283, 1576, 2008,2235M.Eco67Dam (25) gatc 257, 507, 1263, 1280,1286, 1291, 1310,1621, 1699, 1780M.EcoHI (6) ccsgg 1056, 1453, 1613,2384, 2735, 3431M.EcoHK31I (9) yggccr 42, 272, 332, 396,1357, 1531, 1922,1949, 2529M.EcoKDam (25) gatc 257, 507, 1263, 1280,1286, 1291, 1310,1621, 1699, 1780M.EcoKDcm (6) ccwgg 142, 430, 1836, 3649,3662, 3783M.EcoKI (1) aacnnnnnngtgc 2259M.EcoO109I (1) rggnccy 356M.EcoP15I (3) cagcag 3193, 3402, 3286M.EcoP1Dam (25) gatc 257, 507, 1263, 1280,1286, 1291, 1310,1621, 1699, 1780M.EcoPI (6) agacc 890, 1772, 2849, 1470,3000, 3135M.EcoR124II (1) gaannnnnnnrtcg 1741M.EcoRII (6) ccwgg 142, 430, 1836, 3649,3662, 3783M.EcoRV (1) gatatc 3151621, 1699, 1780M.EcoT2Dam (25) gatc 257, 507, 1263, 1280,1286, 1291, 1310,1621, 1699, 1780M.EcoT4Dam (25) gatc 257, 507, 1263, 1280,1286, 1291, 1310,1621, 1699, 1780M.EsaBC1I (22) agct 58, 122, 217, 239, 251,477, 691, 948, 1069,1122M.EsaBC3I (9) tcga 337, 744, 1562, 1718,1742, 1778, 1940,2266, 3710M.EsaBS1I (14) catg 226, 342, 1139, 1320,1665, 1851, 1882,1908, 2474, 2510M.EsaDix3I (9) tcga 337, 744, 1562, 1718,1742, 1778, 1940,2266, 3710M.EsaDix4I (18) ttaa 51, 110, 296, 445, 569,840, 938, 955, 966,978M.EsaDix5I (18) ttaa 51, 110, 296, 445, 569,840, 938, 955, 966,978M.EsaLHCI (25) gatc 257, 507, 1263, 1280,1286, 1291, 1310,1621, 1699, 1780M.FnuDII (12) cgcg 33, 550, 574, 594, 970,1514, 1815, 1847,2360, 2853M.FokI (10) ggatg 463, 1632, 1059, 1242,1304, 1761, 1786,2469, 2756, 2937M.HaeIII (19) ggcc 41, 271, 331, 355, 395,497, 786, 928, 1356,1530M.HaeIV (2) gaynnnnnrtc 1628, 2922M.HapII (21) ccgg 163, 675, 1056, 1183,1354, 1431, 1453,1481, 1612, 1702M.HgaIA (4) gacgc 3130, 3708, 549, 2380 M.HgaIB (4) gacgc 3130, 3708, 549, 2380M.HgiCI (7) ggyrcc 137, 246, 740, 1202,1450, 1485, 2969M.HhaI (30) gcgc 6, 34, 99, 527, 551,564, 573, 595, 621,629M.HhaII (10) gantc 46, 376, 828, 850,1935, 2069, 2922,3439, 3835, 3910M.HindDam (25) gatc 257, 507, 1263, 1280,1286, 1291, 1310,1621, 1699, 1780M.HindI (99) cac 94, 114, 176, 186, 278,372, 414, 581, 586,601M.HindIII (1) aagctt 240M.HinfI (10) gantc 46, 376, 828, 850,1935, 2069, 2922,3439, 3835, 3910M.HinHP1Dam (25) gatc 257, 507, 1263, 1280,1286, 1291, 1310,1621, 1699, 1780M.HinP1I (30) gcgc 6, 34, 99, 527, 551,564, 573, 595, 621,629M.HpaII (21) ccgg 163, 675, 1056, 1183,1354, 1431, 1453,1481, 1612, 1702M.HphIA (6) ggtga 2202, 2443, 768, 2217,2839, 3066M.HphIB (6) ggtga 2202, 2443, 768, 2217,2839, 3066M.Hpy166IV (14) catg 226, 342, 1139, 1320,1665, 1851, 1882,1908, 2474, 2510M.Hpy188I (15) tcnga 255, 721, 1266, 1289,1415, 1759, 2112,2421, 2541, 2552M.Hpy188II (14) catg 226, 342, 1139, 1320,1665, 1851, 1882,1908, 2474, 2510M.Hpy8I (7) gtnnac 776, 831, 848, 1236,2250, 3008, 3496M.Hpy99II (6) gtsac 416, 603, 1571, 1877,2449, 2660M.Hpy99IX (10) gantc 46, 376, 828, 850,1935, 2069, 2922,3439, 3835, 3910M.Hpy99VA (18) cctc 352, 495, 1307, 1371,1507, 1864, 2554,2760, 3638, 3921M.Hpy99VI (25) gatc 257, 507, 1263, 1280,1286, 1291, 1310,1621, 1699, 1780M.Hpy99X (14) catg 226, 342, 1139, 1320,1665, 1851, 1882,1908, 2474, 2510M.Hpy99XI (11) acgt 410, 668, 778, 821,833, 1021, 1565, 1752,2318, 2691M.HpyAI (14) catg 226, 342, 1139, 1320,1665, 1851, 1882,1908, 2474, 2510M.HpyAIIA (11) gaaga 649, 2109, 3160, 3312,493, 1799, 2009, 2127,2236, 2314M.HpyAIII (25) gatc 257, 507, 1263, 1280,1286, 1291, 1310,1621, 1699, 1780M.HpyAIV (10) gantc 46, 376, 828, 850,1935, 2069, 2922,3439, 3835, 3910M.HpyAIX (7) gtnnac 776, 831, 848, 1236,2250, 3008, 3496M.HpyAVIII (30) gcgc 6, 34, 99, 527, 551,564, 573, 595, 621,629M.HpyAX (9) tcga 337, 744, 1562, 1718,1742, 1778, 1940,2266, 3710M.HpyCH4IV (11) acgt 410, 668, 778, 821,833, 1021, 1565, 1752,2318, 2691M.HpyI (14) catg 226, 342, 1139, 1320,1665, 1851, 1882,1908, 2474, 2510M.KpnI (1) ggtacc 246M.LlaDCHIA (25) gatc 257, 507, 1263, 1280,1286, 1291, 1310,1621, 1699, 1780M.LlaDCHIB (25) gatc 257, 507, 1263, 1280,1286, 1291, 1310,1621, 1699, 1780M.LlaKR2I (25) gatc 257, 507, 1263, 1280,1286, 1291, 1310,1621, 1699, 1780M.MboIA (25) gatc 257, 507, 1263, 1280,1286, 1291, 1310,1621, 1699, 1780M.MboIB (25) gatc 257, 507, 1263, 1280,1286, 1291, 1310,1621, 1699, 1780M.MboIIA (11) gaaga 649, 2109, 3160, 3312,493, 1799, 2009, 2127,2236, 2314M.MjaVI (21) ccgg 163, 675, 1056, 1183,1354, 1431, 1453,1481, 1612, 1702M.MlyI (6) gastc 376, 828, 850, 2922,3439, 3910M.MmeIA (3) tccrac 825, 3436, 3620M.MmeIB (3) tccrac 825, 3436, 3620M.MnlIA (18) cctc 352, 495, 1307, 1371,1507, 1864, 2554,2760, 3638, 3921M.MnlIB (18) cctc 352, 495, 1307, 1371,1507, 1864, 2554,2760, 3638, 3921M.MseI (18) ttaa 51, 110, 296, 445, 569,840, 938, 955, 966,978M.MspI (21) ccgg 163, 675, 1056, 1183,1354, 1431, 1453,1481, 1612, 1702M.MthTI (19) ggcc 41, 271, 331, 355, 395,497, 786, 928, 1356,1530M.MthZI (7) ctag 263, 349, 626, 1150,2727, 3062, 3315M.MvaI (6) ccwgg 142, 430, 1836, 3649,3662, 3783M.NgoBIX (3) gtannnnnctc 2261, 3543, 252M.NgoBVIIIA (6) ggtga 2202, 2443, 768, 2217,2839, 3066M.NgoGII (19) ggcc 41, 271, 331, 355, 395,497, 786, 928, 1356,1530M.NgoHVIII (6) ggtga 2202, 2443, 768, 2217,2839, 3066M.NgoMIV (2) gccggc 676, 1951M.NgoMV (16) ggnncc 137, 246, 258, 356,707, 719, 740, 1202,1450, 1485M.NgoMXV (59) gcchr 40, 61, 90, 134, 143,327, 394, 431, 480,785M.NgoPII (19) ggcc 41, 271, 331, 355, 395,497, 786, 928, 1356,1530M.NlaIII (14) catg 226, 342, 1139, 1320,1665, 1851, 1882,1908, 2474, 2510M.NlaX (12) ccngg 142, 430, 1056, 1453,1613, 1836, 2384,2735, 3431, 3649M.NmeSI (1) agtact 2439M.NspHI (3) rcatgy 343, 1852, 3810M.NspI (3) rcatgy 343, 1852, 3810M.PaeR7I (1) ctcgag 338M.PgiI (25) gatc 257, 507, 1263, 1280,1286, 1291, 1310,1621, 1699, 1780M.Phi3TI (19) ggcc 41, 271, 331, 355, 395,497, 786, 928, 1356,1530M.Phi3TII (9) tcga 337, 744, 1562, 1718,1742, 1778, 1940,2266, 3710M.PhiCh1I (25) gatc 257, 507, 1263, 1280,1286, 1291, 1310,1621, 1699, 1780M.PhiMx8I (4) ctssag 310, 338, 1500, 2832 M.PspPI (8) ggncc 355, 498, 786, 1966,2557, 2779, 2796,2875M.PstI (2) ctgcag 310, 1500M.PvuII (4) cagctg 59, 478, 1195, 1555 M.Rho11sI (19) ggcc 41, 271, 331, 355, 395,497, 786, 928, 1356,1530M.RsaI (3) gtac 245, 1754, 2438M.SacI (1) gagctc 252M.Sau3239I (1) ctcgag 338M.Sau3AI (25) gatc 257, 507, 1263, 1280,1286, 1291, 1310,1621, 1699, 1780M.Sau96I (8) ggncc 355, 498, 786, 1966,2557, 2779, 2796,2875M.SauLPI (2) gccggc 676, 1951M.ScaI (1) agtact 2439M.SenPI (12) ccngg 142, 430, 1056, 1453,1613, 1836, 2384,2735, 3431, 3649M.SinI (3) ggwcc 1966, 2557, 2779M.SmaII (25) gatc 257, 507, 1263, 1280,1286, 1291, 1310,1621, 1699, 1780M.SPBetaI (19) ggcc 41, 271, 331, 355, 395,497, 786, 928, 1356,1530M.SPRI (19) ggcc 41, 271, 331, 355, 395,497, 786, 928, 1356,1530M.SscL1I (10) gantc 46, 376, 828, 850,1935, 2069, 2922,3439, 3835, 3910M.Sse9I (15) aatt 108, 183, 200, 288,304, 360, 387, 963,974, 985M.SsoII (12) ccngg 142, 430, 1056, 1453,1613, 1836, 2384,2735, 3431, 3649M.SssI (263) cg 5, 14, 21, 31, 35, 42,64, 76, 87, 98M.Sth368I (25) gatc 257, 507, 1263, 1280,1286, 1291, 1310,1621, 1699, 1780M.StsI (10) ggatg 463, 1632, 1059, 1242,1304, 1761, 1786,2469, 2756, 2937M.Sty1344Dam (25) gatc 257, 507, 1263, 1280,1286, 1291, 1310,1621, 1699, 1780M.StyDam (25) gatc 257, 507, 1263, 1280,1286, 1291, 1310,1621, 1699, 1780M.StyLTI (6) cagag 1207, 1414, 3351,3377, 3697, 3288M.StyLTIII (1) gagnnnnnnrtayg 2418M.StySPI (1) aacnnnnnngtrc 2259M.TaeII (1) tgatca 1292M.TaqI (9) tcga 337, 744, 1562, 1718,1742, 1778, 1940,2266, 3710M.TfiI (4) gawtc 46, 1935, 2069, 3835 M.TpaI (25) gatc 257, 507, 1263, 1280,1286, 1291, 1310,1621, 1699, 1780M.Tsp45I (6) gtsac 416, 603, 1571, 1877,2449, 2660M.TspRI (14) castg 94, 278, 325, 392,1129, 1573, 2496,2523, 2870, 2975M.Tth111I (1) gacnnngtc 1569M.TthHB8I (9) tcga 337, 744, 1562, 1718,1742, 1778, 1940,2266, 3710M.VspI (3) attaat 52, 111, 2746M.XbaI (1) tctaga 350M.XcmI (1) ccannnnnnnnntgg 1269M.XhoI (1) ctcgag 338M.XhoII (11) rgatcy 258, 1264, 1287, 1622,1868, 2275, 2292,3060, 3072, 3158M.XmaIII (3) cggccg 272, 332, 1357 MaeIII (15) gtnac 267, 416, 436, 591,603, 1571, 1877, 2261,2449, 2602MboI (25) gatc 257, 507, 1263, 1280,1286, 1291, 1310,1621, 1699, 1780 MboII (11) gaaga 649, 2109, 3160, 3312,493, 1799, 2009, 2127,2236, 2314MlyI (6) gagtc 850, 3439, 376, 828,2922, 3910MmeI (3) tccrac 825, 3436, 3620MnlI (18) cctc 352, 495, 1307, 1371,1507, 1864, 2554,2760, 3638, 3921MscI (1) tggcca 1531MseI (18) ttaa 51, 110, 296, 445, 569,840, 938, 955, 966,978MslI (4) caynnnnrtg 1888, 2151, 2510,2669MspA1I (7) cmgckg 59, 478, 1195, 1555,2284, 3225, 3470 MwoI (25) gcnnnnnnngc 15, 99, 143, 290, 339,363, 509, 539, 571,615NaeI (2) gccggc 676, 1951NarI (2) ggcgcc 1202, 1450NciI (6) ccsgg 1056, 1453, 1613,2384, 2735, 3431 NcoI (1) ccatgg 1883NgoMIV (2) gccggc 676, 1951NlaIII (14) catg 226, 342, 1139, 1320,1665, 1851, 1882,1908, 2474, 2510 NlaIV (16) ggnncc 137, 246, 258, 356,707, 719, 740, 1202,1450, 1485Nli3877I (1) cycgrg 338NotI (1) gcggccgc 333NsiI (1) atgcat 345NspI (3) rcatgy 343, 1852, 3810PciI (1) acatgt 3810PleI (6) gagtc 850, 3439, 376, 828,2922, 3910PpiI (3) gaacnnnnnctc 835, 3119, 2276Ppu10I (1) atgcat 345PsiI (1) ttataa 907Psp03I (3) ggwcc 1966, 2557, 2779 PspGI (6) ccwgg 142, 430, 1836, 3649,3662, 3783PspOMI (1) gggccc 356PssI (1) rggnccy 356PstI (2) ctgcag 310, 1500PvuI (2) cgatcg 508, 2550PvuII (4) cagctg 59, 478, 1195, 1555 RsaI (3) gtac 245, 1754, 2438RsrII (1) cggwccg 1967SacI (1) gagctc 252SapI (2) gctcttc 1801, 2011Sau96I (8) ggncc 355, 498, 786, 1966,2557, 2779, 2796,2875ScaI (1) agtact 2439SciI (1) ctcgag 338ScrFI (12) ccngg 142, 430, 1056, 1453,1613, 1836, 2384,2735, 3431, 3649SelI (12) cgcg 33, 550, 574, 594, 970,1514, 1815, 1847,2360, 2853SfaNI (11) gcatc 1418, 1673, 1889,2230, 2670, 3722, 346,1740, 1804, 2079SfcI (7) ctryag 310, 370, 558, 1500,2676, 3354, 3545SfoI (2) ggcgcc 1202, 1450SimI (6) gggtc 747, 1053, 1879, 3136,2848, 3617SmlI (7) ctyrag 297, 338, 1842, 2297,3165, 3442, 3704 SpeI (1) actagt 264SphI (2) gcatgc 343, 1852SspD5I (6) ggtga 2202, 2443, 768, 2217,2839, 3066Sth132I (28) cccg 89, 579, 701, 1538,1580, 1613, 2384,2917, 3431, 3475StsI (10) ggatg 463, 1632, 1059, 1242,1304, 1761, 1786,2469, 2756, 2937 StyD4I (12) ccngg 142, 430, 1056, 1453,1613, 1836, 2384,2735, 3431, 3649StyI (2) ccwwgg 1260, 1883TaiI (11) acgt 410, 668, 778, 821,833, 1021, 1565, 1752,2318, 2691TaqI (9) tcga 337, 744, 1562, 1718,1742, 1778, 1940,2266, 3710TaqII (5) gaccga 889, 2400, 1472, 2559,3898TatI (1) wgtacw 2439TauI (18) gcsgc 273, 330, 562, 576,。
pEASY-Blunt Cloning Kit使用说明书

但是载体使用量不变。
● 反应时间绝对不能超过30分钟。 ● 总反应体积不能超过5 μl。 ● 随着克隆片段长度的增加(>3 kb),克隆效率降低。 ● 若有引物二聚体,建议使用PCR纯化试剂盒;紫外照
目录号:CB101 保存:Trans1-T1 Phage Resistant Chemically Competent
Cell-70℃保存六个月,其它-20℃保存六个月。 试剂盒组成 pEASY®- Blunt Cloning Vector (10 ng/μl),Control
Template (5 ng/μl),Control Primers (10 μM),
射不仅容易使DNA发生突变,而且影响克隆效率。
TransGen Biotech
Phone: 010-51296890 Customer Service: 400-898-0321 E-mail : trans @
克隆常见问题分析 (1)克隆效率低 影响克隆效率有许多因素,如扩增目的基因所用引 物,基因的结构,插入片段和载体的比例等。如发 现克隆效率低,尝试用下列方法提高克隆效率。
TransGen Biotech
Phone: 010-51296890 Customer Service: 400-898-0321 E-mail : trans @
pEASY ®-Blunt Cloning Kit
使用前请务必仔细阅读说明书
pEASY ®-Blunt
M13 Reverse Primer
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
● 快速克隆基因,仅需要5分钟反应时间。
● 提供氨苄青霉素和卡那霉素两种筛选标记。
● 对照插入片段克隆效率可达85% 以上。
● 包含LacZ 基因,在含有IPTG,X-gal 的平板培养基
上,可进行蓝白斑筛选。
PCR 产物的制备
(1) 引物要求:引物不能磷酸化。
(2) 酶的选择: Pfu系列的DNA 聚合酶。
M13F (10 μM),M13R (10 μM),Trans1-T1 Phage
Resistant Chemically Competent Cell。Expected PCR
product size: 700 bp。 产品说明 pEASY®- Blunt Cloning Vector 适用于平端克隆。
to 50 μl
Final Concentration as required 0.2 μM 1× 0.2 mM 2.5 units
Not applicable
PCR
94℃ 94℃ 50-60℃ 72℃ 72℃
2-5 min 30 sec 30 sec 1kb/min 10 min
30 cycles
本产品仅供研究,不用于临床诊断。
目录号:CB101 保存:Trans1-T1 Phage Resistant Chemically Competent
Cell-70℃保存六个月,其它-20℃保存六个月。 试剂盒组成 pEASY®- Blunt Cloning Vector (10 ng/μl),Control
Template (5 ng/μl),Control Primers (10 μM),
EcoR V
BstX I
Not I
Xho I
Nsi I Xba I
Apa I
CCC TT
AA GGG CAA TTC TGC AGA TAT CCA TCA CAC TGG CGG CCG CTC GAG CAT GCA TCT AGA GGG CCC AAT
GGG AA PCR Product TT CCC GTT AAG ACG TCT ATA GGT AGT GTG ACC GCC GGC GAG CTC GTA CGT AGA TCT CCC GGG TTA
TransGen Biotech
Phone: 010-51296890 Customer Service: 400-898-0321 E-mail : trans @
pEASY ®-Blunt Cloning Kit
使用前请务必仔细阅读说明书
(3)重组克隆呈现淡蓝色或“fish eye” 插入片段没有影响lacZ 基因读码框,或插入片段太小, 这种情况下菌落呈现淡蓝色,可以进行下游实验。
pEASY ®-Blunt (3929 bp)
LacZα fragment: bases 1-548
Multiple cloning site: bases 234-358 M13 reverse priming site: bases 205-221 T7 promoter priming site: bases 362-381 M13 forward priming site: bases 388-404 f1 origin: bases 546-983 Kanamycin resistance ORF: bases 1317-2111 Ampicillin resistance ORF: bases 2129-2989 pUC origin: bases 3134-3807
对照片段(700 bp)PCR 体系
Components Control Template Control Primers (10 μM) 10×Buffer (Buffer 含Mg2+) 2.5 mM dNTPs EasyPfu DNA Polymerase ddH2O
Volume 1 μl 1 μl 5 μl 4 μl 1 μl
T7 Promoter
M13 Forward Primer
TCG CCC TAT AGT GAG TCG TAT TAC AAT TCA CTG GCC GTC GTT TTA CAA CGT CGT GAC TGG GAA AAC
AGC GGG ATA TCA CTC AGC ATA ATG TTA AGT GAC CTG CAG CAA AAT GTT GCA GCA CTG ACC CTT TTG
射不仅容易使DNA发生突变,而且影响克隆效率。
TransGen Biotech
Phone: 010-51296890 Customer Service: 400-898-0321 E-mail : trans @
克隆常见问题分析 (1)克隆效率低 影响克隆效率有许多因素,如扩增目的基因所用引 物,基因的结构,插入片段和载体的比例等。如发 现克隆效率低,尝试用下列方法提高克隆效率。
a. 纯化PCR 产物。 b. 如插入片段的浓度太低,增加插入片段的量。 c. 用新鲜的PCR 产物。 d. 扩增用平端的酶,如Pfu、Vent DNA Polymerase。 (2)PCR 鉴定重组子失败 当用PCR 方法鉴定重组子,没有得到目的扩增产物, 又没有载体自连,说明PCR 反应失败。重新优化PCR 反应条件或提取质粒,以质粒作模板扩增或酶切鉴定 包含重组子的克隆。
模板,用M13 正向引物和反向引物鉴定重组子。 (3) PCR 反应条件:94℃预变性10 分钟(裂解细胞,
失活核酸酶),94℃变性30 秒,55℃退火30 秒, 72℃延伸( 根据片段的大小确定延伸时间)30 个循环,72℃后延伸10 分钟。确认包含重组子 的克隆,扩大培养,用M13 F,M13 R 引物测序。 (4) 若载体自连,通用引物扩增,自连带大小为200 bp。 限制性酶切分析阳性重组子 挑选白色克隆接种于LB/Amp+ 或 LB/Kan+ 的液体培养基 中,过夜培养。碱裂解法或用试剂盒小量提取质粒,选
(5) 待IPTG,X-gal 被吸收后,取200 μl 菌液铺板,培 养过夜(为得到较多克隆,4000 rpm 离心1 min, 弃掉部分上清,保留100-150 μl,轻弹悬浮菌体, 取全部菌液涂板,培养过夜)。
阳性重组子的鉴定和测序 PCR 方法鉴定阳性重组子 (1) 挑选白色克隆至10 μl 无菌水中,涡旋混合。 (2) 25 μl 反应体系中取1 μl 混合液用作PCR 反应的
4、最佳反应时间
① 片段大小为0.1-1 Kb(含1 Kb):5-10 min
② 片段大小为 1-2 Kb(含2 Kb): 10-15 min
③ 片段大小为 2-3 Kb(含3 Kb): 15-20 min
*片段为胶回收产物,反应时间取最大值。 5、最佳反应温度:25℃
*若片段是高GC结构,可以37℃反应。 (推荐用PCR 仪控温) 转化 (1) 加连接产物于50 μl Trans1-T1感受态细胞中(在
1 μl
轻轻混合,室温 (20℃-37℃) 反应5分钟。反应结束后,
将离心管置于冰上。
注意:
1、最佳插入片段DNA量
载体与片段摩尔比=1:7
*可以粗略的按照“1 Kb 20 ng”的比例计算。
(如1 Kb加20 ng、1.5 Kb加30 ng等)
2、最佳载体使用量:1 μl
无菌水。
pEASY ®-Blunt
M13 Reverse Primer
Hind III
Kpn I
Sac I Bam H I Spe I
BstX I
CAG GAA ACA GCT ATG ACC ATG ATT ACG CCA AGC TTG GTA CCG AGC TCG GAT CCA CTA GTA ACG GCC GCC AGT GTG CTG GAA TTG GTC CTT TGT CGA TAC TGG TAC TAA TGC TGT TCG AAC CAT GGC TCG AGC CTA GGT GAT CAT TGC CGG CGG TCA CAC GAC CTT AAC
感受态细胞刚刚解冻时加入连接产物),轻弹混匀,
冰浴20-30分钟。 (2) 42℃热激30秒,立即置于冰上2分钟。 (3) 加250 μl 平衡至室温的SOC/LB,200 转,37℃孵育1小时。 (4) 取8 μl,500 mM IPTG,40 μl,20 mg/ml X-gal混合,
均匀地涂于准备好的平板上,在37℃放置30 分钟。
(3) 反应条件:为了保证扩增产物的完整性,扩增反应
需要5-10分钟后延伸。反应结束后,电泳检测PCR产物
的量和质量。
克隆反应体系的建立
在微型离心管中依次加入溶液:
PCR 产物
0.5-4 μl
(根据PCR 产物量可适当增减,最多不超过4 μl)
pEASY®- Blunt Cloning Vector
择适宜的限制性内切酶,酶切鉴定重组子。 测序 用M13 F,M13 R,T7 promoter 引物测序,进行序列分析。 注意事项 ● 根据PCR产物的强弱,增减PCR产物的量(0.5-4 μl),
但是载体使用量不变。
● 反应时间绝对不能超过30分钟。 ● 总反应体积不能超过5 μl。 ● 随着克隆片段长度的增加(>3 kb),克隆效率降低。 ● 若有引物二聚体,建议使用PCR纯化试剂盒;紫外照
