考染与银染资料
双向电泳中4种常用染色方法的比较

取、 等电聚焦、 白质转移、D P G 蛋 S S— A E电泳以及染色等
环节 , 个环节 又包括 若 干步骤 。由此可 见 2一 E技 每一 D
术是一个受多种因素控制的技术 , 中任何一种因素没 其
有理 想地控 制好都 有 可能 引起 整 个 实验 的失 败 , 且 这 而 种失 败往往 要等 到 2一 E图谱染 色 显影 之后才 能 看 到。 D 因此 , 2一 E中 , 得到 一张理 想 的 图谱 果 进 行 了对 比 , 筛 选 出适 合 以期 本 实验室 条件 的染色方 法 。
1 材 料 与方 法
11 试 验材料 . 本 实验 所用 的研 究 材料 为 杂交 水 稻威 优 96 1。本实 验染色 所涉及 的试 剂 : 硝酸 银 ( 上海 试 剂一
的质谱分析提供有益的帮助, 除了控制好每一个步骤外 ,
摘
要: 采用 S S— A E电泳技术 , D PG 比较分析 了双向 电泳 中常 用4种染 色方 法的优、 点。结果表 明: 缺 常规银 染法灵敏
度低 ; 的双胺硝酸银银 柒法与质谱 分析不兼容 ; 染方法 一银 染复合染 色法的灵敏度稍低 于双胺硝酸银银 染法, 它的 改进 考 但 质谱 兼容性最高; 改进后的银 染方法灵敏度 高、 背景 清晰、 与质谱 兼容。因此后两种 方法较广泛适用于蛋 白质组学研究。
技术具 有高分 辨率 、 重 复 性 、 量分 析 和制 备 等性 能 , 高 微
它与质谱分析是 当今蛋 白质组学研究 中必不可少的工
具 。2 D 一 E技术所 涉及 的环节 较 多 , 一般 包 括 蛋 白质 提
及实验条件的差异, 同一染色方法往往在不同的实验室
银染
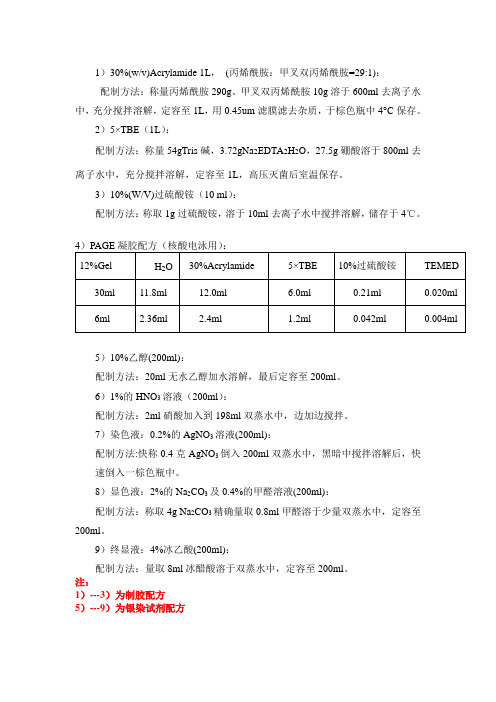
1)30%(w/v)Acrylamide 1L,(丙烯酰胺:甲叉双丙烯酰胺=29:1):配制方法:称量丙烯酰胺290g、甲叉双丙烯酰胺10g溶于600ml去离子水中,充分搅拌溶解,定容至1L,用0.45um滤膜滤去杂质,于棕色瓶中4°C保存。
2)5×TBE(1L):配制方法:称量54gTris碱,3.72gNa2EDTA2H2O,27.5g硼酸溶于800ml去离子水中,充分搅拌溶解,定容至1L,高压灭菌后室温保存。
3)10%(W/V)过硫酸铵(10 ml):配制方法:称取1g过硫酸铵,溶于10ml去离子水中搅拌溶解,储存于4℃。
5)10%乙醇(200ml):配制方法:20ml无水乙醇加水溶解,最后定容至200ml。
6)1%的HNO3溶液(200ml):配制方法:2ml硝酸加入到198ml双蒸水中,边加边搅拌。
7)染色液:0.2%的AgNO3溶液(200ml):配制方法:快称0.4克AgNO3倒入200ml双蒸水中,黑暗中搅拌溶解后,快速倒入一棕色瓶中。
8)显色液:2%的Na2CO3及0.4%的甲醛溶液(200ml):配制方法:称取4g Na2CO3精确量取0.8ml甲醛溶于少量双蒸水中,定容至200ml。
9)终显液:4%冰乙酸(200ml):配制方法:量取8ml冰醋酸溶于双蒸水中,定容至200ml。
注:1)---3)为制胶配方5)---9)为银染试剂配方实验步骤:PCR产物的检测(12% 的PAGE电泳检测):洗净玻璃板、胶条及梳子,晾干,然后用夹子夹好制成灌胶板。
↓用枪吸取 2.36ml三蒸水加入10ml离心管中,再加1.2ml 5×TBE缓冲液,2.4ml 30% Acr,4ul TEMED,最后加42ul APS(过硫酸铵),用移液抢吹打混合混匀,然后缓慢倒入灌胶板中,插上梳子,慢慢放平,让其凝固。
↓待凝胶凝固后,拔掉梳子及玻璃板底部胶条,放入电泳槽,加1×TBE电泳缓冲液并接通电源,60伏,预电泳30min。
快速银染银染步骤及相关药品配制

快速银染银染步骤及相关药品配制银染步骤及相关药品配制一.银染基本步骤1.洗板和擦板电泳槽板先用无水乙醇擦一遍,5分钟后用配制的剥离硅烷(repel)溶液涂抹均匀。
放置15-30分钟玻板(用过的玻板先用NaOH溶液浸泡,直至废胶脱落)先用抹布蘸少许洗洁精在自来水下洗净,再用无水乙醇擦一遍,5分钟后用新鲜配制的亲和硅烷(binding)溶液涂抹均匀。
放置15-30分钟2.装板玻板在下,电泳槽板在上,中间放两根压条(涂抹repel和binding的面相互靠近),对齐后,用三对夹子将波板与电泳槽板夹紧。
3.灌胶用针筒吸取6%的聚丙烯酰胺凝胶溶液,缓慢均匀的从灌胶口将胶灌入,灌好后取下靠近灌胶口的夹子,倒插入梳子,注意不要起汽泡,插好后,再夹上夹子。
凝胶需要1-2个小时。
4.预电泳取下夹子,拔出倒插的梳子,在自来水下将灌胶口冲洗干净,放入电泳槽,卡紧,灌入缓冲液,上下槽各300ml左右,注意用夹子夹紧上槽排放缓冲液的胶管。
80W衡功率电泳半小时。
5.点样用吸管吹干净灌胶口的废胶,将梳子顺插入灌胶口,开始点样,注意不要串样,梳齿不要将胶面弄坏。
6.电泳80W衡功率电泳1小时左右。
7.银染将玻板放入配制好的银染液中,银染半小时左右10.显影从银染液中拿出玻板快速水洗3-5秒,然后放入新鲜配制的显影液中,室温显影,直至条带清晰11.用自来水轻轻冲洗板子正反面几次,洗净残余的NaOH。
二.药品配制1.6%聚丙烯酰胺凝胶(PA):Urea: 420.42gAcr: 60g 丙烯酰胺Bis: 3.16g10×TBE: 50ml加超纯水定容至 1L8% 聚丙烯酰胺PA:尿素 420.42gAcr: 80gBis: 4.212g10*TBE: 100ml加超纯水定容至 1L2. 10×TBETris-base: 216g硼酸:110gEDTA-Na2:14.88g3. binding(亲和硅烷)无水乙醇:2mlBinding原液:8ul冰醋酸:8ul4.Repel (剥离硅烷)三氯甲烷:100mlRepel:2ml5.Loading buffer:甲酰氨:49ml10mM EDTA(PH=8.0):1ml溴酚蓝:0.125g二甲苯青:0.125g7.银染液蒸馏水:1500ml无水乙醇:150ml硝酸银溶液(1g/ml):1500ul 8.显影液蒸馏水:1500ml氢氧化钠:30g甲醛:6ml硫代硫酸钠(10mg/ml):300ul。
银染

快速银染试剂盒Protein Stains Q产品编号:BSP029S-N包装规格:50 Assays产品简介银染是灵敏度较高的一种染色法,是对各种凝胶中的蛋白质进行染色时最常用的方法。
它通过被还原银离子在蛋白质上形成黑色来指示蛋白区带的。
银染的灵敏度比考马斯亮蓝染色高100倍,可以检测低于2 ng的蛋白质。
本试剂盒在保证检测灵敏度的同时,还具有操作方便快速的优点,可在1小时内完成1块凝胶的银染。
运输和保存条件在常温下运输,收到后,将所有试剂放置在2-8°C的环境中保存,保质期一年。
产品组成成份BSP029-NBSP029S-N-1溶液A 300 ulBSP029S-N-2溶液B 20 x 10 mlBSP029S-N-3干粉0.2 gBSP029S-N-4溶液D 5 x 40 mlBSP029S-N-5溶液E 10 ml注意:使用前将干粉用10ml的双蒸水溶解成20倍浓缩的溶液C,再按照需要的体积,稀释为1倍浓度.使用方法1. 取出电泳后的聚丙烯酰胺凝胶,用蒸馏水冲洗凝胶,根据以下程序所需要的试剂量,取一定体积的溶液B2. 和D,用双蒸水稀释成1倍的溶液后再使用.3. 加入20 ml蒸馏水,置于摇床上,震荡5 min。
4. 倾出蒸馏水,加入40%的甲醇20 ml和10 ul溶液A,置于摇床上,震荡10 min。
5. 倾出溶液A,加入20 ml蒸馏水,置于摇床上,震荡5 min,重复1次。
6. 倾出蒸馏水,加入20 ml溶液B,置于摇床上,震荡5 min。
7. 倾出溶液B,加入20 ml蒸馏水,置于摇床上,震荡20s,重复1次。
8. 倾出蒸馏水,加入20 ml溶液C,置于摇床上,震荡10 min。
9. 倾出溶液C,加入20 ml蒸馏水,置于摇床上,震荡1 min.10. 倾出蒸馏水,加入20 ml溶液D和10 ul溶液A,缓慢摇动,直至蛋白质条带具有足够的显色强度(约30 s-1 min)。
银染法的名词解释

银染法的名词解释对于普通人来说,银染法可能是一个陌生的名词,但对于纺织品行业的从业者来说,它却是一个非常重要的工艺。
银染法是一种利用化学方法将银离子引入纺织品中,以达到防菌、抗菌、净化空气等功能的一种技术。
下面,我们将深入探讨银染法的详细内容。
一、银染法的原理银染法利用的是银离子的抗菌性能。
事实上,早在古代,人们就发现银具有抗菌的作用。
在现代,科学家们通过研究发现,银离子能够干扰细菌的细胞膜和核酸的复制,从而抑制细菌的生长和繁殖。
为了将银离子引入纺织品中,银染法应运而生。
二、银染法的流程银染法的流程主要包括:纺织品预处理、银染剂的配制、浸染银染剂、干燥和固定银离子等步骤。
首先,在纺织品进行预处理。
预处理主要包括漂白、洗涤、软化、充电等步骤,以去除纺织品表面的杂质,使其更加适合染色。
接着,配制银染剂。
银染剂通常由银盐和染料组成。
不同的银盐可以产生不同的效果,例如硝酸银可以产生较强的抗菌效果,氧化银则可以产生净化空气的效果。
然后,将纺织品浸泡在银染剂中。
这个步骤中,纺织品会吸收银离子,并且银离子会与纺织品的纤维结合,从而实现功能的目的。
完成浸染后,需要将纺织品进行干燥。
干燥的目的是除去多余的水分,使银离子更好地固定在纺织品上。
最后,采用适当的固定剂将银离子固定在纺织品上。
固定剂能够与银离子进行反应,形成稳定的化学结构,从而增加银离子在纺织品上的使用寿命。
三、银染法的应用银染法在现代纺织品行业中应用广泛。
首先是医疗纺织品领域。
由于银离子的抗菌性能,银染法在生产医用口罩、手术衣等产品时被广泛采用,可以有效地阻止细菌的传播,保护医护人员和患者的健康。
另外,银染法也被应用于家纺领域。
银染法可以制作出带有抗菌功能的床上用品,例如枕头套、被套等。
这些产品可以减少细菌的滋生,为家庭提供一个干净、健康的生活环境。
此外,银染法还可以应用于户外装备领域。
例如,登山服、户外鞋等产品采用了银染法,不仅可以抑制细菌的生长,还有效防止异味产生,让户外运动更加舒适。
银染方法

银染方法1.搪瓷盘中倒入2000ml左右70%乙醇,把胶做好标记卸入乙醇中,摇床上固定15min2.回收乙醇(可重复用3-5次),蒸馏水洗2遍,每遍2-3min,尽可能把水倒净3.190ml蒸馏水中加入3.6%的NaOH4.2ml、20%AgNO33.6ml、氨水2ml混匀配成染色液。
倒入染色液,染色30min。
4.倒掉染色液,蒸馏水洗3遍,每遍2-3min,尽可能把水倒净5.200ml蒸馏水中加入1%柠檬酸钠1ml,甲醛100ul混匀配成显色液。
倒入显色液显色至清晰。
6.倒掉显色液,加蒸馏水停显,并洗2-3遍测序板的硅化处理1:每块玻璃板的面都需硅化处理,以防止凝胶与两块玻璃板紧贴并减少电泳完毕后从胶模中取出凝胶时凝胶发生破裂的可能性。
硅化方法为;将拟硅化的玻璃板置于通风橱中,并戴手套操作,在拟硅化板面上加2-3ml 5%二氯二甲基硅烷(二氯二甲基硅烷5%溶于氯仿中),用纸巾将硅化液涂布均匀,然后用去离子水洗净玻璃板,再用乙醇冲洗后晾干。
如需要从玻璃板上去除原有的硅,可用KOH-甲醇擦拭之。
KOH-甲醇配制为100ml甲醇中加5g片状KOH即可。
测序板的硅化处理2:1.浓度:4%二氯二甲基硅烷2.成分:二氯二甲基硅烷,三氯甲烷4.用途:制备聚丙稀酰胺凝胶时,可用Repel-silane处理小玻板,使凝胶易于剥离。
5.使用方法:测序用玻璃板必须彻底洗净。
先用温水和洗涤剂洗净,用水洗掉洗涤剂,再用去离子水冲洗干净。
最后用乙醇洗板。
1)长玻璃板的处理每次铺胶前均需用亲和硅烷对长玻璃板进行处理。
1、用镜头纸蘸取亲和硅烷溶液少许(1ml左右),涂在长玻璃板上。
要将整块板都涂满、涂匀。
2、4~5分钟后,用95%乙醇洗长玻璃板三次,以去除多余的亲和硅烷。
2)短玻璃板的处理每次铺胶前均需用剥离硅烷对短玻璃板进行硅化处理。
1、处理短玻璃板前先更换手套,以防与亲和硅烷交叉污染。
2、用镜头纸蘸取剥离硅烷溶液适量(1.5ml左右),涂在短玻璃板上。
银染中每步的原理

银染中每步的原理
银染是一种将金属银沉积在物体表面的染色方法,主要用于改变物体的颜色和增加其抗氧化能力。
其原理主要包括以下几个步骤:
1. 表面处理:首先需要对物体表面进行适当的处理,以去除表面的杂质和氧化物,以便银能够更好地沉积在物体表面。
常用的表面处理方法有机械打磨、电化学抛光等。
2. 银溶液制备:制备含有银离子的溶液,通常使用银盐(如硝酸银)和适当的酸性溶液配制而成。
溶液的酸性有助于提供适当的环境,使银离子可以稳定存在,同时可调节酸碱度来控制银沉积的速率和均匀性。
3. 沉积过程:将待染物体放入银溶液中,并加上适当的电压,通过电解作用将银离子还原成金属银,并沉积在物体表面,形成一层均匀的银膜。
电压的选取要根据染色效果和物体材质等因素进行调节。
4. 清洗和后处理:将染色后的物体从银溶液中取出,经过适当的清洗,以去除残留的银离子和其它杂质。
清洗后,还可以进行一些后处理步骤,如漂白、封闭等,以增加染色层的光亮度和耐久性。
总的来说,银染的原理是通过电解作用,将银离子沉积在物体表面,形成一层均匀的银膜。
这一过程的关键是控制电解条件和后处理步骤,以确保银染效果的稳
定和持久。
银染方法

A modified silver staining protocol for visualization of proteins compatible with matrix-assisted laser desorption/ionization and electrospray ionization-mass spectrometryThe growing availability of genomic sequence information,together with improvements in analytical methodology,have enabled high throughput,high sensitivity protein identi-fication.Silver staining remains the most sensitive method for visualization of proteins separated by two-dimensional gel electrophoresis (2-D PAGE).Several silver staining protocols have been developed which offer improved compatibility with subsequent mass spectrometric analysis.We describe a modified silver staining method that is available as a commercial kit (Silver Stain PlusOne;Amersham Pharmacia Biotech,Amersham,UK).The 2-D patterns abtained with this modified protocol are comparable to those from other silver staining methods.Omitting the sensitizing reagent allows higher loading without saturation,which facilitates protein identification and quantita-tion.We show that tryptic digests of proteins visualized by the modified stain afford excellent mass spectra by both matrix-assisted laser desorption/ionization and tandem electrospray ionization.We conclude that the modified silver staining protocol is highly compatible with subsequent mass spectrometric analysis.Keywords:Proteomics /Two-dimensional gel electrophoresis /Silver stain /Mass spectrometry /Protein identification /Matrix assisted laser desorption/ionization ±time of flight /Electrospray ionization ±time of flightEL 4190Jun X.Yan 1Robin Wait 2Tom Berkelman 3Rachel A.Harry 1Jules A.Westbrook 1Colin H.Wheeler 1Michael J.Dunn 11Department of Cardiothoracic Surgery,National Heart and Lung Institute,ImperialCollege School of Medicine,Heart Science Center,Harefield Hospital,Harefield,Middlesex,UK 2Kennedy Institute ofRheumatology,Hammersmith,London,UK 3Amersham Pharmacia Biotech,San Francisco,CA,USAThe increasing availability of genomic sequence informa-tion,together with improvements in protein characteriza-tion by mass spectrometry,have facilitated huge in-creases in the throughput of protein identification.Most commonly,sample components are separated by two-dimensional gel electrophoresis (2-D PAGE)and protein spots are visualized by staining silver,Coomassie blue or SYPRO fluorescent dyes [1±3].Individual spots are then excised from the gel,proteolytically digested,and the masses of the resulting peptides are determined by matrix assisted laser desorption/ionization ±time of flight ±mass spectrometry (MALDI-TOF-MS).The list of peptide masses thus obtained can then be used as a highly spe-cific query to interrogate a protein database [4±9].Recent advances in tandem electrospray ionization-mass spec-trometry (ESI-MS/MS),particularly the development of hybrid quadrupole /orthogonal acceleration TOF instru-ments (Q-TOF),enable routine de novo sequencing of low femtomole levels of peptides [10±13].The ability to determine 10±20amino acid lengths of sequence greatly facilitates cross-species protein identification and retrieval of homologous proteins from genomic and EST data-bases,even when an exactly matching sequence is not present.It is desirable to visualize protein spots in the gel at sensitivities which are roughly comparable to those of the subsequent MALDI-and ESI-MS analyses (usually in the range of nanograms per protein spot).Silver staining has been widely used for this purpose [14,15]since it re-quires relatively inexpensive equipment and reagents and remains one of the most sensitive methods for perma-nently staining proteins in polyacrylamide gels.For protein silver staining,a polyacrylamide gel is soaked in a solution containing soluble silver ions (Ag +)and sub-sequently developed by treatment with a reductant.Pro-tein molecules in the gel promote the reduction of silver ions to metallic silver (Ag 0),which is insoluble and visible.Initial deposition of metallic silver promotes further depo-sition by an autocatalytic process,resulting in exception-ally high sensitivity.There are many published versions of the silver staining process [16,17],which may incorpo-rate,in addition to silver impregnation and development,fixation steps,incubations with sensitivity enhancers (e.g.,glutaraldehyde or formaldehyde),stopping and preservation,and washing steps.The reagents used vary,but the silver reductant is always formaldehyde.The high sensitivity of silver staining comes at the cost of sus-ceptibility to interference from a variety of scources.Correspondence:Dr.Jun X.Yan,Heart Science Center,Hare-field Hospital,Hill End Road,Harefield,Middlesex,UB96JH,UK E-mail:jun.yan@ Fax:+44-(0)1895-828-9003666Electrophoresis 2000,21,3666±3672WILEY-VCH Verlag GmbH,69451Weinheim,20000173-0835/00/1717-3666$17.50+.50/0Exceptional cleanliness must therefore be practiced and reagent and water quality are critical.Silver staining pro-tocols have been developed specifically for visualizingproteins prior to in-gel digestion and mass spectrometric analysis [14,18].Subsequent MS constrains the choice of reagents that can be used during silver staining,because the proteins in the gel must not be chemically modified.Thus many common sensitization reagents (e.g.,glutaraldehyde and strong oxidizing agents)cannot be employed.Since silver staining is a multistep process utilizing numerous reagents,the quality of which is critical,it is often advantageous to purchase a dedicated kit in which the reagents are quality-assured specifically for sil-ver staining.We report here a modified silver staining method that is available as a commercial kit (Silver Stain PlusOne;Amersham Pharmacia Biotech)and we show that it is compatible with subsequent in-gel digestion,MALDI,and ESI analysis.The method is based on that of Heukes-hoven and Dernick [19],but omits the use of glutaralde-hyde in the sensitization step and formaldehyde in the sil-ver impregnation step.The detailed protocol is shown in Table 1.Staining was performed in glass dishes and par-ticular care was taken to avoid contamination by keratin and other extraneous proteins.Electrophoresis 2000,21,3666±3672Silver staining compatible with mass spectrometric analysis 3667Table 1.The modified silver staining protocol using Silver Stain PlusOne kit Step Solution (250mL per gel)Time (min)1.Fix 25mL acetic acid,100mL methanol,125mL milli-Q water 152.Fix25mL acetic acid,100mL methanol,125mL milli-Q water153.Sensitization a)75mL methanol,10ml sodium thiosulfate (5%),17g30sodium acetate 165mL milli-Q water 4.Wash 250mL milli-Q water 55.Wash 250mL milli-Q water 56.Wash 250mL milli-Q water57.Silver a)25mL silver nitrate (2.5%),225mL milli-Q water 208.Wash 250mL milli-Q water 19.Wash 250mL milli-Q water110.Develop6.25g sodium carbonate,100m L formaldehyde,250mL milli-Q water 11.Stop 3.65g EDTA,milli-Q water 1012.Wash 250mL milli-Q water 513.Wash 250mL milli-Q water 514.Wash250mL milli-Q water5a)Omitting the use of glutaraldehyde in the sensitization step and formaldehyde in the sil-ver impregnation step.Working solutions are freshly made immediately prior tostaining.Figure 1.Gel image of normal rat left ventricle using IPG pH 3±10NL 2-D PAGE (12%T)with 100m g total protein loading and silver staining (Owl silver stain kit).P r o t e o m i c s a n d 2-D ENormal human and rat heart left ventricle tissues were used.Sample preparation and2-D PAGE were performed essentially according to Weekes et al.[20].We routinely use100m g total protein loading for analytical gels(pH 3±10NL)and the Owl silver stain kit(Owl Separation Sys-tem,Portsmouth,UK)for visualization.A typical image of one of these gels is shown in Fig.1.To investigate the sensitivity of the PlusOne kit,100,200,300and400m g total protein loading were used.The corresponding gel images are shown in Fig.2.The patterns obtained using the two different kits(Figs.1and2d)are very similar. This,therefore,facilitates comparisons between semipre-parative and analytical gels stained with conventional pro-tocols(e.g.,the Owl kit).Excellent patterns were achieved at higher protein loadings(200,300,and400m g;Fig.2a±c),whereas many spots display negative staining at these loadings when more sensitive staining methods,such as the Owl kit,are used(data not shown).Note that the high-er background obtained from the higher protein loading gels were removed using transform to autoscale the gel image in PDQuest2-D software version6.1(Bio-Rad, Hercules,CA,USA).A400m g total protein loading for IPG3±10strips appears to be optimal in that adequate concentrations of most spots are obtained for MS analy-3668J.X.Yan et al.Electrophoresis2000,21,3666±3672Figure2.Gel images of normal rat left ventricle using IPG pH3±10NL2-D PAGE(12%T)and modi-fied silver staining described in Table1(modified PlusOne silver stain kit)with total protein loading of(a)400m g,(b)300m g,(c)200m g and(d)100m g.sis,while minimizing excessive background and the for-mation of large spot clusters.Higher loadings are possi-ble,however,when using narrow-range IPG strips(data not shown).We investigated the compatibility of the PlusOne modified protocol with ESI-and MALDI-MS.While MALDI is rela-tively tolerant of salts and other contaminants[21,22], the ESI technique is much more susceptible to such inter-ference.Thus,it is necessary to validate the compatibility of the modified stain with both ionization methods.Figure 3shows a gel image of human heart left ventricle(400m g total protein loading)from which20proteins spots were excised for MS characterization.A modified sample prep-aration method was used,which incorporates a destain-ing step[15]to remove silver prior to in-gel digestion with trypsin[14].Aliquots(0.5m L)of the digest supernatant were applied directly to the MALDI target,after which,if necessary,the remainder of the sample was extracted, desalted,and analyzed by ESI-MS/MS.These data are summarized in Table.2.If the results from MALDI mass mapping were ambiguous,ESI-MS/MS was used to gen-erate amino acid sequence data suitable for sequence tag or similarity searching using BLAST.Figure4shows the MALDI mass spectrum of spot19which,when sub-mitted to database searching,retrieved a highly signifi-cant match to ubiquinol cytochrome C reductase.Figure 5a shows a MALDI mass spectrum obtained from spot4, which,when searched,did not find any unambiguous hits.Electrophoresis2000,21,3666±3672Silver staining compatible with mass spectrometric analysis3669Figure3.Gel image of human left ventricle using IPG pH3±10NL2-D PAGE(12%T)with400m gtotal protein loading.The gel was stained using the PlusOne silver stain kit.Protein spots(1±20)labeled on the image were subjected to trypsin digestion and MALDI-TOF-MS or ESI-MS(Table2).This protein was identified by ESI-MS/MS of a doubly charged ion at m/z 777.4,from which 13residues of amino acid sequence were deduced (Fig.5b),which exactly matched the sequence of ATPsynthase a -chain.We conclude that the modified silver staining kit is com-patible with both MALDI-and ESI-MS.Although the sensi-tivity is somewhat lower than other versions,this kit ena-bles higher protein loading,thus facilitating identification by MS.In our laboratory the modified protocol has pro-vided consistent results over a 12-month period and,since the resulting patterns are similar to those produced by the Owl silver stain kit,we have been able to correlate results from semipreparative and analytical gels.Spots of interest are easily located for excision and further charac-terization.A loading of 400m g protein on IPG 3±10strips provides adequate concentrations for successful MALDI analysis of the majority of visible spots.For very low abu-dance proteins,use of the modified stain in conjunction with high protein loadings on narrow pH range IPG strips avoids excessive background staining and spot cluster-ing.We thank the Cardiovascular Disease Group,Rhone-Poulenc Rorer (Collegeville,PA,USA)for providing the rat heart tissue,and Tim Harwood,Amersham PharmaciaElectrophoresis 2000,21,3666±3672Silver staining compatible with mass spectrometric analysis3671T a b l e 2.c o n t i n u e dS p o t E s t i m a t e d I d e n t i f i c a t i o n (a c c e s s i o n n u m b e r )T h e o r e t i c a l A m i n o a c i d P e p t i d e s e q u e n c e i n f o r m a t i o n o b t a i n e d b y E S I -T O F -M S /M S a n a l y s i sN o .p I /M r (D a )p I /M r (D a )c o v e r a g e (%)w i t h M A L D I -T O F -M S 195.7/46200U b i q u i n o l -c y t o c h r o m e c r e d u c t a s e c o m p l e x c o r e p r o t e i n I 5.94/5261939.31.m /z 787.5(3+)A V E L L G D I V Q N C S L E D S Q I E K p r e c u r s o r ,h u m a n (P 31930)2.m /z 901.5(3+)A G Y G P L E Q L P D Y N R3.m /z 1044.3(3+)...F Q G T P L A Q A V E G P S E N V R 4.m /z 1180.7(2+)A V E L L G D I V Q N C S L E D S Q I E K5.m /z 1351.7(2+)Y I I D Q C P A V A G Y Y P I E Q L P D Y N R 205.9/40700A c t i n ,a -c a r d i a c ,h u m a n (P 03996)5.24/4200930.21.m /z 565.9(2+)G Y S F V T T A E R2.m /z 652.7(3+)V A P E E H P T L L T E A P L N P K3.m /z (2+)P y r -E Y D E A G P S I V H RP r o t e i n s p o t s 1±20w e r e e x c i s e d f r o m t h e 2-D g e l s h o w n i n F i g .3a n d a n a l y z e d b y M A L D I -a n d E S I -M S .T h e r e s u l t i n g p e p t i d e m a s s m a p s w e r e s e a r c h e d a g a i n s t S W I S S -P R O T /T r E M B L r e l e a s e 35,u s i n g P r o t e i n P r o b e (M i c r o m a s s ),o r a g a i n s t a n o n r e d u n d a n t d a t a b a s e m a i n t a i n e d b y t h e N a t i o n a l C e n t e r f o r B i o t e c h n o l o g y I n f o r m a t i o n (N C B I )(h t t p ://w w w .n c b i.n l m .n i h .g o v )u s i n g t h e M a s c o t [23]s e a r c h e n g i n e (h t t p ://w w w .m a t r i x s c i e n c e .c o .u k ).A n i n i t i a l m a s s t o l e r a n c e o f 100p p m w a s u s e d ,b u t w a s r e d u c e d t o 50p p m i f e x c e s s i v e n u m b e r s o f h i t s w e r e r e t r i e v e d .A m i n o a c i d s e q u e n c e s o b t a i n e d f r o m E S I -M S /M S w e r e s e a r c h e d a g a i n s t a n o n r e d u n -d a n t d a t a b a s e i n N C B I u s i n g t h e B L A S T p r o g r a m [23].M S /M S s h o w e d t h a t s p o t 1c o n t a i n e d t w o -c o m i g r a t i n g p r o t e i n s .S p o t s 6,8±13,a n d 15±17w e r e n o t a n a l y z e d b y M S /M S b e c a u s e t h e s e a r c h r e s u l t s f r o m t h e M A L D I d a t a w e r e u n a m b i g u o u s .T h e s e q u e n c e c o v e r a g e o f 6,8,9a n d 12a p p e a r s l o w ,b e c a u s e t h e o b s e r v e d p r o -t e i n s p o t s c o r r e s p o n d t o t r u n c a t e d f o r m s ,w h e r e a s t h e c o v e r a g e w a s c a l c u l a t e d f r o m t h e f u l l -l e n g t h s e q u e n c e.Biotech,for conducting this collaboration.JXY acknowl-edges Aventis for their financial support.RAH and JAW thank Proteome Sciences Inc.for their financial support. RW thanks the Wellcome Trust for purchase of the MALDI spectrometer.Work in MJD©s laboratory is sup-ported by the British Heart Foundation.Received May,30,2000References[1]Berggren,K.,Chernolalskaya,E.,Steinberg,T.H.Kemper,C.,Lopez,M.F.,Diwu,Z.,Haugland,R.P.,Patton,W.F.,Electrophoresis2000,21,2509±2521.[2]Steinberg,T.H.,Lauber,W.M.,Berggren,K.,Kemper,C.,Yue,S.,Patton,W.F.,Electrophoresis2000,21,497±508.[3]Steinberg,T.H.,Jones,L.J.,Haugland,R.P.,Singer,V.L.,Anal.Biochem.1996,239,223±237.[4]Henzel,W.J.,Billeci,T.M.,Stults,J.T.,Wong,S.C.,Grim-ley,C.,Watanabe,C.,A1993,90, 5011±5015.[5]Mann,M.,Hojrup,P.,Roeppstorff,P.,Biol,Mass Spectrom.1993,22,338±345.[6]Pappin,D.J.C.,Hojrup,P.,Bleasby,A.J.,Curr.Biol.1993,3,327±332.[7]Liang,X.L.,Bai,J.,Liu,Y.H.,Lubman,D.M.,Anal.Chem.1996,68,1012±1018.[8]Patterson,S.D.,Aebersold,R.,Electrophoresis1995,16,1791±1814.[9]Wheeler,C.H.,Berry,S.L.,Wilkins,M.R.,Corbett,J.M.,Ou,K.,Golley,A.A.,Humphery-Smith,I.,Williams,K.L., Dunn,M.J.,Electrophoresis1996,7,580±587.[10]Morris,H.R.,Paxton,T.,Dell,A.,Langhorne,J.,Berg,M.,Bordoli,R.S.,Hoyes,J.,Bateman,R.H.,Rapid Commun.Mass Spectrom.1996,10,889±896.[11]Shevchenko,A.,Chernushevich,I.,Ens,W.,Standing,K.G.,Thomson,B.,Wilm,M.,Mann,M.,Rapid Commun.Mass Spectrom.1997,11,1015±1024.[12]Borchers,C.,Peter,J.F.,Hall,M.C.,Kunkel,T.A.,Tomer,K.B.,Anal.Chem.2000,72,1163±1168.[13]Kristensen,D.B.,Imamura,K.,Miyamoto,Y.,Yoshizato,K.,Electrophoresis2000,21,430±439.[14]Shevchenko,A.,Wilm,M.,Vorm,O.,Mann,M.,Anal.Chem.1996,68,850±858.[15]Gharahdaghi,F.,Weinberg,C.R.,Meagher,D.A.,Imai,B.S.,Mische,S.M.,Electrophoresis1999,20,601±605.[16]Rabilloud,T.,Electrophoresis1990,11,785±794.[17]Rabilloud,R.,Electrophoresis1992,13,429±439.[18]Arnott,D.,O©Connell,K.L.,King,K.L.,Stults,J.T.,Anal.Biochem.1998,258,1±18.[19]Heukeshoven,J.,Dernick,R.,Electrophoresis1985,6,103±112.[20]Weekes,J.,Wheeler,C.H.,Yan,J.X.,Weil,J.,Eschenha-gen,T.,Scholtysik,G.,Dunn,M.J.,Electrophoresis1999, 20,898±906.[21]Garden,R.W.,Moroz,L.L.,Moroz,T.P.,Shippy,S.A.,Sweedler,J.V.,J.Mass Spectrom.1996,31,1126±1130.[22]Winkler,M.A.,Kundu,S.,Robey,T.E.,Robey,W.G.,J.Chromatogr.A1996,744,177±185.[23]Perkins,D.N.,Pappin,D.J.,Creasy,D.M.,Cottrell,J.S.,Electrophoresis1999,20,3551±3567.[24]Altschul,S.F.,Madden,T.L.,Schäffer,A.A.,Zhang,J.,Zhang,Z.,Miller,W.,Lipman,D.J.,Nuclei Acids Res.1997, 25,3389±3402.[25]Vorm,O.,Mann,M.,J.Am.Soc.Mass Spectrom.1994,5,955±958.3672J.X.Yan et al.Electrophoresis2000,21,3666±3672。
蛋白质银染原理与方法

原理:蛋白质条带的银染是基于蛋白质中各种基团(如琉基、碳基等)与银的结合,检测极限是2~/蛋白条带。
① SDS-PAGE电泳。
注意:银染检测蛋白质的水平是在100ng左右,与CommassieBlue染色比较,银染加样量要少。
否则难以得到清晰的电泳分辨率。
②电泳结束后,戴手套将PAGE胶转移到干净的玻璃培养皿或TIP头盒盖中。
加入25ml FixingSolution(固定液),室温振荡保温30min。
胶需要完全淹没在固定液中。
③彻底弃去固定液,加入25mlIncubation Solution(保育液),室温振荡保温30min。
胶需要完全淹没在保育液中。
④彻底弃去IncubationSolution(保育液),用50ml去离子水洗3次,每次5min。
⑤准备:取10XSilvering Solution, 加入水,混匀后使用。
⑥弃去洗涤的水,加入25mlSilvering Solution (银染液)。
室温振荡保温20min。
⑦彻底弃去银染液,加入25mLDevelopingSolution(显色液),室温振荡保温1~10min。
注意观察目的条带。
如果目的条带出现了,可以考虑终止显色。
显色时间不要过长,否则本底较深。
⑧彻底弃去显色液,加入25mLStopping Solution(终止液),室温振荡保温10min。
⑨彻底弃去StoppingSolution(终止液),用50mL去离子水洗3次,每次5min。
弃去洗涤的水,加入25mL PreservingSolution(保护液),室温振荡保温20min。
银染PAGE胶可以拍照保存,也可以室温玻璃纸干燥。
今天第一次银染,结果出来啥也没有,凝胶就像一块海带一样。
能不能提供详细的银染步骤呢?好像有好几种版本,哪位大侠有经验的希望能点拨一下?hotstoe wrote:1. 取下凝胶,蒸馏水冼胶30秒.2. % 硝酸银染色10分钟.3. 弃去染色液,蒸馏水洗胶1分钟.4.显色液显色15秒,换用新的显色液,显清条带为止.显色液配方: 10克氢氧化钠+克碳酸钠+2m甲醛溶液,定溶到500ml.这个染色方法挺好的,我用过.注意染色时间不能长.我的经验是:1.取下凝胶用去离子水冼胶30秒三次,硝酸银染色10分钟.3.弃去染色液,蒸馏水洗胶1分钟(换2次去离子水)4.显色液显色15秒,换用新的显色液,显清条带为止.不要试图将所有的条带都显出来,要求越高可能导致背景就深,5.染色后要用固定液(冰乙酸)终止染色,6.固定3分钟后再用去离子水洗涤2遍(整个试验比较费去离子水)。
考染与银染

常用的蛋白质染色试剂分为已考马斯亮蓝为代表的有机试剂染色、银染、荧光染色及同位素显色。
其中考马斯亮蓝染色法的应用较为广泛,现将其与其他的蛋白质染色方法(主要是银染法)作一比较,帮助大家更好地去选择合适的蛋白质染色方法。
蛋白质的染色常用的有4类:有机试剂染色、银染、荧光染色及同位素显色。
其中有机试剂染色以考马斯亮蓝染色法(Coomasie brilliant blue,CBB)为代表,在蛋白质分析中常用,但对低丰度蛋白质的显现较差;银染灵敏度虽高,却常与质谱不兼容;荧光染色以SYPRO试剂为主,蛋白质检测灵敏度高,能兼容质谱,但由于需要配备特殊的检测仪器及试剂的昂贵,未被作为常规方法使用;而同位素显色则存在安全性和操作局限性等问题。
因此,筛选简便、节约、检测灵敏度高、质谱兼容的蛋白质着色法是蛋白质组研究所需。
由于考马斯亮蓝染色法的广泛运用,近年来就考马斯亮蓝染色法在提高其灵敏性方面研究者们作了许多改进,方法众多,评价不一。
我们在作双向凝胶电泳时,将常用的几种考马斯亮蓝染色法及银染进行了比较,并就其染色影响因素作出分析。
试剂固相pH梯度干胶条(IPG strip pH3-10NL,IPG strip pH5-8,17cm)、urea、CHAPS、TBP、DTT、Bio-Lyte 3/10 Ampholyte、IAM、SDS、Tris、丙烯酰胺、甲叉双丙烯酰胺、甘氨酸、CBB-G250均为BIO-RAD公司产品。
硫酸铵及磷酸均购自成都科龙试剂厂。
AgNO3为Sigma公司出品。
Protein molecular weight marker #SW0431为MBI公司出品。
甲醛、Na2S2O3与Na2CO3分别为成都天华科技股份有限公司、重庆北碚化学试剂厂和天津市塘沽鹏达化工厂产品。
仪器IPGphor等电聚焦仪、proTEANⅡ垂直电泳仪、Power PAC1000电泳仪、加样溶胀槽、GS-800 Calibrated Densitometer扫描仪、PDQuest凝胶图像分析软件均为BIO-RAD公司产品。
考染和银染单点鉴定

方法:质谱法原理:一、利用MALDI-TOF质谱仪根据肽质量指纹图谱(Peptide Mass Fingerprinting, PMF)与蛋白质数据库中蛋白质的理论PMF比对,就可以鉴定该蛋白质。
优点:速度快,通量高,方法简便。
缺点:灵敏度不足,可检测到的肽段少,对数据库检索是不利的。
肽段分子量精确度往往不够理想,影响鉴定结果。
肽段分子质量在2000以上时,在MALDI-TOF-MS中的分辨率会有所下降。
蛋白质数据库仍不完善。
二、利用ESI-MS质谱仪由于MS/MS具有结构分析功能,可以获得检测到的每一肽段的序列。
根据一级质谱(MS)测得的精确分子质量和串联质谱(MS/MS)所得的序列信息,通过蛋白质数据库的检索鉴定蛋白质种类。
优点:通过串联质谱大大增加了蛋白质鉴定的准确性。
对图谱的精确度和分辨率要求不如PMF高。
采用纳升喷雾提高检测的灵敏度。
缺点:通量与速度不如MALDI。
数据库检索比较费时。
仪器:BrukerAutoflex Ⅱ(MALDI-TOF-TOF-MS);Finnigan LCQ Deca Xp / Finnigan LTQ样品要求:1、防止角蛋白污染2、样品状态:液体、固体或胶内蛋白质(接受考马斯亮兰染色和银染样品鉴定)3、含盐量:挥发性无机盐< 20 mM; 不挥发性无机盐< 5mM4、不少于200 ug /一块胶(银染);5、胶内蛋白质鉴定,蛋白质样品量不少于1mg/一块胶(考马斯亮兰染色)蛋白质鉴定结果常见问题:1、由于角蛋白等蛋白质的污染,造成鉴定为污染蛋白。
2、由于蛋白量过少引起鉴定结果的评分低,可信度不高。
3、由于蛋白质纯度不足,造成鉴定结果为混合蛋白质。
4、由于蛋白质为未知蛋白,造成鉴定无结果。
注意事项:1、推荐采用考马斯亮兰染色。
因为该方法的成功率较高,而银染的成功率则会低很多。
2、银染的方法为快速银染法。
常规银染法不适用质谱鉴定。
3、考马斯亮蓝染色样品的上样量>1mg4、银染法得上样量>0.6mg。
蛋白质银染原理与方法

原理:蛋白质条带的银染是基于蛋白质中各种基团(如琉基、碳基等)与银的结合,检测极限是2~5.0ng/蛋白条带。
① SDS-PAGE电泳。
注意:银染检测蛋白质的水平是在100ng左右,与CommassieBlue染色比较,银染加样量要少。
否则难以得到清晰的电泳分辨率。
②电泳结束后,戴手套将PAGE胶转移到干净的玻璃培养皿或TIP头盒盖中。
加入25ml FixingSolution(固定液),室温振荡保温30min。
胶需要完全淹没在固定液中。
③彻底弃去固定液,加入25mlIncubation Solution(保育液),室温振荡保温30min。
胶需要完全淹没在保育液中。
④彻底弃去IncubationSolution(保育液),用50ml去离子水洗3次,每次5min。
⑤准备:取2.5ml 10XSilvering Solution, 加入22.5mL水,混匀后使用。
⑥弃去洗涤的水,加入25mlSilvering Solution (银染液)。
室温振荡保温20min。
⑦彻底弃去银染液,加入25mLDevelopingSolution(显色液),室温振荡保温1~10min。
注意观察目的条带。
如果目的条带出现了,可以考虑终止显色。
显色时间不要过长,否则本底较深。
⑧彻底弃去显色液,加入25mLStopping Solution(终止液),室温振荡保温10min。
⑨彻底弃去StoppingSolution(终止液),用50mL去离子水洗3次,每次5min。
弃去洗涤的水,加入25mL PreservingSolution(保护液),室温振荡保温20min。
银染PAGE胶可以拍照保存,也可以室温玻璃纸干燥。
今天第一次银染,结果出来啥也没有,凝胶就像一块海带一样。
能不能提供详细的银染步骤呢?好像有好几种版本,哪位大侠有经验的希望能点拨一下?hotstoe wrote:1. 取下凝胶,蒸馏水冼胶30秒.2. 0.1% 硝酸银染色10分钟.3. 弃去染色液,蒸馏水洗胶1分钟.4.显色液显色15秒,换用新的显色液,显清条带为止.显色液配方: 10克氢氧化钠+0.2克碳酸钠+2m甲醛溶液,定溶到500ml.这个染色方法挺好的,我用过.注意染色时间不能长.我的经验是:1.取下凝胶用去离子水冼胶30秒三次,2.0.1% 硝酸银染色10分钟.3.弃去染色液,蒸馏水洗胶1分钟(换2次去离子水)4.显色液显色15秒,换用新的显色液,显清条带为止.不要试图将所有的条带都显出来,要求越高可能导致背景就深,5.染色后要用固定液(冰乙酸)终止染色,6.固定3分钟后再用去离子水洗涤2遍(整个试验比较费去离子水)。
考染与银染资料

常用的蛋白质染色试剂分为已考马斯亮蓝为代表的有机试剂染色、银染、荧光染色及同位素显色。
其中考马斯亮蓝染色法的应用较为广泛,现将其与其他的蛋白质染色方法(主要是银染法)作一比较,帮助大家更好地去选择合适的蛋白质染色方法。
蛋白质的染色常用的有4类:有机试剂染色、银染、荧光染色及同位素显色。
其中有机试剂染色以考马斯亮蓝染色法(Coomasie brilliant blue,CBB)为代表,在蛋白质分析中常用,但对低丰度蛋白质的显现较差;银染灵敏度虽高,却常与质谱不兼容;荧光染色以SYPRO试剂为主,蛋白质检测灵敏度高,能兼容质谱,但由于需要配备特殊的检测仪器及试剂的昂贵,未被作为常规方法使用;而同位素显色则存在安全性和操作局限性等问题。
因此,筛选简便、节约、检测灵敏度高、质谱兼容的蛋白质着色法是蛋白质组研究所需。
由于考马斯亮蓝染色法的广泛运用,近年来就考马斯亮蓝染色法在提高其灵敏性方面研究者们作了许多改进,方法众多,评价不一。
我们在作双向凝胶电泳时,将常用的几种考马斯亮蓝染色法及银染进行了比较,并就其染色影响因素作出分析。
试剂固相pH梯度干胶条(IPG strip pH3-10NL,IPG strip pH5-8,17cm)、urea、CHAPS、TBP、DTT、Bio-Lyte 3/10 Ampholyte、IAM、SDS、Tris、丙烯酰胺、甲叉双丙烯酰胺、甘氨酸、CBB-G250均为BIO-RAD公司产品。
硫酸铵及磷酸均购自成都科龙试剂厂。
AgNO3为Sigma公司出品。
Protein molecular weight marker #SW0431为MBI公司出品。
甲醛、Na2S2O3与Na2CO3分别为成都天华科技股份有限公司、重庆北碚化学试剂厂和天津市塘沽鹏达化工厂产品。
仪器IPGphor等电聚焦仪、proTEANⅡ垂直电泳仪、Power PAC1000电泳仪、加样溶胀槽、GS-800 Calibrated Densitometer扫描仪、PDQuest凝胶图像分析软件均为BIO-RAD公司产品。
PAGE银染方法

一、实验原理银染是一种重要的PAGE染色方法,由于其成本低,所用试剂安全、快速、灵敏度高而被广泛应用。
银染的原理是银离子在碱性pH 环境下被还原成金属银,沉淀在蛋白质的表面上而显色。
由于银染的灵敏度很高,可染出凝胶上低于1 ng/蛋白质点,故广泛的用在2D凝胶分析上,及极低蛋白含量测定的垂直凝胶中。
这里介绍的是一种实验室常用的银染方法,主要是用于垂直凝胶电泳中低丰度蛋白的检测。
二、实验试剂1. 乙醇、冰醋酸、乙酸钠、硫代硫酸钠、硝酸银、碳酸钠、甘氨酸或EDTA钠盐、去离子水、甲醛。
三、实验步骤1. 固定量取100ml 乙醇,25ml 冰醋酸加去离子水到250ml(视胶板大小情况,适当等比例配制,增加固定液量,以下相同。
),使其终浓度达到 40% 乙醇,10% 冰醋酸。
将凝胶板浸入固定液中,固定30min以上。
2. 水洗去离子水浸泡。
3x 10 min。
2. 致敏生物科研提醒:量取75ml 乙醇,17g 乙酸钠或28.2g三水乙酸钠,0.5g硫代硫酸钠加去离子水到体积250ml。
将固定后的凝胶板拿起,浸入致敏液中,浸泡30 min。
4. 水洗去离子水浸泡。
3x 10 min。
5.银染0.625g AgNO3、100 ul 37%甲醛(在使用前加入)加去离子水到终体积250ml。
将胶板浸入银染液中,浸泡30 min。
6.水洗去离子水浸泡。
3x 20 sec。
注意把握时间,水洗时间长显色速度慢,点的颜色偏黄色。
水洗不充分,背景较深。
7.显色6.25 g Na2CO3、50 ul 37% 甲醛(在使用前加入)加去离子水到终体积250ml。
2-15min,显色时间视凝胶大小、厚薄情况而定。
看到各条带,即可将凝胶捞出。
8.终止3.65g EDTA钠盐或者1g 甘氨酸加去离子水到终体积250ml。
浸泡凝胶10 min。
9.保存1% 冰醋酸,4 ℃。
四、注意事项1.银染主要出现在胶的表面,用薄胶(0.5-0.75mm)可以提高灵敏度。
蛋白质银染原理与方法

原理:蛋白质条带的银染是基于蛋白质中各种基团(如琉基、碳基等)与银的结合,检测极限是2~/蛋白条带。
① SDS-PAGE电泳。
注意:银染检测蛋白质的水平是在100ng左右,与CommassieBlue染色比较,银染加样量要少。
否则难以得到清晰的电泳分辨率。
②电泳结束后,戴手套将PAGE胶转移到干净的玻璃培养皿或TIP头盒盖中。
加入25ml FixingSolution(固定液),室温振荡保温30min。
胶需要完全淹没在固定液中。
③彻底弃去固定液,加入25mlIncubation Solution(保育液),室温振荡保温30min。
胶需要完全淹没在保育液中。
④彻底弃去IncubationSolution(保育液),用50ml去离子水洗3次,每次5min。
⑤准备:取10XSilvering Solution, 加入水,混匀后使用。
⑥弃去洗涤的水,加入25mlSilvering Solution (银染液)。
室温振荡保温20min。
⑦彻底弃去银染液,加入25mLDevelopingSolution(显色液),室温振荡保温1~10min。
注意观察目的条带。
如果目的条带出现了,可以考虑终止显色。
显色时间不要过长,否则本底较深。
⑧彻底弃去显色液,加入25mLStopping Solution(终止液),室温振荡保温10min。
⑨彻底弃去StoppingSolution(终止液),用50mL去离子水洗3次,每次5min。
弃去洗涤的水,加入25mL PreservingSolution(保护液),室温振荡保温20min。
银染PAGE胶可以拍照保存,也可以室温玻璃纸干燥。
今天第一次银染,结果出来啥也没有,凝胶就像一块海带一样。
能不能提供详细的银染步骤呢?好像有好几种版本,哪位大侠有经验的希望能点拨一下?hotstoe wrote:1. 取下凝胶,蒸馏水冼胶30秒.2. % 硝酸银染色10分钟.3. 弃去染色液,蒸馏水洗胶1分钟.4.显色液显色15秒,换用新的显色液,显清条带为止.显色液配方: 10克氢氧化钠+克碳酸钠+2m甲醛溶液,定溶到500ml.这个染色方法挺好的,我用过.注意染色时间不能长.我的经验是:1.取下凝胶用去离子水冼胶30秒三次,硝酸银染色10分钟.3.弃去染色液,蒸馏水洗胶1分钟(换2次去离子水)4.显色液显色15秒,换用新的显色液,显清条带为止.不要试图将所有的条带都显出来,要求越高可能导致背景就深,5.染色后要用固定液(冰乙酸)终止染色,6.固定3分钟后再用去离子水洗涤2遍(整个试验比较费去离子水)。
考马斯亮蓝染色的原理和方法

考马斯亮蓝染色的原理和方法在蛋白质染色方法中,目前以考马斯亮蓝染色最为常用。
因为它既克服了氨基黑染色灵敏度不高的限制,又比银染简便易操作。
考染的历史考马斯亮蓝染色最早由Fazekas等在1963年用于醋酸纤维素膜电泳,并认为同样可用于纸电泳,琼脂糖电泳和淀粉凝胶电泳。
1965年Meyer等将此方法应用于聚丙烯酰胺凝胶电泳。
考染的灵敏度考马斯亮蓝染色可以达到0.2~0.5 μg(200~500 ng),最低可检出0.1 μg蛋白;银染的灵敏度比考染高将近100倍,最低可以检出2 ng蛋白。
通过考染条带的深浅粗细,可以大致判断该条带有多少蛋白,如在8.6×6.8 cm(W×L)?.75 mm厚的胶上15~20 μg蛋白大约是下面这样的条带:考马斯亮蓝染料的种类考马斯亮蓝分为R-150、R-250、R-350、G-250等。
其中R-350最为灵敏,R为红蓝色,G 为绿蓝色。
R-250即三苯基甲烷,每个分子含有两个SO3H基团,偏酸性,与氨基黑一样也是结合到蛋白质的碱性基团上。
G-250即二甲花青亮蓝,是一种甲基取代的三苯基甲烷。
推荐一种考染的方法⑴电泳后的凝胶立即浸泡在固定液(500 ml 乙醇,100 ml 冰醋酸,400 ml 蒸馏水)中至少30 min,如有必要可在固定液中浸泡过夜;⑵将固定后的凝胶在热的染色液(0.29 g 考马斯亮蓝溶解在250 ml 脱色液中,在使用前边搅拌边加热至60℃)中浸泡10min,然后用蒸馏水将凝胶淋洗一次;⑶多次变换脱色液(250 ml 乙醇,80 ml冰醋酸,加蒸馏水至1 L),直至凝胶背景脱净为止,为加快脱色,可略加温度;以上各步最好用振荡器(摇床)震荡染色皿。
⑷为了防止凝胶干燥后的龟裂,脱去背景色的凝胶在保存液(25 ml 87% 甘油加225 ml 脱色液)中浸泡30min。
然后将凝胶放在玻璃板上,再用保存液浸湿玻璃纸包住凝胶在室温下晾干。
银染步骤及注意事项

银染步骤是1 固定10冰乙酸min。
2 清洗双蒸水冲洗凝胶次每次1min。
3 染色染色液1g硝酸银.5mL37甲醛1L双蒸水。
现配染色30min。
4 清洗迅速洗凝胶次。
5 显影30g 碳酸钠1.5mL37甲醛200uL 10mg/L硫代硫酸钠。
显影至清晰带纹出现。
成功银染的关键因素包括 1 用超纯水比如NANOpure或Milli-Q纯化或者是双蒸水作银染。
2 用提供的碳酸钠或者ACS试剂级的碳酸钠。
3 在染色后水清洗所用时间的长短很重要用不超过5-10秒的时间清洗胶然后放入显色溶液中一般在水里浸一下就好。
4 甲醛和硫代硫酸钠ul/1ml在使用前及时加入到显色液中。
5 在使用前及时配染色液。
银染的方法种类很多目前有文献报道的就有100 多种。
但是其准确的染色机制还不是特别的清楚。
大致的原理是银离子在碱性pH 环境下被还原成金属银沉淀在蛋白质的表面上而显色。
由于银染的灵敏度很高可染出胶上低于1 ng/蛋白质点故广泛的用在2D 凝胶分析上及极低蛋白含量测定的垂直凝胶中。
这里介绍的是我们实验室成功运用的银染方法对关键操作及要求做了补充主要是用于垂直凝胶电泳中低丰度蛋白的检测如内源性GST-Pulldown、Co-IP 等实验中相互作用蛋白的研究银染操作规程实验目的检测聚丙烯酰胺凝胶中的蛋白质。
实验原理在碱性条件下用甲醛将蛋白带上的硝酸银银离子还原成金属银以使银颗粒沉积在蛋白带上。
染色的程度与蛋白中的一些特殊的基团有关不含或者很少含半胱氨酸残基的蛋白质有时候呈负染。
银染的详细机制还不是非常清楚。
试剂乙醇、冰醋酸、乙酸钠、硫代硫酸钠、硝酸银、碳酸钠、甘氨酸或EDTA.Na2.2H2O、甲醛实验操作程序固定30min或者更长时间o 100ml 乙醇40 乙醇o 25ml冰醋酸10 冰醋酸o 加水到250ml 致敏30min o 75ml 乙醇30 乙醇o 17g 乙酸钠28.2g 三水乙酸钠o 0.5g硫代硫酸钠o 加水到终体积250ml 水洗3 x10min 银染20min o 0.625g AgNO3 o 100 ul 37甲醛在使用前加入o 加水到终体积250ml 水洗2 x 1 min 注意把握时间水洗时间长显色速度慢点的颜色偏黄色显色视情况而定o 6.25 g Na2CO3 o 50 ul 37 甲醛在使用前加入o 加水到终体积250ml 终止10min o 3.65g EDTA.Na2.2H2O 或者1g 甘氨酸o 加水到终体积250ml 保存1 冰醋酸4 ℃注意事项1固定时间较长则加一步水洗30min以免胶太脆而破碎。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
常用的蛋白质染色试剂分为已考马斯亮蓝为代表的有机试剂染色、银染、荧光染色及同位素显色。
其中考马斯亮蓝染色法的应用较为广泛,现将其与其他的蛋白质染色方法(主要是银染法)作一比较,帮助大家更好地去选择合适的蛋白质染色方法。
蛋白质的染色常用的有4类:有机试剂染色、银染、荧光染色及同位素显色。
其中有机试剂染色以考马斯亮蓝染色法(Coomasie brilliant blue ,CBB)为代表,在蛋白质分析中常用,但对低丰度蛋白质的显现较差;银染灵敏度虽高,却常与质谱不兼容;荧光染色以SYPRO试剂为主,蛋白质检测灵敏度高,能兼容质谱,但由于需要配备特殊的检测仪器及试剂的昂贵,未被作为常规方法使用;而同位素显色则存在安全性和操作局限性等问题。
因此,筛选简便、节约、检测灵敏度高、质谱兼容的蛋白质着色法是蛋白质组研究所需。
由于考马斯亮蓝染色法的广泛运用,近年来就考马斯亮蓝染色法在提高其灵敏性方面研究者们作了许多改进,方法众多,评价不一。
我们在作双向凝胶电泳时,将常用的几种考马斯亮蓝染色法及银染进行了比较,并就其染色影响因素作岀分析。
试剂固相pH 梯度干胶条(IPG strip pH3-10NL , IPG strip pH5-8 , 17cm )、urea、CHAPS、TBP、DTT、Bio-Lyte裂解,静置30分钟,15500X g离心30分钟,取上清,进行蛋白质定量。
单向定量电泳取蛋白质分子量标准物,第一次按1 : 2稀释后,以后按1 : 3倍比例逐级稀释,经4级稀释后,取15卩l 上样液上样,性半乳糖苷酶上样量依次分别为1川、0.24 tig 0.062 tig、0.0156 0.004 心作12g/L SDS-聚丙烯酰胺凝胶单向电泳,同时电泳4份胶,显色以确定染色灵敏度。
双向电泳取3001样品液沿水化盘中槽的边缘自左向右线性加入,将17cm IPG胶条胶面朝下置于槽中,静置45分钟后覆盖矿物油,在20 C溶胀11〜16小时。
溶胀后的胶条置于等电聚焦盘中,在Protean IEF Cell中进行等电聚焦,温度设置为20 C,电压模式设置为:快速升压,250 V,0.5小时;500 V,0.5小时;10000 V,60000 Vh (伏特X小时)。
取IEF后的IPG胶条,先后分别置于含DTT平衡液1 (6mol/L urea , 2g/L SDS ,0.05mol/L Tris pH 8.8 , 200ml/L gylcecol , 2g/L )及含碘乙酰胺平衡液2 (同平衡液1,但其中DTT 换为2.5g/L碘乙酰胺)中轻微摇荡各10分钟。
将平衡好的IPG胶条置于预先灌制的12g/L SDS-聚丙烯酰胺凝胶上,封固,在15 'C低温水循环条件下,15 mA/gel电泳15分钟,然后以25mA/gel恒流电泳。
显色取出SDS-聚丙烯酰胺凝胶,经超纯水清洗2次,每次1分钟,4块胶分别按4种不同的染色方法显色。
下列每步操作皆在摇床上进行。
4种染色法成份组成及操作如下:①传统考马斯亮蓝染色法:清洗后的凝胶经固定液(45.4%甲醇、4.6%乙酸)固定1小时,随后用染色液(45.4%甲醇、4.6%乙酸、0.1% CBB G250 )着色过,再经洗脱液(5%甲醇、7.5%乙酸)过脱色至背景清晰。
②胶体考马斯亮蓝染色法(colloidalCoomassie brilliant blue ,cCBB )染色法:凝胶先经超纯水漂洗后,再用colloidal Coomassie blue G250染色液(1.6%磷酸、8%硫酸铵、0.02% CBB G250、20%乙醇)着色过,洗脱液为超纯水,换液3〜4次直至背景清晰。
③改良考马斯亮蓝染色法(modified Coomassie brilliant blue ,mCBB )染色法[6];操作方法同胶体考马斯亮蓝染色法,只是染色液组成不同(0.12% CBB G250、10%硫酸铵、10%磷酸、20%乙醇),着色1小时即可。
④银染(silver staining ):凝胶漂洗后先被固定液(50%甲醇+ 5%乙酸)固定1 小时,随后分别用50%甲醇和超纯水清洗各10分钟,0.02 mg/L Na2S2O3 致敏1分钟,超纯水清洗2次,每次1分钟,再用预冷的0.15% AgNO3着色20分钟,超纯水再次清洗2次,每次1分钟,2% Na2CO3 + 0.04%甲醛显色20〜30秒,当溶液变黄以后除去,换新鲜的溶液后继续显色直至显色适度后,以5%乙酸终止显色。
成像、分析、质谱鉴定脱色后,凝胶经GS-800 Calibrated Densitometer 扫描成像,用PDQuest软件进行分析。
切取蛋白质点,胰酶消化,行质谱鉴定。
结果4种染色法的蛋白质检测灵敏性分析伕半乳糖苷酶的上样量依次分别为1、0.24、0.062、0.0156和0.004 qg经传统考马斯亮蓝染色后的条带在62ng水平隐隐可见;而胶体考马斯亮蓝染色法的灵敏度稍高,62ng条带明显可见;改良考马斯亮蓝染色法的效果更好些,在15.6ng水平可见条带的显示;而银染法灵敏度最高,在15.6ng水平可见清晰条带的显现,同时在4ng 可见隐约条带。
4种染色法的比较显示,灵敏度最高的是银染法,可达纳克级水平,其次为改良考马斯亮蓝染色法,10纳克级蛋白质能被检测。
而胶体考马斯亮蓝染色法和经典考马斯亮蓝染色法稍差,检测水平仅达几十纳克。
4种染色法的双向电泳蛋白质点显示比较来自NB4细胞的全蛋白经pH 3-10NL的等电点梯度分离,SDS-PAGE二维垂直电泳后被3种考染法着色成像的显示可以看岀传统考马斯亮蓝染色法的蛋白质点显现最少,胶体考马斯亮蓝染色法的蛋白质点显现较多,而改良考马斯亮蓝染色法的蛋白质点显现更多。
凝胶背景以后二者为更清晰。
经PDQuest软件进行分析,3幅图的蛋白质点显现数分别为483、679、890。
NB4细胞的全蛋白经pH 5-8 IPG分离,分别经改良考马斯亮蓝染色法与银染法着色成像的蛋白点显示,二者上样量一致,蛋白点的显现数量相当,但银染的蛋白点明显显示着色重。
这与单向电泳的效果相同。
4种染色法的可操作性比较在比较其蛋白质检测灵敏性的同时,对染色法的操作简便性、安全性以及蛋白质经质谱鉴定的成功率也作了对比。
比较显示,考马斯亮蓝染方法简便,但耗时较长,经改进后可以做到无毒操作,质谱兼容性较高。
银染步骤繁多,但耗时短;操作过程中有甲醇、甲醛等毒性物的接触,质谱兼容性低。
即使我们选择的银染法被文献报道为质谱兼容的,但仍然表现为低的蛋白鉴定率,不及考马斯亮蓝染。
讨论凝胶染色方法对蛋白质分辨率的影响提高双向凝胶电泳的分辨率是蛋白质组学研究尚待解决的课题及技术发展方向,影响蛋白质分辨率的因素较多,其中凝胶染色方法对蛋白质分辨率的影响不可忽视,它影响了实验的显像结果,直接干预了后续的质谱鉴定。
目前,在蛋白质组学领域中广泛应用的是考马斯亮蓝染色法和银染法两种。
考马斯亮蓝(Coomassie brilliant blue ,CBB G-250 )的化学名称为二甲花青亮蓝,偏酸性,与蛋白质的碱性基团结合,颜色由棕黄色转为深兰色。
考马斯亮蓝染色法可以达到微克级检测水平,着色程度与蛋白量的线性动力学相关范围广,适合定量分析。
该法由于较低的成本、使用方便及与下游的质谱鉴定技术的良好相容性,而得到了非常广泛的使用。
然而微克级的检测水平使它漏检了相当多的低丰度蛋白质,日益成为蛋白质组学的瓶颈,因此大量提高染色灵敏度的研究及各种染色液的配方和方法应运而生,经文献报道的方法众多,评价不一。
我们的试验选择了3种常用的考马斯亮蓝染色法进行比较,测得传统考马斯亮蓝染色法的灵敏度可达几百纳克,胶体考马斯亮蓝染色法能检测到几十纳克蛋白,改良考马斯亮蓝染色法则可达几纳克蛋白水平。
考马斯亮蓝染色法经过改进能获得高的蛋白质检测灵敏度,甚至可与银染媲美。
银染法是利用银离子与氨基酸共价结合,还原剂的加入使与胶内蛋白质结合的银离子形成金属银而显色。
文献报道检测限度可低于1 ng的蛋白质,其灵敏度比传统考马斯亮蓝染色法高约100倍,被广泛应用于蛋白质组学研究,但银染步骤复杂,繁琐,且着色线性动力学的覆盖范围窄,导致蛋白质差异显示的不准确性,同时由于游离银离子及相关试剂的存在,给后续分析及鉴定带来一定的实际困难。
我们的实验只采用了一种银染法,结果显示银染法的灵敏性高于所有考马斯亮蓝染色法,4ng的蛋白质条带也能显现,但蛋白质鉴定成功率低。
蛋白质检测灵敏度的影响因素蛋白质着色显示的灵敏性既取决于染色试剂,如上述分析,又与染色过程中涉及的有机溶剂及离子有关。
在考马斯亮蓝染色法和银染法中都存在如甲醇、乙醇、乙酸、甲醛等有机溶剂的使用。
甲醇和乙醇在染色里起固定作用,把蛋白质固定在凝胶中或阻滞它们在凝胶中扩散。
同时也去除电泳过程中遗留的干扰染色过程的物质,如去垢剂、还原剂和缓冲液等成分。
乙酸的作用既是辅助固定蛋白质同时又维持染液的酸性度,以利于考马斯亮蓝与蛋白质结合。
他们的存在有利于获得背景清晰、信噪比高的图像。
由于3种有机溶剂的相似作用,我们在胶体考马斯亮蓝染色法及改良考马斯亮蓝染色法里,保留乙醇作为唯一的固定清洁剂,用磷酸替代乙酸发挥酸化作用,结果显示两种考马斯亮蓝染色法获得的图像背景比传统考马斯亮蓝染色法的要清晰,条带也锐利。
在胶体考马斯亮蓝染色法中,酸性的乙醇介质中加入硫酸铵,使得着色剂形成胶体染色颗粒,胶本身可不被显著着色,蛋白染色灵敏度得以提高。
而在改良考马斯亮蓝染色法中,在高酸、高离子环境下,蛋白质的葡糖胺和天冬酰胺酸质子化,更利于与染色剂发生结合。
因此2种染色法呈现出背景清晰、蛋白检测多的图像,且在改良考马斯亮蓝染色法中由于高酸、高离子存在,灵敏度岀现提高,几接近银染水平。
甲醛在银染中发挥还原剂作用,使结合在蛋白质上的银还原而显色。
甲醛浓度影响银染效果,浓度一般为400〜500卩I/L甲醛浓度较高时胶颜色发黄,背景色混杂,难以发现目的点;浓度较低不仅染色时间延长,而且不易着色。
银的络合剂硫代硫酸钠防止自由银离子还原为金属银,可减少非特异染色,降低背景染色,其浓度一般为0.1〜0.2mg/L。
该络合剂用量过多,胶颜色过深;少则不足以螯合掉游离的银离子,胶颜色发黑。
质谱兼容性的影响因素考马斯亮蓝G-250在一定的pH时,这种染料-蛋白质复合物被完全解聚。
适应于蛋白质的质谱鉴定;而银的方法,以提高银染鉴定的成功率。
操作的安全性与简便性由于有机溶剂的相似作用,我们在改进的考马斯亮蓝染色法里,抛弃有毒的甲醇及气味难闻的乙酸,同时在步骤上,我们采用染色和固定一步法进行,减少了第一步固定的时间,仅用超纯水清洗,缩短了整个染色及人与有毒物接触的时间。
